扩展功能
文章信息
- 黄杰, 周瑜, 刘与之, 朱丹, 宋旭颢, 陈本平, 张修月, 岳碧松
- HUANG Jie, ZHOU Yu, LIU Yuzhi, ZHU Dan, SONG Xuhao, CHEN Benping, ZHANG Xiuyue, YUE Bisong
- 基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析
- Characteristics of Microsatellites in Arborophila rufipectus Genome Sequences Using 454 GS FLX
- 四川动物, 2015, 34(1): 8-14
- Sichuan Journal of Zoology, 2015, 34(1): 8-14
- 10.3969/j.issn.1000-7083.2015.01.002
-
文章历史
- 收稿日期:2014-06-16
- 接受日期:2014-09-15
2. 四川老君山国家级自然保护区管理局, 四川屏山 645350
2. Laojunshan National Nature Reserve Management Bureau, Pingshan, Sichuan Province 645350, China
四川山鹧鸪Arborophila rufipectus,隶属鸡形目Galliformes雉科Phasianidae山鹧鸪属Arborophila,别名砣砣鸡、笋鸡,是中国特有珍稀鸟类,主要分布于四川省中南部的屏山、马边、甘洛及其毗连的山脉,分布范围极其狭窄(Liao et al.,2007a,2007b;戴波等,2014)。由于其分布范围不足于1800 km2,种群数量不到2000只,栖息地破碎化极其严重,不仅被列为国家Ⅰ级重点保护动物,国际自然保护联盟(IUCN)濒危物种红皮书也将其列为濒危等级,国际鸟盟(Bird Life International)以及世界雉类协会(WPA)鹑类专家组还将该物种列入最优先的保护领域并将其纳入了鹑类保护组行动计划(King & Li,1988;Collar et al.,2001;IUCN,2014)。为了有效保护四川山鹧鸪这一珍稀濒危物种,近几十年来,许多学者都致力于对其的不断探索,并取得了一些基础信息,但是这些研究都主要集中于四川山鹧鸪的生境状况、生态习性及分布情况等宏观方面(Liao et al.,2007a,2007b,2008;Dai et al.,2009;廖文波,胡锦矗,2010;戴波等,2014),对其分子生物学领域的研究却鲜有报道(He et al.,2009;Liu et al.,2012)。显然,目前取得的资料还远远不能满足对四川山鹧鸪进行科学有效的保护和管理的需要。黄杰等: 基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析
微卫星(microsatellite),又被称为简单序列重复(simple sequence repeats)或短串联重复(short t and em repeats),由于其具有符合孟德尔遗传规律、丰富的多态性、共显性遗传、分布广且均匀、数量大、易于检测等特点,被认为是优良的分子遗传标记,倍受研究者们的青睐。近年来常被用于检测物种遗传变异和诸如种群间基因流、遗传多样性、种群结构、物种交配方式以及合作繁殖等生态学特征(于颖等,2009;Huang et al.,2013;Page et al.,2014)。然而,就鸟类而言,除了火鸡Meleagris gallopavo、日本鹌鹑Coturnix japonica、红原鸡Gallus gallus、珍珠鸟Poephila guttata、朱鹮Nipponia nippon、丹顶鹤Grus japonensis、山蓝鸭Hymenolaimus malacorhynchos等具有相关微卫星的报道外(Reed et al.,2000;Kayang et al.,2002;He et al.,2006;Abdelkrim et al.,2009;Zou et al.,2010),目前还没有关于四川山鹧鸪微卫星的任何相关研究报道。
本研究通过第二代测序技术——罗氏454公司的GS FLX测序平台,对四川山鹧鸪核基因组进行浅测序,利用生物信息学方法开发的微卫星搜索软件扫描其基因组序列,搜索和输出微卫星序列,首次对其核基因组微卫星序列进行了初步的统计和分析,旨在对四川山鹧鸪基因组水平上的微卫星重复序列的种类、数量等分布特征进行初步探索,以期深化对四川山鹧鸪基因组的认识和了解,为以后开发和筛选大量高质量微卫星标记提供数据支持,也为利用微卫星标记更加有效地保护四川山鹧鸪奠定了基础。 1 材料和方法 1.1 样品收集和基因组DNA的提取
用于基因组浅测序的样本来自本实验室,是一只于2008年5月2日采自四川省屏山县四川老君山国家级自然保护区自然死亡的四川山鹧鸪(28.69983°N,104.03253°E),其肌肉样本浸泡在95%的酒精中,-20 ℃保存。本研究采用酚-氯仿法提取基因组DNA(Sambrook & Russell,2002)。1.2 454 GS FLX高通量测序
将提取的DNA样本于2013年12月送北京诺禾致源生物信息科技有限公司,采用罗氏454 GS FLX高通量测序技术对四川山鹧鸪进行基因组测序。首先利用超声波或震荡离心等方法把纯化好的DNA样本打断成长度200~500 bp小片段,在这些小片段的两端加上不同的接头,构建单链DNA文库。高通量测序的基本步骤:纯化基因组DNA;片段化回收小于500 bp的部分;补平末端;5磷酸化和3加上一个A;连接上测序接头(Adaptors);纯化连接产物,将没连接上的和Adaptors去掉;PCR扩增;去除掉引物;纯化成最后的产物;文库制备产物的检测;文库测序。1.3 微卫星分析
采用本实验室开发的微卫星搜索及统计软件——MSDB v2.4(http://msdb.biosv.com)(Du et al.,2013),对四川山鹧鸪基因组浅测序的序列进行搜索和统计。本研究所使用的搜索标准如下:(1)单碱基重复次数≥12,二碱基重复次数≥7,三碱基重复次数≥5,四、五、六碱基重复次数≥4,且搜索的微卫星均为完美型微卫星(perfect microsatellites or pure microsatellites)(Du et al.,2013;汪自立等,2013);(2)考虑到碱基互补配对原则以及起始碱基的排列差异,将所有可循环的序列及其互补序列归为一类,如ACG核心重复序列代表所有CGA、GAC、TGC、GCT和CTG的微卫星。总体来说,单碱基重复有A、C 2种独特类型;二碱基重复有4种独特类型;三碱基重复有10种独特类型;四碱基重复有33种独特类型,五碱基重复有102种独特类型;六碱基重复有350种独特类型。
关于微卫星序列分布特征的统计术语定义如下:(1)重复类型:指微卫星序列中基本重复单元是由几个碱基组成,按照重复单元的碱基组成数目,微卫星序列可以分为6种重复类型:即单碱基(mononucleotide)、二碱基(dinucleotide)、三碱基(trinucleotide)、四碱基(tetranucleotide)、五碱基(pentanucleotide)和六碱基(hexanucleotide)。(2)重复拷贝类别:指各重复类型具体由哪些碱基构成,例如三碱基重复类型ACG和AGC分别属于不同的重复拷贝类别,而GCT、CTG和TGC则属于相同的重复拷贝类别。(3)重复拷贝数:指一个微卫星序列中其核心序列的重复次数,例如(AGGC)17,这个AGGC拷贝类别的重复拷贝数是17。(4)密度(bp/Mb):指一个物种基因组中每Mb序列包含的微卫星序列长度(bp)。(5)丰度(个/Mb):指一个物种基因组中每Mb序列包含的微卫星序列数量。2 结果与分析 2.1 各类型微卫星重复序列数目和相应的百分比
采用454 GS FLX Titanium高通量测序技术测序,得到7 106 456对clean paired-end reads,长度为250 bp,总数据量3.55 Gb,利用paired-end reads部分重叠进行拼接最后得到6 842 637条短序列,平均长度303 bp,最大长度370 bp,GC含量为43.54%。将这些序列直接用MSDB v2.4微卫星搜索软件搜索,共找到335 263个重复序列。所有的重复类型如表 1所示,单碱基重复类型数目最多,为197 913个,丰度为95.28个/Mbp,占重复序列总数目的59.03%;其次是四碱基重复类型,为45 575个,丰度为21.94个/Mbp,占13.59%;再次分别是三碱基重复类型为28 131个,丰度为13.54个/Mbp,占8.39%;六碱基重复类型为25 309个,丰度为12.18个/Mbp,占7.55%;两碱基重复类型为21 746个,丰度为97.3个/Mbp,占6.49%;最少的是五碱基重复类型,为16 591个,丰度为7.99个/Mbp,仅占4.95%(表 1)。
| 微卫星分布 | 单碱基 | 二碱基 | 三碱基 | 四碱基 | 五碱基 | 六碱基 | 总计 |
| 重复序列数量/No. | 197 913 | 21 746 | 28 131 | 45 575 | 16 591 | 25 307 | 335 263 |
| 丰度/ No./Mbp | 95.28 | 10.47 | 13.54 | 21.94 | 7.99 | 12.18 | |
| 密度/ bp/Mb | 1464.69 | 180.77 | 236.82 | 456.4 | 242.23 | 470.18 | |
| 各重复类型所占比例/% | 59.03 | 6.49 | 8.39 | 13.59 | 4.95 | 7.55 |
同一种类型的重复序列中,各重复拷贝类别所占的比例也不相同(表 2)。单碱基重复类型中,我们扫描出拷贝类别为A的重复数目最为丰富,有187 963个,占单碱基重复序列总数目的94.97%,C重复拷贝类别的数量很少,只有9950个,仅占了5.03%;两碱基重复类型中,AC重复拷贝类别最多,占两碱基重复序列总数目的56.87%,有12 366个,其次是AG(23.63%)和AT(18.44%),CG类别数量最少,仅有230个,占1.06%;三碱基重复类型中,10种重复拷贝类别中数量最多的是AAC,有10 216个,占三碱基重复序列总数目的36.31%,其他类型如表 2所示。四碱基、五碱基和六碱基重复类型的重复拷贝类别均较多,在此我们列举了数量较多的部分拷贝类别。四碱基重复类型中,AAAC重复拷贝类别数量最多,为17 970个,占39.43%,其次AAAT(26.21%)、AAAG(10.06%)、AAGG(5.45%)、AGGG(2.16%)和AGAT(1.86%)。其他的类别数量相对较少,数量均小于1000个。五碱基重复类型中,AAACA重复拷贝类别数量最多,有4538个,占27.35%,其次是AAAGA(11.07%)、AATAT(5.33%)、AAGGG(4.27%)、AAGAG(3.31%)和CCCGG(3.07%),其他重复拷贝类别数量均不到500个。六碱基重复类型中,重复拷贝类别数量最多的是AGGGTT,有23 086个,占91.22%。
| 微卫星重复类型 | 常见重复拷贝类别(比例/%) | |
| 单碱基重复类型 | A(94.97) | C(5.03) |
| 二碱基重复类型 | AC(56.87) | AT(18.44) |
| AG(23.63) | CG(1.06) | |
| 三碱基重复类型 | AAC(36.31) | AAT(15.78) |
| AGG(14.71) | CCG(8.75) | |
| ACG(6.42) | AGC(6.26) | |
| ACC(4.12) | AAG(4.30) | |
| ACT(1.76) | AGT(1.59) | |
| 四碱基重复类型 | AAAC(39.43) | AAAT(26.21) |
| AAAG(10.06) | AAGG(5.45) | |
| AGGG(2.16) | AGAT(1.86) | |
| 五碱基重复类型 | AAACA(27.35) | AAAGA(11.07) |
| AATAT(5.33) | AAGGG(4.27) | |
| AAGAG(3.31) | CCCGG(3.07) | |
6种不同重复类型微卫星的拷贝数分布情况也有差异。单碱基重复的分布范围为12~188次,主要分布在12~38次,有196 382个,占单碱基重复类型总数的99.23%。由于单碱基数量较多,如图 1所示,我们列举了二至六碱基重复类型微卫星拷贝数分布情况。二碱基重复的拷贝数分布范围为4~78次,主要分布在7~17次,共有21 443个,占98.61%;三碱基重复的拷贝数分布范围为5~33次,主要分布在5~16次,共有28 114个,占99.94%;在四碱基重复中,拷贝数分布范围为4~48次,主要分布在4~20次,共计45 068个,占98.89%;五碱基重复的拷贝数分布范围为4~38次,主要分布在4~19次,共有16 151个,占了98.89%;六碱基重复拷贝数的分布范围为4~32次,主要分布在4~16次,共有24 773个,占97.89%。
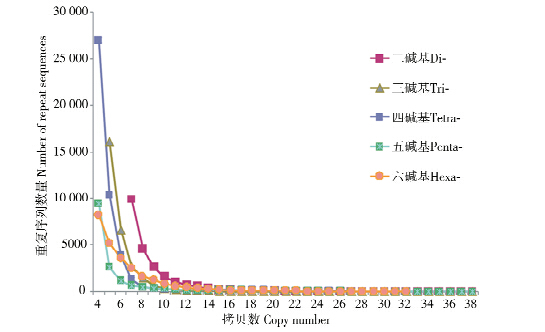 |
| 图 1 不同重复拷贝类型微卫星的重复次数分布 Fig. 1 Distribution of the copy number in the different microsatellite motifs |
如图 2所示,在四川山鹧鸪基因组中微卫星数量最多的前16种重复拷贝类别依次为A、AGGGTT、AAAC、AC、AAAT、AAC、C、AG、AAAG、AAACA、AAT、AGG、AT、AGC、AAGG、CCG,其中超过半数都是三碱基和四碱基重复类型,而AAACA、AGGGTT分别是这16种重复拷贝类别唯一的五碱基、六碱基重复类型。这些重复拷贝类别的微卫星数量都在2000个以上,总数是308 780个,占所搜索到的微卫星重复拷贝类别总数的92.10%,而其他所有拷贝类别仅占7.90%(表 3)。这16种类型中,重复拷贝次数为4~30的微卫星数量有305 238个,占总数的98.85%。
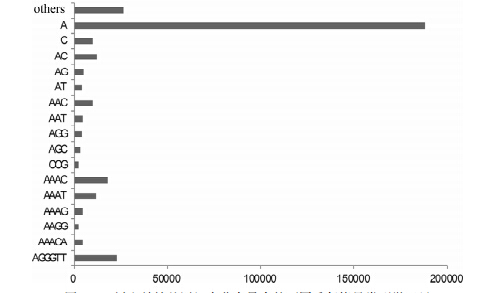 |
| 图 2 四川山鹧鸪基因组中分布最多的不同重复拷贝类型微卫星 Fig. 2 Distribution of the most frequent microsatellite motifs of Arborophila rufipectus |
| 类型Motif | 重复次数Copy repeats | 最长重复 Longest motif | ||||
| ≤11 | 12~20 | 21~30 | ≥31 | 总数 Total no. (比例/%) | ||
| A | — | 170 382 | 14 463 | 3118 | 187 963(56.06) | 1×(A)188 |
| AGGGTT | 21 503 | 1366 | 189 | 28 | 23 086(6.70) | 14×(AGGGTT)32 |
| AAAC | 17 967 | 3 | — | — | 17 970(5.36) | 3×(AAAC)12 |
| AC | 10 680 | 1599 | 83 | 4 | 12 366(3.69) | 1×(AC)44 |
| AAAT | 11 946 | 1 | — | — | 11 947(3.56) | 1×(AAAT)12 |
| AAC | 10 101 | 115 | — | — | 10 216(3.05) | 3×(AAC)17 |
| C | — | 8901 | 704 | 345 | 9950(2.97) | 1×(C)131 |
| AG | 4614 | 482 | 28 | 15 | 5139(1.53) | 1×(AG)78 |
| AAAG | 3390 | 776 | 350 | 31 | 4584(1.38) | 1×(AAAG)48 |
| AAACA | 4516 | 21 | 1 | — | 4538(1.35) | 1×(AAACA)22 |
| AAT | 4417 | 21 | — | — | 4438(1.32) | 1×(AAT)17 |
| AGG | 4090 | 49 | — | — | 4139(1.24) | 4×(AGG)15 |
| AT | 3958 | 53 | — | — | 4011(1.20) | 1×(AT)19 |
| AGC | 3514 | 11 | — | 1 | 3526(1.05) | 1×(AGC)33 |
| AAGG | 2011 | 383 | 88 | 2482(0.74) | 1×(AAGG)35 | |
| CCG | 2453 | 9 | — | — | 2462(0.73) | 2×(CCG)15 |
| Total | 105 160 | 184 172 | 15 906 | 3542 | 308 780(92.10) | |
不同重复拷贝类型微卫星重复次数的分布各不相同。在检测到的所有重复拷贝类型中,(A)n是分布最多的微卫星类型,占全部微卫星的56.06%,重复拷贝次数主要分布在12~30次。但是,四川山鹧鸪基因组中最长的微卫星类型是AGGGTT,最长重复次数是32次,即核心序列总长度为192 bp,共发现14个这种重复拷贝类型的微卫星。短的重复类型(单碱基、二碱基、三碱基)比长的重复单元(四碱基、五碱基、六碱基)的分布更广泛。如果不考虑单碱基重复类型,(AGGGTT)n不仅是数量最多,同时也是重复次数分布最广的微卫星拷贝类型,重复次数分布范围为4~32次。3 讨论
一个物种基因组中的微卫星序列有着十分重要的意义,其序列分布特征可以作为衡量不同物种基因组差异的一个指示(马秋月等,2013)。本研究采用454测序技术对四川山鹧鸪基因组序列进行测序,利用微卫星搜索软件搜索并分析了其基因组完美型微卫星的分布频率以及基本重复类型的特点。发现在四川山鹧鸪基因组中,单碱基重复类型数量上占优势(59.03%),这与包括人在内的灵长类(Tóth et al.,2000;Subramanian et al.,2003)、大熊猫(李午佼等,2014)、牛和绵羊(戚文华等,2013)等多种高等脊椎动物基因组中优势微卫星类型一致。但是在一些基因组相对较小的昆虫和真菌中,三碱基重复类型是数量最多的微卫星类型(Wang et al.,2008;Castagnone-Sereno et al.,2010;汪自立等,2013)。这表明,高等生物基因组倾向于单碱基微卫星类型占主导地位。
在不考虑单碱基的情况下,四川山鹧鸪基因组中四碱基重复类型的数量占优势,这与黄杰等(2012)关于红原鸡的研究结果相一致,而其他脊椎动物则是二碱基重复类型占优势,所以,鸟类基因组可能具有四碱基微卫星的偏好。另外,四碱基重复类型中,重复拷贝类别最为丰富的是AAAC,其次是AAAT、AAAG,这4种拷贝类型总和占了75%以上,由此可见,AAAB(B代表除A以外的任何碱基)重复拷贝类别在四川山鹧鸪基因组中含量最丰富。这一结果与Tóth等(2000)对啮齿类、灵长类、维管束植物及真菌类的研究结果一致。红原鸡基因组微卫星分析也存在这种情况(黄杰等,2012)。四碱基微卫星在PCR扩增过程中不易产生滑带(Stutter b and s)或阴影带(Shadow b and s)导致错误的基因型,因而被认为是比二碱基和三碱基微卫星更精确和可靠的微卫星遗传标记(Archie et al.,2003)。因此,本研究搜索出的大量四碱基微卫星也为以后筛选多态性微卫星标记奠定了数据基础。
四川山鹧鸪基因组微卫星中,单碱基重复类型以(A)n为主(94.97%),二碱基重复类型中GC含量也较低,真菌(Kim et al.,2008)、人、果蝇、拟南芥、酵母(Katti et al.,2001)以及多数真核生物(Hong et al.,2007;Sharma et al.,2007)的微卫星GC含量也较低。其他5种重复类型中,重复拷贝类别较丰富的微卫星均富含A或T,很少有G或C(图 3)。普遍认为,DNA复制过程中滑移是微卫星产生机制,而DNA序列发生滑动复制的概率与序列GC含量成负相关(Schlotterer & Tauze,1992;Zhao et al.,2011;汪自立等,2013)。这可能是因为C≡G之间的键比A=T难断裂,GC含量高的序列复制过程不易发生滑动,因此,多数微卫星均是A/T含量较丰富,而CG/GC重复类型微卫星频率较低。另外,根据分布频率最高的重复拷贝类型微卫星的拷贝数分布情况发现(表 3),不同重复拷贝类型微卫星重复次数的分布情况虽然各不相同,但是不同重复拷贝类型中,均可以发现重复拷贝次数少的类型所占该类型微卫星总数的比例最高。
对于缺乏分子生物学研究的四川山鹧鸪这一濒危珍稀鸟类而言,微卫星标记的开发对于其在个体识别、数量调查、种群遗传多样性评估、遗传结构分析、连锁图谱构建以及遗传追踪等研究领域都有着十分重要的意义。本研究首次对四川山鹧鸪基因组测序并扫描出了大量微卫星序列,为高质量四川山鹧鸪微卫星标记的开发提供了十分充足的数据基础。另外,根据微卫星的重复次数、类型以及侧翼序列等特征,下一步我们还将选择出需要的微卫星序列并设计合适的引物,筛选出高质量多态性的微卫星标记。
| 戴波, 陈本平, 岳碧松, 等. 2014. 四川山鹧鸪栖息地分析与预测[J]. 四川动物, 33(3): 329-336. |
| 黄杰, 杜联明, 李玉芝, 等. 2012. 红原鸡全基因组中微卫星分布规律研究[J]. 四川动物, 31(3): 358-363. |
| 李午佼, 李玉芝, 杜联明, 等. 2014. 大熊猫和北极熊基因组微卫星分布特征比较分析[J]. 四川动物, 33(6): 874-878. |
| 廖文波, 胡锦矗. 2010. 四川山鹧鸪生态习性研究进展[J]. 绵阳师范学院学报, 29(2): 67-71. |
| 马秋月, 戴晓港, 陈赢男, 等. 2013. 枣基因组的微卫星特征[J]. 林业科学, 49(12): 81-87. |
| 戚文华, 蒋雪梅, 肖国生, 等. 2013. 牛和绵羊全基因组微卫星序列的搜索及其生物信息学分析[J]. 畜牧兽医学极, 44(11): 1724-1733. |
| 汪自立, 黄杰, 杜联明, 等. 2013. 二斑叶螨和肩突硬蜱基因组微卫星分布规律研究[J]. 四川动物, 32(2): 481-486. |
| 于颖, 杨孔, 王永, 等. 2009. 藏鸡微卫星文库的构建与微卫星标记筛选[J]. 四川动物, 28(3): 358-362. |
| Abdelkrim J, Robertson BC, Stanton JL, et al. 2009. Fast, cost-effective development of species-specific microsatellite markers by genomic sequencing[J]. Biotechniques, 46(3): 185-192. |
| Archie EA, Moss CJ, Alberts SC. 2003. Characterization of tetranucleotide microsatellite loci in the African Savannah Elephant (Loxodonta africana africana)[J]. Mol Ecol Notes, 3: 244-246. |
| Castagnone-Sereno P, Danchin EGJ, Deleury E, et al. 2010. Genome-wide survey and analysis of microsatellites in nematodes, with a focus on the plant-parasitic species Meloidogyne incognita[J]. BMC Genomics, 11: 598. |
| Collar NJ, Andreev AV, Chan S, et al. 2001. Threatened birds of Asia: The Bird Life International Red Data Book[M]. Cambridge, UK: Bird Life International: 836-843. |
| Dai B, Dowell SD, Garson PJ, et al. 2009. Habitat utilisation by the threatened Sichuan partridge, Arborophila rufipectus: consequences for managing newly protected areas in southern China[J]. Bird Conserv Int, 19(2): 187-198. |
| Du LM, Li YZ, Zhang XY, et al. 2013. MSDB: A user-friendly program for reporting distribution and building databases of microsatellites from genome sequences[J]. J Hered, 104(1): 154-157. |
| He LP, Wan QH, Fang SG, et al. 2006. Development of novel microsatellite loci and assessment of genetic diversity in the endangered Crested Ibis, Nipponia nippon[J]. Conserv Genetics, 7(1): 157-160. |
| He LW, Dai B, Zeng B, et al. 2009. The complete mitochondrial genome of the Sichuan Hill Partridge (Arborophila rufipectus) and a phylogenetic analysis with related species[J]. Gene, 435(1-2): 23-28. |
| Hong CP, Piao ZY, Kang TW, et al. 2007. Genomic distribution of simple sequence repeats in Brassica rapa[J]. Mol Cells, 23(3): 349-356. |
| Huang J, Li YZ, Li P, et al. 2013. Genetic quality of the Miyaluo captive forest musk deer (Moschus berezovskii) population as assessed by microsatellite loci[J]. Biochem Syst Ecol, 47: 25-30. |
| IUCN. 2014. The IUCN Red List of Threatened Species (Version 2014. 2)[EB/OL]. http://www.iucnredlist.org. 2014. |
| Katti MV, Ranjekar PK, Gupta VS. 2001. Differential distribution of simple sequence repeats in eukaryotic genome sequences[J]. Mol Biol Evol, 18(7): 1161-1167. |
| Kayang BB, Inoue-Murayama M, Hoshi T, et al. 2002. Microsatellite loci in Japanese quail and cross-species amplification in chicken and guinea fowl[J]. Genet Sel Evol, 34(2): 233-253. |
| Kim TS, Booth JG., Gauch Jr HG, et al. 2008. Simple sequence repeats in Neurospora crassa: distribution, polymorphism and evolutionary inference[J]. BMC Genomics, 9: 31. |
| King B, Li GY. 1988. China's most endangered Galliformes[J]. Oryx, 22(4): 216-217. |
| Liao WB, Hu JC, Li C. 2007a. Habitat utilization during the pairing season by the common hill-partridge Arborophila torqueola in Baiposhan Natural Reserve, Sichuan, China[J]. Orn Sci, 6(2): 87-94. |
| Liao WB, Hu JC, Li C, et al. 2008. Roosting behaviour of the endangered Sichuan Hill-partridge Arborophila rufipectus during the breeding season[J]. Bird Conserv Internatn, 18(3): 260-266. |
| Liao WB, Li C, Hu JC, et al. 2007b. Habitat utilization of the Sichuan Hill-partridge (Arborophila rufipectus) in the non-breeding period in Laojunshan Nature Reserve[J]. Zool Res, 28(2): 172-178. |
| Liu ZF, He LW, Yuan HG, et al. 2012. CR1 retroposons provide a new insight into the phylogeny of Phasianidae species (Aves: Galliformes) [J]. Gene, 502(2): 125-132. |
| Page RB, Sankamethawee W, Pierce AJ, et al. 2014. High throughput sequencing enables discovery of microsatellites from the puff-throated bulbul (Alophoixus pallidus) and assessment of genetic diversity in Khao Yai National Park, Thailand[J]. Biochem Syst Ecol, 55: 176-183. |
| Reed KM, Roberts MC, Murtaugh J, et al. 2000. Eight new dinucleotide microsatellite loci in turkey (Meleagris gallopavo)[J]. Anim Genet, 31(2): 140. |
| Sambrook J, Russell DW. 2002. Molecular Cloning: a Laboratory Manual, 3rd ed[M]. Beijing: Science Press. |
| Schlotterer C, Tautz D. 1992. Slippage synthesis of simple sequence DNA[J]. Nucleic Acids Res, 20(2): 211-215. |
| Sharma PC, Grover A, Kahl G. 2007. Mining microsatellites in eukaryotic genomes[J]. Trends Biotechnol, 25(1): 490-498. |
| Subramanian S, Mishra RK, Singh L. 2003. Genome-wide analysis of microsatellite repeats in humans: their abundance and density in specific genomic regions[J]. Genome Biol, 4(2): R13. |
| Tóth G, Gaspari Z, Jurka J. 2000. Microsatellites in different eukaryotic genomes: survey and analysis[J]. Genome Res, 10(7): 967-981. |
| Wang SZ, Lorenzen MD, Beeman RW, et al. 2008. Analysis of repetitive DNA distribution patterns in the Tribolium castaneum genome[J]. Genome Biol, 9: R61. |
| Zhao XY, Tan ZY, Feng HP, et al. 2011. Microsatellites in different Potyvirus genomes: Survey and analysis[J]. Gene, 488(1-2): 52-56. |
| Zou HF, Dong HY, Kong WY, et al. 2010. Characterization of 18 polymorphic microsatellite loci in the red-crowned crane (Grus japonensis), an endangered bird[J]. Anim Sci J, 81(4): 519-522." |
 2015, Vol. 34(1)
2015, Vol. 34(1)




