2. 河南中医药大学 第一附属医院脑病介入科,郑州 450004
2. Department of Encephalopathy Intervention, The First Affilated Hospital of Henan University of TCM, Zhengzhou 450004, China
人血清白蛋白是人体血液中最丰富的载体蛋白,在血浆中的质量浓度为42 g/L,约占血浆总蛋白的60%,具有贮运内源和外源化合物的作用[1]。许多药物能够与人血清白蛋白进行可逆性地结合,结合后的药物复合物不易穿透血脑屏障、毛细血管壁等多种生物膜,从而对药物在体内的分布与代谢产生重要影响[2]。研究人血清白蛋白与药物的相互作用,可以了解药物在人体内的运载、代谢过程及其作用机制,对药物化学和药物分子设计具有重要意义。
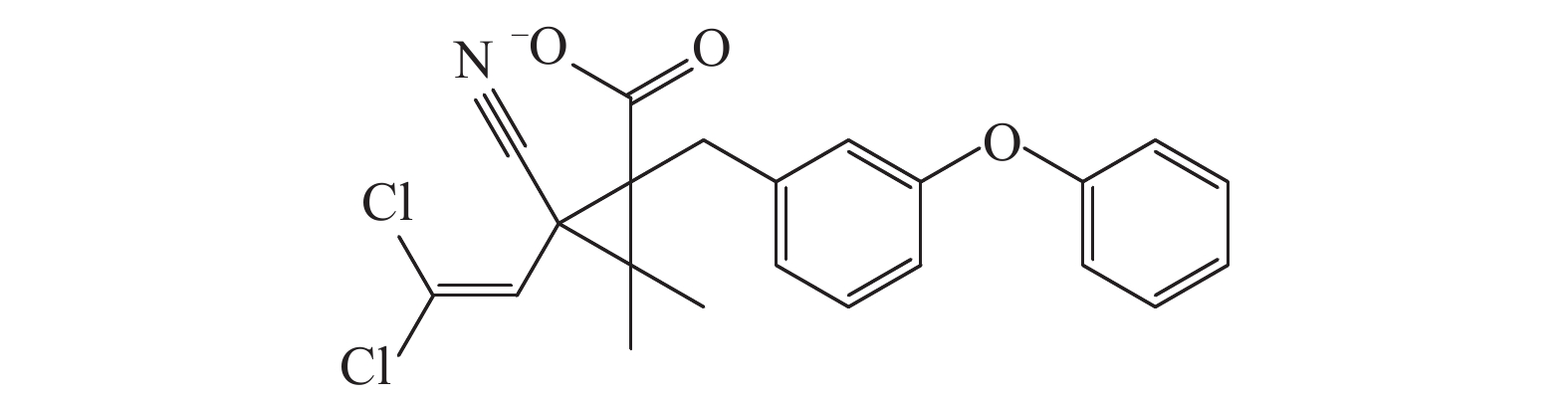
氯氰菊酯 (cypermethrin),结构式见图式 1,分子式为C22H19C12NO3,相对分子质量为416.32,是中国广泛使用的Ⅱ型拟除虫菊酯类杀虫剂,具有较高的胃毒性和触杀作用,主要用于农业害虫和卫生害虫等的防治[3]。
|
图式1 氯氰菊酯的化学结构 Scheme1 Structural formula of cypermethrin |
虽然人体接触低剂量的氯氰菊酯不足以引起明显的中毒症状,但由于其具有一定的蓄积性,蓄积过量会导致呼吸困难、血压下降、阵发性抽搐及出现意识障碍等[4]。研究发现,氯氰菊酯可导致雄性大鼠生殖激素和生育力发生明显变化[5],也可作用于大鼠血液系统和淋巴系统,引起抗体红细胞及白蛋白效价降低[6-7],并可与牛血清白蛋白结合,进而影响氯氰菊酯的分布和代谢[8]。可见氯氰菊酯的毒性作用机理涉及生物体的多种系统,但这些研究多局限于实验动物 (主要是大鼠),缺乏氯氰菊酯在人体内降解和代谢机理的证据。
分子模拟技术是预测蛋白与小分子作用和结合模式的一种有效方法。徐洪亮[9]运用分子模拟方法成功预测了泽兰黄素与牛血清白蛋白的结合模式;徐飞等[10]运用分子模拟方法,研究了泽泻醇类化合物与人血清白蛋白相互作用的分子机理。笔者以人血清白蛋白 (HSA) 为模型,综合运用分子对接、分子动力学、结合自由能计算及丙氨酸突变扫描等分子模拟方法,研究了氯氰菊酯与HSA的相互作用机制,以获取二者作用的结合位点、氢键构成、作用力类型以及关键氨基酸等信息,旨在为阐明氯氰菊酯在人体内的运输和代谢机制提供理论依据。
1 材料与方法 1.1 模型的准备HSA的三维结构从Protein Data Bank数据库中下载,登录号2BX8,分辨率为0.27 nm (2.7 Å)。将HSA结构导入YASARA模拟软件,去除水分子、加氢和添加缺失原子[11],在该软件中构建氯氰菊酯的分子结构。
1.2 分子对接运用AutoDock4.2进行HSA与氯氰菊酯的分子对接,使用Addsol程序计算HSA的溶剂参数,运用Autogrid程序计算网格。在对接过程中,将氯氰菊酯分子设为全柔性结构,旋转键由AutoDockTools程序识别并设为可以任意旋转;使用拉马克遗传算法 (LGA) 和局部能量搜索相结合 (GALS) 的方法,对对接构象进行搜索,并进行100次对接计算,依据聚类分析结果和Amber评分函数打分的高低保留10个构象,选取自由能最低的构象进行分析。
1.3 分子动力学模拟在郑州师范学院生物物种资源研究重点实验室的YASARA分子模拟平台完成本体系分子模拟,受体和配体的结构均来源于1.2节中分子对接结果。将体系置于TIP3P显性立方体水分子模型中,溶质原子与边界之间的距离为0.5 nm (5 Å),模拟采用Amber14力场和周期性边界条件,并向体系添加抗衡离子 (Na+和Cl–) 保持其电中性。分子动力学模拟在pH 7.4和298 K条件下进行,采用NVT正则系宗和郎格万方法控制体系温度,模拟时长约35 ns,其中积分步长为2.5 fs,采样间隔为50 ps。以上程序均在Centos系统下运行。
1.4 结合自由能的计算和能量分解MM-PBSA (molecular mechanics Poisson-Boltzmann surface area)[12]和分子力学广义波恩表面积法 (MM-GBSA,molecular mechanics generalized Born surface area) 是采用分子力学与连续介质模型相结合来计算自由能的方法。其中:MM-GBSA方法是通过配体和受体复合物结构的能量最小化计算两者的结合自由能,无需对分子动力学模拟的轨迹进行采样,因此计算更为高效;而MM-PBSA方法是基于对分子动力学模拟的轨迹进行采样,计算受体和配体的结合自由能,结果更为精确[13-14]。本研究选择在Centos系统下,运行程序软件Amber12的MMPBSA.py模块,采用MM-PBSA法计算HSA和氯氰菊酯的结合自由能,选取平衡后10 ns动力学轨迹的100个构象进行分析。按公式 (1)~(4) 计算体系自由能。
| $\Delta {G_{{\rm{bind}}}} = \Delta {G_{{\rm{complex}}}} - \Delta {G_{{\rm{receptor}}}} - \Delta {G_{{\rm{ligand}}}}$ | (1) |
| $\Delta G = \Delta {E_{{\rm{gas}}}} + \Delta {G_{{\rm{solve}}}} - T\Delta S$ | (2) |
| $\Delta {E_{{\rm{gas}}}} = \Delta {E_{{\rm{ele}}}} + \Delta {E_{{\rm{vdw}}}}$ | (3) |
| $\Delta {G_{{\rm{solve}}}} = \Delta {G_{{\rm{pb}}}} + \Delta {G_{{\rm{nopolar}}}}$ | (4) |
其中:ΔEgas为气相中内能,ΔGsolve为溶剂化自由能,TΔS为熵变值,ΔEele为静电作用能,ΔEvdw为范德华力作用能,ΔGpb为极性溶剂化能,ΔGnopolar为非极性溶剂化能。由于本研究主要研究受体和配体的结合模式,故平移、旋转和振动过程中的熵变对结合自由能的影响可以忽略不计[15]。
运用Amber12软件包,分解HSA单个氨基酸总能量和侧链能量值[16]。每个氨基酸残基的能量贡献被拆解为4个能量项。按公式 (5) 计算。
| $\Delta G = \Delta {E_{{\rm{ele}}}} + \Delta {E_{{\rm{vdw}}}} + \Delta {G_{{\rm{pb}}}} + \Delta {G_{{\rm{nopolar}}}}$ | (5) |
为评估在HSA和氯氰菊酯结合自由能中能量贡献值较大的氨基酸残基的贡献,就对接复合物的动力学轨迹进行丙氨酸突变扫描[17],比较野生型和突变型复合物自由能的变化 (ΔΔGbind)。方法同1.4节,按 (6) 式计算。
| $\Delta \Delta {G_{{\rm{bind}}}} = \Delta {G_{{\rm{bind}}\text{-}{\rm{mutant}}}} - \Delta {G_{{\rm{bind}}\text{-}{\rm{wildtype}}}}$ | (6) |
若ΔΔGbind为正值,表明此氨基酸的突变对配体和受体的结合是不利的;若ΔΔGbind为负值,表明该氨基酸的突变有利于配体和受体的结合。
2 结果与分析 2.1 分子对接HSA与氯氰菊酯进行刚性对接,设置Gid Box包含整个HSA空间结构,对接体系格点为102 × 120 × 90,间距为0.037 5 nm,经聚类分析后,依据结构的多变性和结合能量的高低得到最优结合模式,如图1所示。可见,氯氰菊酯结合于HSA三维晶体结构圆筒疏水腔的底部,在两者相互作用的二维图中显示,氯氰菊酯的O原子与HSA的氨基酸残基Arg209形成了1个氢键,距离为0.31 nm (3.1 Å)。

|
A. 整体图 (overall view);B. 局部图 (locality view);C. 二维图 (2D view)。 图 1 HSA与氯氰菊酯结合模式 Fig. 1 Binding mode of HSA and cypermethrin |
2.2 分子动力学模拟结果
为验证分子对接结果的可靠性和对接复合物的稳定性,运用分子动力学模拟,分析了对接复合物中HSA和单体HSA主链原子的均方根偏差 (RMSD)(图2) 及HSA中氨基酸残基均方根涨落 (RMSF)(图3)。由图2中可以看出:在20 ns时复合物RMSD值趋于收敛平衡,表明体系达到稳定状态[18],稳定状态时单体HSA的RMSD值高于复合物中HSA的RMSD值,表明HSA与氯氰菊酯结合后更加稳定,说明分子对接的结果是可靠的。

|
图 2 分子动力学模拟中主链原子均方根偏差的变化 Fig. 2 The change of RMSD of backbone atoms in molecular dynamics simulation |

|
图 3 分子动力学模拟中氨基酸残基均方根涨落的变化 Fig. 3 The change of RMSF of amino acid residues in molecular dynamics simulation |
为研究分子对接复合物的柔性及其结构的局部运动特征,计算了分子动力学模拟过程中的RMSF值。氨基酸的RMSF值越大,表明其在结合过程中柔性越强。分析发现:在整个模拟过程中复合物的RMSF平均值仅为0.14 nm (1.4 Å),波动比较大的数值位于HSA的末端和氨基酸残基编号300左右区域,表明这两个区域柔性较强;200~250左右的区域数据波动较小,表明该区域柔性较小,为HSA的保守区域。
2.3 结合自由能计算结果基于Amber12程序和MM-PBSA方法计算得人血清白蛋白与氯氰菊酯结合自由能为 –83.43 kJ/mol (表1),表明两者形成了稳定复合物,标准方差为0.39 (小于0.5),说明计算结果是可靠的。对结合自由能进行分解发现,范德华力、静电作用力和非极性溶剂化能可促进两者的结合,能量贡献值分别为 –99.33、–35.61和 –16.28 kJ/mol,其中范德华力是主要驱动力;极性溶剂化能是主要的抑制力,能量贡献值为67.78 kJ/mol (见表1)。
|
|
表 1 HSA和氯氰菊酯的结合自由能和能量分项 Table 1 The binding free energy and subentry of HSA and cypermethrin complex |
2.4 单个氨基酸残基的能量分解结果
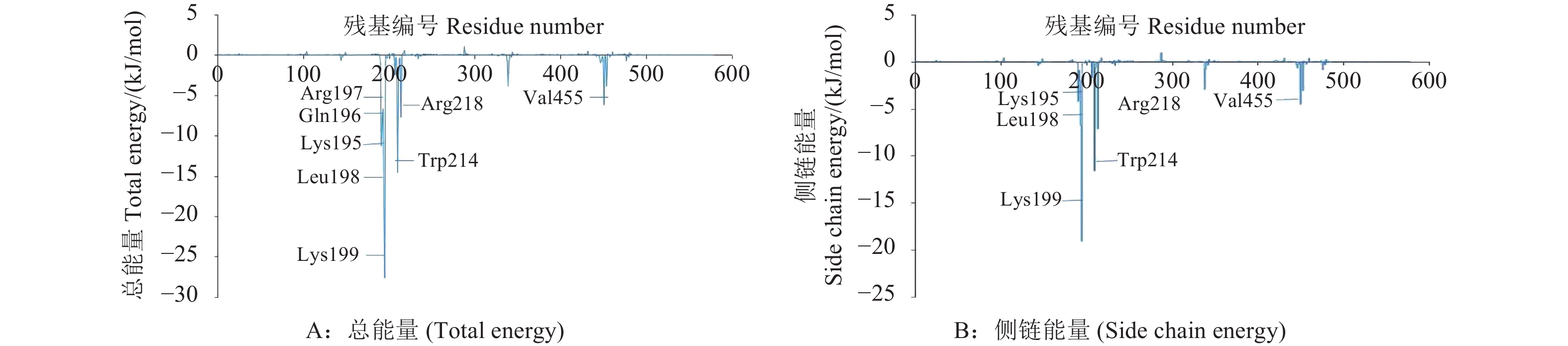
为研究HSA单个氨基酸残基在结合过程中的能量贡献及分布,运用Amber12程序分析了单个氨基酸的总能量和侧链能量,结果见表2和图4。共有8个氨基酸的能量贡献值超过6 kJ/mol,分别为Lys195、Gln196、Arg197、Leu198、Lys199、Trp214、Arg222和Val455,其中Leu198和Lys199的能量贡献值超过18 kJ/mol,这些氨基酸集中在残基200附近区域,这与RMSF的分析结果相吻合,说明这一区域是HSA的保守区域;图4中展示了侧链能量的分布,统计分析发现与总能量基本一致,可见其能量主要分布在侧链上。
HSA中与氯氰菊酯形成氢键的氨基酸Arg209,虽然其有较强的静电作用力 (–38.24 kJ/mol),但是其极性溶剂化能高达36.18 kJ/mol,导致Arg209对总能量的贡献值仅为 –0.13 kJ/mol,所以其对HSA和氯氰菊酯结合的影响不大。
|
|
表 2 HSA和氯氰菊酯复合物单个氨基酸残基能量分解的分项值 Table 2 The decomposed energy subentry value of per amino acid residue of HSA-cypermethrin complex |
|
图 4 单个氨基酸残基的能量分解图 Fig. 4 The energy decomposition spectrum of per amino acid residue |
2.5 关键氨基酸丙氨酸突变扫描结果
假设局部的构象变化不影响配体和受体结合的模式,根据单个氨基酸能量分解结果,选取能量贡献值最大的4个氨基酸Lys195、Leu198、Lys199和Trp214和形成氢键的Arg209进行丙氨酸突变扫描,结果见表3。从ΔΔGbind值可见,Lys195、Leu198、Lys199和Trp214的突变均不利于HSA与氯氰菊酯的结合,而Arg209突变对结合自由能的影响较小。
|
|
表 3 HSA与氯氰菊酯结合突变体的结合自由能 Table 3 The calculated binding free energy of complex mutant of HSA and cypermethrin |
3 小结与讨论
蛋白质与杀虫剂结合可有效降低杀虫剂在自由状态下的浓度,并将其转运至处置位点,促进杀虫剂的代谢,减轻相应毒性[16]。药物与生物体血清白蛋白的结合速率和结合能力决定了药物在血浆中的储留量,结合能力越强,血浆中游离的药物分子越少,其生理活性越低[19]。本研究中,HSA与氯氰菊酯的结合自由能为 –83.43 kJ/mol,表明二者可以形成稳定的复合物,分子动力学模拟结果也证实了复合物的稳定性,说明人体可能对氯氰菊酯具有较强的代谢能力。
HSA的晶体结构含有3个结构域:Domain I (1~195)、Domain II (196~383) 和Domain III (384~585)[20],每个结构域含有A和B两个亚结构,大多数药物结合在IIA和IIIA部位,其中IIIA活性最强[21],本研究中RMSF分析结果也显示,HSA柔性最高的区域在III区,但分子对接和残基能量分解结果发现,HSA与氯氰菊酯结合时主要的能量贡献残基集中于195~210区域,即IIA区域,说明氯氰菊酯结合位点在IIA区,不在活性最高的IIIA区,这与华发令 (Warfarin) 和HSA的结合模式是一致的[22]。可见,HSA与氯氰菊酯的结合模式不是蛋白活性位点单一因素决定的,而是多种因素共同作用的结果。
Moreira等[23]认为,在突变扫描中,ΔΔGbind ≥ 4 kcal/mol (16.72 kJ/mol) 的氨基酸为“hot spots (热点)”,2 kcal/mol ≤ ΔΔGbind < 4 kcal/mol时的氨基酸为“warm spots (暖点)”,ΔΔGbind < 2 kcal/mol (8.36 kJ/mol) 时的氨基酸为“null spots (非点)”。本研究中选取能量贡献大于2 kcal/mol (8.36 kJ/mol) 的氨基酸残基进行丙氨酸突变扫描,结果显示:仅Lys199的ΔΔGbind大于4 kcal/mol(16.74 kJ/mol),为“热点”,说明其对HSA与氯氰菊酯的结合至关重要;与Lys199能量贡献相近的残基Leu198和Trp214的ΔΔGbind均小于2 kcal/mol (8.36 kJ/mol),由于氨基酸残基的能量贡献包括与蛋白内部其他氨基酸的作用和与外部小分子的作用两部分,鉴于Leu198和Trp214的突变对结合自由能的影响较少,说明两者的能量贡献主要是与蛋白内部其他氨基酸的作用能,对保持复合物结构的稳定性具有一定的贡献,但对结合小分子贡献较小。值得注意的是,形成氢键的残基Arg209无论是分解的能量贡献值还是ΔΔGbind都较小,可能是由于Arg209突变之后其功能会被其他氨基酸取代,所以其对HSA与氯氰菊酯的整体结合能力影响不大。
综上所述,本研究运用分子模式方法研究了氯氰菊酯与HSA结合的分子机制,为了解氯氰菊酯在人体内代谢的生理过程提供了依据,也为开发新型更加环保、低毒的杀虫剂探索了新的思路。
| [1] |
马萍, 迟艳华, 庄稼, 等. 骨螺紫及其铜配合物与人血清白蛋白相互作用的光谱学研究[J]. 高等学校化学学报, 2009, 30(8): 1509–1515.
MA P, CHI Y H, ZHUANG J, et al. Spectroscopic studies on the interaction of murexide and murexide-copper complex with human serum albumin[J]. Chem J Chin Univ, 2009, 30(8): 1509–1515. |
| [2] |
魏言春, 宋佳兴. 人血清白蛋白药物输运过程中的催化作用研究[J]. 激光生物学报, 2013, 22(1): 53–57.
WEI Y C, SONG J X. Catalytic effects of human serum albumin to the bound drugs during plasma transmission[J]. Acta Laser Biology Sinica, 2013, 22(1): 53–57. |
| [3] |
董园. 农药氯氰菊酯的生物降解规律研究[D]. 南宁: 广西大学, 2012.
DONG Y. Pesticide of cis-cypermethrin biodegradation law research[D]. Nanning: Guangxi University, 2012. |
| [4] |
李文艳, 杨宏莉, 蒋雪, 等. 氯氰菊酯和高效氯氰菊酯的生殖毒性研究进展[J]. 环境与健康杂志, 2007, 27(7): 652–654.
LI W Y, YANG H L, JIANG X, et al. Research progress on reproductive toxicity of cypermethrin and beta-cypermethrin [J]. J Environ Health, 2007, 27(7): 652–654. |
| [5] | ELHCTICHA A, DA’AS S I, KHAMAS W, et al. Evaluation of the toxic potentials of cypcrmcthrin pesticide on some reproductive and fertility parameters in the male rats[J]. Arch Environ Contaminat Toxicol, 2001, 41(4): 522–528. doi:10.1007/s002440010280 |
| [6] |
鲍清, 杨光, 任亚浩, 等. 乙体氯氰菊酯对小鼠大脑皮质 γ-氨基丁酸水平及相关酶和受体的影响
[J]. 实用预防医学, 2017, 24(11): 1300–1302.
BAO Q, YANG G, REN Y H, et al. Effects of beta-cypermethrin on γ-aminobutyric acid level, relevant enzyme and receptor in the cerebral cortex of mice [J]. Pract Prev Med, 2017, 24(11): 1300–1302. doi:10.3969/j.issn.1006-3110.2017.11.006 |
| [7] |
李海斌, 李君. 氯氰菊酯毒作用研究进展[J]. 环境与健康杂志, 2007, 24(5): 372–374.
LI H B, LI J. Advance on cypermethrine toxicity research[J]. J Environ Health, 2007, 24(5): 372–374. |
| [8] |
崔永. 农药毒死蜱和氯氰菊酯的遗传毒性研究[D]. 杭州: 浙江大学, 2006.
CUI Y. Investigations on genotoxicity of pesticides: chlorpyrifos and cypermethrin[D]. Hangzhou: Zhejiang University, 2006. |
| [9] |
徐洪亮. 小分子药物与牛血清白蛋白相互作用研究[D]. 长春: 吉林大学, 2013.
XU H L. Study on the interaction between small-molecular drugs and bovine serum albumin[D]. Changchun: Jilin University, 2013. |
| [10] |
徐飞, 张林群, 何立巍, 等. 泽泻醇类化合物与血清白蛋白相互作用的分子机理研究[J]. 化学学报, 2011, 69(19): 2228–2234.
XU F, ZHANG L Q, HE L W, et al. Molecular mechanism on the interaction between alisols and human serum albumin[J]. Acta Chimica Sinica, 2011, 69(19): 2228–2234. |
| [11] | BAADEN M, SANSOM M S P. OmpT: molecular dynamics simulations of an outer-membrane enzyme T[J]. Biophys J, 2004, 87(5): 2942–2953. doi:10.1529/biophysj.104.046987 |
| [12] | KOLLMAN P A, MASSOV I, REYES C, et al. Calculating Structures and free energies of complex molecules: combining molecular mechanics and continuum models[J]. Acc Chem Res, 2000, 33(12): 889–897. doi:10.1021/ar000033j |
| [13] | LYNE P D, LAMB M L, SAEH J C. Accurate prediction of the relative potencies of members of a series of kinase inhibitors using molecular docking and MM-GBSA scoring[J]. J Med Chem, 2006, 49(16): 4805–4808. doi:10.1021/jm060522a |
| [14] | DAS D, KOH Y, et al. Prediction of potency of protease inhibitors using free energy simulations with polarizable quantum mechanics-based lieand charees and a hybrid water model[J]. J Chem Inf Model, 2009, 49(12): 2851–2862. doi:10.1021/ci900320p |
| [15] | GOHIKE H, KIEL C, CASE D A. Insights into protein–protein binding by binding free energy calculation and free energy decomposition for the Ras–Raf and Ras–Ral GDS complexes[J]. J Mol Biol, 2003, 330(4): 891–913. doi:10.1016/S0022-2836(03)00610-7 |
| [16] | SHI S H, CHEN J Z, HU G D, et al. Molecular insight into the interaction mechanisms of inhibitors BEC and BEG with HIV-1 protease by using MM-PBSA method and molecular dynamics simulation[J]. J Mol Struct: Theochem, 2009, 913(1-3): 22–27. doi:10.1016/j.theochem.2009.07.010 |
| [17] | YANG X Q, LIU J Y, CHEN M H, et al. Key amino acid associated with acephate detoxification by Cydia pomonella carboxylesterase based on molecular dynamics with alanine scanning and site-directed mutagenesis [J]. J Chem Inf Model, 2014, 54(5): 1356–1370. doi:10.1021/ci500159q |
| [18] |
刘吉元. 蛋白质与配体相互作用分子模拟研究[D]. 杨凌: 西北农林科技大学, 2014.
LIU J Y. In silico study of protein-ligand interactions[D]. Yangling: Northwest A & F University, 2014. |
| [19] |
张建军, 高缘, 孙婉瑾. 白蛋白作为药物载体的研究[J]. 化学进展, 2011, 23(8): 1747–1754.
ZHANG J J, GAO Y, SUN W J. Albumin as drug carriers[J]. Progress Chem, 2011, 23(8): 1747–1754. |
| [20] | HE W Y, LI Y, TIAN J N, et al. Spectroscopic studies on binding of shikonin to human serum albumin[J]. J Photochem Photobiol A: Chem, 2005, 174(1): 53–61. doi:10.1016/j.jphotochem.2005.03.016 |
| [21] | CONG H, CHEN X M, CAI C, et al. Resonance light scattering spectrum of the alizarin green-CTMAB-nucleic acids system and determination of nucleic acids at nanogram levels[J]. Spectroscopy, 2007, 22(2): 64–72. |
| [22] | WANG W, DONINI O, REYES C M, et al. Biomolecular simulations: Recent developments in force fields, simulations of enzyme catalysis, protein-ligand, protein-protein, and protein-nucleic acid noncovalent interactions[J]. Annu Rev Biophys Biomol Struct, 2001, 30(1): 211–243. doi:10.1146/annurev.biophys.30.1.211 |
| [23] | MOREIRA I S, FERNANDES P A, RAMOS M J. Computational alanine scanning mutagenesis-An improved methodological approach for protein-DNA complexes[J]. J Comput Chem, 2007, 28(3): 644–654. doi:10.1002/(ISSN)1096-987X |
 2018, Vol. 20
2018, Vol. 20


