γ-氨基丁酸 (γ-aminobutyric acid,GABA) 受体是广泛分布于昆虫和脊椎动物中枢神经系统中的一种抑制性受体,受内源性配体 GABA 及一些重要药物的调节[1, 2, 3]。GABA 受体不仅与人的癫痫、焦虑、失眠、帕金森综合征及酒精中毒等神经性疾病有关,同时还是农药如阿维菌素、有机氯类和氟虫腈等的作用靶标[4, 5]。
氟虫腈是作用于 GABA 受体的一种苯基吡唑类杀虫剂,通过与昆虫 GABA 受体结合,阻断氯离子通道,最终导致昆虫神经和肌肉过度兴奋而死亡[6, 7, 8, 9]。其对半翅目、鳞翅目、鞘翅目害虫以及对环戊二烯类、菊酯类、氨基甲酸酯类杀虫药剂产生抗性的害虫均有较高的生物活性。
关于氟虫腈与昆虫 GABA 受体之间相互作用的研究非常重要。与放射性同位素标记法相比[10, 11, 12],荧光标记法具备高度灵敏性和极宽的动态响应时间,可高灵敏和高选择性地检测复杂生物分子,现已在蛋白质、核酸、细胞检测及免疫分析等方面得到广泛应用[13] 。该方法也已成功应用于 GABA 受体的研究中,尤其在药物与 GABA 受体相互作用的位点与活性方面,现已合成了 Ro7-NBD、Ro7-BD607、Ro7-AMCA、Biotin-1012S、Dansyl-Ro7 和 FITC-fipronil 等荧光探针,并用于研究药物与哺乳动物和鱼类GABA受体的结合作用[14, 15, 16, 17, 18, 19]。目前,尚未见采用荧光标记法研究昆虫GABA受体与配体相互作用的报道。
半胱酰胺环受体超家族 (Cys-loop receptor) 的成员有乙酰胆碱受体、GABA 受体及谷氨酸、甘氨酸和五羟色胺 3 受体[20, 21]。GABAA 受体是 GABA 受体的一个亚型[22]。由于半胱酰胺环家族中各成员之间的序列同源性较高,因此,可以基于已知三维结构的受体成员来构建 GABAA 受体模型,现多以乙酰胆碱受体为模板[23, 24]。目前在已鉴定出的昆虫 GABA 受体的 3 个亚基中,只有 RDL 亚基可形成功能性 GABA 门控氯离子通道[25, 26, 27, 28]。目前,有关昆虫 GABA 受体的晶体结构尚未见报道,但其三维结构可由同源建模得到,可通过分子对接研究受体与配体的相互作用[29] 。研究者利用同源建模的方法,以不同的模板模拟了昆虫 GABA 受体的三维结构,并研究了其与部分杀虫剂的相互作用及以 GABA 受体为靶点的杀虫剂对哺乳动物和昆虫的选择性[23, 30, 31, 32]。鉴于此,本研究利用荧光标记法和分子模拟法,分别研究了氟虫腈与昆虫 GABA 受体的相互作用,拟从实验和理论两方面揭示氟虫腈对昆虫产生强毒性的原因。
1 材料与方法 1.1 试剂与仪器改良型Bradford蛋白浓度测定试剂盒 (上海生工生化有限公司);三羟甲基氨基甲烷、蔗糖、异硫氰酸荧光素和 99.9% 氟虫腈 (fipronil) 原药等 (阿拉丁试剂有限公司);家蝇 Musca domestica L. 由南方农药创制中心 (上海基地) 提供;试验用水为蒸馏水。
TU 2120 型紫外分光光度计 (北京普析通用仪器设备有限公司);Cary Eclipse 荧光分光光度计 (美国VARIAN公司);L-80XP 超速离心机(美国Beckman公司);超滤管 (美国Millipore公司);优普超纯水制造器 (成都超纯科技有限公司)。
1.2 试验方法 1.2.1 家蝇脑 GABA 受体粗提物的制备参考文献[33]方法。取新鲜家蝇脑 6 g,加入冰冷的 Tris-HCl (pH=7.5,50 mmol/L) 缓冲液,0~4 ℃ 下研磨匀浆,1 000 g 离心 15 min;保留上清液,沉淀继续离心分离,重复 3 次。合并上清液,1.5×105 g 离心 40 min;沉淀复悬于上述缓冲液中,1.5×105g 离心 60 min,重复 3 次。取沉淀于液氮中保存,备用。采用 Bradford 法[19]测定受体粗提物中的蛋白浓度。
1.2.2 氟虫腈荧光探针 (FITC-氟虫腈) 的制备参考文献[19]方法。取异硫氰酸荧光素 155.6 mg (0.4mmol)、氟虫腈 349.6 mg (0.8 mmol) 和三乙胺 200 μL,加入 6 mL 重蒸DMF中,溶解后于 40 ℃ 下避光搅拌过夜。采用制备型薄层层析分离,以 V (氨水):V (甲醇):V (氯仿)=2:33:65 的混合液作洗脱液,以 V (氨水):V (甲醇):V (氯仿)=5:50:45 的混合液作提取液。过滤,60 ℃ 真空旋干,得到橘红色固体产物,其结构式见图式 1[19]。
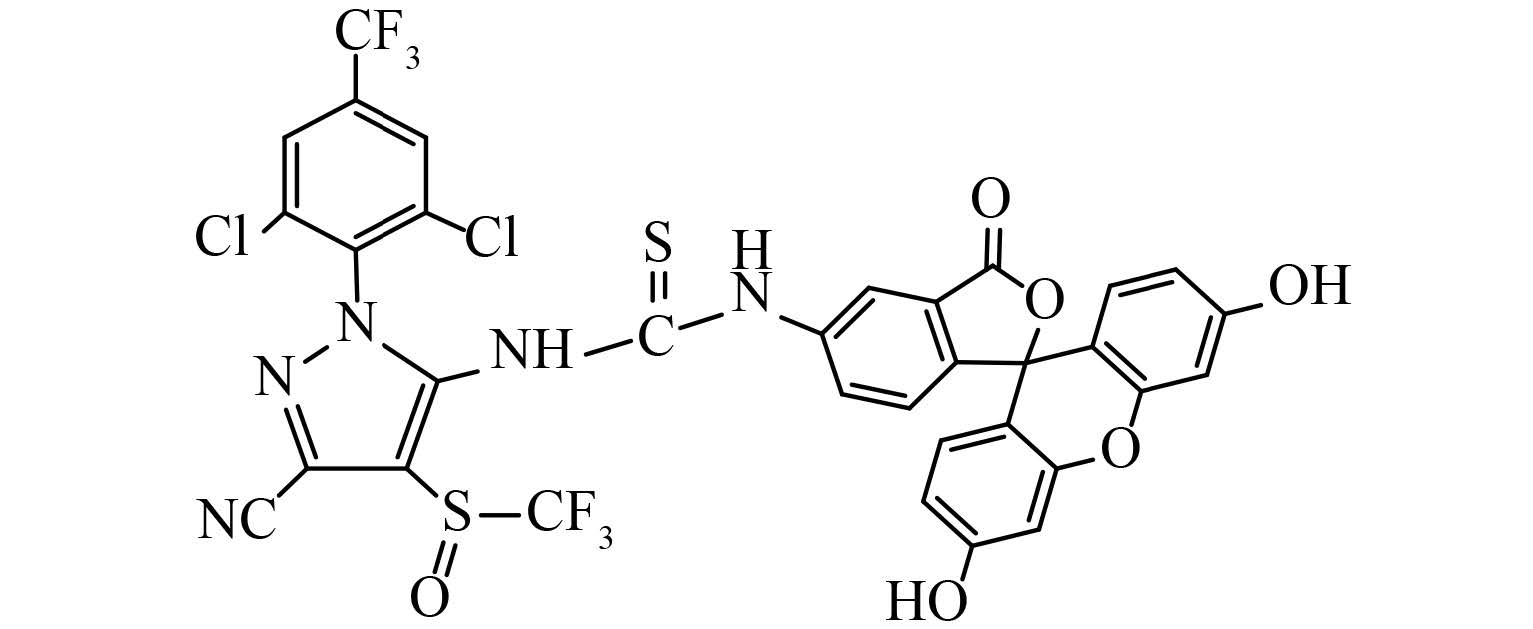
|
图式 1 氟虫腈荧光配体 Scheme 1 Structures of FITC-fipronil |
取 50 μL FITC-氟虫腈溶液 (终浓度为 2.5~ 250nmol/L) 置于 EP 管中,加入 930 μL 组织蛋白溶液 (含 318.68 μg 蛋白)。测定配体与受体的总结合时加入 20 μL Tris-HCl 缓冲液 (pH=7.5,50nmol/L);测定非特异性结合时加入 20 μL 1mmol/L 氟虫腈溶液,总体积为 1 mL。振荡 1min 后,在暗处 4 ℃ 条件下孵育 50 min,而后在 4 ℃、20 000 g 下离心 15 min;沉淀用 1 mL 冰 Tris-HCl 缓冲液 (pH=7.5,50 nmol/L) 冲洗,沉淀复悬于 1 mL 上述冰 Tris-HCl 缓冲液中,将溶液在 4 ℃、2×104 g 下离心 15min;沉淀用 1 mL 50% 的甲醇水溶液解离 100min;再次在 4 ℃、2×104 g 下离心 15 min;收集上清液 (S),测定其荧光强度。激发波长为 490 nm (入射和发射的狭缝宽均为 10 nm),在荧光光度计上记录 490~600 nm 波长范围内的发射光谱,平行重复 3 次。
1.3 计算方法采用Discovery Studio V3.1软件[34]进行分子模拟计算,若无另外说明,运行的参数均为软件默认的设置。
1.3.1 同源建模从 Swiss-Prot/TrEMBL 数据库[35]中得出果蝇 RDL 亚基的氨基酸序列 (序列登记号为:P25123)。由于 GABAA 受体与烟碱型乙酰胆碱受体属于同一蛋白家族,两者亚基的相似度为 60%,故烟碱型乙酰胆碱受体可以用来构建 GABAA 受体结构。目前,已有许多关于以乙酰胆碱受体为模板,构建 Cys-loop 受体的报道[36, 37, 38, 39, 40]。本研究选用电鳐烟碱型乙酰胆碱受体[24] (PDB ID:IOED) 为模板,删除目标序列胞外区的氨基酸和 M3 与 M4 之间的胞内 LOOP 区,采用 Align sequence 程序对果蝇 RDL 亚基的氨基酸序列与模板序列进行比对,采用同源建模 (homology modeling) 构建每个目标亚基的初始三维模型。使用 Discovery Studio 中的 Modeller 模块对同源建模得到的果蝇 RDL 受体进行优化,然后用拉式图 (Ramachandran plot) 对受体结构进行验证[31]。
1.3.2 分子对接对接前先对小分子氟虫腈加力场 (CHARMm)[41] 优化,再采用 CDOCKER 程序[42]进行分子对接计算。依据电生理学和突变试验证实的关键氨基酸残基确定受体的活性口袋,定义对接活性腔。而后将小分子配体放入活性口袋中进行构象搜索。最后综合考虑对接构象的能量、匹配度、氢键数及构象等,选择最佳构象。
1.3.3 复合物分子动力学模拟 (MD simulation)采用CHARMm力场对 RDL receptor-fipronil 复合物体系进行限制性动力学模拟,第一步:加显性溶剂环境,即将复合物体系的质心置于立方体水溶剂盒子的中心,水分子选用 TIP3P 模型。为保持体系电中性,再加抗衡离子 (阳离子为钠离子,阴离子为氯离子),55 个钠离子和 65 个氯离子,复合物到盒子壁的最小距离为 0.7 nm。第二步:对整个体系采用 1 fs 的积分步长,依次使用最陡下降法和共轭梯度法各进行 5 000 步的能量优化,以消除分子间的不合理碰撞。第三步:用 2 fs 的积分步长对体系进行 3 010 ps 的分子动力学模拟,包含升温 10 ps (温度:50~300 K)、平衡 2ns、采样 1 ns,每隔 1 000 步记录 1 次轨迹。在此模拟中,用 PME (Particle Mesh Ewald electrostatics) 方法处理静电[43],用 SHAKE 算法限制键长[44],范德华相互作用截取半径为 1.4 nm。
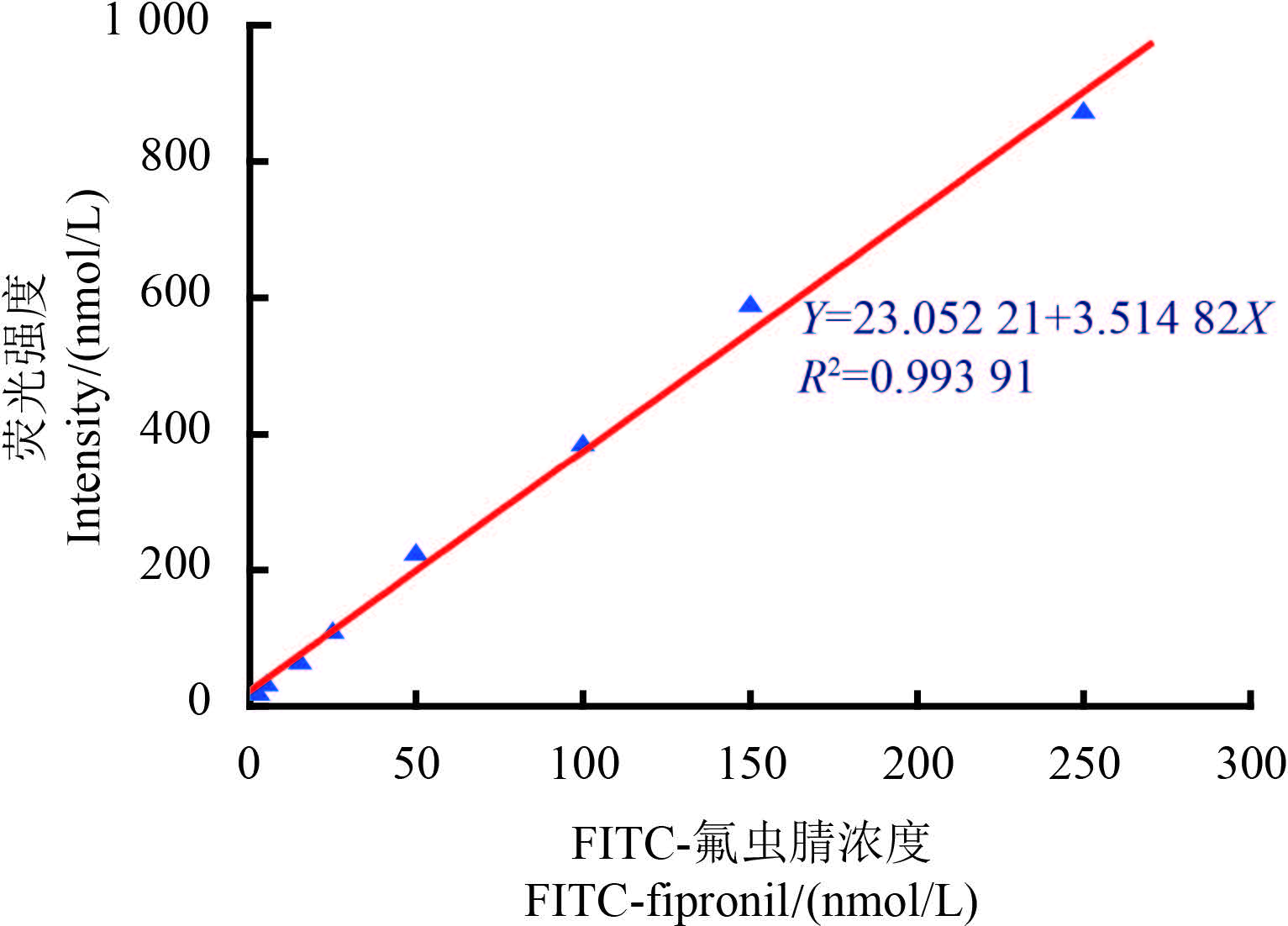
2 结果与讨论 2.1 荧光标记法测定结合常数FITC 激发和发射波长分别为 490 和 521 nm,荧光配体 FITC-氟虫腈的激发和发射波长分别为 490 和 515 nm,可见 FITC 在连接配体前后其激发及发射波长发生的变化不大。因受受体蛋白背景荧光的影响,荧光配体的荧光强度测定受到干扰。Janssen 等[45]通过醋酸缓冲液 (pH=4) 解离结合在受体蛋白上的 CLDEF 荧光配体,利用反相高效液相色谱法测得的 Bmax 和 Kd 值与放射性同位素标记法测得的结果相似。杨姗[19]等通过 50% 甲醇水溶液解离结合在 GABA 受体上的 Ro-1986,最终得到 Ro-1986 对受体的 Kd 值 67 nmol/L。异硫氰酸荧光素在酸性溶液中不稳定[46],荧光易消失,所以本研究采用 50% 甲醇水溶液对其进行解离。图 1 为 50% 甲醇水溶液解离下荧光强度随荧光配体浓度变化的标准曲线。
|
图 1 氟虫腈荧光配体在50%甲醇水溶液中的荧光强度与配体浓度的标准曲线 Fig. 1 Fluorescence intensity vs. FITC-fipronil concentration in 50% methanol |
由图 1 可知:荧光强度随荧光配体浓度增加而逐渐增强,两者呈线性相关。Scatchard 方程可用于药物与蛋白质、核酸等受体相互作用的研究中,为受体亲和力及亲和常数的定量研究提供了有效方法[47],其表达式如下:

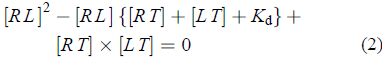
[RL]:平衡时配基-受体复合物浓度,nmol/L
[L]:平衡时游离配基浓度,nmol/L
[RT]:受体的初始浓度,nmol/L
[LT]:总配基浓度,nmol/L
Kd:解离平衡常数,nmol/L
(2) 式是双曲线的一元二次方程,当 [RT]、Kd 不变时,[RL] 随 [LT] 的增大开始增加很快,后趋于平缓。Kd 值是衡量配基与受体相互结合能力的一个重要物理量,Kd 值越小,表示结合能力越大,即亲和能力越大。通过饱和试验研究 FITC-氟虫腈与家蝇脑的 GABA 受体结合特性,绘制饱和曲线可求出 [RT] 及 Kd 值。
图 2a 为 FITC-氟虫腈的饱和曲线。由图可知,随 FITC-氟虫腈浓度增加 (0~300 nmol/L),FITC-氟虫腈与受体结合逐渐趋向饱和。图 2b 为 Scatchard 图,由该图可得 [RT]和Kd 值。由 Scatchard 图得出的 [RT] 和 Kd 值与饱和曲线求出的相同,[RT] 值为 (21.3±2.5) pmol/mg protein,Kd 值为 (109±9) nmol/L。这与前人研究的结论一致 (荧光标记法得到的 Kd 值与放射性标记法得到的 Kd 值常常有几倍的差距,虽然放射性同位素标记法在研究 GABA 受体中具有其自身的优越性,但存在对人体伤害大、产生的放射性废物不易处理等问题,使得荧光标记法越来越被人们所运用[11, 19])。Abalis 等[48]利用放射性同位素标记法研究得出,氟硝西泮与家蝇胸部 GABA 受体 Kd 值为 24.3 nmol/L;Rosario 等[49]研究得出 [3H]GABA 与果蝇中枢神经系统 GABA 受体 Kd值为 23nmol/L。由此可知,氟虫腈与昆虫 GABA 受体有较高的亲和力,进而对昆虫产生强毒性。
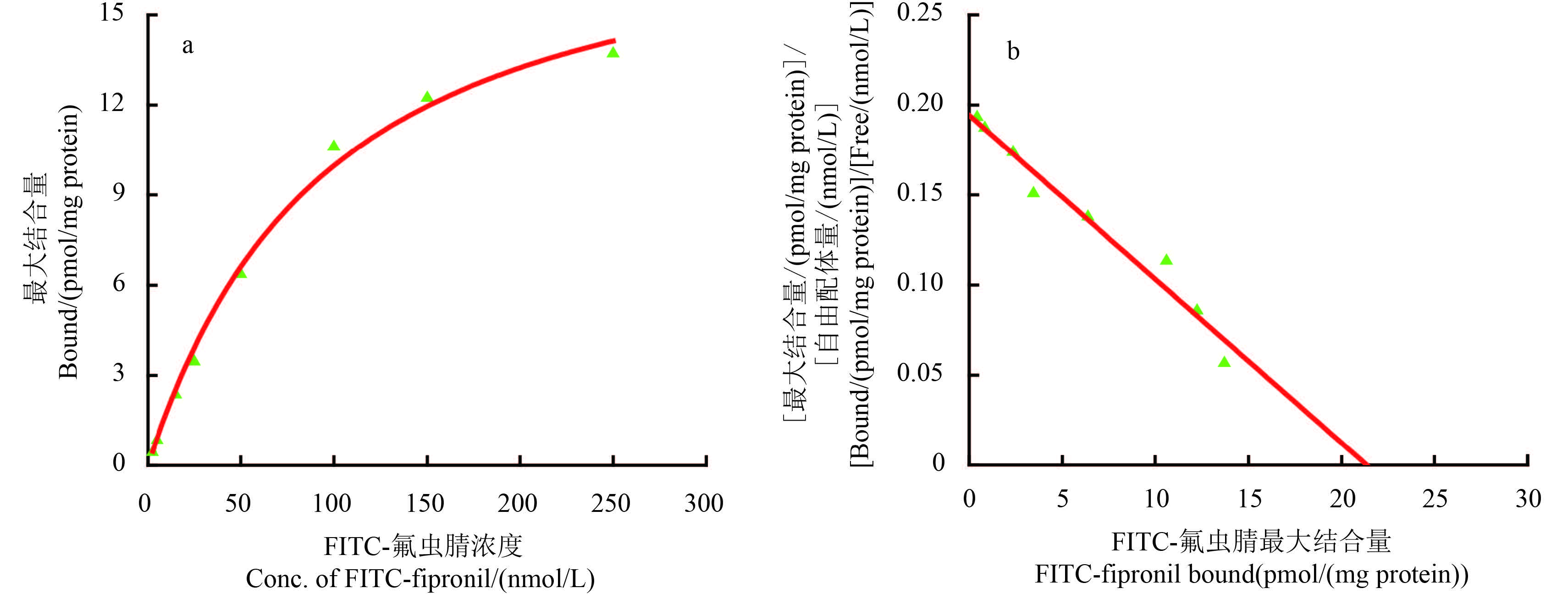
|
图 2 氟虫腈荧光配体与受体结合的饱和曲线(a)和Scatchard图(b) Fig. 2 Saturability (a) and Scatchard analysis(b) of FITC-fipronil binding to the receptor |
模板烟碱型乙酰胆碱受体与果蝇 RDL 亚基比对的一致性为 22.4%。图 3 为烟碱型乙酰胆碱受体 β 亚基与果蝇 RDL 亚基的序列比对,蓝色阴影为相似氨基酸,颜色越深,相似度越高。
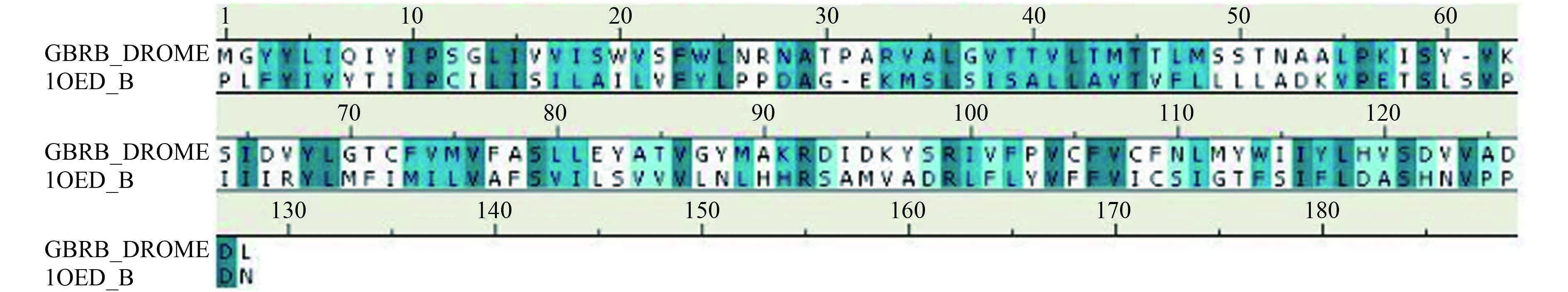
|
图 3 果蝇RDL亚基与模板的序列比对 Fig. 3 Alignment of the acid sequence of fruit fly RDLR subunit with the template |
图 4a 为果蝇 RDL 受体跨膜区模型,5 个亚基均一致。利用 PROCHECK 程序对所建模型进行评价,其结果通过程序生成的 Ramachandran plot 来检测蛋白主链构型中氨基酸残基 ψ-Ψ 二面角的合理性。图 4b 为 Ramachandran plot 图,分析得出,大量氨基酸残基位于 ψ=-60°、Ψ=-45° 处,与受体结构中有较多的 α 螺旋结构的特征吻合。从拉式图可看出,有 99.7% 的氨基酸残基位于允许范围,只有 0.3% 落在了不合理区 (拉式图说明:位于蓝色线内表示有利区的氨基酸,位于紫红色线内表示可行区的氨基酸,紫红色线外表示不可行区的氨基酸)。由此表明构建的模型合理。
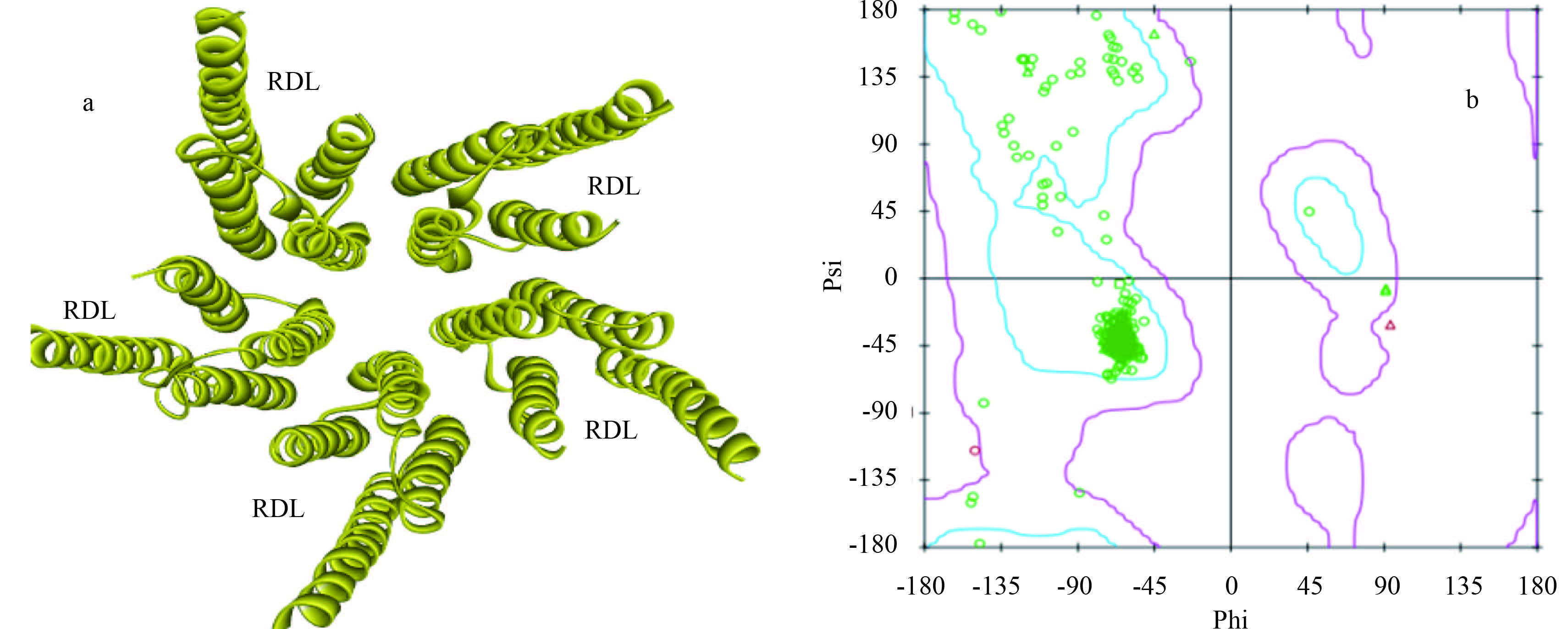
|
图 4 果蝇RDL受体跨膜区结构(a)及果蝇RDL受体模型的Ramachandran plot 图(b) Fig. 4 Transmembrane domain of fruit fly RDL receptor (a) and Ramachandran plot of fruit fly RDL receptor (b) |
只有确定了起决定性作用的氨基酸残基,才能定义小分子配体与受体的活性腔。由文献可知,在果蝇 RDL 受体同源五聚体中,Ala301、Thr305 及 Leu308 与非竞争性拮抗剂的结合起关键性作用[50]。因此利用位于受体通道内侧的氨基酸残基—— Ala301、Thr305 及 Leu308定义半径为 1.5 nm 的对接活性腔,见图 5。

|
图 5 氟虫腈与RDL受体的结合位点 Fig. 5 Fipronil binding sites in RDL receptor |
果蝇 RDL 受体与氟虫腈的 CDOCKER 相互作用能为 –137.93 kJ/mol,图 6a 为氟虫腈与果蝇 RDL 受体的对接结果。在果蝇 RDL 受体中,氟虫腈位于通道 Ala301~ Leu308 的中间部位,苯环及其取代基朝向胞外区,吡唑环朝向胞内区一端。图 6a 显示,氟虫腈的三氟甲基亚磺酰基朝向 Ala301,氨基和吡唑环在受体的极性 Thr305 旁边,苯环和取代的三氟甲基在 Leu308 旁边;图 6b 为相互作用时氟虫腈周围 0.5 nm 内的氨基酸,其中氟虫腈用球棍状表示,氢键以绿色虚线标注。表 1 为氟虫腈与该受体形成的氢键信息。受体中的氨基酸 Thr 分别与氟虫腈的三氟甲基亚磺酰基的 F 原子和 S 原子形成氢键,氢键的距离均小于 0.3 nm。
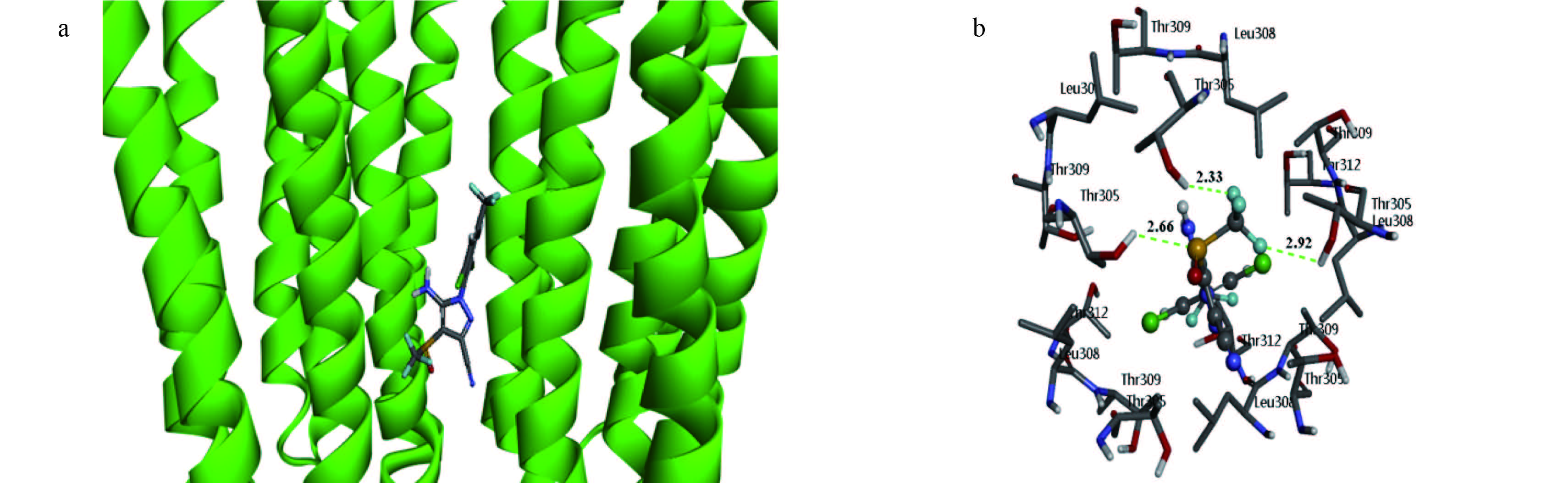
|
图 6 氟虫腈与RDL受体的作用位置(a)及氟虫腈与RDL受体作用时 0.5 nm 内的氨基酸(b) Fig. 6 Interaction position of fipronil in RDL receptor (a) and residues in RDL receptor with 0.5 nm of fipronil (b) |
|
|
表 1 氟虫腈与RDL受体作用的氢键 Table 1 Hydrogen bonds formed between fipronil and RDL receptor |
由图 7 可知:FITC-氟虫腈与 RDL 受体作用是其氟虫腈部分与之产生了 3 条氢键,其 CDOCKER 相互作用能为 –137.93 kJ/mol,这与氟虫腈与 RDL 受体作用产生的结果一致。由图 8 知,FITC 与 RDL受体对接无法产生构象,表明 FITC-氟虫腈与 RDL 受体发生作用的是氟虫腈。对接结果显示,果蝇 RDL 受体中 Ala301 至 Leu308 间的区域内有与氟虫腈结合的位点,这与已有文献报道[32, 50]基本一致。由 CDOCKER 相互作用能和所形成的氢键数目及长度可知,配体氟虫腈与 RDL 受体形成的复合物较稳定,表明氟虫腈与 RDL 受体具有高亲和力,进而产生毒性。
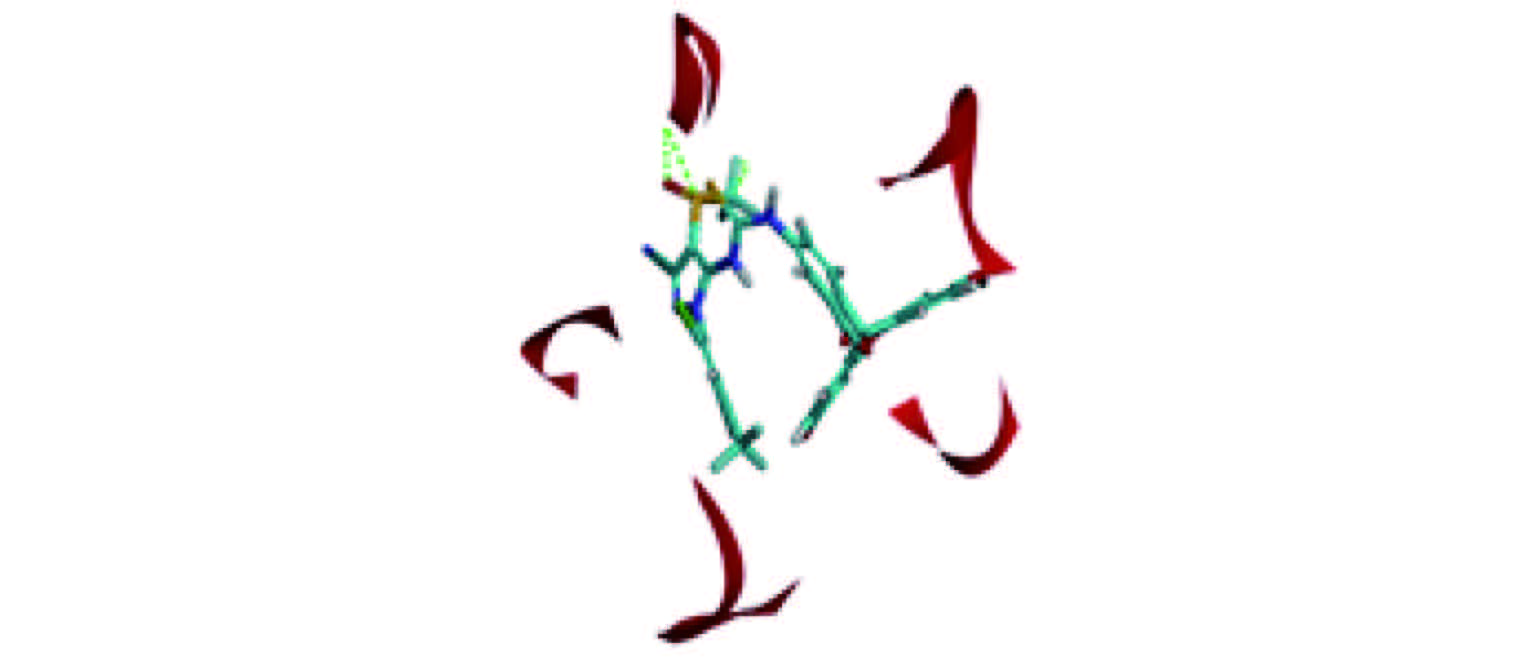
|
图 7 FITC-氟虫腈与RDL受体作用 Fig. 7 Interaction of FITC-fipronil in RDL receptor |
|
图 8 FITC与RDL受体的对接 Fig. 8 Docking of FITC with RDL receptor |
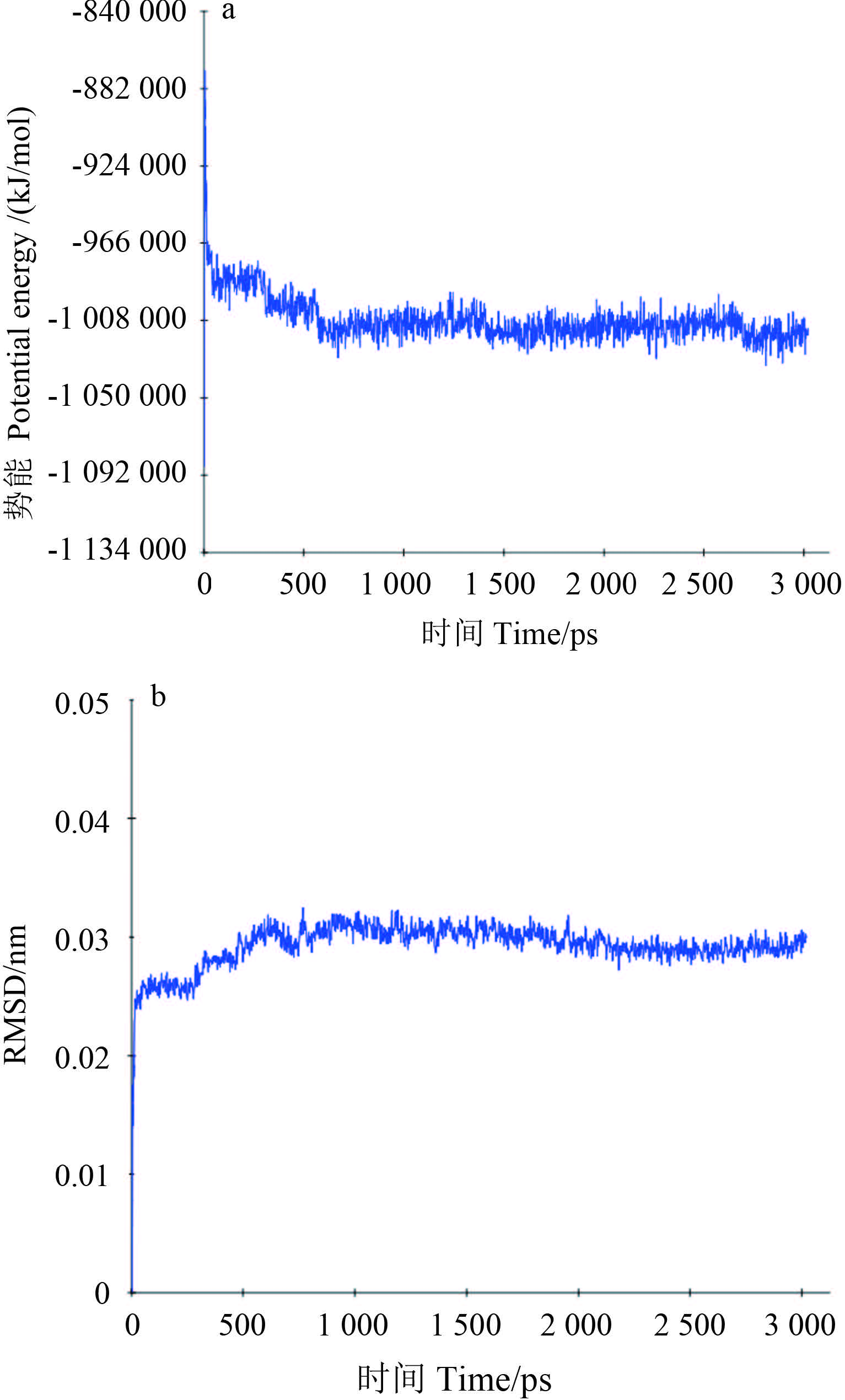
为考查对接结果的稳定性,将对接得到的 RDL 受体-氟虫腈复合物的最佳构象进行 MD 模拟,结果见图 9。表 2 给出了 500 ps 后氟虫腈与 RDL 受体形成的氢键。由图 9a、9b 可知,RDL 受体-氟虫腈复合物的势能在 600 ps 后趋向平衡,达到稳定状态,体系的 RMSD 值基本稳定,在 0.032nm 附近波动。由表 2 知,在动力学模拟后,复合物体系形成了 6 个氢键,其中频率大于 40% 的有 2 个:A 链 Thr312 羟基 H 原子与氟虫腈苯环上三氟甲基的F原子形成的,频率为 59.0%;D 链 Thr309 羟基 H 原子与氟虫腈苯环上取代的 Cl 原子形成的,频率为 45.6%。这些氢键出现的几率较大,对维持复合物的稳定性起到了很大作用,从氢键稳定性的角度解释了氟虫腈对果蝇高毒性的机理,这与荧光标记试验结论一致。两种方法均表明氟虫腈与昆虫 GABA 受体有较强的相互作用,显示出了它们之间具有高亲和性,进而产生毒性。
|
图 9 RDL 受体-氟虫腈复合物势能(a)和 RMDS值(b) 随时间变化的情况 Fig. 9 Potential energy variation(a) and RMSD variation(b) of fruit fly RDL receptor-fipronil complex varying with time |
|
|
表 2 RDL 受体-氟虫腈复合物MD模拟后的氢键 Table 2 Hydrogen bonds formed between RDL recepor-fipronil complex by MD simulation |
本研究采用荧光标记法及分子模拟法研究了氟虫腈对昆虫 GABA 受体的作用。荧光标记法得出的亲和常数值证明了氟虫腈与昆虫 GABA 受体之间具有高亲和性;分子模拟法中的 CDOCKER 相互作用能、氢键及 MD 模拟结果也表明氟虫腈与昆虫 GABA 受体之间存在高亲和性。两种方法均验证了氟虫腈与昆虫 GABA 受体之间具有的较强亲和性是导致氟虫腈对昆虫产生高毒性的原因。本研究为研制高效绿色农药提供了理论指导。
| [1] | ENNA S J, MÖHLER H. The GABA receptors[M]. Totowa, New Jersey:Humana Press, 2007:28-29. |
| [2] | KLEIN R L, HARRIS R A. Regulation of GABAA receptor structure and function by chronic drug treatments in vivo and with stably transfected cells[J]. Jap J Pharmacol, 1996, 70(1):1-15. |
| [3] | MICHELS G, MOSS S J. GABAA receptors:properties and trafficking[J]. Crit Rev Biochem Mol Biol, 2007, 42(1):3-14. |
| [4] | CASIDA J E. Insecticide action at the GABA-gated chloride channel:recognition, progress, and prospects[J]. Arch Insect Biochem Physiol, 1993, 22(1-2):13-23. |
| [5] | PRADA C, UDIN S B, WIECHMANN A F, et al. Stimulation of melatonin receptors decreases calcium levels in Xenopus tectal cells by activating GABAC receptors[J]. J Neurophysiol, 2005, 94(2):968-978. |
| [6] | SCHOFIELD P R, Darlison M G, Fujita N, et al. Sequence and functional expression of the GABAA receptor shows a ligand-gated receptor super-family[J]. Nature, 1987, 328(6127):221-227. |
| [7] | ANZELIUS M, EKSTRÖM P, MÖHLER H, et al. Immunocytochemical localization of GABAA receptor β2/β3-subunits in the brain of Atlantic salmon (Salmo salar L.)[J]. J Chem Neuroanat, 1995, 8(3):207-221. |
| [8] | MEHTA A K, TICKU M K. An update on GABAA receptors[J]. Brain Res Rev, 1999, 29(2/3):196-217. |
| [9] | IKEDA T, NAGATA K, KONO Y, et al. Fipronil modulation of GABAA receptor single-channel currents[J]. Pest Manag Sci, 2004, 60(5):487-492. |
| [10] | CASALOTTI S O, STEPHENSON F A, BARNARD E A. Separate subunits for agonist and benzodiazepine binding in the γ-aminobutyric acid A receptor oligomer[J]. J Biol Chem, 1986, 261(32):15013-15016. |
| [11] | BRAESTRUP C, ALBRECHTSEN R, SQUIRES R F. High densities of benzodiazepine receptors in human cortical areas[J]. Nature, 1977, 269(5630):702-704. |
| [12] | NIELSEN M, BRAESTRUP C, SQUIRES R F. Evidence for a late evolutionary appearance of brain-specific benzodiazepine receptors:an investigation of 18 vertebrate and 5 invertebrate species[J]. Brain Res, 1978, 141(2):342-346. |
| [13] | LIN C G, ZhANG G L, YANG J H. Fluorimetric determination of nucleic acids with terbium and lutetium[J]. Microchem J, 2002, 71(1):9-14. |
| [14] | DENG Y L, PALMER C J, CASID J E. House fly brain γ-aminobutyric acid-gated chloride channel:target for multiple classes of insecticides[J]. Pestic Biochem Phys, 1991, 41(1):60-65. |
| [15] | SATTELLE D B, HOLYOKE C W, SCHNEE M E, et al. A bicyclic dinitrile radioligand for studying GABA-gated Cl- channels[J]. J Physiol, 1995, 483.P:193P |
| [16] | GANT D B, CHALMERS A E, WOLFF M A, et al. Fipronil:action at the GABA receptor[J]. Rev Toxicol, 1998, 2:147-156. |
| [17] | NISHIWAKI H, NAKAGAWA Y, TAKEDA D Y, et al. Binding activity of substituted benzyl derivatives of chloronicotinyl insecticides to housefly-head membranes, and its relationship to insecticidal activity against the housefly Musca domestica[J]. Pest Manag Sci, 2000, 56(10):875-881. |
| [18] | ALAM M S, KAJIKI R, HANATANI H, et al. Synthesis and structure-activity relationships of 1-phenyl-1H-1,2,3-triazoles as selective insect GABA receptor antagonists[J]. J Agric Food Chem, 2006, 54(4):1361-1372. |
| [19] | 杨姗, 张博, 张元, 等. 荧光标记法研究鱼类GABA受体与Ro7-1986/1和Fipronil的相互作用[J]. 高等学校化学学报, 2015, 36(3):456-462. YANG S, ZHANG B, ZHANG Y, et al. Interaction of GABA receptor in the fish brain with Ro7-1986/1 and Fipronil by fluorescent-labeled ligand[J]. Chem J Chin Uinv, 2015, 36(3):456-462. |
| [20] | RAYES D, DE ROSA M J, SINE S M, et al. Number and locations of agonist binding sites required to activate homomeric cys-loop receptors[J]. J Neurosci, 2009, 29(18):6022-6032. |
| [21] | ACCARDI M V, BEECH R N, FORRESTER S G. Nematode cys-loop GABA receptors:biological function, pharmacology and sites of action for anthelmintics[J]. Invertebr Neurosci, 2012, 12(1):3-12. |
| [22] | BERTRAND S, NG G Y K, PURISAI M G, et al. The anti-convulsant, antihyperalgesic agent gabapentin is an agonist at brain γ-aminobutyric acid type B receptors negatively coupled to voltage-dependent calcium channels[J]. J Pharmacol Exp Ther, 2001, 298(1):15-24. |
| [23] | MCGONIGLE I, LUMMIS S C R. RDL receptors[J]. Biochem Soc Trans, 2009, 37(6):1404-1406. |
| [24] | MIYAZAWA A, FUJIYOSHI Y, UNWIN N. Structure and gating mechanism of the acetylcholine receptor pore[J]. Nature, 2003, 423(6943):949-955. |
| [25] | FFRENCH-CONSTANT R H, MORTLOCK D P, SHAFFER C D, et al. Molecular cloning and transformation of cyclodiene resistance in Drosophila:an invertebrate γ-aminobutyric acid subtype A receptor locus[J]. Proc Natl Acad Sci USA, 1991, 88(16):7209-7213. |
| [26] | ARONSTEIN K, AULD V, FFRENCH-CONSTANT R. Distribution of two GABA receptor-like subunits in the Drosophila CNS[J]. Invert Neurosci, 1996, 2(2):115-120. |
| [27] | HARVEY R J, SCHMITT B, HERMANS-BORGMEYER I, et al. Sequence of a Drosophila ligand-gated ion-channel polypeptide with an unusual amino-terminal extracellular domain[J]. J Neurochem, 1994, 62(6):2480-2483. |
| [28] | HENDERSON J E, SODERLUND D M, KNIPPLE D C. Characterization of a putative γ-aminobutyric-acid (GABA) receptor β-subunit gene from Drosophila melanogaster[J]. Biochem Biophys Res Commun, 1993, 193(2):474-482. |
| [29] | JORGENSEN W L. Rusting of the lock and key model for protein-ligand binding[J]. Science, 1991, 254(5034):954-955. |
| [30] | CHENG J, JU X L, CHEN X Y, et al. Homology modeling of human α1β2γ2 and house fly β3 GABA receptor channels and Surflex-docking of fipronil[J]. J Mol Model, 2009, 15(9):1145-1153. |
| [31] | 汪小芬, 任天瑞, 姚建华. 氟虫腈与斑马鱼GABAA及果蝇RDL受体作用的差异性研究[J]. 农药学学报, 2014, 16(3):259-270.WANG X F, REN T R, YAO J H. Differences of interactions between fipronil with zebrafish GABAA and Drosophila melanogaster RDL receptor[J]. Chin J Pestic Sci, 2014, 16(3):259-270. |
| [32] | ZHENG N, CHENG J G, ZHANG W H, et al. Binding difference of fipronil with GABAARs in fruitfly and zebrafish:insights from homology modeling, docking, and molecular dynamics simulation studies[J]. J Agric Food Chem, 2014, 62(44):10646-10653. |
| [33] | JANSSEN M J, STEGEMAN M. Solubilized benzodiazepine receptors for use in receptor assays[J]. J Pharm Biomed Anal, 1996, 14(8-10):989-996. |
| [34] | Accelrys Inc. BIOVIA discovery studio[CP/OL]. (2008-12-07)[2015-10-09]. http://accelrys.com/products/discovery-studio/. |
| [35] | US. National Library of Medicine. NCBI databases[DB/OL]. (1988-11-04)[2015-10-09]. http://www.ncbi.nlm.nih.gov/guide. |
| [36] | LAW R J, LIGHTSTONE F C. Modeling neuronal nicotinic and GABA receptors:important interface salt-links and protein dynamics[J]. Biophys J, 2009, 97(6):1586-1594. |
| [37] | CHANGEUX J P. The nicotinic acetylcholine receptor:the founding father of the pentameric ligand-gated ion channel superfamily[J]. J Biol Chem, 2012, 287(48):40207-40215. |
| [38] | ASHBY J A, MCGONIGLE I V, PRICE K L, et al. GABA binding to an insect GABA receptor:a molecular dynamics and mutagenesis study[J]. Biophys J, 2012, 103(10):2071-2081. |
| [39] | LUMMIS S C R. Locating GABA in GABA receptor binding sites[J]. Biochem Soc Trans, 2009, 37(6):1343-1346. |
| [40] | HARRISON N J, LUMMIS S C R. Locating the carboxylate group of GABA in the homomeric rho GABAA receptor ligand-binding pocket[J]. J Biol Chem, 2006, 281(34):24455-24461. |
| [41] | BROOKS B R, BRUCCOLERI R E, OLAFSON B D, et al. CHARMM:A program for macromolecular energy, minimization, and dynamics calculations[J]. J Comput Chem, 1983, 4(2):187-217. |
| [42] | WU G S, ROBERTSON D H, BROOKS III C L, et al. Detailed analysis of grid-based molecular docking:a case study of CDOCKER-A CHARMm-based MD docking algorithm[J]. J Comput Chem, 2003, 24(13):1549-1562. |
| [43] | ESSMANN U, PERERA L, BERKOWITZ M L, et al. A smooth particle mesh Ewald method[J]. J Chem Phys, 1995, 103(19):8577-8593. |
| [44] | KRÄUTLER V, VAN GUNSTEREN W F, HÜNENBERGER P H. A fast SHAKE algorithm to solve distance constraint equations for small molecules in molecular dynamics simulations[J]. J Comput Chem, 2001, 22(5):501-508. |
| [45] | JANSSEN M J, ENSING K, DE ZEEUW R A. A fluorescent receptor assay for benzodiazepines using coumarin-labeled desethylflumazenil as ligand[J]. Anal Chem, 2001, 73(13):3168-3173. |
| [46] | 李勇, 何俊, 但德忠, 等. 异硫氰酸荧光素标记人血清白蛋白[J]. 华西药学杂志, 2001, 16(1):25-26. LI Y, HE J, DAN D Z, et al. Labeling of human serum albumin with fluorescein isothiocyanate[J]. West China J Pharm Sci, 2001, 16(1):25-26. |
| [47] | MA J K H, JUN H W, LUZZI L A. Determination of equilibrium constants and binding capacities using a modified scatchard method in drug-protein binding studies[J]. J Pharm Sci, 1973, 62(12):2038-2040. |
| [48] | ABALIS I M, ELDEFRAWI M E, ELDEFRAWI A T, et al. Biochemical identification of putative GABA/benzodiazepine receptors in house fly thorax muscles[J]. Pestic Biochem Phys, 1983, 20(1):39-48. |
| [49] | ROSARIO P, BARAT A, RAMIREZ G. Characterization of binding sites for[3H] GABA in Drosophila melanogaster CNS membranes[J]. Neurochem Int, 1989, 15(1):115-120. |
| [50] | CHEN L G, DURKIN K A, CASIDA J E. Structural model for γ-aminobutyric acid receptor noncompetitive antagonist binding:Widely diverse structures fit the same site[J]. Proc Nat Acad Sci USA, 2006, 103(13):5185-5190. |
 2016, Vol. 18
2016, Vol. 18


