2. 中国科学院 上海有机化学研究所, 上海 200032
2. Shanghai Institute of Organic Chemistry, Chinese Academy of Sciences, Shanghai 200032, China
氟虫腈(fipronil,CAS:120068-37-3,结构式见Scheme 1)是首个应用于有害生物防治的苯基吡唑类杀虫剂,能与昆虫γ-氨基丁酸受体(GABAR)结合,阻断氯离子通道,进而引起神经和肌肉过度兴奋而死亡[1,2]。环境毒理学研究发现,氟虫腈对生态环境具有潜在的危险性,对水生生物(如鱼、虾和蟹等)有很高的毒性[3,4]。
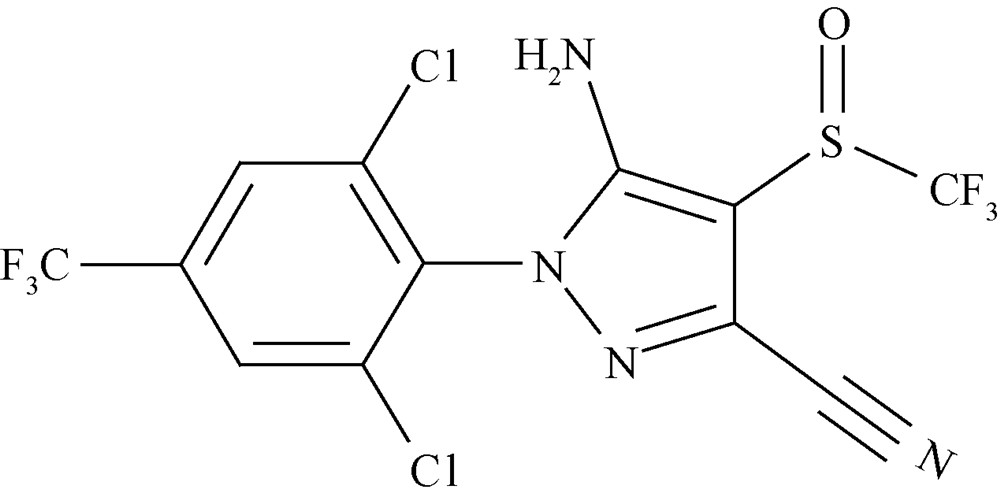 | Scheme 1 |
GABAR是脊椎动物中枢神经系统、昆虫中枢系统及周缘神经系统中的抑制性受体,受内源性配体γ-氨基丁酸以及药理学上和临床上一些重要药物的调节[5]。GABAA受体(GABAAR)是GABAR中最重要的受体之一,是木防己苦毒宁(picrotoxinin)和一些杀虫剂(如林丹、硫丹和氟虫腈类)的主要作用位点,这些化合物作为非竞争性拮抗剂与GABAAR结合,阻断受体通道,导致动物中枢神经系统兴奋过度[6,7,8]。α1β2γ2GABAAR是脊椎动物GABAAR中最主要的亚型[9],5个亚基中心形成一个直径约为0.5 nm的氯离子通道[10]。昆虫GABAR是一些杀虫剂的重要靶标。目前,放射性配体结合试验和电生理学技术已经在昆虫毒力学研究中得到应用,昆虫RDL(resistance to dieldrin)受体(RDLR)具有GABAR的功能,但在药理学方面与脊椎动物的GABAAR存在差异[11]。目前,仅发现3种昆虫GABAR的亚基,分别为RDL亚基、GRD亚基和LCCH3亚基[12]。其中,RDL亚基在表达系统中形成功能性的同源五聚体,可以作为昆虫GABAR的模型来研究其药理学特征[13]。
目前,有关GABAR的研究报道主要集中在电生理方面,脊椎动物斑马鱼(Danio rerio)和昆虫GABAR的晶体结构尚未见报道,GABAR的结构主要还是通过同源建模获得。GABAR与配体的相互作用研究主要是通过分子对接[14]的方法来实现。
Alam等采用3D-QSAR方法对所合成的一系列GABAR拮抗剂(苯基-1,2,3-三唑类化合物)的结构-活性关系进行了研究,并通过分子对接的方法研究了这些化合物对β3同源五聚体和异源GABAAR的选择性[15]。Law等用分子对接的方法讨论了木防己苦毒宁、TBPS(t-butylbicyclophosphorothio-nate)、EBOB(ethynylbicycloorthobenzoate)以及硫丹等GABAR非竞争性拮抗剂的作用位点[16]。Campagna-Slater等通过同源建模研究了GABAAR的离子通道的结构,并对作用于GABAAR上的药物位点进行了讨论[17]。次素琴等通过构建人的β3同源五聚体来模拟昆虫GABAR的结构,并研究了部分杀虫剂与β3同源五聚体模型的相互作用[18]。程瑾等还构建了家蝇β3同源五聚体模型,并研究了部分以GABAR为靶点的杀虫剂对哺乳动物和昆虫的选择性[19]。McGonigle等采用乙酰胆碱结合蛋白为模板建立了RDL受体(RDLR)胞外区的三维结构[20]。
巨修炼等采用同源建模和分子对接的方法,构建了斑马鱼GABAAR的一种亚型(ρ亚基组成的同源五聚体),并探讨了氟虫腈及其衍生物与受体之间的相互作用[21]。这些工作只是初步研究了斑马鱼GABAAR与配体的相互作用,但没有研究斑马鱼GABAAR与配体复合物体系的微观运动,斑马鱼GABAAR和果蝇RDLR与氟虫腈结合的差异性研究也未见报道。
在此,本文将介绍氟虫腈与黑腹果蝇Drosophila melanogaster RDLR和斑马鱼Danio rerio α1β2γ2GABAAR(以下用GABAAR表述)作用的差异。即采用同源建模方法,构建斑马鱼GABAAR和果蝇RDLR,比较两受体与氟虫腈作用位点的差异;采用分子对接的方法,研究氟虫腈与两受体的作用模式;采用分子动力学方法比较氟虫腈与两受体作用的稳定性,从分子水平解释氟虫腈阻断果蝇RDLR和斑马鱼GABAAR氯离子通道的作用机理。
1 计算方法
计算工作均在DELL precision T3500工作站上完成,采用Discovery Studio V3.1软件包[22]进行分子模拟计算,除特殊说明外,运行的参数均为软件默认设置。
1.1 同源模建从Swiss-Prot/TrEMBL数据库[23]中得到斑马鱼α1、β2、γ2亚基和果蝇RDL亚基的氨基酸序列(序列登记号分别为:AAI24698,AAI15079,XP_687331和P25123)。 鉴于GABAAR与烟碱型乙酰胆碱受体属于同一蛋白家族,二者亚基的相似度为60%,因此,烟碱型乙酰胆碱受体可以用于构建GABAAR结构。目前,已有许多关于以乙酰胆碱受体作为模板构建Cys-Loop受体的报道[24,25,26,27,28]。本研究选取电鳐烟碱型乙酰胆碱受体[29](PDB ID:1OED,分辨率0.4 nm)作为模板,删除目标序列胞外区的氨基酸和M3与M4之间的胞内LOOP区,采用Align sequence程序对斑马鱼α1、β2、γ2亚基及果蝇RDL亚基的氨基酸序列与模板序列进行比对,依据比对结果和电鳐乙酰胆碱受体的三维结构,采用同源模建(homology modeling)模块搭建每个目标亚基的初始三维模型。每个亚基生成5个模型,选取PDF total energy值[30]和DOPE Score值[31]均较低的模型作为初始模型。之后利用叠合模块(Superimpose) 将优化后的亚基分别与模板亚基进行叠合组装,即得到具有相对空间排列的斑马鱼GABAA异源五聚体受体和果蝇RDL同源五聚体受体模型。
1.2 模型优化评估采用CHARMm力场[32]对同源模建的初始结构进行steepest descent和conjugate gradient 能量最小化各5 000步,使结构的势能收敛值达到0.042 kJ/(mol·nm)。然后,对能量最小化的受体结构进行分子动力学(MD)模拟。主要包括:2 ps的体系升温过程(温度从50~300 K),轨迹记录频率(output frequency)为每隔100步记录1次;2 ns的平衡过程和500 ps的系统采样过程,轨迹记录频率为每隔1 000步记录1次,模拟中采用的积分步长为1 fs。在该模型中,采用隐性溶剂环境。优化过程采用Procheck[33]程序评估受体结构的合理性。
1.3 分子对接在CHARMm力场下优化氟虫腈的结构,采用CDOCKER程序[34]进行分子对接计算。根据电生理学研究和突变试验证实的关键氨基酸残基确定受体的活性口袋,定义对接活性球。将配体放入活性口袋内进行多构象搜索。利用CHARMm力场对复合物进行模拟退火,以优化对接构象。将得到的配体构象进行聚类分析(RMSD≤0.1 nm),分析对接能量较低的前30个构象,根据聚类情况和对接打分选取最佳构象。
1.4 受体-配体复合物分子动力学模拟采用CHARMm力场对受体-氟虫腈复合物体系进行限制性动力学模拟。主要包括以下步骤:首先对体系添加显性溶剂环境,即将复合物体系的质心置于立方体水溶剂盒子的中心,水分子选用TIP3P模型。为保持体系电中性,其中添加抗衡离子(阳离子为Na+,阴离子为Cl-)。在斑马鱼GABAAR-氟虫腈体系中添加57个Cl-和37个Na+,在果蝇RDLR-氟虫腈体系中添加65个Cl-和55个Na+,复合物至盒子壁的最小距离为0.7 nm。然后对整个体系采用1 fs的积分步长,依次用最陡下降法和共轭梯度法各进行5 000步的能量优化,以消除分子间的不合理碰撞。最后用2 fs的积分步长,对体系进行3 010 ps的分子动力学模拟,包括升温10 ps(温度:50~300 K);平衡2 ns;采样1 ns,每隔1 000步记录1次轨迹。在复合物MD模拟中,用PME(Particle Mesh Ewald electrostatics)方法[35]处理静电作用,并采用SHAKE算法[36]限制键长,范德华相互作用截断半径取1.4 nm。
2 结果与讨论 2.1 受体结构构建
斑马鱼GABAAR的α1、 β2和γ2亚基以及果蝇RDL亚基与模板(烟碱型乙酰胆碱受体)比对的一致性分别为18.6%、18.0%、22.0%和22.4%。图 1a给出了斑马鱼GABAAR的γ2亚基和果蝇RDL亚基与模板烟碱型乙酰胆碱受体的β亚基的序列比对。图中蓝色阴影表示相似氨基酸,颜色越深相似度越高。图 1b为斑马鱼GABAAR各亚基和果蝇RDL亚基第二跨膜区(M2)的氨基酸序列。
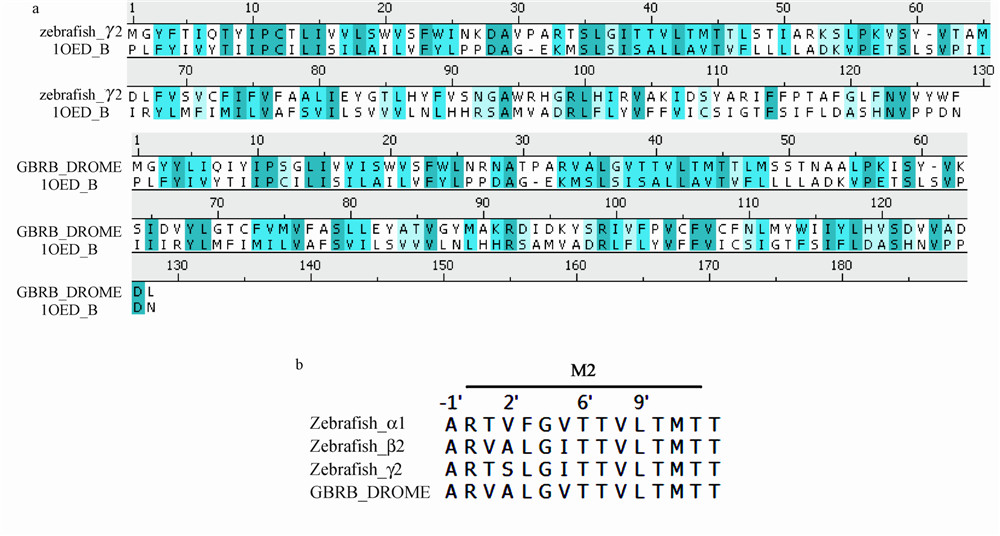 | 图 1 (a)斑马鱼GABAAR γ2亚基和果蝇RDL亚基同模板的序列比对;(b)斑马鱼GABAAR和果蝇RDLR亚基M2区的氨基酸序列Fig. 1 (a) Alignments of the acid sequence of zebrafish GABAAR γ2 subunit and fruitfly RDLR subunit with the template; (b) Amino acids sequence of M2 region in zebrafish GABAAR and fruitfly RDLR |
图 1b显示,斑马鱼GABAAR各亚基和果蝇RDL亚基中6′和9′位的氨基酸相同,分别是Thr和Leu。斑马鱼GABAAR α1、β2和γ2亚基2′位的氨基酸分别为Val、Ala和Ser,而果蝇RDL亚基的2′位为Ala,可见两受体2′位的氨基酸存在差异。两受体M2区2′~9′位的氨基酸显示,其氨基酸序列具有较高的相似度。
为考察分子动力学模拟轨迹的稳定性和收敛情况,对斑马鱼GABAAR和果蝇RDLR的构象基于α-碳原子的均方根偏差值(root mean square deviation,RMSD)和势能进行了分析。图 2显示了2个受体的RMSD值和势能随时间变化的趋势。图 2a和2b显示:两个受体结构的RMSD值均在500 ps前,上升较快,受体结构变化较大;500 ps之后,RMSD基本稳定,其中斑马鱼GABAAR的RMSD值稳定在0.2 nm左右,果蝇RDLR的RMSD值基本稳定在0.22 nm。
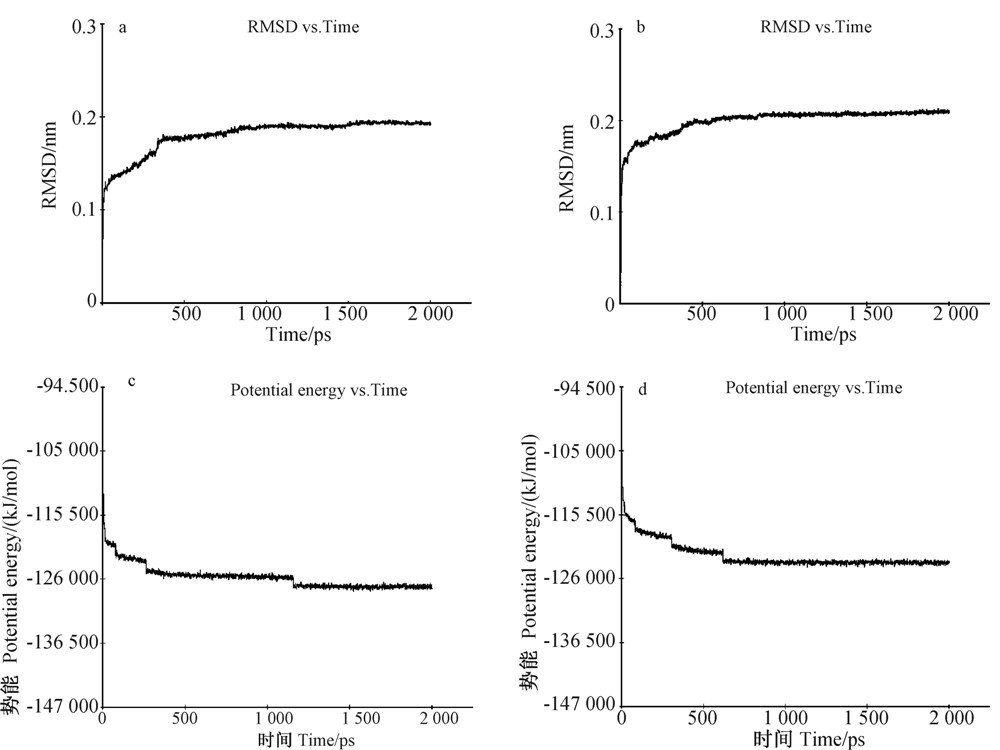 | (a)斑马鱼GABAAR结构的RMSD值随时间变化图; (b)果蝇RDLR结构的RMSD值随时间变化图;(c)斑马鱼GABAAR的势能随时间变化图; (d)果蝇RDLR的势能随时间变化图 (a) RMSD fluctuation of zebrafish GABAAR by MD simulation; (b) RMSD fluctuation of fruitfly RDLR by MD simulation; (c) Potential energy fluctuation of zebrafish GABAAR by MD simulation; (d) Potential energy fluctuation of fruitfly RDLR by MD simulation 图 2 受体结构的分子动力学模拟结果Fig. 2 MD simulation results of receptors structure |
图 2c显示,斑马鱼GABAAR的结构在500 ps之后势能趋于稳定,但大约在1 200 ps略有波动,随之又趋于稳定;图 2d显示,果蝇RDLR结构的势能在600 ps之后趋于稳定。经分子动力学模拟后,斑马鱼GABAAR结构的势能比果蝇RDLR结构的要低,说明斑马鱼GABAAR结构更为稳定。
图 3给出了优化后的GABAR模型。图 3a为斑马鱼GABAAR跨膜区模型,α1亚基为绿色,β2亚基为紫色,γ2亚基为黄色;图 3b为果蝇RDLR跨膜区模型,5个亚基均为黄色。
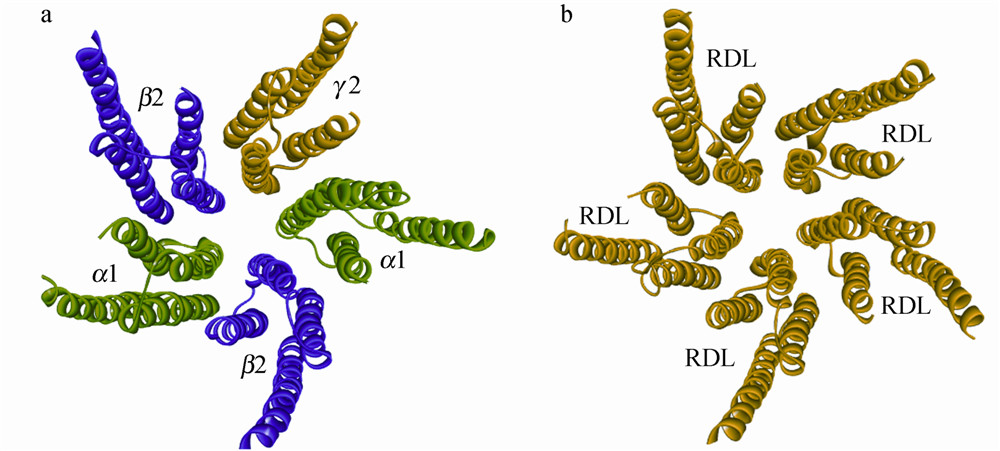 | (a)斑马鱼GABAAR跨膜区结构;(b)果蝇RDLR跨膜区结构 (a) Transmembrane domain of zebrafish GABAAR;(b) Transmembrane domain of fruitfly RDLR图 3 GABAR跨膜区结构Fig. 3 Structrues of GABAR |
图 4a和4b分别为斑马鱼GABAAR和果蝇RDLR结构与模板的叠合结果,其中模板为红色。斑马鱼GABAAR和果蝇RDLR模型与模板叠合,得到基于α-碳原子的RMSD值分别为0.23和0.21 nm,它们的RMSD值在合理范围内[37]。
 | (a)斑马鱼GABAAR(黄色)与模板(红色)的叠合; (b)果蝇RDLR(绿色)与模板(红色)的叠合(a) Superimpose of zebrafish GABAAR (yellow) and template(red);(b) Superimpose of fruitfly RDLR (green) and template (red)图 4 受体模型与模板的叠合Fig. 4 Superimpose of GABAR and template |
利用PROCHECK程序对所建目标模型进行评估,其结果通过程序生成的Ramachandran plot[38]来检测蛋白主链构型中氨基酸残基φ-ψ二面角的合理性。优化后的模型的Ramachandran plot见图 5。分析结果显示,在2个Ramachandran plot中,大量的氨基酸残基位于φ=-60°、ψ=-45°处,与受体结构中有较多α-螺旋结构的特征吻合。根据氨基酸残基构象统计,在斑马鱼GABAAR中,99.5%的氨基酸位于拉氏图的允许范围,仅0.5%的残基落在不合理区;果蝇RDLR有99.7%的氨基酸位于拉氏图的允许范围,仅0.3%的残基落在不合理区。这个结果表明,获得的模型是合理的。
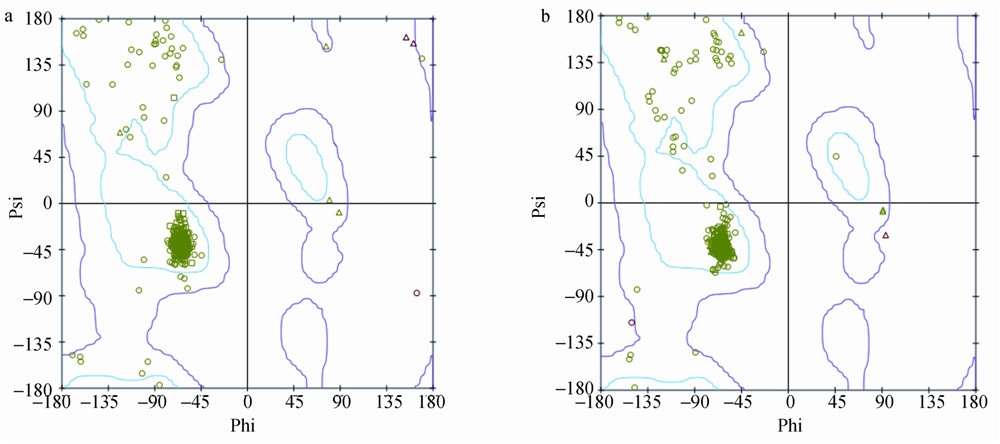 | 图 5 (a)斑马鱼GABAAR模型的Ramachandran图; (b)果蝇RDLR模型的Ramachandran图Fig. 5 (a) Ramachandran plot of zebrafish GABAAR; (b) Ramachandran plot of fruitfly RDLR |
2.2 活性位点分析
据报道,GABAR非竞争性拮抗剂的结合位点位于受体通道第二跨膜区中间部位[39]。定点突变数据和膜片钳技术研究表明:在GABAAR的α1亚基中,Val284、Thr288、Thr294和Ser299可能与PTX(Picrotoxinin)之间存在相互作用;β2亚基的Thr279和Thr285以及γ2亚基的Thr310和Thr316经突变后,被阻断的氯离子通道能自发开放[40,41,42]。在果蝇RDLR同源五聚体中,Ala301、Thr305和Leu308对非竞争性拮抗剂的结合起关键作用[43]。基于这些研究结果,利用两受体位于通道内侧的氨基酸残基:α1亚基的Val284、Thr288和Leu291,β2亚基的Ala275、Thr279和Leu282,γ2亚基的Thr310、Leu313,RDL亚基的Ala301、Thr305和Leu308,分别定义半径为1.2 nm的对接活性球,结果见图 6。
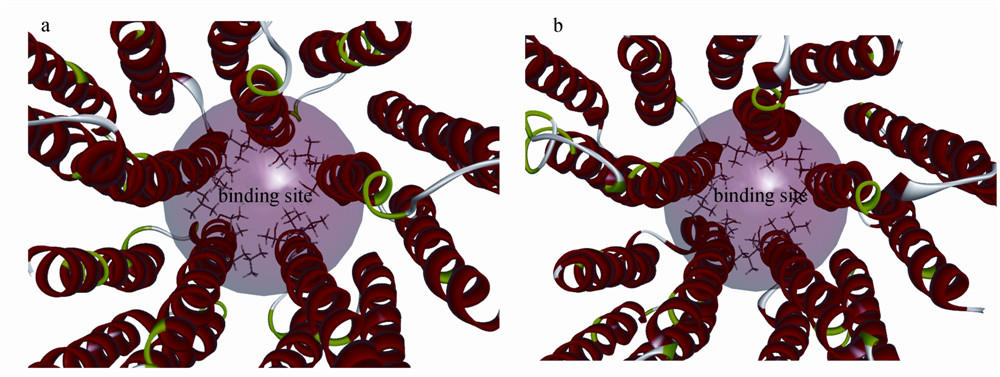 | 图 6 (a)斑马鱼GABAAR与氟虫腈的结合位点; (b)果蝇RDLR与氟虫腈的结合位点Fig. 6 (a) Fipronil binding sites in zebrafish GABAAR; (b) Fipronil binding sites in fruitfly RDLR |
图 7显示了斑马鱼GABAAR和果蝇RDLR第2跨膜区的叠合图,由两受体第2跨膜区叠合得到基于α-碳原子的RMSD值为0.06 nm。斑马鱼GABAAR(紫色)和果蝇RDLR(绿色)M2区的关键氨基酸用棒状表示;两受体各亚基M2区中Leu的构象一致性较高;斑马鱼GABAAR A(β2)和D(β2)链中的氨基酸与果蝇RDLR的相同。但是,对应位置的氨基酸构象存在差异;斑马鱼GABAAR B(γ2)链Ser306、C(α1)链Val284和E(α1)链Val284对应果蝇RDLR的Ala301,两受体在该位置的氨基酸不同,构象的差异很大。
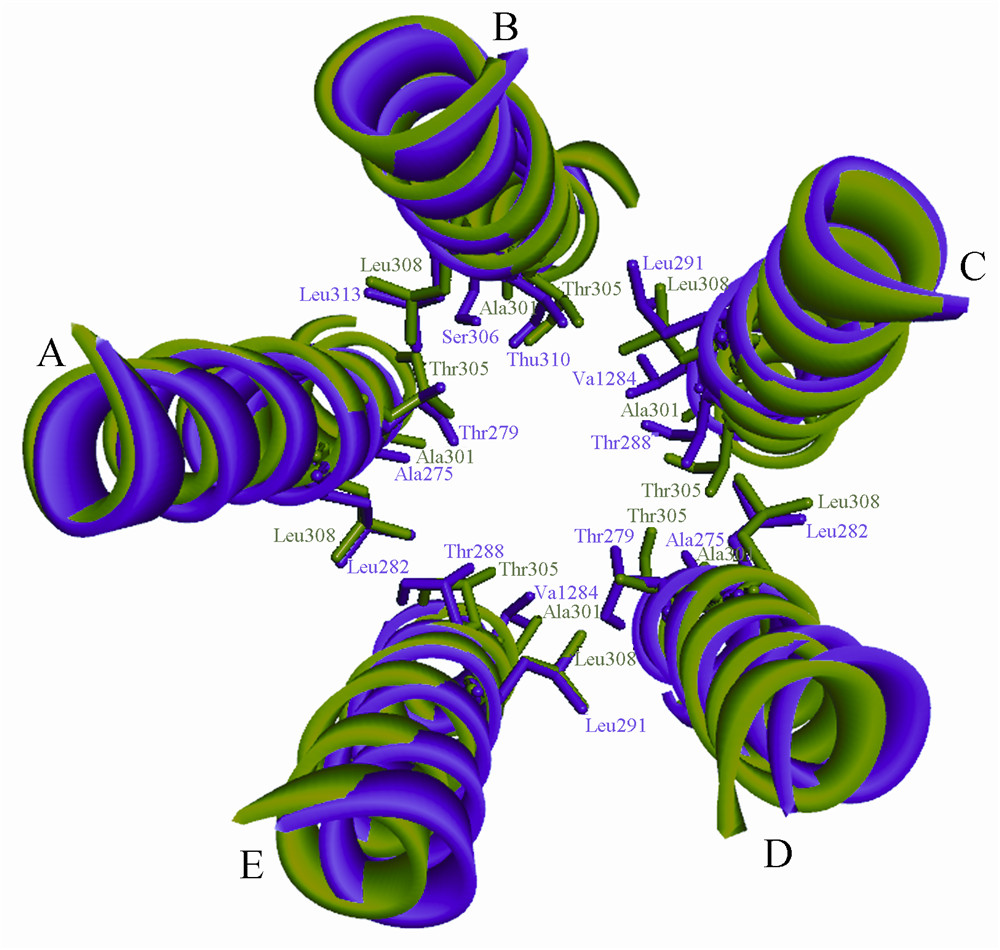 | 图 7 斑马鱼GABAAR(紫色)与果蝇RDLR(绿色)M2区的叠合Fig. 7 Superimpose of the M2 region between zebrafish GABAAR(purple) and fruitfly RDLR(green) |
2.3 氟虫腈与受体的作用
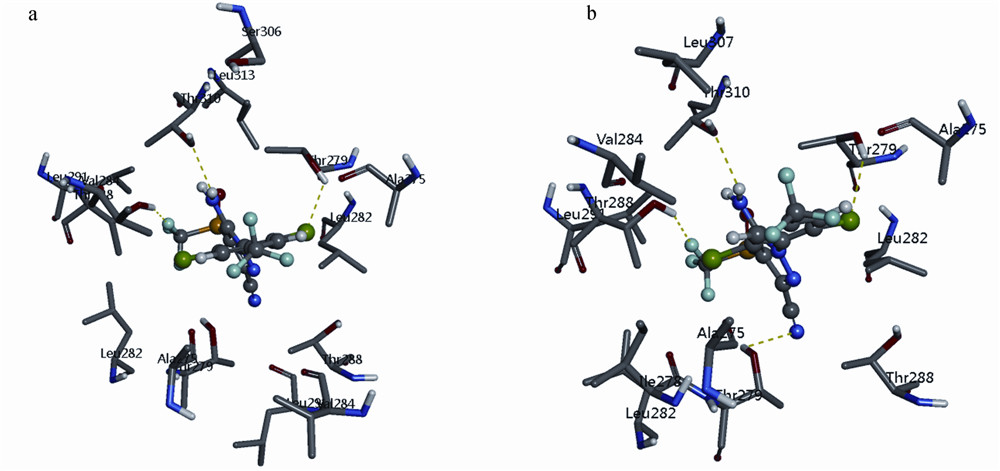
斑马鱼GABAAR和果蝇RDLR与氟虫腈的Cdocker相互作用能(Cdocker Interaction Energy)分别为-121.55和-137.93 kJ/mol,氟虫腈与果蝇RDLR的Cdocker相互作用能比斑马鱼GABAAR要高。图 8a和8b分别为氟虫腈与斑马鱼GABAAR及果蝇RDLR的对接结果。结果显示,氟虫腈在两受体中的作用位置存在差异。在斑马鱼GABAAR中,氟虫腈位于斑马鱼GABAAR通道的末端,十分靠近胞内区,苯环及环上的取代基朝向胞内区一侧,吡唑环朝向胞外区;而在果蝇RDLR中,氟虫腈位于通道Ala301~Leu308的中间部位,苯环及环上的取代基朝向胞外区,吡唑环朝向胞内区一端。图 8a显示,氟虫腈的苯环位于Ala275和Val284等疏水性氨基酸附近,吡唑环和三氟甲基亚磺酰基靠近Thr288、Thr279和Thr310;图 8b显示,氟虫腈的三氟甲基亚磺酰基朝向Ala301,吡唑环和氨基位于受体的极性Thr305附近,而苯环及取代的三氟甲基则位于Leu308附近。
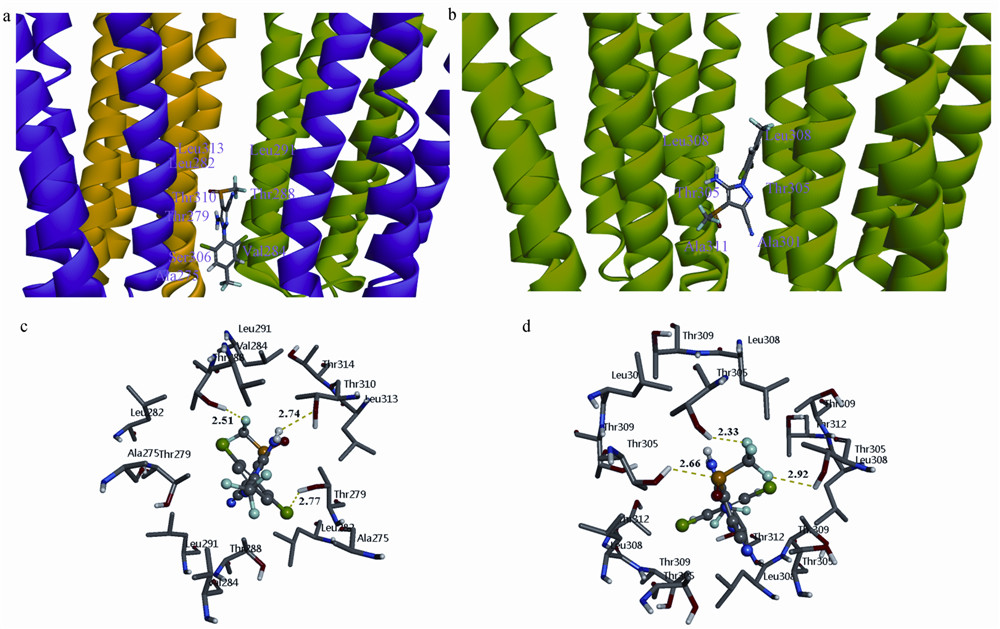 | (a) 氟虫腈与斑马鱼GABAAR的作用位置; (b)氟虫腈与果蝇RDLR的作用位置; (c) 氟虫腈与斑马鱼GABAAR作用时,0.5 nm以内的氨基酸; (d) 氟虫腈与果蝇RDLR作用时,0.5 nm以内的氨基酸. (氟虫腈以球棍状显示,氢键以绿色虚线标注,为清晰起见,仅显示氨基酸的极性氢原子) (a) Interaction position of fipronil in the chloride channel of zebrafish GABAAR; (b) Interaction position of fipronil in the chloride channel of fruitfly RDLR; (c) Residues in zebrafish GABAAR within 0.5 nm of fipronil; (d) Residues in fruitfly RDLR within 0.5 nm of fipronil. (fipronil(ball and stick),H-bonds are shown as green dashed lines,only amino acid residues with polar hydrogen atoms are shown) 图 8 氟虫腈与受体的作用Fig. 8 Interactions between receptors and fipronil |
图 8c和8d分别显示了氟虫腈与斑马鱼GABAAR及果蝇RDLR作用时,氟虫腈周围0.5 nm以内的氨基酸;表 1列出了氟虫腈与这2个受体分别形成的氢键。比较2个对接结果发现,氟虫腈在两受体结合口袋中所呈现的构象存在差异,氟虫腈与两受体形成氢键的氨基酸都是Thr,但与斑马鱼GABAAR形成氢键的原子是三氟甲基亚磺酰基的F原子、氨基的H原子以及苯环上取代的Cl原子;而氟虫腈与果蝇RDLR形成氢键的原子则是S原子和三氟甲基亚磺酰基的F原子。不过,两受体与氟虫腈形成的氢键数目相同,且氢键的距离均小于0.3 nm。
| 表 1 受体与氟虫腈作用的氢键Table 1 Hydrogen bonds formed between GABAR and fipronil |
氟虫腈与斑马鱼GABAAR的对接结果表明,斑马鱼GABAAR 中β2亚基的Thr279、α1亚基的Thr288和γ2亚基的Thr310对氟虫腈的结合十分关键,这与已有的报道[41,42]基本一致。对接结果说明了氟虫腈对斑马鱼产生毒性的原因。
氟虫腈与果蝇RDLR的对接结果表明:果蝇RDLR氟虫腈的结合位点位于Ala301到Leu308之间的区域内,这与Chen等[43]的研究结果基本一致,但在本研究中,Ala301与氟虫腈的距离大于0.5 nm,与氟虫腈没有直接的相互作用,为此,推测该氨基酸可能在维持结合口袋的稳定性方面起作用;氟虫腈与果蝇RDLR的Thr305形成了氢键,这与文献报道结果[44]一致。
2.4 受体-配体复合物稳定性
为考察对接结果的合理性和稳定性,将对接产生的受体-氟虫腈的最佳构象进行MD模拟,结果如图 9所示;表 2给出了500 ps后氟虫腈与两受体形成的氢键。图 9a和9b显示,斑马鱼GABAAR-氟虫腈复合物和果蝇RDLR-氟虫腈复合物的势能分别在800 ps之后和600 ps之后逐渐趋向平衡,达到稳定状态。
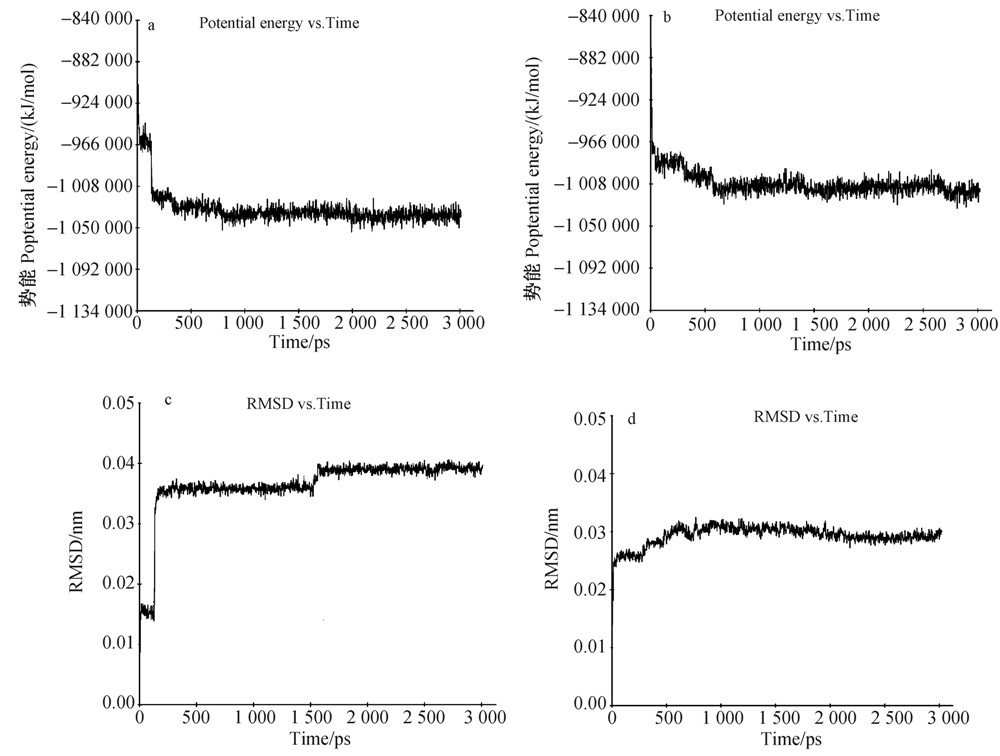 | (a)斑马鱼GABAAR-氟虫腈的势能随时间的变化; (b)果蝇RDLR-氟虫腈复合物的势能随时间的变化; (c)斑马鱼GABAAR-氟虫腈的RMSD值随时间的变化; (d)果蝇RDLR-氟虫腈的RMSD值随时间的变化 (a) Potential energy variation of zebrafish GABA\-AR-fipronil complex; (b) Potential energy variation of fruitfly RDLR-fipronil complex; (c) RMSD variation of zebrafish GABA\-AR-fipronil complex; (d) RMSD variation of fruitfly RDLR-fipronil complex图 9 受体-氟虫腈复合物MD模拟结果Fig. 9 Receptor-fipronil complexes by MD simulation |
| 表 2 复合物进行分子动力学模拟后的氢键Table 2 Hydrogen bonds formed between receptor and fipronil by MD simulation |
图 9c显示,斑马鱼GABAAR与氟虫腈复合物体系在200 ps左右时,RMSD值在0.036 nm附近,这主要是因为受体中3个亚基α1、β2和γ2与氟虫腈之间形成了3个氢键的结果;在1 500 ps之后,体系的RMSD值稳定在0.04 nm附近。模拟轨迹表明,此构象变化对应于斑马鱼GABAAR另一条β2亚基上Thr279的羟基H原子与氟虫腈的氰基N原子之间形成了概率为31.2%的氢键作用(见图 10和表 2),使得受体构象发生了变化,RMSD值随之改变。 图 9d显示,果蝇RDLR-氟虫腈复合物体系在600 ps之后,体系的RMSD值基本稳定,在0.032 nm附近波动。模拟的轨迹表明,果蝇RDLR与氟虫腈复合物体系的构象在MD模拟过程中的变化大于斑马鱼GABAAR与氟虫腈复合物体系。另外,由于果蝇RDLR与氟虫腈形成的氢键数目较多(见表 2),从而引起体系的构象变化,即RMSD值出现波动。
 | (a) 500 ps与1 500 ps之间的氟虫腈与斑马鱼GABAAR复合物平均结构; (b) 1 500 ps之后的氟虫腈与斑马鱼GABAAR复合物平均结构(a) The complex structure between 500 ps and 1 500 ps; (b) The complex structure after 1 500 ps图 10 分子动力学模拟产生的氟虫腈与斑马鱼GABAAR复合物结构Fig. 10 The complex structures of zebrafish GABA\-AR-fipronil by MD simulation |
表 2的信息显示,斑马鱼GABAAR-氟虫腈复合体系在动力学模拟后的氢键主要有4个,分别是:α1亚基Thr288的羟基H原子与氟虫腈中三氟甲基亚磺酰基上的1个F原子形成的,频率为67.2%;2个 β2亚基Thr279的羟基H原子分别与氟虫腈苯环上的1个Cl原子和氰基N原子形成了氢键,频率分别是67.6%和31.2%;另外,γ2亚基Thr310的羟基O原子与氟虫腈的氨基H原子也形成了概率为44.4%的氢键。这些氢键出现的几率较大,说明斑马鱼GABAAR-氟虫腈复合物体系十分稳定[45],从氢键稳定性的角度解释了氟虫腈对斑马鱼高毒的机理[46]。在动力学模拟后,果蝇RDLR-氟虫腈复合体系形成了6个氢键,其中概率大于40%的氢键只有2个,分别是由氟虫腈苯环上三氟甲基的1个F原子和苯环上取代的1个Cl原子分别与A链上Thr312和D链上Thr309的羟基H原子形成的,概率分别为59.0%和45.6%,说明这些氢键作用对维持这2个受体-氟虫腈复合物的稳定起到了主要作用。
表 3给出了受体-氟虫腈复合物经MD模拟后的平均相互作用能。斑马鱼GABAAR和果蝇RDLR与氟虫腈的总相互作用能(ETotal)分别为-554.23和-464.06 kJ/mol,静电相互作用能(EEle)分别为-450.20和-334.99 kJ/mol,范德华作用能(Evdw)分别为104.03和129.07 kJ/mol,说明氟虫腈在识别和结合两受体的过程中,静电作用占主导地位。斑马鱼GABAAR与氟虫腈的总相互作用能和静电相互作用能均比果蝇RDLR与氟虫腈的作用能要低,说明斑马鱼GABAAR-氟虫腈体系比果蝇RDLR-氟虫腈体系更稳定。
| 表 3 分子动力学模拟后的平均相互作用能Table 3 Average interaction energy of two systems by MD simulation |
本研究结果表明,氟虫腈与斑马鱼GABAAR和果蝇RDLR的作用模式和效果存在差异。斑马鱼GABAAR和果蝇RDLR与氟虫腈作用位点的氨基酸序列一致性较高,但构象差异较大。氟虫腈结合在斑马鱼GABAAR通道末端,靠近胞内区;氟虫腈与果蝇RDLR的结合位点则位于M2区Ala301~Leu308之间的区域内。受体-氟虫腈复合物的分子动力学(MD)模拟轨迹研究表明:氟虫腈与斑马鱼GABAAR 之间形成了4个氢键——α1亚基的Thr288(1个氢键)、β2亚基的Thr279(2个氢键)以及γ2亚基的Thr310(1个氢键),其中有2个氢键的概率大于60%;而果蝇RDLR的Thr305、Thr309以及Thr312与氟虫腈形成了6个氢键,但只有A链的Thr312与氟虫腈形成了概率大于50%的氢键,稳定性比前者低;另外,斑马鱼GABAAR与氟虫腈的相互作用能也低于果蝇RDLR与氟虫腈之间的相互作用能。
| [1] | MOFFAT A S. New chemicals seek to outwit insect pests[J]. Science, 1993, 261(5121): 550-551. |
| [2] | IKEDA T, NAGATA K, KONO Y, et al. Fipronil modulation of GABAA receptor single-channel currents[J]. Pest Management Science, 2004, 60(5): 487-492. |
| [3] | STEHR C M, LINBO T L, INCARDONA J P, et al. The developmental neurotoxicity of fipronil: notochord degeneration and locomotor defects in zebrafish embryos and larvae[J]. Toxicol Sci, 2006, 92(1): 270-278. |
| [4] | WANG X, ZHOU S, DING X, et al. Effect of triazophos, fipronil and their mixture on miRNA expression in adult zebrafish[J]. J Environ Sci Health B, 2010, 45(7): 648-657. |
| [5] | ENNA S J. The GABA receptors[M]. Totowa New Jersey: Humana Press, 2007: 28-29. |
| [6] | JANNA L K, ISABELLE M P. The Cys-loop pentameric ligand-gated ion channel receptors: 50 years on[J]. Can J Physiol Pharmacol, 2012, 90(6): 771-782. |
| [7] | ZHAO X L, VINCENT L S. The role of GABA and glutamate receptors in susceptibility and resistance to chloride channel blocker insecticides[J]. Pestic Biochem Physiol, 2010, 97(2): 153-160. |
| [8] | ELDEFRAWI M E, ELDEFRAWI A T. Insecticide actions on GABA receptors and voltage-dependent chloride channels[M]//Insecticide Action. US: Springer, 1989: 1-11. |
| [9] | MCKERNAN R M, WHITING P J. Which GABAA-receptor subtypes really occur in the brain?[J]. Trends Neurosci, 1996, 19(4): 139-143. |
| [10] | HASHIMOTO T. GABA receptor chloride ion channel[J]. Nihon Jinsho. Japanese Journal of Clinical Medicine, 1998, 56(7): 1824-1829. |
| [11] | DAVID B S, SARAH C R L, JAMES F H W, et al. Pharmacology of insect GABA receptors[J]. Neurochemical Research, 1991, 16(3): 363-374. |
| [12] | 卢文才, 何林, 薛传华, 等. 昆虫γ-氨基丁酸受体研究现状[J]. 昆虫知识, 2009, 46(1): 152-158. LU Wencai, HE Lin, XUE Chuanhua, et al. Advances in research of gamma-aminodutyric acid receptors of insects[J]. Chin Bull Entomol, 2009, 46(1): 152-158. (in Chinese) |
| [13] | NEIL S M, STEVEN D B, DAVID B S. Stable expression of a functional homo-oligomeric drosophila GABA receptor in a drosophila cell line[J]. Proc Roy Soc Lond B: Biolog Sci, 1994, 258(1353): 307-314. |
| [14] | JORGENSEN W L. Rusting of the lock and key model for protein-ligand binding[J]. Science, 1991, 254(5034): 954-955. |
| [15] | ALAM M S, HUANG J, OZOE F, et al. Synthesis, 3D-QSAR, and docking studies of 1-phenyl-1H-1,2,3-triazoles as selective antagonists for β 3 overα1 β 2γ2 GABA receptors[J]. Bioorg Med Chem, 2007, 15(15): 5090-5104. |
| [16] | LAW R J. LIGHTSTONE F C. GABA receptor insecticide non-competitive antagonist may bind at allosteric modulator sites[J]. Intern J Neurosci, 2008, 118(5): 705-734. |
| [17] | CAMPAGNA-SLATER V, WEAVER D F. Molecular modelling of the GABAA ion channel protein[J]. J Mol Graph Model, 2007, 25(5): 721-730. |
| [18] | CI S Q, REN T R, SU Z G. Modeling the interaction of fipronil-related non-competitive antagonists with the GABA β 3-receptor[J]. J Mol Model, 2007, 13(3): 457-464. |
| [19] | CHENG J, JU X L, CHEN X Y, et al. Homology modeling of humanα1 β 2γ2 and house fly β 3 GABA receptor channels and Surflex-docking of fipronil[J]. J Mol Model, 2009, 15(9): 1145-1153. |
| [20] | MCGONIGLE I, LUMMIS S C. RDL receptors[J]. Biochem Soc Trans, 2009, 37(Pt6): 1404-1406. |
| [21] | 巨修炼, 王黎丽, 李科. 斑马鱼A型γ-氨基丁酸受体同源模建及分子对接[J]. 武汉工程大学学报, 2013, 35(6): 20-29. JU Xiulian, WANG Lili, LI Ke. Homology modeling and docking of zebrafishγ-aminobutyric acid receptor[J]. J Wuhan Inst Tech, 2013, 35(6): 20-29. (in Chinese) |
| [22] | Accelrys Inc. Discovery Studio [CP/OL]. (2008-12-07). http://accelrys.com/products/ discovery-studio/ |
| [23] | US. National Library of Medicine. NCBI data base [DB/OL]. (1988-11-04). http://www. ncbi.nlm.nih.gov/guide. |
| [24] | LAW R J, LIGHTSTONE F C. Modeling neuronal nicotinic and GABA receptors: important interface salt-links and protein dynamics[J]. Biophys J, 2009, 97(6): 1586-1594. |
| [25] | CHANGEUX J P. The nicotinic acetylcholine receptor: the founding father of the pentameric ligand-gated ion channel super family[J]. J Biol Chem, 2012, 287(48): 40207-40215. |
| [26] | ASHBY J A, MCGONIGLE I V, PRICE K L, et al. GABA binding to an insect GABA receptor: a molecular dynamics and mutagenesis study[J]. Biophys J, 2012, 103(10): 2071-2081. |
| [27] | LUMMIS S C R. Locating GABA in GABA receptor binding sites[J]. Biochem Soc Trans, 2009, 37(6): 1343-1346. |
| [28] | HARRISON N J, LUMMIS S C R. Locating the carboxylate group of GABA in the homomeric rho GABAA receptor ligand-binding pocket[J]. J Biol Chem, 2006, 281(34): 24455-24461. |
| [29] | MIYAZAWA A, FUJIYOSHI Y, UNWIN N. Structure and gating mechanism of the acetylcholine receptor pore[J]. Nature, 2003, 423(6943): 949-955. |
| [30] | ŠALI A, OVERINGTON J P. Derivation of rules for comparative protein modeling from a database of protein structure alignments[J]. Protein Sci, 1994, 3(9): 1582-1596. |
| [31] | SHEN M Y, SALI A. Statistical potential for assessment and prediction of protein structures[J]. Protein Sci, 2006, 15(11): 2507-2524. |
| [32] | BERNARD R B, ROBERT E B, BARRY D O, et al. CHARMm: A program for macromolecular energy, minimization, and dynamics calculations[J]. J Comput Chem, 1983, 4(2): 187-217. |
| [33] | LASKOWSKI R A, MACARTHUR M W, MOSS D S, et al. PROCHECK-a program to check the stereochemical quality of protein structures[J]. J Appl Crystallogr, 1993, 26(2): 283-291. |
| [34] | WU G, ROBERTSON D H, BROOKS C L, et al. Detailed analysis of grid-based molecular docking: a case study of CDOCKER-a CHARMm-based MD docking algorithm[J]. J Comput Chem, 2003, 24(13): 1549-1562. |
| [35] | ULRICH E, PERERA L, BERKOWITZ M L. A smooth particle mesh Ewald method[J]. J Phys Chem, 1995, 103(19): 8577-8593. |
| [36] | VINCENT K, WILFRED F G, PHILIPPE H H. A fast SHAKE algorithm to solve distance constraint equations for small molecules in molecular dynamics simulations[J]. J Comput Chem, 2001, 22(5): 501-508. |
| [37] | CHANG Y T, LOEW G H. Homology modeling and substrate binding study of human CYP4A11 enzyme[J]. Proteins: Structure, Function, and Genetics, 1999, 34(3): 403-415. |
| [38] | HOOFT R W W, CHRIS S, GERRIT V. Objectively judging the quality of a protein structure from a Ramachandran plot[J]. Bioinformatics, 1997, 13(4): 425-430. |
| [39] | CHEN L G, DURKIN K A, CASIDA J E. Spontaneous mobility of GABAA receptor M2 extracellular half relative to noncompetitive antagonist action[J]. J Biol Chem, 2006, 281(50): 38871-38878. |
| [40] | PERRET P, SARDA X, WOLFF M. Interaction of non-competitive blockers within theγ-aminobutyric acid type A chloride channel using chemically reactive probes as chemical sensors for cysteine mutants[J]. J Biol Chem, 1999, 274(36): 25350-25354. |
| [41] | GURLET D, AMIN J, ROSS P C, et al. Point mutations in the M2 region of the alpha, beta, or gamma subunit of the GABAA channel that abolish block by picrotoxin[J]. Receptors & Channels, 1995, 3(1): 13-20. |
| [42] | XU M, DOUGLAS F C, MYLES H A. Interaction of picrotoxin with GABAA receptor channel-lining residues probed in Cysteine mutants[J]. Biophys J, 1995, 69(5): 1858-1867. |
| [43] | CHEN L G, DURKIN K A, CASIDA J E. Structural model forγ-aminobutyric acid receptor noncompetitive antagonist binding: Widely diverse structures fit the same site[J]. Proc Natl Acad Sci USA, 2006, 103(13): 5185-5190. |
| [44] | HISANO K, OZOE F, HUANG J, et al. The channel-lining 6' amino acid in the second membrane-spanning region of ionotropic GABA receptors has more profound effects on 4'-ethynyl-4-n-propylbicycloorthobenzoate binding than the 2' amino acid[J]. Invert Neurosci, 2007, 7(1): 39-46. |
| [45] | 安康, 柴晓杰, 薛飞, 等. VEGFR-2与抑制剂Sunitinib的分子对接及分子动力学研究[J]. 化学学报, 2012, 70(10): 1232-1236. AN Kang, CHAI Xiaojie, XUE Fei, et al. Study on docking and molecular dynamics simulation between VEGFR-2 and the inhibitor sunitinib[J]. Acta Chimica Sinica, 2012, 70(10): 1232-1236. (in Chinese) |
| [46] | 赵于丁, 徐敦明, 范青海, 等. 10种农药对斑马鱼的毒性与安全评价[J]. 安徽农业科学, 2007, 35(22): 6801-6802. ZHAO Yuding, XU Dunming, FAN Qinghai, et al. Toxicity of ten pesticides to brachydanio rerio and safety evaluation[J]. J Anhui Agric Sci, 2007, 35(22): 6801-6802. (in Chinese) |
 2014, Vol. 16
2014, Vol. 16


