许多研究均表明,第一次“绿色革命”与植物激素相关的基因编码是密不可分的[1]。植物激素是一类具有调节植物生长发育功能的有机内源物质。天然植物激素可分为生长素、赤霉素、细胞分裂素、脱落酸和乙烯(结构见图1)。[14]有些植物激素的稳定性较差,如生长素见光易分解[2],有些则合成成本过高,如脱落酸[3],这些因素都极大地限制了其在生产实际中的应用。尽管人们已经设计合成了一些人工植物激素,如2,4-D(2,4-二氯苯酚代乙酚),但是其综合性能仍无法与天然植物激素相比[4],主要原因是对植物激素调控的分子机理的研究相对比较滞后。
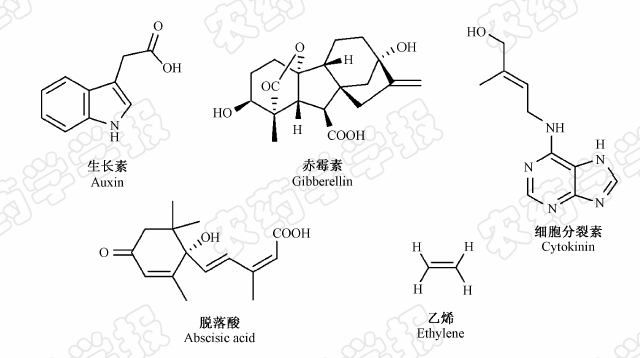 | 图 1 具五种植物激素的化学结构 Fig.1 Chemical structures of five plant hormones |
随着化学、生物学等研究技术的不断进步,与植物激素调控相关的分子机理的研究也取得了重要进展。人们相继发现了多种植物激素的受体,尤其是生长素、赤霉素和脱落酸受体三维晶体结构的解析,无疑对植物激素调控的分子机理研究及新型人工植物激素的合理设计具有重要意义。但目前尚未见有关这方面研究进展的系统综述,为此,笔者拟对这3种植物激素的受体结构及相关分子机理的研究进行综述,旨在为新型人工植物激素的生物合理设计提供参考。
尽管生长素是最早(1880年)被发现的一种植物激素,但是100多年过去了,对生长素调控分子机理的研究却一直没有较大进展,生长素受体蛋白始终未被确证。过去很多研究组均将目光锁定在生长素结合蛋白(auxin binding protein 1, ABP1)上,2002年Napier研究小组报道了ABP1与生长素复合物的三维晶体结构[5],这无疑是其受体研究过程中的一个重要发现,但研究者并未发现ABP1在调控植物生长中所起的作用。对此,Tomic研究小组对ABP1蛋白的结构进行了广泛的模拟研究,从模拟的结构来看,在与生长素结合前后,ABP1蛋白口袋周围的构象并未发生显著变化,因此,他们认为ABP1蛋白的下游一定存在另一个信号传导蛋白,而ABP1蛋白端基的构象变化很可能是该信号传导的关键,这种变化是由生长素间接导致的[6]。但是在后来的很长一段时间里,研究者都未能发现ABP1的下游蛋白。 其他一些研究组在进行生长素参与调控的泛素化降解途径研究中发现,生长素可以促使某些蛋白质降解,从而激活有关基因的表达[7]。20世纪末,Estelle研究小组首先从拟南芥Arabidopsis thaliana中筛选到了一种蛋白突变体,由于其具有抑制生长素转运的能力,因此被命名为抑制运输响应蛋白1(transport inhibitor response protein 1, TIR1)[8]。随着研究的不断深入,他们后来发现TIR1对生长素的转运并无抑制作用,相关分子克隆研究结果表明,TIR1属于F-box结构蛋白,该类蛋白在泛素和蛋白酶体协同降解蛋白质途径中,可以特异性地识别底物蛋白而参与调控转录,因此他们认为TIR1蛋白可能是生长素信号传导中的一个重要成员[9]。随后,很多研究组的研究结果均证明,生长素可以通过促进生长素转录抑制蛋白(auxin/indole-3-acetic acid, Aux/IAA transcriptional repressor proteins)的泛素化降解而起到调节转录的作用[10, 11, 12] 。Estelle研究小组也进一步证明了TIR1是一种泛素连接酶,Aux/IAA转录抑制蛋白可以通过与生长素响应因子(auxin response factor, ARF)蛋白形成二聚体,从而抑制其活性。生长素可以通过增强TIR1蛋白与Aux/IAA转录抑制蛋白的相互作用而促进其泛素化降解,从而解除Aux/IAA对ARF的抑制,进而启动相关基因的表达[10, 11, 12] 。2005年,Estelle和Leyser两个研究组同时报道了生长素可以有效促进TIR1蛋白与IAA的相互作用,从而证明了TIR1就是生长素受体[4, 5, 6, 7, 8, 9, 10, 11, 12, 13]。该发现在生长素受体研究史上具有里程碑的意义。
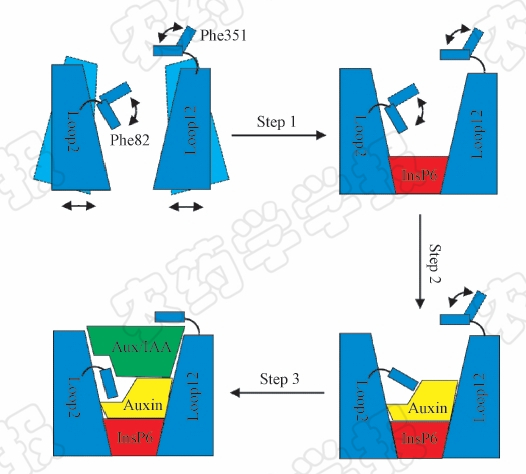
然而生长素究竟怎样调控TIR1蛋白与Aux/IAA转录抑制蛋白的相互作用呢?郑宁研究小组于2007年首次报道了拟南芥TIR1蛋白与Aux/IAA复合物的三维晶体结构(图2)[14]。TIR1蛋白是由富含亮氨酸重复序列组成的中通桶型结构,其内外两层分别由β折叠和α螺旋交替而成。内层在结合了肌醇六磷酸辅因子(InsP6)后形成单一开口的空腔,生长素结合于空腔的内部,从而为Aux/IAA转录抑制蛋白的结合提供了适宜的分子表面,即生长素在促进TIR1与Aux/IAA转录抑制蛋白的相互作用中扮演了“分子胶水”的角色[14]。杨光富研究小组的研究结果表明,肌醇六磷酸充当了TIR1蛋白的构象稳定剂,而生长素则可以调控Phe82侧链构象的变化,Phe82和Phe351侧链构象的变化对于Aux/IAA转录抑制蛋白的识别起到了关键作用(图3)[15]。由此可见,由生长素及其受体介导的分子调控机制已逐渐被人们所认识,但这并不意味着已经揭示了生长素调节植物生长所涉及的所有信号通路的机制,还有很多亟待解决的问题值得去探讨。
 | 图 2 TIR1、生长素以及Aux/IAA复合物的晶体结构 Fig.2 Crystal structure of the TIR1 complexed with auxin and Aux/IAA |
 | 图 3 生长素与受体感知过程中关键氨基酸的作用 Fig.3 The roles of key amino acid in the perception of auxin and reception |
赤霉素(GAs)属于双萜类化合物,已经发现的赤霉素类物质多达136种,其中只有少数几种具有植物生长调节活性。从赤霉素的化学结构((图1)来看,其具有很强的疏水性,因而在赤霉素受体的早期研究中,很多学者认为赤霉素与疏水性的哺乳动物类固醇激素具有相似的作用机制,即在疏水作用下,赤霉素可以跨过植物细胞膜与DNA或RNA发生作用,从而影响基因的转录和表达[16]。但持反对意见的学者认为,赤霉素不会与DNA或RNA发生直接的相互作用,一定存在着一种赤霉素结合蛋白(GA-binding protein, GBP)[17]。随后,Stoddart研究小组首次从莴苣胚轴的蛋白提取物中发现了能够与赤霉素发生相互作用的蛋白,这是第一次发现赤霉素结合蛋白[18]。此后一系列的赤霉素结合蛋白陆续被发现,其中,Nakajima研究小组在秧豆苗中发现的赤霉素结合蛋白对活性赤霉素分子具有特异性识别的功能,因此是一种潜在的赤霉素受体[19]。 后来研究者们发现,转录调节因子——DELLA蛋白可以抑制赤霉素的信号传导[20],由此开始了对赤霉素介导DELLA蛋白的泛素化降解机理研究。DELLA蛋白位于细胞核内,属于GRAS蛋白家族,[JP2]是一类转录抑制因子,通过调节其他基因的表达而实现其对赤霉素信号传导的抑制作用[21]。DELLA蛋白有着高度保守的序列,在拟南芥中已经发现了5种DELLA蛋白:GA INSENSITIVE (GAI)、REPRESSOR of ga (RGA)、 RGA-LIKE 1(RGL1)、RGA-LIKE 2 (RGL2)和RGA-LIKE3 (RGL3)蛋白,相当于水稻中的(Slender Rice 1, SLR1)蛋白、小麦中的Reduced Height Genes (Rht)蛋白等[22]。其中Rht蛋白就是第一次“绿色革命”中与小麦增产密切相关的蛋白[23]。后来研究者们进一步发现,拟南芥的SLY1蛋白突变体呈现出对赤霉素不敏感的表型,同时RGA蛋白的表达水平变高。这些研究表明,赤霉素能够介导RGA与SCFSLY1E3连接酶复合物的结合,从而促使其泛素化降解,与植物生长相关的基因表达因此被激活,该过程是赤霉素调控植物生长分子机理的关键[24]。 2005年,Ueguchi-Tanaka研究小组发现,GID1蛋白(gibberellin insensitive Dwarf 1)的缺失可以导致水稻对赤霉素不敏感,并证明GID1蛋白就是赤霉素的受体[25]。GID1蛋白可与活性赤霉素分子结合,而结合了赤霉素分子的GID1蛋白可与DELLA蛋白发生相互作用,促进DELLA蛋白的降解,从而使相关基因的表达被激活。2008年,Matsuoka和Hakoshima两个研究小组分别报道了水稻和拟南芥GID1蛋白的三维晶体结构((图4)。根据GID1-GA复合物的晶体结构,他们认为赤霉素可以诱导GID1蛋白的氮端变构,从而影响DELLA蛋白与GID1蛋白的相互作用,促进DELLA蛋白的泛素化降解,进而影响植物生长[26,27]。这就是已被人们广为接受的赤霉素诱导GID1蛋白的“变构学说”((图5A),但是变构状态的apo-GID1蛋白的晶体结构一直未能得到。 最近,杨光富研究小组在深入分析GID1-GA复合物晶体结构的基础上,通过对GID1蛋白的构象进行微秒级的分子动力学模拟,首次发现了赤霉素与受体GID1蛋白结合的新通道,并提出了“非变构学说”的赤霉素作用新机制,即赤霉素在通过新通道与GID1蛋白结合时,并不介导其氮端结构域发生构象变化,而是通过稳定GID1与DELLA蛋白之间的氢键,从而介导DELLA蛋白的泛素化降解(图5B)[28]。他们运用PMF方法计算了2种机制下赤霉素进入GID1蛋白活性口袋的活化能能垒,从而证实了“非变构学说”是一种能量上更为有利的作用机制[28]。
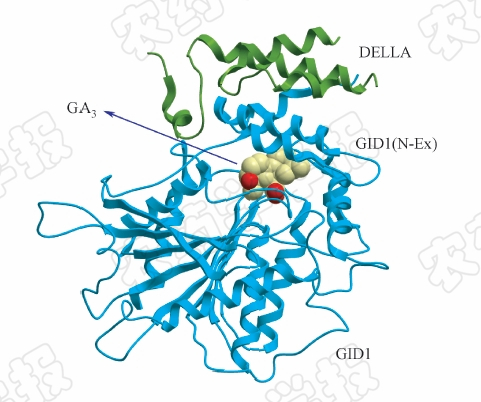 | 图 4 GA\-3、GID1以及DELLA复合物的晶体结构 Fig.4 Structure of the GA\-3-GID1-DELLA complex |
 | 图 5 赤霉素调控的GID1与DELLA结合的分子机理 Fig. 5 Molecular mechanism of GA induced binding of GID1 and DELLA |
脱落酸是一种具有倍半萜结构的植物激素,简称ABA,具有促进种子成熟与休眠的功能,对植物根的生长和叶的脱落具有促进作用。其生理作用主要是导致休眠及促进落叶,对细胞的分裂与伸长具有抑制作用,还可以调节植物对多重环境压力,如寒冷、干旱和多盐等的适应能力。植物体中有着与脱落酸相关的复杂信号网络,其中涉及到激酶、磷酸酯酶、G蛋白、泛素化途径及RNA转录因子等[29]。其中脱落酸受体(ABAR)是传递和接收其信号的起始,同时也是惟一直接感受其信号的单元,因此寻找和发现脱落酸受体对于其调控分子机理的研究具有十分重要的作用。 对脱落酸受体的研究始于20世纪70年代,Meldrum研究小组发现脱落酸可以通过控制气孔的开关来调节植物体内水分的含量,进而调节植物体适应各种复杂环境的能力,因此他们认为其结合蛋白有可能位于质膜上[30]。但之后的30多年里,人们一直未能发现脱落酸受体。直到2006年,张大鹏研究小组从蚕豆叶中纯化得到了一种可以调节气孔开合的蛋白,通过序列分析发现,该蛋白可以编码镁离子鳌合酶的H亚基(H subunit of Mg-chelatase, CHLH)[31]。镁离子螯合酶的H亚基蛋白是叶绿素生物合成以及植物质体向细胞核信号传导中的重要组分[31]。ABAR/CHLH可特异性地结合脱落酸,在种子萌发、生长和气孔运动的信号通路中起调控作用,是一种潜在的脱落酸受体[32]。脱落酸受体的发现无异于找到了控制植物气孔运动和种子发育的命脉,但是有关ABAR/CHLH感知脱落酸信号的分子机理还有待进一步探索。 2007年,马力耕研究小组在拟南芥中也发现了一种脱落酸受体,即一种参与传递脱落酸信号的G蛋白偶联受体(GCR2),其G蛋白由α、 β和γ 3个亚基组成[33]。与之前不同的是,该蛋白是一种细胞膜外的脱落酸受体。他们认为GCR2和Gα亚基共同调控脱落酸下游信号,并发现脱落酸与GCR2结合会导致GCR2-Gα蛋白复合物解离,从而释放Gα而激活下游的反应[33]。2009年Assmann研究小组在拟南芥中又发现了2种GPCR型G蛋白,并发现这2种蛋白能与拟南芥G蛋白α亚基的GPA1相互作用。因这2种蛋白均具有GTP酶活性而被命名为GPCR-type G proteins (GTG1和GTG2)[34]。他们还发现GTG1和GTG2蛋白缺失型拟南芥对脱落酸的敏感性较差,由此推断GTG蛋白不仅是一类新型的G蛋白,还是一类细胞膜脱落酸受体[34]。
2009年,Grill和Cutler 两个研究小组发现,拟南芥中脱落酸可以通过一类pyrabactin resistance(PYR/PYL)蛋白来抑制蛋白磷酸酶PP2C的活性,因此认为PYR/PYL是一类潜在的脱落酸受体,但对其活性位点及调节机理尚不清楚[35,36]。由于报道的脱落酸受体种类很多,其争议也很大,因此相关的结构生物学研究至关重要。同年,多个研究组相继报道了apo-PYL蛋白、脱落酸-PYL蛋白复合物、以及脱落酸-PYL蛋白-PP2C复合物的高分辨率晶体结构(图6)[37.38, 39, 40,41]。通过结构比较和分析,PYL蛋白被确认为脱落酸受体,一种新颖的“门闩锁”机制在脱落酸信号传导中起到了非常重要的作用[38]。PYL蛋白有一个内层的结合腔,腔口的2条柔性链起到了类似“门和闩”的功能:未结合脱落酸时,2条柔性链呈敞开状态;当脱落酸进入活性腔后,2条柔性链发生构象变化,从敞开变为关闭状态,就像门闩一样将脱落酸关在活性腔中。由脱落酸导致的构象变化可以形成一个适于PP2C蛋白结合的分子表面,于是PYL蛋白与PP2C蛋白结合而抑制其活性,PYL蛋白中的2条柔性链在脱落酸信号传导及抑制PP2C活性方面起了至关重要的作用((图7)[39]。
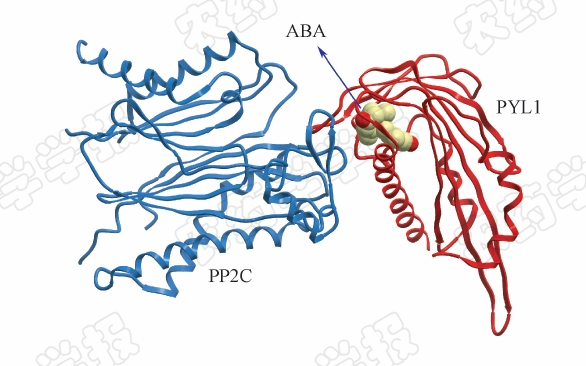 | 图 6 PYL1-ABA-PP2C复合物的晶体结构 Fig.6 Crystal structure of the PYL1-ABA-PP2C complex |
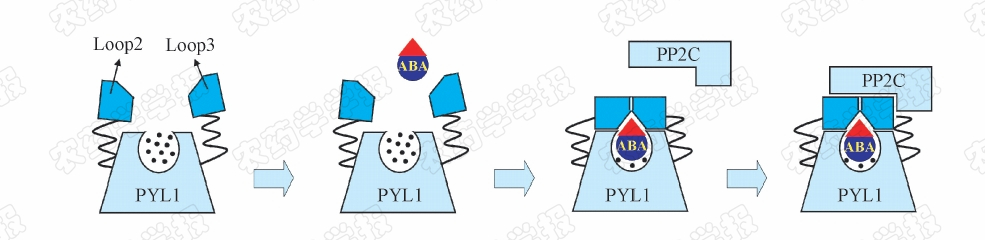 | 图 7脱落酸调控的PYL1与PP2C结合的分子机理 Fig.7 Molecular mechanism of ABA induced binding of PYL1 and PP2C |
本文综述了生长素、赤霉素和脱落酸3种天然植物激素受体结构的发现及其作用机制的研究进展。虽然目前已经发现并证实了多种植物激素受体蛋白,其中有些已得到其晶体结构,但并不能说明每种植物激素只有唯一的受体,因此分离及鉴定新的受体蛋白依然具有很重要的意义。目前我们也只是了解了植物激素信号传导过程中一个小小的环节,其调控植物生长的完整机制(即从激素的合成到发挥作用,再到代谢分解)并未被完全认识,植物激素受体作用机制研究之路还很漫长。此项研究的一个重要意义在于:其可以使人们真正了解植物生长的奥秘所在,有助于设计更为高效的人工植物激素,从而开启利用外源物调控植物生长的新时代。
| [1] | SALAMINI F. Plant Biology. Hormones and the green revolution[J]. Science, 2003, 302(5642): 71-72. |
| [2] | CALLIS J. Plant biology-Auxin action[J]. Nature, 2005, 435(7041): 436-437. |
| [3] | CONSTANTINO M G, LOSCO P, CASTELLANO E E. A novel synthesis of (+/-)-abscisic acid[J]. J Org Chem 1989, 54(3): 681-683. |
| [4] | DHARMASIRI N, DHARMASIRI S, ESTELLE M. The F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435(7041): 441-445. |
| [5] | WOO E J, MARSHALL J, BAULY J, et al. Crystal structure of auxin-binding protein 1 in complex with auxin[J]. EMBO J, 2002, 21(12): 2877-2885. |
| [6] | BERTOŠA B, KOJI Ć-PRODI Ć B, WADE R C, et al. Mechanism of auxin interaction with auxin binding protein (ABP1): a molecular dynamics simulation study[J]. Biophys J 2008, 94(1): 27-37. |
| [7] | WORLEY C K, ZENSER N, RAMOS J, et al. Degradation of Aux/IAA proteins is essential for normal auxin signalling[J]. Plant J, 2000, 21(6): 553-562. |
| [8] | RUEGGER M, DEWEY E, HOBBIE L, et al. Reduced naphthylphthalamic acid binding in the tir3 mutant of Arabidopsis is associated with a reduction in polar auxin transport and diverse morphological defects[J]. Plant Cell, 1997, 9(5): 745-757. |
| [9] | RUEGGER M, DEWEY E, GRAY W M, et al. The TIR1 protein of Arabidopsis functions in auxin response and is related to human SKP2 and yeast grr1p[J]. Genes Dev, 1998, 12(2): 198-207. |
| [10] | GRAY W M, KEPINSKI S, ROUSE D, et al. Auxin regulates SCF(TIR1)-dependent degradation of AUX/IAA proteins[J]. Nature, 2001, 414(6861): 271-276. |
| [11] | TIWARI S B, WANG X J, HAGEN G, et al. AUX/IAA proteins are active repressors, and their stability and activity are modulated by auxin[J]. Plant Cell, 2001, 13(12): 2809-2822. |
| [12] | ZENSER N, ELLSMORE A, LEASURE C, et al. Auxin modulates the degradation rate of Aux/IAA proteins[J]. Proc Natl Acad Sci USA, 2001, 98(20): 11795-11800. |
| [13] | KEPINSKI S, LEYSER O. The arabidopsis F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435(7041): 446-451. |
| [14] | TAN X, CALDERON-VILLALOBOS L I, SHARON M, et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase[J]. Nature, 2007, 446(7136): 640-645. |
| [15] | HAO G F, YANG G F. The role of Phe82 and Phe351 in auxin-induced substrate perception by TIR1 ubiquitin ligase: a novel insight from molecular dynamics simulations[J]. PLoS One, 2010, 5(5): e10742. |
| [16] | UEGUCHI-TANAKA M, NAKAJIMA M, MOTOYUKI A, et al. Gibberellin receptor and its role in gibberellin signaling in plants[J]. Annu Rev Plant Biol, 2007, 58:183-198. |
| [17] | LAMARCO K, THOMPSON C C, BYERS B P, et al. Identification of ets-and notch-related subunits in GA binding protein[J]. Science, 1991, 253(5021): 789-792. |
| [18] | STODDART J L, WILLIAMS P D. Interaction of [(3)H]gibberellin A 1 with a sub-cellular fraction from lettuce (Lactuca sativa L.) hypocotyls: requirement for protein synthesis[J]. Planta, 1979, 147(3): 264-268. |
| [19] | NAKAJIMA M, TAKITA K, WADA H, et al. Partial purification and characterization of a gibberellin-binding protein from seedlings of Azukia angularis[J]. Biochem Biophys Res Commun, 1997, 241(3): 782-786. |
| [20] | DILL A, JUNG H S, SUN T P. The DELLA motif is essential for gibberellin-induced degradation of RGA[J]. Proc Natl Acad Sci USA, 2001, 98(24): 14162-14167. |
| [21] | FU X, RICHARDS D E, AIT-ALI T, et al. Gibberellin-mediated proteasome-dependent degradation of the barley DELLA protein SLN1 repressor[J]. Plant Cell, 2002, 14(12): 3191-3200. |
| [22] | ECKARDT N A. Foolish seedlings and DELLA regulators: the functions of rice SLR1 and arabidopsis RGL1 in GA signal transduction[J]. Plant Cell, 2002, 14(1): 1-5. |
| [23] | PENG J, RICHARDS D E, HARTLEY N M, et al.‘Green revolution’genes encode mutant gibberellin response modulators[J]. Nature, 1999, 400(6741): 256-261. |
| [24] | WEN C K, CHANG C. Arabidopsis RGL1 encodes a negative regulator of gibberellin responses[J]. The Plant Cell, 2002, 14(1): 87-100. |
| [25] | UEGUCHI-TANAKA M, ASHIKARI M, NAKAJIMA M, et al. GIBBERELLIN INSENSITIVE DWARF1 encodes a soluble receptor for gibberellin[J]. Nature, 2005, 437(7059): 693-698. |
| [26] | SHIMADA A, UEGUCHI-TANAKA M, NAKATSU T, et al. Structural basis for gibberellin recognition by its receptor GID1[J]. Nature, 2008, 456(7221): 520-523. |
| [27] | MURASE K, HIRANO Y, SUN T P, et al. Gibberellin-induced DELLA recognition by the gibberellin receptor GID1[J]. Nature, 2008, 456(7221): 459-463. |
| [28] | HAO G F, YANG S G, YANG G F, et al. Computational gibberellin-binding channel discovery unraveling the unexpected perception mechanism of hormone signal by gibberellin receptor[J]. J Comput Chem, 2013, 34(24): 2055-2064. |
| [29] | PEI Z M, MURATA Y, BENNING G, et al. Calcium channels activated by hydrogen peroxide mediate abscisic acid signalling in guard cells[J]. Nature, 2000, 406(6797): 731-734. |
| [30] | BRIERLEY J B, HORTON R W, MELDRUM B S. Physiological observations during prolonged epileptic seizures in primates and their relation to subsequent brain damage[J]. J Physiol, 1972, 222(1): 69-70. |
| [31] | SHEN Y Y, WANG X F, WU F Q, et al. The Mg-chelatase H subunit is an abscisic acid receptor[J]. Nature, 2006, 443(7113): 823-826. |
| [32] | ZHANG D P, WU Z Y, LI X Y, et al. Purification and identification of a 42-kilodalton abscisic acid-specific-binding protein from epidermis of broad bean leaves[J]. Plant Physiol, 2002, 128(2): 714-725. |
| [33] | LIU X, YUE Y, LI B, et al. A G protein-coupled receptor is a plasma membrane receptor for the plant hormone abscisic acid[J]. Science, 2007, 315(5819): 1712-1716. |
| [34] | PANDEY S, NELSON D C, ASSMANN S M. Two novel GPCR-type G proteins are abscisic acid receptors in Arabidopsis[J]. Cell, 2009, 136(1): 136-148. |
| [35] | MA Y, SZOSTKIEWICZ I, KORTE A, et al. Regulators of PP2C phosphatase activity function as abscisic acid sensors[J]. Science, 2009, 324(5930): 1064-1068. |
| [36] | PARK S Y, FUNG P, NISHIMURA N, et al. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins[J]. Science, 2009, 324(5930): 1068-1071. |
| [37] | SANTIAGO J, DUPEUX F, ROUND A, et al. The abscisic acid receptor PYR1 in complex with abscisic acid[J]. Nature, 2009, 462(7273): 665-668. |
| [38] | MELCHER K, NG L M, ZHOU X E, et al. A gate-latch-lock mechanism for hormone signalling by abscisic acid receptors[J]. Nature, 2009, 462(7273): 602-608. |
| [39] | MIYAZONO K, MIYAKAWA T, SAWANO Y, et al. Structural basis of abscisic acid signalling[J]. Nature, 2009, 462(7273): 609-614. |
| [40] | YIN P, FAN H, HAO Q, et al. Structural insights into the mechanism of abscisic acid signaling by PYL proteins[J]. Nat Struct Mol Biol, 2009, 16(12): 1230-1236. |
| [41] | NISHIMURA N, HITOMI K, ARVAI A S, et al. Structural mechanism of abscisic acid binding and signaling by dimeric PYR1[J]. Science, 2009, 326(5958): 1373-1379. |
 2014, Vol.16
2014, Vol.16 


