文章信息
- 任晶, 王政淇, 曹阿莉, 蒋忠纯, 张楠, 张瑞福, 沈其荣
- REN Jing, WANG Zhengqi, CAO Ali, JIANG Zhongchun, ZHANG Nan, ZHANG Ruifu, SHEN Qirong
- 促生解淀粉芽胞杆菌SQR9定殖对黄瓜根系基因表达的转录组学研究
- Transcriptomic analysis of colonization by plant growth-promoting rhizobacterium Bacillus amyloliquefaciens SQR9 on gene expression of cucumber roots
- 南京农业大学学报, 2021, 44(2): 296-304
- Journal of Nanjing Agricultural University, 2021, 44(2): 296-304.
- http://dx.doi.org/10.7685/jnau.202005009
-
文章历史
- 收稿日期: 2020-05-07
植物根际中存在着数量庞大、种类繁多的微生物, 其中, 植物根际促生细菌(plant growth-promoting rhizobacteria, PGPR)是一类能在根际定殖, 并具有促生、协助植物抗逆或生防功能的有益微生物[1]。PGPR菌株作为生产微生物肥料的主要菌种, 在农业上广泛应用, 在提高作物产量和改善作物抗病、抗逆能力上具有较好的效果[2]。有关PGPR菌株的研究主要集中在其促生/生防的作用机制及与植物的互作机制(定殖机制)方面[3]。PGPR的促生机制主要包括提供/活化养分和分泌促生活性物质[4]; 生防机制主要包括产生拮抗物质[5]和与病原菌竞争营养/空间的直接作用以及诱导植物系统抗性的间接作用[6]。PGPR菌株在植物根际的成功定殖是其充分发挥促生/生防功能的前提[2], 该过程主要包括向根际趋化和在根表聚集形成生物膜[7]。促生菌根际趋化或根表成膜能力减弱后, 其根际定殖能力也显著下降[8]。
目前关于PGPR菌株作用机制和定殖机制的研究多从菌株自身方面入手, 而从植物响应促生菌的角度了解上述问题的相关报道还比较少[9-10]。如解淀粉芽胞杆菌SQR9可系统性诱导黄瓜分泌更多的色氨酸, 并以其为底物分泌合成吲哚乙酸(indole-3-acetic acid, IAA), 进而促进植物根系发育和对养分的吸收[11]。Verhagen等[12]通过基因芯片研究拟南芥与荧光假单胞菌WCS417r(诱导系统抗性菌株)的互作过程, 发现菌株WCS417r接种后能诱导叶片中系统抗性相关基因的表达, 从而帮助拟南芥抵御病原菌丁香假单胞菌DC3000侵染叶片。拟南芥在受到病原菌侵染后根系会分泌苹果酸, 其可特异性吸引有益的枯草芽胞杆菌FB17并增强其定殖, 菌株FB17能够诱导植物系统抗性, 从而抵御病原菌[13]。
解淀粉芽胞杆菌SQR9是本实验室分离的1株兼具促生和生防效果的根际有益菌, 在促进黄瓜生长和防控黄瓜土传枯萎病方面效果明显[14]。菌株SQR9可通过活化养分(分泌植酸酶溶解有机磷)和产生植物激素(IAA等)及挥发性物质等发挥促生功能, 通过产生拮抗物质(如抗生素)和诱导系统抗性等机制防控土传病害, 菌株能趋化黄瓜根际并在根表形成生物膜。本研究通过转录组测序技术研究接种菌株SQR9后黄瓜根系基因的表达谱变化, 以期从植物角度进一步扩展和深入了解促生菌的作用机制。
1 材料与方法 1.1 试验材料黄瓜品种为‘津春4号’(天津科润农业科技股份有限公司)。解淀粉芽胞杆菌(Bacillus amyloliquefaciens)SQR9(CGMCC accession No. 5808, 中国普通微生物菌种保藏管理中心)由本实验室提供。低盐LB液体培养基(1 L): 氯化钠3 g, 酵母粉5 g, 蛋白胨10 g, pH7.0。PBS缓冲液(1 L): 氯化钠8 g、氯化钾0.2 g、磷酸二氢钾0.24 g, 磷酸氢二钠1.44 g, pH7.4。MS培养液(1 L): MS培养基4.74 g(青岛海博生物科技公司)。MS固体培养基(1 L): MS培养基4.74 g, 蔗糖7.5 g, 培养基中添加4.5 g·L-1的琼脂, pH7.0。
1.2 试验方法 1.2.1 菌株悬液制备将-80 ℃甘油管保存的菌株SQR9在LB固体平板上划线活化, 37 ℃培养12 h后, 挑取单菌落接种于3 mL新鲜的液体LB试管, 37 ℃、170 r·min-1振荡培养10 h。将该种子液以1%(体积分数)接种量转接至液体LB摇瓶中, 37 ℃、170 r·min-1振荡培养至对数中期(D600=1.0, 约5 h), 4 ℃离心收集菌体, 菌体用无菌PBS缓冲液洗涤2次, 用等体积无菌1/4 MS营养液重悬后待用。
1.2.2 黄瓜幼苗培养及接种将黄瓜种子在温水中浸泡(50 ℃左右), 然后放置于37 ℃恒温培养箱中培养6~8 h, 用70%(体积分数)乙醇消毒1 min, 无菌水洗涤3~4次, 再用2%(体积分数)次氯酸钠消毒8 min, 无菌水洗涤5~6次, 用无菌滤纸将种子表面水分吸干, 放置于MS固体培养基上, 30 ℃避光催芽。种子露白后移到装有固体MS培养基的无菌组培瓶中, 在28 ℃、光/暗培养时间为16 h/8 h的温室中培养; 黄瓜幼苗生长至2片子叶时转移至装有50 mL无菌1/4 MS营养液的三角瓶中, 继续在温室振荡培养72 h。黄瓜苗培养到2叶1心时, 选取长势一致的幼苗, 转移至装有225 mL无菌1/4 MS营养液的三角瓶中, 设置2个处理: 1)对照(CK), 添加25 mL无菌1/4 MS营养液; 2)接种菌株SQR9处理(SQR9), 添加25 mL SQR9菌悬液到三角瓶中, 终浓度为D600=0.1。每个处理设置3个生物学重复, 每个生物学重复包含6株黄瓜幼苗, 处理72 h后取样分析。
1.2.3 黄瓜根系RNA的提取、质量检测和测序文库构建剪取处理72 h的黄瓜根系组织, 用锡箔纸包好并编号, 迅速放置液氮罐中冷冻, 委托上海欧易生物医学科技有限公司完成RNA提取、测序和文库构建, 并使用Illumina测序仪进行测序。
1.2.4 测序数据分析和差异表达基因筛选测序数据已上传至NCBI的SRA数据库(https://www.ncbi.nlm.nih.gov/sra, Accession No.: SRR11850235-SRR11850240)。利用Hisat 2软件[15]将测序数据与参考基因组(CsaV3_1, http://www.cucurbitgenomics.org/organism/20)进行序列比对; 采用Trimmomatic软件[16]对原始数据进行质量预处理, 并对整个质控过程中的reads进行统计汇总。利用DESeq[17]筛选差异表达基因。
1.2.5 差异表达基因的GO(gene ontology)[18]和KEGG(Kyoto encyclopedia of genes and genomes)[19]富集分析采用Blast2GO和topGOR包进行GO功能分析, 得到所有显著性差异基因、上调差异基因以及下调差异基因富集的功能模块并绘图; 利用KEGG数据库对差异蛋白编码基因进行Pathway分析(结合KEGG注释结果), 并用超几何分布检验方法计算每个pathway条目中差异基因富集的显著性。
1.2.6 实时定量PCR分析选取部分基因通过Real-time PCR对转录组结果进行验证, 利用反转录试剂盒(TaKaRa)对1.2.3节中获得的RNA进行反转录, 得到的cDNA采用核酸定量仪Thermo Scientific NANODROP 1000(NanoDrop Technologies, USA)进行定量。Real-time PCR靶标基因和所用引物见表 1。试剂盒为SYBR Premix Ex TaqTM(Perfect Real Time, TaKaRa), 扩增仪为ABI(Applied BIO Systems)PRISM 7500 Real-time PCR System。反应体系(20 μL): SYBR Premix Ex TaqTM(2×)10 μL, 正、反向引物(10 μmol·L-1)各0.4 μL, ROX reference dye Ⅱ(50×)0.4 μL, DNA 2 μL, ddH2O 6.8 μL。反应程序: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 30个循环; 95 ℃ 15 s, 60 ℃ 1 min; 95 ℃ 15 s。每个样品设3个重复, 以无菌水代替模板DNA为阴性对照。实时监测并记录荧光信号的变化。
| 引物名称Primers name | 引物序列Primer sequences (5′→3′) | 引物名称Primers name | 引物序列Primer sequences (5′→3′) | |
| WRKY29-F | CCGATCCCGAAGTCTTTAT | PDC1-F | CTCCGATGCTTTCAAACCGTTA | |
| WRKY29-R | GAAACTTCTTCCGGGTGG | PDC1-R | TGGCAGTATCAATCAACTCGT | |
| ABI4-F | TCTTCACCAACAACCTCGTAT | CYP71A1-F | AGACGTTGAGATTGAAGGTTAC | |
| ABI4-R | TCTGAAGGTAGAGGTGGAAGT | CYP71A1-R | GAGGGTCTCTTTGAATGGC | |
| CsaV3_6G047740-F | GAGAAGAATGTGGACATGACG | PCO1-F | GAGAATGGAAACGAGAGCG | |
| CsaV3_6G047740-R | TGATACCCATCATCTTGCAGT | PCO1-R | AGACCGTCTGATCTTTCTACA | |
| ERF1B-F | TTGTATGAGAGGAGGGTTAGC | TPPJ-F | TTCATGTCCGAAGCTATGAGG | |
| ERF1B-R | CACCGATACTTCATTTCCCG | TPPJ-R | TCTCTGCATCTGCCACTTAC | |
| UGD1-F | CCTGGTCTTGATGAAGTAGTGA | ADH1-F | ACCGATTTGAAAGAAGGCGA | |
| UGD1-R | TATATCTGCCTCTGAGACGTG | ADH1-R | TCTCTTCTGACTTACAGTGACG | |
| SS-F | TTTGGGACAGAACACTCTCAC | CYP86A2-F | TTGCATGATTGGATCTCCGATA | |
| SS-R | GGTAAGGCCAGACTTCAAATC | CYP86A2-R | TTTAGCGAAGAAAGGGAAAGC | |
| ASP1-F | TTGCCTGATCCTACATGGTC | PUB22-F | GAAGATTCTGAGAGTTTCGTCG | |
| ASP1-R | ATGATGATAGTAGCGGTAGGT | PUB22-R | AATCCGCACCATTTCCTG |
采用Excel 2017软件和GraphPad Prism 7.0软件处理分析数据。
2 结果与分析 2.1 黄瓜根系转录组测序的整体信息分别提取菌株SQR9接种72 h和不接菌对照的黄瓜根系RNA用于测序。2个处理的有效测序片段(clean reads)在40 Mb以上, 有效碱基质量值大于30的碱基数目占总碱基数目(Q30)的94%以上, GC含量在43%左右。使用Hisat2软件将测序后经过滤得到的clean reads比对到黄瓜参考基因组序列(CsaV3_1), 比对率(在参考序列上有唯一比对位置的测序序列数量占有效测序数量的比例)超过93%(表 2), 说明转录组测序质量较高, 可用于后续分析。
| 样品 Sample |
原始测序片段/Mb Raw reads |
有效测序片段/Mb Clean reads |
碱基质量值 大于30/% Qphred 30 |
GC含量/% GC content |
比对率/% Uniquely mapped |
| CK_1 | 50.37 | 48.27 | 94.18 | 43.71 | 95.23 |
| CK_2 | 44.09 | 42.24 | 94.08 | 43.69 | 95.13 |
| CK_3 | 52.92 | 50.87 | 94.30 | 43.67 | 95.10 |
| SQR9_1 | 54.83 | 52.55 | 94.12 | 43.59 | 94.17 |
| SQR9_2 | 50.88 | 48.95 | 94.33 | 43.51 | 94.22 |
| SQR9_3 | 54.86 | 52.68 | 94.20 | 43.61 | 93.93 |
通过DESeq软件比较分析SQR9处理组与对照组之间的基因表达差异。按照同时满足表达倍数差异(fold change, FC)≥2和显著性P≤0.05的要求, 共筛选到484个显著差异表达基因。其中SQR9处理后有300个基因表达显著上调, 184个基因表达显著下调, 上调基因数量是下调基因的1.6倍(图 1)。对处理组和对照组黄瓜根系的差异表达基因聚类分析(图 2)表明: 纵列上聚类在同一个簇中的基因可能具有相似的生物学功能; 功能相近的基因具有相近的表达变化趋势, 且处理间的重复性良好。
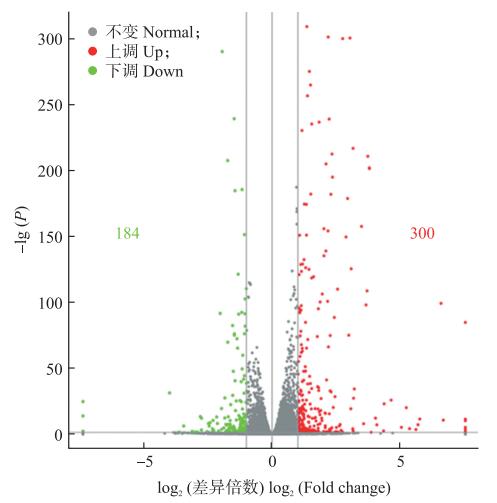
|
图 1 接种SQR9与对照处理中黄瓜根系差异表达基因的火山图 Fig. 1 Volcano of differentially expressed genes ofcucumber roots treated with SQR9 andcontrol treatment |

|
图 2 接种SQR9与对照处理中黄瓜根系差异基因表达谱聚类分析 Fig. 2 Cluster analysis of differential expression genesof cucumber roots treated with SQR9 and thecontrol treatment |
采用Real-time PCR对菌株SQR9处理的黄瓜根系部分差异表达基因进行验证, 发现所选基因在对照和处理组间的表达趋势与转录组测序结果基本一致(表 3), 说明本次测序结果可信度较高。
| 基因 Gene |
基因差异倍数(FC) Fold changes of genes expression |
基因 Gene |
基因差异倍数(FC) Fold changes of genes expression |
|||
| 转录组测序 RNA-seq |
定量PCR Real-time PCR |
转录组测序 RNA-seq |
定量PCR Real-time PCR |
|||
| CsaV33_4G006110: WRKY29 | 3.50±0.25 | 2.89±0.46 | CsaV33_3G033550: PDC1 | 4.05±0.86 | 5.31±0.36 | |
| CsaV33_6G001910: ABI4 | 51.92±0.48 | 21.85±0.63 | CsaV33_1G008960: CYP71A1 | -7.69±0.05 | -1.49±0.04 | |
| CsaV33_6G047740 | -5.00±0.07 | -2.94±0.10 | CsaV33_4G012050: PCO1 | 6.25±2.34 | 8.12±1.00 | |
| CsaV33_7G027380: ERF1B | 2.35±0.35 | 6.06±3.33 | CsaV33_UNG144090: TPPJ | 2.20±0.66 | 2.48±0.09 | |
| CsaV33_1G002790: UGD1 | 4.31±0.40 | 4.26±0.12 | CsaV33_7G024960: ADH1 | 3.57±0.30 | 10.08±0.41 | |
| CsaV33_1G041400: SS | 2.86±0.21 | 3.78±0.28 | CsaV33_5G002710: CYP86A2 | 2.78±0.31 | 2.52±0.17 | |
| CsaV33_3G035690: ASP1 | 3.83±0.60 | 4.70±0.27 | CsaV33_6G038570: PUB22 | 2.19±0.53 | 3.02±0.24 | |
GO富集分析可获得差异表达基因所参与的生物过程(biological process, BP)、分子功能(molecular function, MF)和细胞组分(cellular component, CC)的信息。在SQR9处理组与对照组之间的差异表达基因中, 共筛选到325个基因参与了BP、MF和CC的过程, 其中上调基因为179个, 下调基因为146个(图 3)。参与BP的差异基因主要集中在细胞过程、代谢过程、单组织过程、应激反应和生物调节, 差异基因数分别为170、150、148、125和84。在MF类别中, 差异基因被富集的功能主要是结合和催化活性, 差异基因数分别187和159。在CC中, 差异基因主要存在于细胞、细胞部分、细胞器和膜等方面。由此可知, 解淀粉芽胞杆菌SQR9接种后主要影响黄瓜根系各细胞组织的生长、初级和次级代谢以及环境响应等方面, 且很多差异表达基因同时参与2个甚至更多的生物学途径。

|
图 3 接种SQR9与对照处理中黄瓜根系差异表达基因的GO功能分析 Fig. 3 GO analysis of differential expression genes(DEG)of cucumber roots treatedwith SQR9 and the control treatment |
由图 4可知: 与对照相比, KEGG通路共富集到143个差异表达基因, 主要分布在4个大类中, 分别是代谢、遗传信息过程、环境信息过程和细胞过程。其中代谢大类涉及11个代谢途径, 包括129个差异表达基因(78个上调, 51个下调)。上调基因主要集中在脂质代谢、能量代谢、碳水化合物代谢、其他次级代谢物合成和氨基酸代谢通路中; 下调基因主要集中在脂质代谢、碳水化合物代谢和氨基酸代谢通路中, 这表明在菌株SQR9诱导下, 黄瓜根系各组织的代谢通路发生了显著变化, 影响黄瓜的生长。

|
图 4 接种SQR9与对照处理中黄瓜根系差异基因的KEGG功能分类 Fig. 4 KEGG classification of DEG of cucumber roots treated with SQR9 and the control treatment |
从表 4可见: SQR9处理黄瓜根系差异表达基因参与KEGG通路前20的条目中(根据差异基因数目排序), 所涉及的差异基因参与的通路均属于代谢大类, 包括氨基酸代谢、碳水化合物代谢和次级代谢物生物合成等途径。其中氨基酸代谢途径主要涉及色氨酸、甘氨酸、丝氨酸和苏氨酸等的代谢; 碳水化合物代谢途径包括糖酵解途径(EMP途径)、淀粉和蔗糖代谢途径以及氨基糖和核糖代谢途径, 其中涉及14个上调基因, 8个下调基因, 表明菌株SQR9通过促进黄瓜植物组织中的糖代谢, 为各组织的生长提供能量, 有利于植物生长发育。次级代谢物的生物合成途径主要包括苯丙烷生物合成和类黄酮生物合成通路, 其中涉及20个上调基因和5个下调基因, 这些基因在植物的生长发育和应答胁迫中发挥重要作用, 如避免逆境胁迫伤害和提高植物抗病性等[5, 20]。
| 代谢分类 Metabolism classification |
通路名称 Term of pathway |
差异基因数 DEG number |
P值 P-value |
| 氨基酸代谢Amino acid metabolism | ko00260:甘氨酸, 丝氨酸和苏氨酸的代谢 Glycine, serine and threonine metabolism |
4 | 0.019 3 |
| 氨基酸代谢Amino acid metabolism | ko00270:半胱氨酸和蛋氨酸代谢 Cysteine and methionine metabolism |
4 | 0.034 7 |
| 氨基酸代谢Amino acid metabolism | ko00330:精氨酸和脯氨酸代谢 Arginine and proline metabolism |
3 | 0.023 5 |
| 氨基酸代谢Amino acid metabolism | ko00350:酪氨酸代谢Tyrosine metabolism | 5 | 0.000 2 |
| 氨基酸代谢Amino acid metabolism | ko00360:苯丙氨酸代谢Phenylalanine metabolism | 3 | 0.026 5 |
| 氨基酸代谢Amino acid metabolism | ko00380:色氨酸代谢Tryptophan metabolism | 4 | 0.000 8 |
| 辅助因子和维生素的代谢 Metabolism of cofactors and vitamins |
ko00830:视黄醇代谢Retinol metabolism | 4 | 0 |
| 能量代谢Energy metabolism | ko00680:甲烷代谢Methane metabolism | 4 | 0.003 3 |
| 能量代谢Energy metabolism | ko00910:氮代谢Nitrogen metabolism | 5 | 0.000 1 |
| 其他氨基酸的代谢 Metabolism of other amino acids |
ko00460:氰氨基酸代谢Cyanoamino acid metabolism | 3 | 0.025 0 |
| 其他次级代谢产物的生物合成 Biosynthesis of other secondary metabolites |
ko00940:苯丙烷生物合成Phenylpropanoid biosynthesis | 23 | 0 |
| 其他次级代谢产物的生物合成 Biosynthesis of other secondary metabolites |
ko00941:类黄酮生物合成Flavonoid biosynthesis | 3 | 0.000 7 |
| 其他次级代谢产物的生物合成 Biosynthesis of other secondary metabolites |
ko00945:二苯乙烯类、二芳基庚烷和姜醇的生物合成 Stilbenoid, diarylheptanoid andgingerol biosynthesis |
3 | 0.000 1 |
| 碳水化合物代谢Carbohydrate metabolism | ko00010:糖酵解/糖异生Glycolysis/gluconeogenesis | 6 | 0.007 4 |
| 碳水化合物代谢Carbohydrate metabolism | ko00500:淀粉和蔗糖代谢Starch and sucrose metabolism | 10 | 0.009 9 |
| 碳水化合物代谢Carbohydrate metabolism | ko00520:氨基糖和核苷酸糖代谢 Amino sugar and nucleotide sugar metabolism |
6 | 0.013 5 |
| 异生物素的生物降解和代谢 Xenobiotics biodegradation and metabolism |
ko00980:细胞色素P450对异生物素的代谢 Metabolism of xenobiotics by cytochrome P450 |
4 | 0.001 7 |
| 脂质代谢Lipid metabolism | ko00071:脂肪酸降解Fatty acid degradation | 4 | 0.002 3 |
| 脂质代谢Lipid metabolism | ko00140:类固醇激素的合成Steroid hormone biosynthesis | 4 | 0 |
| 脂质代谢Lipid metabolism | ko00592:α-亚麻酸代谢α-linolenic acid metabolism | 5 | 0.002 0 |
参与氨基酸代谢途径的代表性差异基因主要表现为上调(图 5-A): CsaV3_2G009060基因编码肉桂酸4-羟化酶(cinnamate 4-hydroxylase, C4H), C4H是合成酚酸类物质的关键酶, 具有抗胁迫的作用; ASP1基因编码天冬氨酸转氨酶蛋白(aspartate aminotransferase, ASPAT), ASPAT是植物初级氮素同化的关键酶, 可提高植物的氮素利用效率; ADH1基因编码醇脱氢酶(alcohol dehydrogenase, ADH), ADH属于锌结合脱氢/还原蛋白超家族, 在乙醇发酵途径中发挥重要作用; PCO1基因编码半胱氨酸氧化酶蛋白(plant cysteine oxidases, PCO), PCO可以感知细胞内氧浓度的变化。

|
图 5 接种SQR9与对照处理中黄瓜根系代表性差异基因表达情况 Fig. 5 Representative differential expression genes of cucumber roots treated with SQR9 and the control treatment A. 氨基酸代谢; B. 碳水化合物代谢; C. 次级代谢物的生物合成; D.信号转导和防御。每个基因颜色代表SQR9处理和对照间的差异倍数对数值。 A. Amino acid metabolism; B. Carbohydrate metabolism; C. Biosynthesis of secondary metabolites; D. Signal transduction and defense response. The color of each gene represents the logarithm of fold change between the inoculation of SQR9 and the control treatment. |
碳水化合物代谢途径有22个差异显著表达基因, 其中14个基因在菌株SQR9接种后显著上调(上调3~5倍), 部分代表性基因以热图形式展示(图 5-B)。UGD1基因编码UDP-葡萄糖-6-脱氢酶家族蛋白, 参与细胞壁合成; SS基因编码蔗糖合酶蛋白, 催化蔗糖的合成和分解, 是糖代谢中的关键酶; PDC1基因编码丙酮酸脱羧酶蛋白, 参与糖酵解途径; TPPJ基因编码海藻-6-磷酸磷酸酶蛋白; FBA6基因编码果糖二磷酸醛缩酶, 是卡尔文循环途径中的关键酶。
次级代谢物的生物合成大类主要包括苯丙烷代谢通路和类黄酮生物合成通路, 共有25个差异基因属于该代谢途径。从图 5-C可知: CsaV3_4G027010(Peroxidase 11, PER11)、CsaV3_5G033660(Peroxidase 21, PER21)、CsaV3_6G002170(Peroxidase 51, PER51)和CsaV3_6G018990(Peroxidase 52, PER52)是编码过氧化物酶(参与植物抗病抗逆过程)的基因, 除PER21基因表达下调外其他基因均在菌株SQR9接种后显著上调; CsaV3_2G009070(编码肉桂酸4-羟化酶)、COMT1(编码咖啡酸3-O-甲基转移酶)、CCR2(编码肉桂酰辅酶A还原酶)和SHT(编码亚精胺羟肉桂酰基转移酶)等基因表达均显著上调; BGLU12(编码β-葡萄糖苷酶)和CsaV3_3G034620(编码肌钙蛋白还原酶)基因表达下调。
在信号转导和防御反应大类中, 差异基因主要编码转录因子和一些功能酶。如图 5-D所示: WRKY29基因(编码乙烯途径中的转录因子)、PUB22基因(编码E3泛素转移酶蛋白)和CYP86A2基因(编码细胞色素P450蛋白)表达上调, 其中WRKY转录因子在各种生物胁迫防卫反应中发挥重要作用。ERF1B(ethylene-responsive factor, ERF)和ABI4(abscisic acid-insensitive 4)是编码乙烯途径中转录因子的基因, 与对照组相比其表达显著上调。IAA8基因编码IAA信号响应途径抑制子, 其基因表达显著下调; SAUR21和SAUR77基因编码IAA途径下游响应蛋白, 其表达显著上调。上述3个IAA途径基因的表达变化表明菌株SQR9产生的外源IAA激活了植物内源的IAA信号途径, 从而促进黄瓜根系发育和植株生长。
3 讨论与结论解淀粉芽胞杆菌SQR9是一株具有促进黄瓜、玉米等植物生长和防控病原菌侵染功能的根际促生菌。黄瓜植株接种解淀粉芽胞杆菌SQR9后, 其根系的差异表达基因主要涉及氨基酸代谢、碳水化合物代谢、次级代谢物的生物合成以及信号转导与防御4个途径, 促进植物的生长发育, 提高宿主的抗病抗逆能力; 相关研究结果从植物角度阐述根际促生菌的作用机制。
在植物生长方面, 菌株SQR9主要调控黄瓜植株的氨基酸代谢、碳水化合物代谢和生长素信号途径。在氨基酸代谢方面, 黄瓜根部接种菌株SQR9后, 其根部天冬氨酸转氨酶(ASPAT)的编码基因表达上调。王宇晨等[21]发现ASPAT1基因在杨树根、茎和叶中的表达无差异, 而ASPAT2—ASPAT9基因在根部的表达量较高; 其中ASPAT4基因和ASPAT8基因可提高植物氮素利用效率。因此, 菌株SQR9能激活ASPAT从而提高黄瓜的氮素利用能力并促进植株生长发育。碳水化合物代谢方面, 响应菌株SQR9后, 黄瓜根部UGD1、PDC1和FBA6等基因表达上调。UGD是细胞壁多糖、纤维素和半纤维素合成的前提, 参与细胞壁的形成过程[22]; 郑玉成等[23]发现茶树尿苷二磷酸-葡萄糖-脱氢酶基因(CsUGD)基因影响茶树的生长发育, 并且是茶树多糖合成途径的关键合成酶。丙酮酸脱羧酶(PDC)参与糖酵解途径并催化丙酮酸脱羧生成乙醛[24]。果糖二磷酸醛缩酶(FBA)则是卡尔文循环的关键酶; 蔡冰冰[25]发现过表达番茄中的SlFBA4和SlFBA7对植株生长发育及光合作用有促进作用。因此, 菌株SQR9对黄瓜根系UGD1、PDC1和FBA6等基因的激活有利于黄瓜的生长发育。除此之外, 前期研究已证明IAA合成是菌株SQR9促进植物生长的主要机制。已知高浓度IAA与IAA受体结合后形成复合体, 其与Aux/IAA抑制子高效结合并促使后者泛素化, 泛素化后的抑制子被26S蛋白酶快速降解, 从而解除对下游IAA响应因子的抑制; 随后ARF激活下游的IAA信号途径基因表达, 发挥促进侧根形成和根系发育等作用[26]。本试验中, 编码IAA信号响应途径抑制子的IAA8基因表达在接种菌株SQR9后显著下调, 而编码该途径下游的IAA响应因子的SAUR21和SAUR77基因表达显著上调, 说明黄瓜植株对菌株SQR9产生的外源IAA产生响应, 诱导植物IAA响应途径中下游基因的表达, 有利于植物的生长。这一结果与前期实验室发现IAA合成是菌株SQR9主要促生机制的现象相吻合[27]。巩子毓[28]在田间试验中发现含有菌株SQR9的生物有机肥处理的黄瓜产量连续5季高于普通有机肥处理和化肥处理, 并且连续施用生物有机肥能有效改善黄瓜品质(提高可溶性糖和蛋白质含量)。以上分析表明菌株SQR9通过激活黄瓜根部的氨基酸代谢、碳水化合物代谢(EMP途径和卡尔文循环等)和IAA信号途径促进黄瓜植株的生长发育, 并提高黄瓜产量和品质。
在调控植物免疫和抗逆响应方面, Wu等[29]发现解淀粉芽胞杆菌SQR9能够激活植物的水杨酸(SA)、茉莉酸(JA)/乙烯(ET)等信号通路, 从而激发宿主的诱导性系统抗性, 提高其对后续接种病原菌的抵抗力。本研究中转录组测序发现, 与对照比较接种菌株SQR9处理72 h后黄瓜根部WRKY29和ERF1编码基因的表达水平提高。植物WRKY转录因子在植物防御和环境胁迫下起重要作用。ERF转录因子属于AP2/ERF大家族, AtERF1参与ET和JA介导的防御信号途径, 并可诱导下游防御相关基因的表达[30]。说明菌株SQR9可通过激活黄瓜根部免疫途径基因的表达而诱导植物免疫, 提高宿主的抗病能力。除此之外, 在植物抗逆方面, 转录组测序结果表明接种SQR9能够诱导黄瓜根部C4H和ADH1基因的表达。Kim等[31]发现洋麻HcC4H基因可以应对损伤、水杨酸和盐等非生物胁迫, 从而提高植物的耐胁迫能力。Kato-Noguchi[32]发现玉米幼苗受到高盐胁迫时, 会通过提高ADH活性而增强根部代谢, 以抵御高盐胁迫。因此, 推断菌株SQR9对C4H以及ADH1等抗逆基因的激活有助于提高宿主抵抗生物和非生物逆境胁迫能力。
综上所述, 后续研究将进一步探索黄瓜植株地上部响应菌株SQR9的转录组特征, 并与本文的地下部数据进行整合分析, 以期阐明菌株SQR9对黄瓜促生和抗病的作用机制。
| [1] |
Vacheron J, Desbrosses G, Bouffaud M, et al. Plant growth-promoting rhizobacteria and root system functioning[J]. Frontiers in Plant Science, 2013, 4: 356. |
| [2] |
Lugtenberg B, Kamilova F. Plant-growth-promoting rhizobacteria[J]. Annual Review of Microbiology, 2009, 63: 541-556. DOI:10.1146/annurev.micro.62.081307.162918 |
| [3] |
Boonaert C J P, Dufrene Y F, Rouxhet P G. Encyclopedia of Environmental Microbiology[M]. New York: Wiley, 2003: 450-465.
|
| [4] |
Egamberdiyeva D. The effect of plant growth promoting bacteria on growth and nutrient uptake of maize in two different soils[J]. Applied Soil Ecology, 2007, 36: 184-189. DOI:10.1016/j.apsoil.2007.02.005 |
| [5] |
Xu Z H, Shao J H, Li B, et al. Contribution of bacillomycin D in Bacillus amyloliquefaciens SQR9 to antifungal activity and biofilm formation[J]. Applied and Environment Microbiology, 2013, 79(3): 808-815. DOI:10.1128/AEM.02645-12 |
| [6] |
Rolfe S A, Griffiths J, Ton J. Crying out for help with root exudates: adaptive mechanisms by which stressed plants assemble health-promoting soil microbiomes[J]. Current Opinion in Microbiology, 2019, 49: 73-82. DOI:10.1016/j.mib.2019.10.003 |
| [7] |
Zhang N, Yang D Q, Wang D D, et al. Whole transcriptomic analysis of the plant-beneficial rhizobacterium Bacillus amyloliquefaciens SQR9 during enhanced biofilm formation regulated by maize root exudates[J]. BMC Genomics, 2015, 16(1): 1-20. DOI:10.1186/1471-2164-16-1 |
| [8] |
Feng H C, Zhang N, Fu R X, et al. Recognition of dominant attractants by key chemoreceptors mediates recruitment of plant growth-promoting rhizobacteria[J]. Environment Microbiology, 2019, 21(1): 402-415. DOI:10.1111/1462-2920.14472 |
| [9] |
Jones J D, Dangl J L. The plant immune system[J]. Nature, 2006, 444(7117): 323-329. DOI:10.1038/nature05286 |
| [10] |
Ausubel F M. Are innate immune signaling pathways in plants and animals conserved?[J]. Nature Immunology, 2005, 6(10): 973-979. DOI:10.1038/ni1253 |
| [11] |
Liu Y P, Chen L, Zhang N, et al. Plant-microbe communication enhances auxin biosynthesis by a root-associated bacterium Bacillus amyloliquefaciens SQR9[J]. Molecular Plant-Microbe Interactions, 2016, 29(4): 324-330. DOI:10.1094/MPMI-10-15-0239-R |
| [12] |
Verhagen B W M, Glazebrook J, Zhu T, et al. The transcriptome of rhizobacteria-induced systemic resistance in Arabidopsis[J]. Molecular Plant-Microbe Interactions, 2004, 17(8): 895-908. DOI:10.1094/MPMI.2004.17.8.895 |
| [13] |
Rudrappa T, Czymmek K J, Paré P W, et al. Root-secreted malic acid recruits beneficial soil bacteria[J]. Plant Physiology, 2008, 148(3): 1547-1556. DOI:10.1104/pp.108.127613 |
| [14] |
Cao Y, Zhang Z H, Ling N, et al. Bacillus subtilis SQR9 can control Fusarium wilt in cucumber by colonizing plant roots[J]. Biology and Fertility of Soils, 2011, 47(5): 495-506. DOI:10.1007/s00374-011-0556-2 |
| [15] |
Kim D, Langmead B, Salzberg S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 |
| [16] |
Bolger A M, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [17] |
Anders S, Huber W. Differential Expression of RNA-Seq Data at the Gene Level: the DESeq Package[M]. Heidelberg: Germany, 2012.
|
| [18] |
Alexa A, Rahnenfuhrer J. TopGO: enrichment analysis for gene ontology[EB/OL]. [2020-04-05]. http://www.bioconductor.org/packages/2.11/bioc/html/topGO.html.
|
| [19] |
Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2008, 36(Suppl 1): D480-D484. |
| [20] |
Colquhoun T A, Kim J Y, Wedde A E, et al. PhMYB4 fine-tunes the floral volatile signature of Petunia×hybrida through PhC4H[J]. Journal of Experimental Botany, 2011, 62(3): 1133-1143. DOI:10.1093/jxb/erq342 |
| [21] |
王宇晨, 曲春浦, 刘关君. 杨树天冬氨酸转氨酶基因家族鉴定及表达分析[J]. 南方农业学报, 2019, 50(3): 506-514. Wang Y C, Qu C P, Liu G J. Identification and expression analysis of poplar aspartate aminotransaminase (ASPAT) gene family[J]. Journal of Southern Agriculture, 2019, 50(3): 506-514 (in Chinese with English abstract). |
| [22] |
Rudrappa T, Czymmek K J, Paré P W, et al. Root-secreted malic acid recruits beneficial soil bacteria[J]. Plant Physiology, 2008, 148(3): 1547-1556. DOI:10.1104/pp.108.127613 |
| [23] |
郑玉成, 王鹏杰, 林浥, 等. 茶树尿苷二磷酸-葡萄糖-脱氢酶基因的克隆及其表达分析[J]. 中草药, 2018, 49(23): 5640-5648. Zheng Y C, Wang P J, Lin Y, et al. Cloning and expression of UGD-glucose-dehydrogenase gene CsUGD in Camellia sinensis[J]. Chinese Traditional and Herbal Drugs, 2018, 49(23): 5640-5648 (in Chinese with English abstract). DOI:10.7501/j.issn.0253-2670.2018.23.025 |
| [24] |
Tadege M, Dupuis I, Kuhlemeier C. Ethanolic fermentation: new functions for an old pathway[J]. Trends in Plant Science, 1999, 4(8): 320-325. DOI:10.1016/S1360-1385(99)01450-8 |
| [25] |
蔡冰冰. 番茄果糖-1, 6-二磷酸醛缩酶基因表达特性及其对光合作用的调控[D]. 泰安: 山东农业大学, 2017. Cai B B. Expression pattern and regulation of fructose 1, 6-diphosphate aldolase gene on photosynthesis in tomato[D]. Tai'an: Shandong Agricultural University, 2017(in Chinese with English abstract). |
| [26] |
Tan X, Calderon-Villalobos L I A, Sharon M, et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase[J]. Nature, 2007, 446(7136): 640-645. DOI:10.1038/nature05731 |
| [27] |
邵佳慧. 解淀粉芽孢杆菌SQR9吲哚乙酸合成途径及促生效应的研究[D]. 南京: 南京农业大学, 2014: 13-14. Shao J H. Indole-3-acetic acid synthesis pathways and plant-growth-promotion effects of Bacillus amyloliquefaciens SQR9[D]. Nanjing: Nanjing Agricultural University, 2014: 13-14(in Chinese with English abstract). |
| [28] |
巩子毓. 连续施用生物有机肥促进黄瓜产量及改善土壤微生物区系研究[D]. 南京: 南京农业大学, 2017. Gong Z Y. Continuous application of bio-organic fertilizer promoted cucumber yields and altered soil microflora[D]. Nanjing: Nanjing Agricultural University, 2017(in Chinese with English abstract). |
| [29] |
Wu G W, Liu Y P, Xu Y, et al. Exploring elicitors of the beneficial rhizobacterium Bacillus amyloliquefaciens SQR9 to induce plant systemic resistance and their interactions with plant signaling pathways[J]. Molecular Plant-Microbe Interactions, 2018, 31(5): 560-567. DOI:10.1094/MPMI-11-17-0273-R |
| [30] |
Lorenzo O, Piqueras R, Sánchez-Serrano J J, et al. ETHYLENE RESPONSE FACTOR 1 integrates signals from ethylene and jasmonate pathways in plant defense[J]. The Plant Cell, 2003, 15(1): 165-178. DOI:10.1105/tpc.007468 |
| [31] |
Kim J, Choi B, Natarajan S, et al. Expression analysis of kenaf cinnamate 4-hydroxylase (C4H) ortholog during developmental and stress responses[J]. Plant Omics, 2013, 6(1): 65-72. |
| [32] |
Kato-Noguchi H. Osmotic stress increases alcohol dehydrogenase activity in maize seedlings[J]. Biologia Plantarum, 2000, 43(4): 621-624. DOI:10.1023/A:1002864318871 |




