文章信息
- 宋蒙飞, 王星, 张开京, 陈劲枫, 娄群峰
- SONG Mengfei, WANG Xing, ZHANG Kaijing, CHEN Jinfeng, LOU Qunfeng
- 黄瓜DnaJ基因家族鉴定及对高温胁迫的表达响应
- Identification of DnaJ gene family in cucumber and its expression response to high temperature stress
- 南京农业大学学报, 2021, 44(2): 267-277
- Journal of Nanjing Agricultural University, 2021, 44(2): 267-277.
- http://dx.doi.org/10.7685/jnau.202006038
-
文章历史
- 收稿日期: 2020-06-24
DnaJ蛋白(又称J蛋白)是一类保守的辅伴蛋白家族, 因包含1个标志性高保守的J结构域而得名, 在生物体内作为热激蛋白(HSP70)的辅伴分子而存在[1]。而HSP70是生物在高温环境下合成的一组蛋白质, 存在于从细菌到人类的活生物体中, 是生物响应高温胁迫的主要因子[2]。作为HSP70的辅伴侣分子, DnaJ蛋白在胁迫条件下能促进HSP70的ATP酶活性, 并参与维持细胞内蛋白的折叠、复合物解聚等多种生命活动进程, 在生命体抵抗和适应逆境胁迫方面发挥着重要作用[3]。
DnaJ蛋白通常情况下包含4个保守的结构域: N端J结构域、甘氨酸/丙氨酸结构域、含4个CxxCXGXG重复基序的锌指结构域和C末端结构域, DnaJ蛋白可根据其含有的结构域差异分为不同亚型(常见为3种类型)[4]。DnaJ蛋白广泛存在于生命体内不同部位, 并因其在细胞内定位的不同而具有不同的功能, 例如在拟南芥中已鉴定的120个DnaJ蛋白亚细胞分别定位于线粒体、叶绿体、内质网、细胞核和质膜上[5]。目前研究最多的为定位于叶绿体的DnaJ蛋白, 其在叶绿体中可参与PS Ⅱ光合系统组装或修复、类囊体膜建成、叶绿体分裂发育、叶绿体转录、蛋白转运等过程[6-8]。DnaJ蛋白另一重要功能是能够参与植物胁迫响应进程, 已有报道表明DnaJ蛋白在植物抗旱、抗高温、抗冷、抗盐以及抗病方面均发挥重要作用[3]。
高温容易造成植物体内酶系统紊乱、细胞膜系统破坏、各组分改变等一系列生理生化变化, 从而影响光合及呼吸作用, 改变植物的源库关系, 使作物的生长发育受到严重的影响, 最终造成减收、减产等一系列问题[9-10]。黄瓜(Cucumis sativus L.)是重要的瓜类蔬菜作物之一, 适宜生长温度为16~26 ℃, 属喜温作物, 但不耐热[11]。目前在设施栽培或者南方地区露地栽培中, 高温已成为设施黄瓜栽培生产的主要障碍[12]。在黄瓜苗期、营养生长期、初花期、结果期遇到高温环境会影响黄瓜正常生长发育, 降低黄瓜产量和品质, 最终导致商品性降低[13-15]。高温胁迫已经给黄瓜生产带来了巨大的损失, 是我国黄瓜生产中急需解决的问题之一。目前关于黄瓜耐热性的研究主要集中在分子标记的开发上, 研究者利用黄瓜耐热及不耐热材料通过连锁性分析鉴定了多个与耐高温相关的QTL位点及连锁标记[16], 但是关于黄瓜耐高温相关基因家族的分析尚未见报道。
目前在拟南芥、水稻、棉花中均有关于DnaJ基因家族的研究报道[5, 17-18]。但是, 在黄瓜中关于DnaJ基因家族的研究尚未见报道。本研究基于已公布的黄瓜基因组信息对黄瓜DnaJ基因家族进行全基因组解析, 注释该基因家族包括基因组成、结构、染色体分布和系统发育分析等信息, 同时, 通过公共数据库、RNA-seq数据、实时定量PCR分析DnaJ基因的表达模式, 并探讨其在植物响应高温环境胁迫中的功能, 旨在为探明黄瓜响应高温胁迫应答机制提供理论参考。
1 材料与方法 1.1 黄瓜DnaJ基因家族全基因组鉴定首先, 从葫芦科作物基因组数据库(http://cucurbitgenomics.org/)下载黄瓜‘9930’参考基因组相关信息文件(包括基因组序列、基因编码序列、蛋白序列)[19], 从pfam数据库数(http://pfam.xfam.org/)下载DnaJ蛋白HMM(隐马尔可夫模型)种子文件(编号: PF00226)[20]。其次, 参考崔梦杰等[21]的方法, 以种子文件为模板, 通过HMMER 3.1软件的hmmsearch功能搜索蛋白质数据库, 参数设为默认值。以初次筛选得到的黄瓜DnaJ蛋白序列为模板, 通过clusterW软件将这些序列进行多序列对比, 利用HMMER 3.1软件中hmmbuild功能将对比结果建立黄瓜DnaJ蛋白特异hmm模型。最后, 通过hmmsearch功能以黄瓜DnaJ蛋白特异hmm模型为模板再次搜索黄瓜蛋白数据库, 所得结果通过NCBI的CDD数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)检查蛋白质的保守结构域后[22], 手动筛除不含DnaJ特异J结构域核心区序列(组氨酸/脯氨酸/天冬氨酸, 即His/Pro/Asp, HPD结构)的蛋白编号, 最终得到黄瓜DnaJ蛋白家族基因。
1.2 黄瓜DnaJ基因家族染色体位置、蛋白特性及结构分析黄瓜DnaJ基因家族染色体位置信息通过葫芦科基因组数据库Browser功能获得, 并通过Circos软件(http://circos.ca/)表现为图形模式; 以基因名称搜索黄瓜基因组文件得到DnaJ基因家族蛋白全长序列, 通过序列分析工具在线软件(http://www.detaibio.com/sms2/protein_mw.html)计算蛋白相对分子质量、等电点(PI值); 利用在线软件WOLF PRORT(https://www.genscript.com/wolf-psort.html?src=leftbar)预测蛋白亚细胞定位; 使用MEME在线软件(http://meme-suite.org/tools/meme)分析蛋白motif特征[23], motif搜索个数为10, 其他参数默认, 通过TBtools展示分析结果。
1.3 黄瓜DnaJ基因家族进化及复制模式分析参考Miernyk[5]的方法获得拟南芥DnaJ基因家族信息, 并在拟南芥数据库下载拟南芥DnaJ基因家族蛋白序列。将黄瓜及拟南芥DnaJ基因家族J蛋白结构域序列通过Neighbor-joining进行聚类分析, 使用MEGA 7.0软件构建系统发育树[24], Bootstrap值设置为1 000。通过在线软件EvolView(https://www.evolgenius.info/evolview/)进行图形调整。以黄瓜DnaJ基因家族蛋白序列为模板, 通过MCScanX软件进行基因组内复制事件鉴定[25], 并通过Circos软件表现为图形模式。
1.4 黄瓜DnaJ基因家族表达分析在NCBI的SRA数据库中下载黄瓜基因在各组织的表达水平原始数据[26], 通过salmon软件计算黄瓜基因表达量[27], 根据基因名称获得黄瓜DnaJ基因家族在黄瓜各组织表达量, 通过Heml软件绘制热图。
以华北密刺型黄瓜自交系品种‘长春密刺’为试验材料, 于光照培养箱中培养黄瓜幼苗至5~6片真叶期, 对其进行高温胁迫处理。高温处理: 昼/夜时间为16 h/8 h, 温度为42 ℃/32 ℃; 对照: 昼/夜时间为16 h/8 h, 温度为28 ℃/24 ℃。分别于0、6、12、24、48 h时取黄瓜叶片, 液氮速冻后保存于-80 ℃, 每个处理3个重复。利用RNA提取试剂盒(TaKaRa)提取植物叶片总RNA, 利用Prime ScriptTM RT Reagent试剂盒(TaKaRa)反转录合成cDNA, 使用SYBR Premix Ex TaqTM试剂盒(TaKaRa)进行荧光定量反应。考虑到具有高度相似性核心结构的基因可能具有相同的调控功能, 因此分别在黄瓜DnaJ基因家族各亚簇组别中随机挑选1个基因(共9个基因), 参考其编码RNA序列设计定量PCR引物, 并检测基因在各处理中的相对表达量。定量PCR体系、反应程序及计算方法采用Song等[28]的方法。
2 结果与分析 2.1 黄瓜DnaJ基因家族鉴定及蛋白序列信息分析以PF00226的HMM文件为索引, 对黄瓜基因组进行DnaJ基因筛选, 在去除冗余基因后, 共得到81个黄瓜DnaJ基因(表 1)。这些基因所编码的蛋白序列中至少含有1个完整的J结构域核心区序列(HPD结构)。根据其在染色体上的位置信息, 重新命名了这些基因名称(CsDnaJ1—CsDnaJ81)(表 1)。对DnaJ基因编码的蛋白序列进行分析, 发现DnaJ基因编码的蛋白氨基酸长度为113~2 551 aa, 其中66个(80.2%)蛋白序列长度都大于200 aa。另外, 其蛋白相对分子质量为(12.10~279.60)×103, 等电点(PI)为4.56~11.20。亚细胞位置预测显示, 大部分蛋白定位于细胞核(42/81, 51.8%)及叶绿体(20/81, 24.7%)中, 其余分别定位于细胞质(10)、细胞膜(3)、线粒体(2)、质体(2)、内质网(1)及液泡(1)中。
| 基因序号 Gene ID |
新命名 Newname |
染色体 Chromosome |
位置 Position |
分组 Group |
氨基酸长度 The length of amino acid |
相对分子质量/103 Relativemolecular mass |
等电点 PI |
亚细胞定位 Subcellularlocalization |
| Csa1G001510 | CsDnaJ1 | Chr 1 | 266 604~269 244 | Ⅳ | 842 | 94.27 | 9.33 | 细胞核Nucleus |
| Csa1G013230 | CsDnaJ2 | Chr 1 | 1 782 802~1 785 241 | Ⅴ | 589 | 67.46 | 5.11 | 细胞核Nucleus |
| Csa1G031710 | CsDnaJ3 | Chr 1 | 3 284 188~3 303 460 | Ⅸ | 388 | 42.62 | 8.85 | 叶绿体Chloroplast |
| Csa1G042670 | CsDnaJ4 | Chr1 | 4 430 048~4 431 545 | Ⅸ | 235 | 27.09 | 5.41 | 叶绿体Chloroplast |
| Csa1G045610 | CsDnaJ5 | Chr 1 | 5 036 912~5 039 959 | Ⅲ | 347 | 38.70 | 9.62 | 细胞质Cytoplasm |
| Csa1G064710 | CsDnaJ6 | Chr 1 | 6 813 658~6 818 208 | Ⅱ | 300 | 35.20 | 9.65 | 质体Plastid |
| Csa1G320350 | CsDnaJ7 | Chr 1 | 13 738 487~13 741 140 | Ⅳ | 362 | 40.74 | 7.87 | 细胞核Nucleus |
| Csa1G397110 | CsDnaJ8 | Chr 1 | 14 711 311~14 728 796 | Ⅸ | 2 551 | 279.60 | 6.12 | 细胞核Nucleus |
| Csa1G535800 | CsDnaJ9 | Chr 1 | 19 204 081~19 207 106 | Ⅳ | 468 | 53.53 | 4.61 | 细胞核Nucleus |
| Csa1G572990 | CsDnaJ10 | Chr 1 | 21 351 042~21 354 512 | Ⅳ | 848 | 94.57 | 8.85 | 细胞核Nucleus |
| Csa1G637960 | CsDnaJ11 | Chr 1 | 25 490 096~25 498 071 | Ⅳ | 252 | 29.89 | 9.78 | 细胞核Nucleus |
| Csa1G642540 | CsDnaJ12 | Chr 1 | 25 607 133~25 609 719 | Ⅶ | 414 | 45.83 | 6.16 | 细胞核Nucleus |
| Csa2G022810 | CsDnaJ13 | Chr 2 | 2 736 153~2 738 850 | Ⅸ | 265 | 29.86 | 5.20 | 叶绿体Chloroplast |
| Csa2G033330 | CsDnaJ14 | Chr 2 | 3 197 987~3 200 621 | Ⅴ | 162 | 18.22 | 5.35 | 细胞核Nucleus |
| Csa2G058620 | CsDnaJ15 | Chr 2 | 4 319 569~4 324 459 | Ⅶ | 334 | 37.39 | 5.53 | 叶绿体Chloroplast |
| Csa2G249270 | CsDnaJ16 | Chr 2 | 12 158 601~12 162 192 | Ⅶ | 340 | 38.12 | 6.55 | 细胞质Cytoplasm |
| Csa2G264570 | CsDnaJ17 | Chr 2 | 12 787 491~12 788 095 | Ⅸ | 146 | 16.06 | 8.46 | 叶绿体Chloroplast |
| Csa2G301500 | CsDnaJ18 | Chr 2 | 14 466 098~14 470 314 | Ⅵ | 284 | 31.79 | 10.14 | 细胞核Nucleus |
| Csa2G353470 | CsDnaJ19 | Chr 2 | 16 349 111~16 352 145 | Ⅵ | 245 | 29.25 | 9.81 | 细胞核Nucleus |
| Csa2G360030 | CsDnaJ20 | Chr 2 | 16 976 993~16 979 050 | Ⅳ | 174 | 19.23 | 4.65 | 叶绿体Chloroplast |
| Csa2G369750 | CsDnaJ21 | Chr 2 | 18 070 524~18 082 899 | Ⅷ | 468 | 51.47 | 7.67 | 细胞核Nucleus |
| Csa2G372140 | CsDnaJ22 | Chr 2 | 18 428 099~18 431 824 | Ⅴ | 135 | 15.91 | 4.79 | 细胞核Nucleus |
| Csa2G408950 | CsDnaJ23 | Chr 2 | 21 233 928~21 236 619 | Ⅲ | 343 | 38.05 | 9.46 | 细胞质Cytoplasm |
| Csa3G020050 | CsDnaJ24 | Chr 3 | 2 083 620~2 090 586 | Ⅴ | 540 | 60.09 | 9.40 | 细胞质Cytoplasm |
| Csa3G129700 | CsDnaJ25 | Chr 3 | 8 368 761~8 370 568 | Ⅸ | 218 | 25.07 | 10.00 | 细胞核Nucleus |
| Csa3G165080 | CsDnaJ26 | Chr 3 | 10 876 613~10 878 005 | Ⅳ | 268 | 30.78 | 9.20 | 细胞核Nucleus |
| Csa3G184060 | CsDnaJ27 | Chr 3 | 12 941 508~12 946 289 | Ⅱ | 686 | 76.82 | 5.40 | 叶绿体Chloroplast |
| Csa3G223330 | CsDnaJ28 | Chr 3 | 14 791 608~14 794 730 | Ⅴ | 648 | 74.17 | 8.23 | 细胞核Nucleus |
| Csa3G710780 | CsDnaJ29 | Chr 3 | 26 888 164~26 889 931 | Ⅸ | 157 | 17.51 | 6.52 | 叶绿体Chloroplast |
| Csa3G710790 | CsDnaJ30 | Chr 3 | 26 897 124~26 898 835 | Ⅸ | 157 | 17.64 | 5.26 | 叶绿体Chloroplast |
| Csa3G730990 | CsDnaJ31 | Chr 3 | 27 445 080~27 449 407 | Ⅷ | 573 | 62.81 | 9.95 | 液泡Vacuole |
| Csa3G731180 | CsDnaJ32 | Chr 3 | 27 558 672~27 560 991 | Ⅲ | 344 | 37.88 | 9.33 | 细胞膜Cytomembrane |
| Csa3G782800 | CsDnaJ33 | Chr 3 | 30 422 428~30 425 931 | Ⅰ | 276 | 31.20 | 4.97 | 叶绿体Chloroplast |
| Csa3G806290 | CsDnaJ34 | Chr 3 | 30 865 483~30 869 971 | Ⅵ | 268 | 31.04 | 9.69 | 细胞核Nucleus |
| Csa3G810550 | CsDnaJ35 | Chr 3 | 31 089 653~31 092 665 | Ⅵ | 283 | 32.56 | 8.70 | 细胞核Nucleus |
| Csa3G843760 | CsDnaJ36 | Chr 3 | 34 066 279~34 069 137 | Ⅸ | 317 | 35.61 | 9.86 | 叶绿体Chloroplast |
| Csa3G878950 | CsDnaJ37 | Chr 3 | 37 009 441~37 011 982 | Ⅲ | 336 | 36.84 | 9.09 | 细胞质Cytoplasm |
| Csa3G890060 | CsDnaJ38 | Chr 3 | 37 991 530~37 994 560 | Ⅴ | 138 | 16.25 | 4.68 | 细胞核Nucleus |
| Csa3G891630 | CsDnaJ39 | Chr 3 | 38 046 989~38 048 804 | Ⅳ | 336 | 37.63 | 8.19 | 细胞核Nucleus |
| Csa4G006450 | CsDnaJ40 | Chr 4 | 1 129 977~1 137 749 | Ⅷ | 506 | 54.50 | 9.12 | 叶绿体Chloroplast |
| Csa4G011640 | CsDnaJ41 | Chr 4 | 1 683 695~1 688 164 | Ⅳ | 786 | 88.33 | 9.73 | 细胞核Nucleus |
| Csa4G064050 | CsDnaJ42 | Chr 4 | 5 150 715~5 158 029 | Ⅸ | 558 | 63.67 | 8.38 | 细胞核Nucleus |
| Csa4G095550 | CsDnaJ43 | Chr 4 | 6 246 049~6 250 698 | Ⅷ | 446 | 47.93 | 9.43 | 叶绿体Chloroplast |
| Csa4G292450 | CsDnaJ44 | Chr 4 | 11 482 044~11 483 605 | Ⅸ | 113 | 12.18 | 10.88 | 叶绿体Chloroplast |
| Csa4G343090 | CsDnaJ45 | Chr 4 | 14 208 070~14 217 583 | Ⅶ | 1 369 | 150.61 | 6.22 | 细胞核Nucleus |
| Csa4G496260 | CsDnaJ46 | Chr 4 | 17 293 149~17 303 290 | Ⅳ | 988 | 109.80 | 6.22 | 细胞核Nucleus |
| Csa4G593910 | CsDnaJ47 | Chr 4 | 19 036 303~19 037 201 | Ⅸ | 199 | 22.36 | 9.46 | 细胞质Cytoplasm |
| Csa4G665650 | CsDnaJ48 | Chr 4 | 23 260 271~23 282 496 | Ⅶ | 399 | 44.17 | 5.93 | 细胞质Cytoplasm |
| Csa5G139530 | CsDnaJ49 | Chr 5 | 3 617 391~3 620 332 | Ⅳ | 760 | 85.67 | 9.54 | 细胞核Nucleus |
| Csa5G175710 | CsDnaJ50 | Chr 5 | 7 327 362~7 329 584 | Ⅸ | 466 | 51.28 | 9.53 | 内质网Endoplasmic reticulum |
| Csa5G467910 | CsDnaJ51 | Chr 5 | 16 420 635~16 426 713 | Ⅷ | 408 | 46.07 | 8.74 | 细胞核Nucleus |
| Csa5G523160 | CsDnaJ52 | Chr 5 | 18 508 684~18 512 685 | Ⅸ | 302 | 34.23 | 8.73 | 叶绿体Chloroplast |
| Csa5G571570 | CsDnaJ53 | Chr 5 | 19 994 469~19 995 680 | Ⅴ | 139 | 15.95 | 4.56 | 细胞核Nucleus |
| Csa5G577340 | CsDnaJ54 | Chr 5 | 20 263 640~20 267 696 | Ⅷ | 345 | 38.54 | 6.09 | 细胞核Nucleus |
| Csa5G585980 | CsDnaJ55 | Chr 5 | 20 909 760~20 912 016 | Ⅲ | 135 | 14.89 | 10.55 | 细胞核Nucleus |
| Csa5G604140 | CsDnaJ56 | Chr 5 | 22 402 004~22 404 623 | Ⅲ | 317 | 35.57 | 9.66 | 细胞质Cytoplasm |
| Csa5G610320 | CsDnaJ57 | Chr 5 | 23 699 983~23 704 976 | Ⅷ | 410 | 45.38 | 6.54 | 细胞核Nucleus |
| Csa5G613470 | CsDnaJ58 | Chr 5 | 24 102 123~24 104 968 | Ⅶ | 414 | 46.07 | 6.44 | 细胞核Nucleus |
| Csa5G623520 | CsDnaJ59 | Chr 5 | 24 863 348~24 866 050 | Ⅸ | 113 | 12.10 | 11.20 | 叶绿体Chloroplast |
| Csa6G067950 | CsDnaJ60 | Chr 6 | 4 818 083~4 821 462 | Ⅳ | 941 | 107.28 | 5.89 | 细胞核Nucleus |
| Csa6G190200 | CsDnaJ61 | Chr 6 | 12 300 916~12 303 900 | Ⅷ | 274 | 31.51 | 5.14 | 细胞核Nucleus |
| Csa6G306340 | CsDnaJ62 | Chr 6 | 14 649 575~14 651 979 | Ⅳ | 349 | 39.94 | 8.53 | 细胞核Nucleus |
| Csa6G324860 | CsDnaJ63 | Chr 6 | 15 126 474~15 131 174 | Ⅸ | 731 | 81.36 | 8.31 | 质体Plastid |
| Csa6G344270 | CsDnaJ64 | Chr 6 | 15 645 519~15 650 234 | Ⅶ | 386 | 43.46 | 5.51 | 细胞质Cytoplasm |
| Csa6G367120 | CsDnaJ65 | Chr 6 | 16 836 152~16 842 153 | Ⅰ | 394 | 45.03 | 6.67 | 细胞核Nucleus |
| Csa6G399770 | CsDnaJ66 | Chr 6 | 17 734 003~17 735 932 | Ⅸ | 272 | 32.05 | 9.89 | 叶绿体Chloroplast |
| Csa6G434360 | CsDnaJ67 | Chr 6 | 20 551 854~20 561 950 | Ⅶ | 1 342 | 148.43 | 5.46 | 细胞核Nucleus |
| Csa6G445030 | CsDnaJ68 | Chr 6 | 20 678 413~20 683 581 | Ⅵ | 499 | 54.76 | 6.85 | 叶绿体Chloroplast |
| Csa6G502820 | CsDnaJ69 | Chr 6 | 25 402 473~25 408 232 | Ⅳ | 1 434 | 160.40 | 4.97 | 细胞核Nucleus |
| Csa6G505950 | CsDnaJ70 | Chr 6 | 25 811 706~25 812 690 | Ⅸ | 165 | 18.19 | 10.06 | 叶绿体Chloroplast |
| Csa6G518110 | CsDnaJ71 | Chr 6 | 27 357 927~27 360 377 | Ⅲ | 350 | 38.05 | 9.66 | 细胞膜Cytomembrane |
| Csa6G524620 | CsDnaJ72 | Chr 6 | 28 237 753~28 241 178 | Ⅳ | 470 | 52.82 | 4.88 | 细胞核Nucleus |
| Csa7G014590 | CsDnaJ73 | Chr 7 | 910 221~914 594 | Ⅵ | 291 | 32.60 | 9.81 | 细胞核Nucleus |
| Csa7G032270 | CsDnaJ74 | Chr 7 | 1 819 347~1 823 445 | Ⅳ | 709 | 78.92 | 8.79 | 细胞核Nucleus |
| Csa7G039200 | CsDnaJ75 | Chr 7 | 2 096 427~2 098 557 | Ⅲ | 340 | 37.60 | 9.43 | 细胞膜Cytomembrane |
| Csa7G061190 | CsDnaJ76 | Chr 7 | 3 563 962~3 564 459 | Ⅶ | 166 | 18.58 | 10.41 | 叶绿体Chloroplast |
| Csa7G190690 | CsDnaJ77 | Chr 7 | 6 926 212~6 926 975 | Ⅸ | 121 | 13.69 | 9.55 | 线粒体Chloroplast |
| Csa7G405850 | CsDnaJ78 | Chr 7 | 15 519 490~15 520 689 | Ⅴ | 131 | 14.88 | 8.61 | 细胞核Nucleus |
| Csa7G407530 | CsDnaJ79 | Chr 7 | 15 637 312~15 641 532 | Ⅷ | 345 | 38.83 | 6.52 | 细胞质Cytoplasm |
| Csa7G428930 | CsDnaJ80 | Chr 7 | 16 580 272~16 585 384 | Ⅷ | 448 | 49.04 | 8.79 | 线粒体Mitochondrion |
| Csa7G448690 | CsDnaJ81 | Chr 7 | 18 324 127~18 326 791 | Ⅴ | 244 | 27.42 | 6.93 | 细胞核Nucleus |
| 注: Ⅰ—Ⅳ为根据进化关系分析所产生的亚簇组别号。Ⅰ-Ⅳ is the subcluster group number produced by evolutionary relationship analysis. | ||||||||
利用MEME软件对黄瓜DnaJ基因家族蛋白序列进行分析, 结果发现所有蛋白都含有motif 1, 其为J结构域核心序列, 含有稳定的HPD结构, 是J蛋白与HSP70蛋白相互作用的关键结构; 而motif 2为J结构的C端螺旋结构, motif 1与motif 2结合形成完整的J结构域。由图 1可知: 66个黄瓜DnaJ蛋白都含有完整的J结构, 而其余15个蛋白只含有HPD结构及不完全的α螺旋结构所构成的J结构域; 而motif 4(81个DnaJ蛋白中共9个蛋白含有该结构域)为甘氨酸/苯丙氨酸富集结构域序列(G/F结构域), motif 5(共4个)为CxxCXGXG重复基序的锌指结构域序列, motif 3(共10个)或motif 6(共12个)为DnaJ-C末端结构域序列。在其他作物中可以根据结构域特征将DnaJ蛋白分为3类, 而黄瓜中只含有其中2类, 即: Ⅰ只有J结构域(共72个); Ⅱ含有J结构域、G/F结构域及C末端结构域(共9个)。另外, 一些DnaJ蛋白同样存在其他非DnaJ蛋白特有结构域。
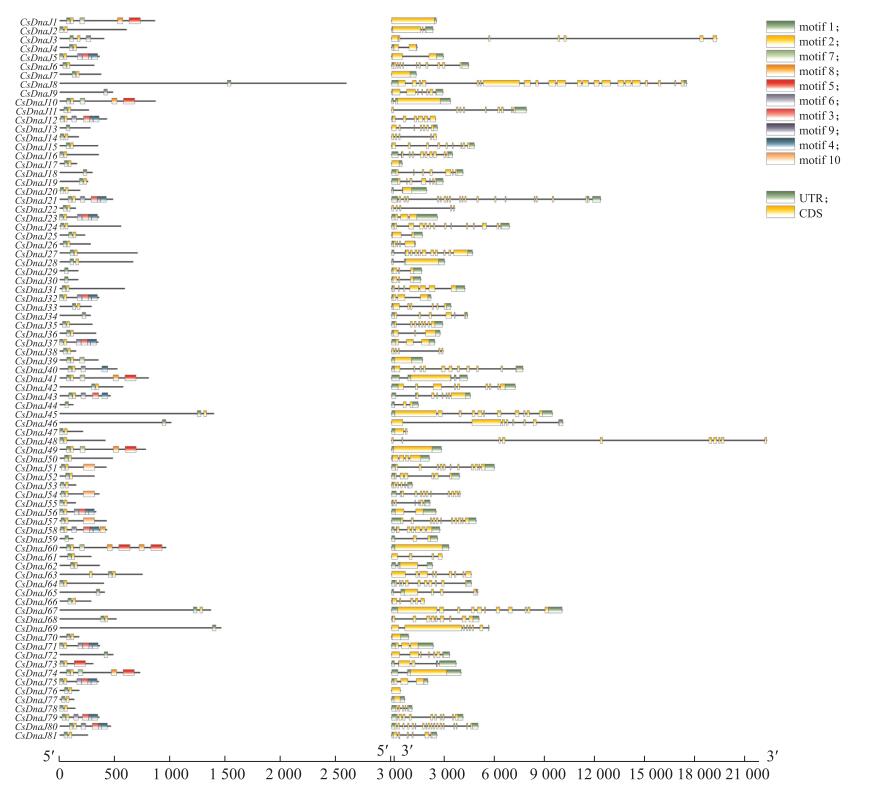
|
图 1 CsDnaJ基因motif结构及基因结构分析 Fig. 1 Analysis of motif structure and gene structure of CsDnaJ gene |
通过对黄瓜DnaJ基因家族mRNA编码序列比对可知, 黄瓜DnaJ基因均含有1~22个外显子, 其中基因CsDnaJ8含有外显子最多, 为22个, 而14个CsDnaJ基因(17.3%)只含有1个外显子, 无内含子序列。
2.3 黄瓜DnaJ基因家族保守结构域及系统进化分析对黄瓜DnaJ基因编码蛋白进行对比分析发现, 这些蛋白均含有保守的J结构域, 由85个氨基酸构成, 其中第1~3位(DYY)、5~8位(ILGV)、17~18位(AS)、20~32位(EELKKAYRKLALK)、34~38位(HPDKN)、65(F)、68(I)、71~78(AYEVLSDP)、80~81(KR)及84~85位(YD)氨基酸序列高保守。但核心结构域中其他序列仍存在部分差异, 可能造成其行使功能的不同(图 2)。
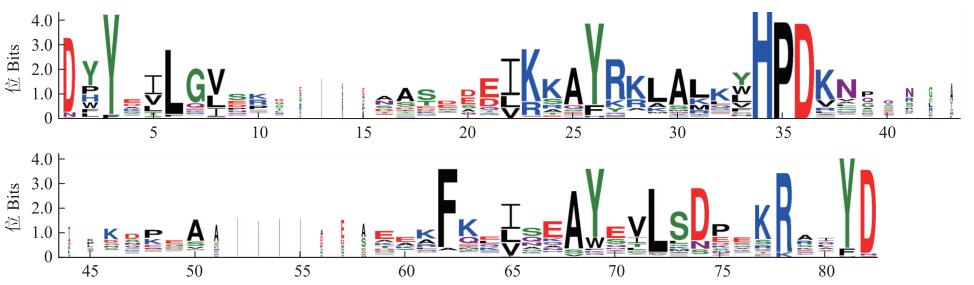
|
图 2 黄瓜DnaJ基因家族的J保守结构域序列标签 Fig. 2 The conserved domain logo of J domain in cucumber DnaJ gene family |
利用黄瓜DnaJ基因家族及拟南芥DnaJ基因家族的核心结构域(J结构域)序列进行聚类分析并构建系统进化树, 结果(图 3)显示, 黄瓜DnaJ蛋白与拟南芥DnaJ蛋白可明显区分为9个亚簇, 每个亚簇均存在拟南芥同源蛋白。其中Ⅸ组含有黄瓜DnaJ蛋白最多, 为19个(23.5%), Ⅰ和Ⅱ最少, 为2个(2.7%)。
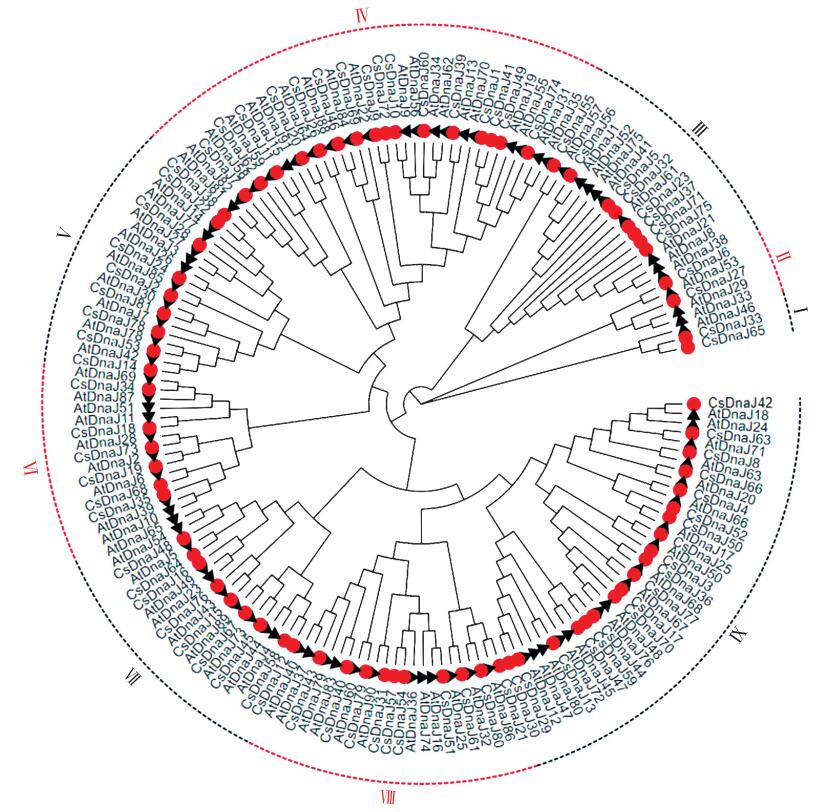
|
图 3 黄瓜及拟南芥DnaJ基因家族进化分析 Fig. 3 Evolution analysis of DnaJ gene family in cucumber and Arabidopsis thaliana |
由图 4可见: 染色体定位分析发现CsDnaJ基因在黄瓜7条染色体均存在且呈无规则分布, 其中Chr 3含有DnaJ基因最多, 为16个, Chr 4、Chr 7最少, 为9个, 这可能与染色体长度相关。
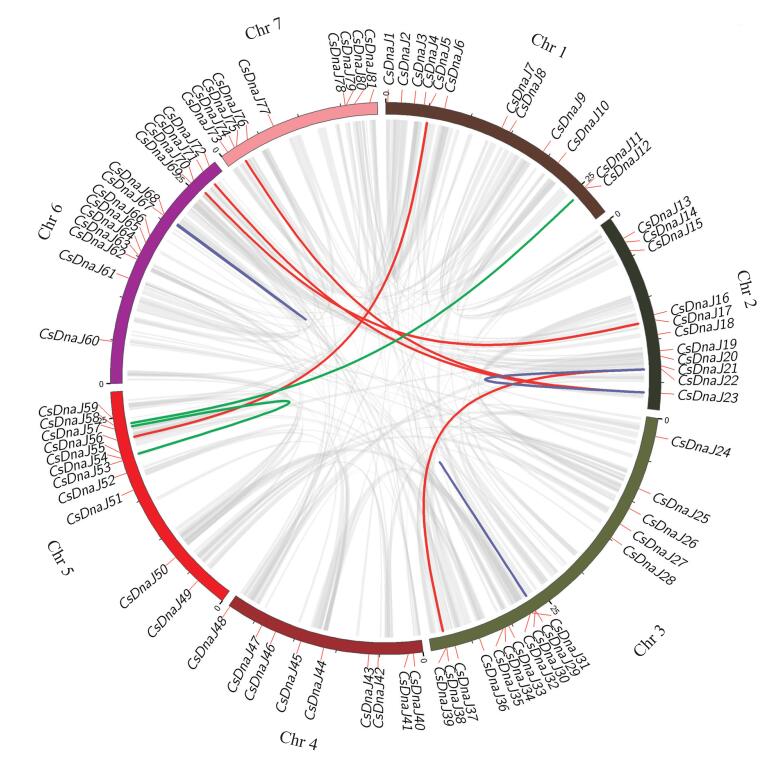
|
图 4 黄瓜DnaJ基因家族染色体定位及共线性分析 Fig. 4 Chromosome location and collinearity analysis of DnaJ gene family in cucumber 红线为片段重复事件, 紫线为串联重复事件, 绿线为随机重复事件。 The fragment repeat events are represented by red lines, the purple line are series repeat events, and the green lines are random repeat events. |
通过共线性分析发现, 黄瓜DnaJ基因家族共存在17个共线基因, 分为10对复制基因, 其中存在5对片段重复事件, 3对串联重复事件, 2对随机重复事件。对复制基因对的KA/KS值计算发现, 其值均小于1, 为0.03~0.72, 表明CsDnaJ在进化过程中主要受纯化选择的作用, 可能与人类长期对优质黄瓜品种的驯化和培育有关。
2.5 黄瓜DnaJ基因家族组织表达特异性由图 5可知: 黄瓜DnaJ基因家族在各组织中均有表达, 其中, 在叶、茎、子房中的表达量略高于在雄花、雌花、根、雄花及卷须中的表达量。各基因间的表达量差异较大, 例如基因CsDnaJ20、CsDnaJ39、CsDnaJ71在各组织中表达量均较低(小于1), 而基因CsDnaJ20、CsDnaJ35、CsDnaJ45在各组织中的表达量均较高(大于10)。这些高表达基因分别属于第Ⅳ、Ⅵ、Ⅶ亚组, 且多数定位于叶绿体中。黄瓜DnaJ家族基因的组织表达特异性表明, 不同DnaJ家族基因成员在黄瓜各组织具有不同的调控作用, 且个别基因的高表达也表明其在DnaJ基因家族调控黄瓜生长发育过程中的重要性。
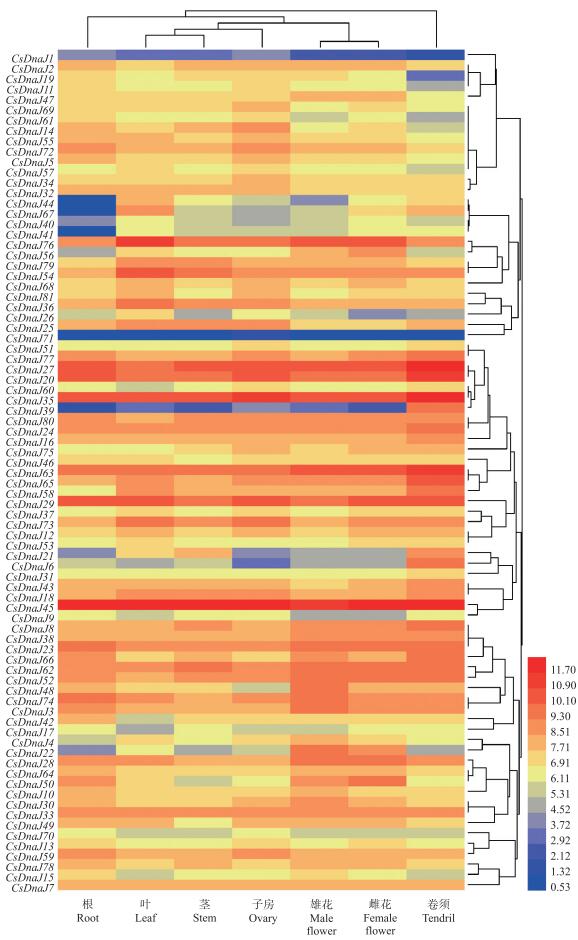
|
图 5 黄瓜DnaJ基因家族组织表达分析 Fig. 5 Tissue expression analysis of DnaJ gene family in cucumber |

|
图 6 高温胁迫下黄瓜表型观察 Fig. 6 Phenotype observation of cucumber under high temperature stress A. 黄瓜植株高温处理前生长状态; B.黄瓜植株经高温处理后生长状态对比。 A. Growth state of cucumber plants before high temperature treatment; B. Comparison of growth state of cucumber plants after high temperature. |
由图 6可见: 在高温处理48 h后, 黄瓜植株出现明显萎蔫状态。由图 7可见: 9个DnaJ基因皆出现上调表达趋势, 且与对照组相比大部分基因表达量具有显著性差异。9个DnaJ基因在不同处理时期表达模式有所差异: 5个基因(CsDnaJ33、CsDnaJ6、CsDnaJ37、CsDnaJ14、CsDnaJ19)在高温处理初期(6 h)表达量达到最高值, 其后逐渐下降; CsDnaJ72基因在高温处理12 h时表达量最高, 其他处理时间基本持平; CsDnaJ16基因在不同处理时间表达量相近; CsDnaJ31基因在处理前期表达量持续上升, 至24 h达到最高点, 其后下降; CsDnaJ29基因在处理前期表达量变化不显著, 在48 h时上升达到最高值。DnaJ基因家族在高温胁迫下的高表达现象表明了植物在高温环境下可以通过诱导DnaJ基因高表达来进一步响应胁迫环境, 维持植物体的正常发育状态。这也同样表明DnaJ蛋白作为应激蛋白在黄瓜响应胁迫环境下起重要调节作用。
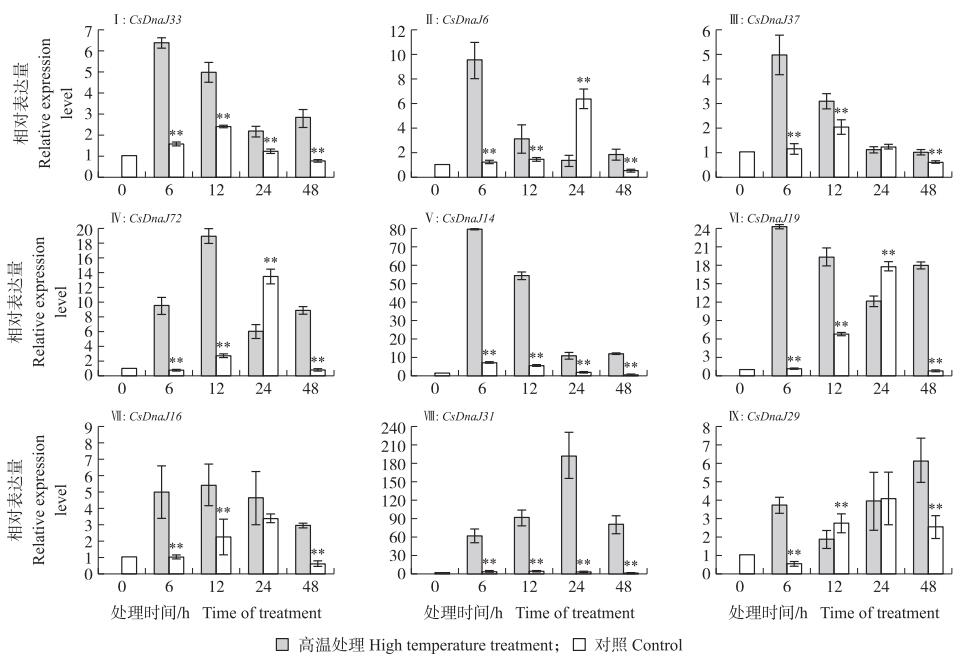
|
图 7 黄瓜DnaJ基因家族在高温胁迫下表达分析 Fig. 7 Expression analysis of DnaJ gene family in cucumber under high temperature stress **P < 0.01. |
DnaJ蛋白作为热激蛋白家族中的一类蛋白, 大部分以胁迫响应因子而广泛存在于植物组织细胞中, 在植物面临生物及非生物胁迫响应中扮演重要的角色[1]。DnaJ蛋白家族基因的表达能够显著提高植物耐热性, 降低植物受高温影响造成的细胞损伤[3]。对DnaJ蛋白家族基因的鉴定及分析有助于深入研究其在胁迫环境下的调控功能。
本试验利用生物信息学手段在作物黄瓜中鉴定了81个DnaJ基因家族成员, 而在拟南芥、水稻、棉花中分别鉴定到91、101、119个DnaJ基因家族成员[5, 17-18], 这几种植物在基因家族成员数量及蛋白特性上均存在高度相似性。这些植物的DnaJ家族成员蛋白序列中均存在由HPD三肽核心结构所构成的高保守J结构域, 但该保守结构域同样存在部分氨基酸差异。由于黄瓜DnaJ家族蛋白序列长度差异较大, 因此对黄瓜及拟南芥DnaJ家族核心J结构域蛋白序列信息进行进化分析, 发现黄瓜DnaJ家族蛋白与拟南芥相似, 同样可以分为9个亚簇, 说明DnaJ家族基因间进化比2个物种间的进化早, 且同一簇内蛋白可能行使相似的功能。
同样对黄瓜DnaJ家族蛋白中其他结构域进行分析, 发现黄瓜中存在G/F结构、CxxCxGxG锌指结构以及DnaJ-C末端结构, 在其他植物中根据DnaJ基因含有结构域的不同可将其分为3种类型[29], 而黄瓜DnaJ蛋白只含有其中2种类型, 缺乏类型Ⅰ即4个结构域都含有的DnaJ蛋白, 这可能会造成物种间对环境温度适应、胁迫反应的差异。DnaJ基因的功能与其所在的亚细胞位置密切关联[7-8]。本研究中通过对黄瓜DnaJ蛋白的亚细胞位置预测, 发现大部分蛋白定位于细胞核与叶绿体内, 目前在植物中对DnaJ蛋白的研究主要集中在定位于叶绿体的DnaJ蛋白, 其能够参与叶绿体发育、光合作用、蛋白转运等多个进程[6-8]。黄瓜中DanJ蛋白的亚细胞定位预测结果同样说明DnaJ蛋白对黄瓜叶绿体发育至关重要。
基因复制在基因家族形成、物种适应中起重要作用, 并可以为具有新功能的基因提供原始遗传材料[30]。黄瓜DnaJ基因家族的共线性分析发现在黄瓜DnaJ蛋白家族进化过程中同样出现多次基因复制现象, 包含串联复制、片段复制及随机复制事件。其中, 片段复制出现最多(5次), 说明其在黄瓜DnaJ基因家族形成过程中起重要作用, 但总体复制现象仅占全部基因的21%(17/81), 这说明存在着其他的进化方式。
本试验结果表明, 黄瓜DnaJ基因在植物体内各组织均有表达, 说明DnaJ基因在黄瓜的生长发育过程发挥着不可缺少的作用。但基因间的表达模式并不相同, 这可能是不同的DnaJ基因在黄瓜器官组织中可能发挥不同的作用模式所造成的。另外, 一些基因在各组织中均呈低水平表达, 可能是因为这些基因功能冗余或者发生了转录沉默/转录后沉默效应, 这种现象及效应在家族基因行使功能中较为常见[31]。
DnaJ蛋白可以作为细胞内感应胁迫环境的主要因子, 在诸如高温、低温、强光等逆境条件下会大量表达[32-33], 并以此参与植物响应环境胁迫进程。本试验通过定量PCR验证了DnaJ基因在高温环境的表达水平, 结果表明高温环境可以诱导DnaJ基因高表达, 因此黄瓜DnaJ被高温环境所激活, 引起表达上调从而参与植物响应胁迫进程。另外, 不同的DnaJ基因在随胁迫环境处理时间所表现出的表达模式、表达差异倍数具有明显差异, 这反映了不同DnaJ基因在胁迫响应进程中调控功能的差异。下一步可通过基因转育或转基因方法将DnaJ基因响应胁迫功能应用于黄瓜抗胁迫品种的选育中。
| [1] |
樊芳菲, 杨暹, 康云艳, 等. 植物DnaJ蛋白的研究进展[J]. 分子植物育种, 2018, 16(6): 2028-2034. Fan F F, Yang X, Kang Y Y, et al. Research progress on DnaJ proteins in plants[J]. Molecular Plant Breeding, 2018, 16(6): 2028-2034 (in Chinese with English abstract). |
| [2] |
祁茂冬, 谢鑫, 魏凤菊. 禾本科植物HSP70研究进展[J]. 植物生理学报, 2019, 55(8): 1054-1062. Qi M D, Xie X, Wei F J. Research progress of HSP70s in Poaceae[J]. Plant Physiology Journal, 2019, 55(8): 1054-1062 (in Chinese with English abstract). |
| [3] |
张驰, 刘丹丹, 刘建中. 植物J蛋白的生物学功能及其作用机制[J]. 浙江大学学报(农业与生命科学版), 2018, 44(3): 275-282. Zhang C, Liu D D, Liu J Z. Biological functions and action mechanisms of J-domain proteins in plants[J]. Journal of Zhejiang University (Agriculture and Life Sciences), 2018, 44(3): 275-282 (in Chinese with English abstract). |
| [4] |
Verma A K, Tamadaddi C, Tak Y, et al. The expanding world of plant J-domain proteins[J]. Critical Reviews in Plant Sciences, 2019, 38(5/6): 382-400. |
| [5] |
Miernyk J A. The J-domain proteins of Arabidopsis thaliana: an unexpectedly large and diverse family of chaperones[J]. Cell Stress & Chaperones, 2001, 6(3): 209-218. |
| [6] |
孔凡英, 邓永胜, 孟庆伟. 叶绿体J蛋白研究进展[J]. 植物生理学报, 2011, 47(3): 235-243. Kong F Y, Deng Y S, Meng Q W. Research advancement of J proteins in chloroplast[J]. Plant Physiology Journal, 2011, 47(3): 235-243 (in Chinese with English abstract). |
| [7] |
Wang G D, Kong F Y, Zhang S, et al. A tomato chloroplast-targeted DnaJ protein protects Rubisco activity under heat stress[J]. Journal of Experimental Botany, 2015, 66(11): 3027-3040. DOI:10.1093/jxb/erv102 |
| [8] |
Kong F Y, Deng Y S, Zhou B, et al. A chloroplast-targeted DnaJ protein contributes to maintenance of photosystem II under chilling stress[J]. Journal of Experimental Botany, 2014, 65(1): 143-158. DOI:10.1093/jxb/ert357 |
| [9] |
Wahid A, Gelani S, Ashraf M, et al. Heat tolerance in plants: an overview[J]. Environmental and Experimental Botany, 2007, 61(3): 199-223. DOI:10.1016/j.envexpbot.2007.05.011 |
| [10] |
王改妮, 白万鹏, 王锁民. 植物响应高温胁迫的信号转导和转录调控机制研究进展[J]. 分子植物育种, 2020, 18(24): 8109-8118. Wang G N, Bai W P, Wang S M. Advances in research of signal transduction and transcriptional regulatory mechanism of plants in response to heat stress[J]. Molecular Plant Breeding, 2020, 18(24): 8109-8118 (in Chinese with English abstract). |
| [11] |
陈雪倩, 韩静, 任仲海. 黄瓜响应高温胁迫转录组分析[J]. 分子植物育种. http://kns.cnki.net/kcms/detail/46.1068.S.20200511.1658.013.html. Chen X Q, Han J, Ren Z H. Transcriptome analysis of cucumber in response to high temperature stress[J]. Molecular Plant Breeding. http://kns.cnki.net/kcms/detail/46.1068.S.20200511.1658.013.html. |
| [12] |
郝婷, 丁小涛, 金海军, 等. 高温对黄瓜生长的影响及设施栽培对策[J]. 上海农业学报, 2014, 30(2): 125-131. Hao T, Ding X T, Jin H J, et al. Effects of high temperature on cucumber growth and countermeasures in protected culture[J]. Acta Agriculturae Shanghai, 2014, 30(2): 125-131 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-3924.2014.02.029 |
| [13] |
徐晓昀. 高温胁迫对黄瓜叶片光合特性的影响[D]. 兰州: 甘肃农业大学, 2007. Xu X Y. Effects of high temperature on photosynthetic characteristics in cucumber leaves[D]. Lanzhou: Gansu Agricultural University, 2007(in Chinese with English abstract). |
| [14] |
Baninasab B, Ghobadi C. Influence of paclobutrazol and application methods on high-temperature stress injury in cucumber seedlings[J]. Journal of Plant Growth Regulation, 2011, 30(2): 213-219. DOI:10.1007/s00344-010-9188-2 |
| [15] |
Ding X T, Jiang Y P, He L Z, et al. Exogenous glutathione improves high root-zone temperature tolerance by modulating photosynthesis, antioxidant and osmolytes systems in cucumber seedlings[J]. Scientific Reports, 2016, 6: 35424. DOI:10.1038/srep35424 |
| [16] |
孙亚玲, 臧传江, 姚甜甜, 等. 我国黄瓜耐热性研究进展[J]. 中国果菜, 2018, 38(10): 57-62. Sun Y L, Zang C J, Yao T T, et al. Research advances on heat tolerance of cucumber in China[J]. China Fruit & Vegetable, 2018, 38(10): 57-62 (in Chinese with English abstract). |
| [17] |
赵旭, 刘向国, 王爱荣. 水稻DnaJ蛋白的生物信息学分析[J]. 亚热带农业研究, 2011, 7(3): 206-211. Zhao X, Liu X G, Wang A R. Bioinformatic analysis of the DnaJ protein family in rice[J]. Subtropical Agriculture Research, 2011, 7(3): 206-211 (in Chinese with English abstract). |
| [18] |
徐珍珍, 郭琪, 刘静, 等. 雷蒙德氏棉DnaJ蛋白的生物信息学预测及盐胁迫下的表达分析[J]. 棉花学报, 2015, 27(5): 391-400. Xu Z Z, Guo Q, Liu J, et al. Genome-wide identification and expression pattern analysis of the DnaJ protein family in Gossypium raimondii under salt stress using a bioinformatics method[J]. Cotton Science, 2015, 27(5): 391-400 (in Chinese with English abstract). |
| [19] |
Huang S W, Li R Q, Zhang Z H, et al. The genome of the cucumber, Cucumis sativus L.[J]. Nature Genetics, 2009, 41(12): 1275-1281. DOI:10.1038/ng.475 |
| [20] |
Finn R D, Coggill P, Eberhardt R Y, et al. The Pfam protein families database: towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44(D1): D279-D285. DOI:10.1093/nar/gkv1344 |
| [21] |
崔梦杰, 王晨, 冷翔鹏, 等. 葡萄SBP-box转录因子家族的生物信息学分析及其应答激素调控葡萄果实成熟的作用[J]. 南京农业大学学报, 2018, 41(3): 429-439. Cui M J, Wang C, Leng X P, et al. Bioinformatic analysis of the SBP-box transcription factor and function of their regulation modes on berry ripening process by responding to hormones[J]. Journal of Nanjing Agricultural University, 2018, 41(3): 429-439 (in Chinese with English abstract). DOI:10.7685/jnau.201706045 |
| [22] |
Marchler-Bauer A, Bo Y, Han L Y, et al. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research, 2017, 45(D1): D200-D203. DOI:10.1093/nar/gkw1129 |
| [23] |
Bailey T L, Boden M, Buske F A, et al. MEME Suite: tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37(Suppl 2): W202-W208. |
| [24] |
Kumar S, Stecher G, Tamura K. MEGA 7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [25] |
Wang Y P, Tang H B, DeBarry J D, et al. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research, 2012, 40(7): e49. DOI:10.1093/nar/gkr1293 |
| [26] |
Li Z, Zhang Z H, Yan P C, et al. RNA-Seq improves annotation of protein-coding genes in the cucumber genome[J]. BMC Genomics, 2011, 12(1): 1-11. DOI:10.1186/1471-2164-12-1 |
| [27] |
Patro R, Duggal G, Kingsford C. Salmon: accurate, versatile and ultrafast quantification from RNA-seq data using lightweight-alignment[J]. bioRxiv, 2015. DOI:10.1101/021592 |
| [28] |
Song M F, Zhang M R, Cheng F, et al. An irregularly striped rind mutant reveals new insight into the function of PG1β in cucumber (Cucumis sativus L.)[J]. Theoretical and Applied Genetics, 2020, 133(2): 371-382. DOI:10.1007/s00122-019-03468-0 |
| [29] |
Fan F F, Yang X, Cheng Y, et al. The DnaJ gene family in pepper (Capsicum annuum L.): comprehensive identification, characterization and expression profiles[J]. Frontiers in Plant Science, 2017, 8: 689. DOI:10.3389/fpls.2017.00689 |
| [30] |
Cannon S B, Mitra A, Baumgarten A, et al. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J]. BMC Plant Biology, 2004, 4: 10. DOI:10.1186/1471-2229-4-10 |
| [31] |
Jury J A, Frayne J, Hall L. Sequence analysis of a variety of primate fertilin alpha genes: evidence for non-functional genes in the Gorilla and man[J]. Molecular Reproduction and Development, 1998, 51(1): 92-97. DOI:10.1002/(SICI)1098-2795(199809)51:1<92::AID-MRD11>3.0.CO;2-W |
| [32] |
Scarpeci T E, Zanor M I, Carrillo N, et al. Generation of superoxide anion in chloroplasts of Arabidopsis thaliana during active photosynthesis: a focus on rapidly induced genes[J]. Plant Molecular Biology, 2008, 66(4): 361-378. DOI:10.1007/s11103-007-9274-4 |
| [33] |
Piippo M, Allahverdiyeva Y, Paakkarinen V, et al. Chloroplast-mediated regulation of nuclear genes in Arabidopsis thaliana in the absence of light stress[J]. Physiological Genomics, 2006, 25(1): 142-152. DOI:10.1152/physiolgenomics.00256.2005 |




