文章信息
- 严无瑕, 仇美华, 刘妍, 薛妍生, 陶丽丽, 徐志辉, 沈标, 张瑞福, 沈其荣
- YAN Wuxia, QIU Meihua, LIU Yan, XUE Yansheng, TAO Lili, XU Zhihui, SHEN Biao, ZHANG Ruifu, SHEN Qirong
- 解淀粉芽胞杆菌SQR9亲缘识别与辨别相关基因的筛选
- Screening of genes related to kin recognition and discrimination in Bacillus amyloliquefaciens SQR9
- 南京农业大学学报, 2021, 44(1): 119-126
- Journal of Nanjing Agricultural University, 2021, 44(1): 119-126.
- http://dx.doi.org/10.7685/jnau.202003038
-
文章历史
- 收稿日期: 2020-03-20
2. 江苏省耕地质量与农业环境保护站, 江苏 南京 210029
2. Jiangsu Station for Protection of Arable Land Quality and Agricultural Environment, Nanjing 210029, China
近年来, 社会微生物学的研究正引起研究学者的广泛关注, 部分科学家不仅仅以微生物个体为研究对象, 而是瞄准微生物整个种群及其内部的复杂关系[1]。群居的生活方式意味着微生物个体之间存在广泛的合作和冲突的行为[2]。微生物之间广泛存在利他、合作、欺骗和竞争性行为, 其中利他行为最受关注。利他行为对实施者产生负面作用, 对行为接受者产生正面作用。例如, Spoering等[3]研究铜绿假单胞菌生物膜耐药性时发现:绝大多数生物膜中的菌株并不具备耐药性, 只有少部分菌株即使受到高浓度的抗生素冲击后仍能存活, 这部分菌株生长速度相对缓慢, 相对于种群中其他菌株来说, 将自己的遗传基因传给下一代的机会很小, 他们的分工是在极端情况下, 防止整个种群的灭亡, 是一种利他的合作行为。
根据汉密尔顿原则, 微生物必须通过一定的机制来保证受益者的亲缘关系, 一方面保证个体的无私行为是有意义的, 另一方面防止合作关系中的欺骗行为[4]。微生物种群内通过亲缘识别来维持和稳定合作行为[5]。Wall[6]首次提出微生物“亲缘识别”作用模型, 近亲菌种通过亲缘识别(kin recognition, 主要通过细胞膜蛋白识别)和非亲缘排斥(non-kin exclusion, 主要通过分泌毒素)2个过程完成微环境内的亲缘种群富集(kin enrichment), 保护了近亲菌种合作行为的有序进行, 其中非亲缘排斥过程也称为亲缘辨别。研究较为深入的亲缘识别机制是黏细菌中的外膜置换系统(out membrane exchange, OME)[6]。黏细菌细胞间的识别蛋白TraA/B相互匹配后不仅能实现蛋白、脂多糖等“公共物资”的交换, 还能修复功能缺陷的细胞[7]。细菌素是一类常见的毒素, 其靶细胞作用范围较窄, 一般针对近缘或同种的不同菌株, 例如大肠杆菌素(colicins)[8]。细菌素经传递系统(如五型、六型分泌系统等)或细胞溶解等方式释放后, 会与靶细胞上的特定细胞表面受体结合, 并通过各种机制杀死它们。若靶细胞内包含相应的免疫蛋白, 则可被识别为同类而免受细菌素的毒害; 若靶细胞内无相应的免疫蛋白, 则被识别为非同类细胞而被杀死[8-9]。
芽胞杆菌(Bacillus)是一种典型的广泛存在于土壤中的革兰氏阳性细菌, 它们之间存在亲缘辨别现象, 但其亲缘识别的机制尚未深入阐明。研究发现, 1 cm3土壤中分离的枯草芽胞杆菌依据不同的亲缘关系能在游动(swarming)培养基上形成边界(boundary)、半融合(intermediate)和融合(merge)状态, 亲缘关系近的菌株通过亲缘协作过程(kin recognition system)加强合作(融合), 而亲缘关系远的菌株通过非亲缘排斥过程(non-kin exclusion system)抵御对方对自身资源的侵蚀[10]。解淀粉芽胞杆菌(Bacillus amyloliquefaciens)SQR9是本实验室从黄瓜种植发病区的健康植株根际分离的1株根际促生菌, 在盆栽和大田试验中表现出显著的促生和生防效果[11-12]。在实验室条件下, 菌株SQR9拮抗根际病原微生物、根际定殖和促进植物生长的分子机制都已基本阐述清楚[11, 13], 但菌株SQR9的亲缘识别与辨别的研究还很薄弱。本研究首先利用实验室已有突变体并通过转座子突变筛选了大量的解淀粉芽胞杆菌SQR9的突变体, 研究它们与亲缘菌株之间Swarming对峙表型, 最后通过基因敲除验证, 鉴定出解淀粉芽胞杆菌SQR9中参与亲缘识别与辨别系统的部分基因。
1 材料与方法 1.1 材料 1.1.1 供试菌株和质粒解淀粉芽胞杆菌(Bacillus amyloliquefaciens)SQR9、FZB42、T-5和DSM7, 模式菌株枯草芽胞杆菌(Bacillus subtilis)168, 菌株SQR9kz(携带质粒pUBXC[14], 在木糖诱导下易转化, 具有零霉素抗性基因)及本实验室保存的野生型菌株SQR9的突变体库。质粒pMarA[15](含有转座子TnYLB-1), 具有红霉素(Em)和卡那霉素(Km)抗性基因, 为大肠杆菌-芽胞杆菌温敏型穿梭载体。
1.1.2 试剂壮观霉素(Spc)、红霉素(Em)、卡那霉素(Km)、零霉素(Zeo)和木糖均购自于南京鼎思生物技术有限公司; Prime STAR HS DNA Ploymerase、2×Fast Taq Master Mix、T4 DNA连接酶均购自于宝生物技术有限公司(TaKaRa)。细菌基因组提取试剂盒、One Step Cloning试剂盒、质粒小量提取试剂盒、DNA凝胶回收试剂盒和PCR清洁回收试剂盒购自于南京诺唯赞生物科技有限公司, DL-4-氯苯丙氨酸购自于美国Sigma公司。
1.1.3 引物所有引物均由生工生物工程(上海)股份有限公司合成。
1.1.4 供试培养基LB培养基:蛋白胨10 g, 酵母粉5 g, 氯化钠3 g, 蒸馏水1 000 mL。Swarming培养基:15 mmol·L-1硫酸铵, 8 mmol·L-1七水硫酸镁, 27 mmol·L-1氯化钾, 7 mmol·L-1柠檬酸钠, 50 mmol·L-1 Tris-HCl(pH7.5), 1 μmol·L-1硫酸铁, 10 μmol·L-1硫酸锰, 0.6 mmol·L-1磷酸二氢钾, 0.86 mmol·L-1赖氨酸, 0.78 mmol·L-1色氨酸, 0.2%葡萄糖。MSgg固体培养基:5 mmol·L-1磷酸钾缓冲液(pH7.0), 100 mmol·L-1 MOPS溶液(pH7.0), 2 mmol·L-1氯化镁, 700 mmol·L-1氯化钙, 50 mmol·L-1氯化锰, 50 mmol·L-1氯化铁, 1 mmol·L-1氯化锌, 2 mmol·L-1硫胺, 0.5%甘油, 0.5%谷氨酸, 50 mg·mL-1色氨酸, 50 mg·mL-1苯丙氨酸。
1.2 试验方法 1.2.1 平板游动对峙试验参照文献[16-18], 将LB平板上保存的解淀粉芽胞杆菌及其突变体接种到3 mL新鲜配制的Swarming培养液中, 37 ℃振荡培养过夜。将菌液稀释到10-4(D600=0.5), 接种1 μL菌液于Swarming培养基上, 每个培养基接种2个菌株。待菌液风干后, 于37 ℃培养箱正置培养24 h。观察并拍照。
1.2.2 生物膜平板对峙试验将LB平板上保存的解淀粉芽胞杆菌及其突变体接种到3 mL新鲜配制的MSgg培养液中, 37 ℃振荡培养过夜。将菌液稀释到10-4(D600=0.5), 接种2 μL菌液于MSgg固体培养基上, 每个培养基接种2种菌株。待菌液风干后, 于30 ℃培养箱正置培养3~7 d。观察并拍照。
1.2.3 利用转座子TnYLB-1构建解淀粉芽胞杆菌SQR9的突变体文库取10 μL SQR9kz种子液接种至含有零霉素的LB液体培养基中, 于37 ℃、170 r·min-1培养至D600为0.5左右, 然后添加终浓度为5 mg·L-1的木糖, 于37 ℃、170 r·min-1诱导1 h后, 取200 μL菌液分装至无菌的2 mL离心管中, 加入5 μL pMarA质粒, 于30 ℃、100 r·min-1复苏3~4 h, 将菌悬液涂布于含有零霉素和卡那霉素双抗生素的LB平板上(Zeo终质量浓度为20 μg·mL-1, Km为5 μg·mL-1), 于30 ℃恒温培养箱中倒置培养过夜。挑取转化子, 接种至含20 μg·mL-1 Zeo、5 μg·mL-1 Km和1 μg·mL-1 Em的LB液体培养基中, 于30 ℃、170 r·min-1培养12 h, 提取质粒DNA。以原始pMarA质粒DNA为阳性对照, 菌株SQR9基因组为阴性对照, 利用引物oAtnpFwd、oAtnpRev和oITR分别扩增出转化子质粒Himar1片段和转座子TnYLB-1片段, 并对扩增产物进行凝胶电泳检测。将验证正确的转化子SQR9-pMarA接种至含有1 μg·mL-1 Em的LB液体培养基中, 于30 ℃、200 r·min-1培养12 h, 接种10 μL菌液于含有红霉素的LB液体培养基中, 于30 ℃、200 r·min-1培养18 h, 将菌液稀释至10-5~10-6倍, 并取200 μL涂布于含5 μg·mL-1 Km的LB固体培养基上, 置于50 ℃高温培养箱培养过夜。随机挑选3 000个单菌落, 接种于每孔含100 μL LB液体培养基的无菌96孔细胞培养板中, 于37 ℃、140 r·min-1培养3~4 h, 每孔加入100 μL 30%无菌甘油, -80 ℃存放[16]。
1.2.4 SQR9转座子突变体文库的筛选与插入位点分析提取筛选出的目标突变体基因组, 用限制性内切酶TaqⅠ于65 ℃水浴锅反应16 h对其进行酶切, 用PCR清洁回收试剂盒进行纯化, 回收目标突变株基因组片段。将纯化后的酶切DNA产物用T4 DNA连接酶于16 ℃自连环化12~16 h。取上述酶连反应产物10 μL作为DNA模板, 采用引物oIPCR1和oIPCR2进行PCR扩增, 并将片段送至南京金斯瑞生物科技有限公司测序, 测序引物为oIPCR3(表 1), 通过序列比对, 找到转座子在突变体中的插入位点。
| 引物名称Primers | 引物序列Primer sequence(5′→3′) | 用途Usage |
| ΔflhO-UF ΔflhO-UR |
ATCGTGCGGCTGTTCAGCTG TATTTCAGAGGTGGAAGACCAAAATGGGGGACATTCA |
上游片段扩增 Amplification of up stream fragments |
| ΔflhO-BF ΔflhO-BR |
TGAATGTCCCCCATTTTGGTCTTCCACCTCTGAAATA CGCAGTTTAAACAGGCGTAATAAGTCATTGTGCAGGCCGCCAA |
目标基因相邻基因片段Back扩增 Amplification of adjacent fragments of target gene Back |
| ΔflhO-PSF ΔflhO-PSR |
TTGGCGGCCTGCACAATGACTTATTACGCCTGTTTAAACTGCG CGCAAATGAAATCGGCAAAGTGTAACCGTTCGTATAATGTATGCT |
反向筛选标记PS片段扩增 Amplification of reverse screening marker fragments PS |
| ΔflhO-DF ΔflhO-DR |
AGCATACATTATACGAACGGTTACACTTTGCCGATTTCATTTGCG TTGTTAAAAGGATTATATAC |
下游片段扩增 Amplification of down stream fragments |
| ΔflhO-VF ΔflhO-VR |
GCGATTTTGAGAAAGCCGCA GGTGAACGTACCGCTGAAGA |
突变体验证 Mutant verification |
| oAtnpFwd oAtnpRev |
CCCGGTCAATGGAGCAATTCGGACGATTGACAAGC CCCGGTCAAGTCGACGCAGATTCCGGTCTAACAAAG |
PA-Haimar1扩增 Amplification of PA-Haimar1 |
| oIPCR1 oIPCR2 |
GCTTGTAAATTCTATCATAATTG AGGGAATCATTTGAAGGTTGG |
突变位点扩增 Amplification of mutation sites |
| oIPCR3 | GCATTTAATACTAGCGACGCC | 突变位点测序Sequencing of mutation sites |
| oITR | CCTGCAGTAACAGGTTGGCTGATAAGTCCCCGGTCT | 转座元件TnYLB-1扩增 Amplification of transposition element TnYLB-1 |
基于PCR原理和反筛标记pheS的解淀粉芽胞杆菌基因敲除方法对菌株SQR9中的鞭毛合成相关基因flhO进行敲除[19]。利用ΔflhO-UF/UR、ΔflhO-DF/DR和ΔflhO-BF/BR引物, 以菌株SQR9基因组为模板, 分别扩增出Up同源臂、Down同源臂和Back同源臂及PS片段, 各片段均采用高保真DNA聚合酶进行PCR扩增。将扩增得到的4个片段序列产物进行融合PCR。在含零霉素的LB试管中接种SQR9kz单菌落, 于37 ℃、170 r·min-1培养12 h。取出10 μL菌液转接于新鲜的含20 μg·mL-1零霉素的LB试管中, 于37 ℃、170 r·min-1培养5 h(D600=0.5), 添加终浓度为5 mg·L-1的木糖, 于37 ℃、170 r·min-1诱导培养1 h后, 吸取200 μL菌液至2 mL无菌离心管, 加入40 μL上述4个融合片段, 混合均匀, 于37 ℃、100 r·min-1培养7~8 h, 将菌悬液涂布于含有壮观霉素的LB平板上(终质量浓度为100 μg·mL-1), 于37 ℃恒温培养箱中倒置培养过夜。在含壮观霉素的LB试管中接种转化子, 于37 ℃、170 r·min-1培养至对数前期(D600=0.5), 取出10 μL菌液涂布于含有20 mmol·L-1 DL-4-氯苯丙氨酸的无机盐固体培养基平板上, 于37 ℃恒温培养箱中倒置过夜培养, 挑取单菌落, 提取基因组, 以此为模板, 利用ΔflhO-VF/VR验证引物进行扩增, 判断是否发生正确的双交换同源重组, 并将片段送至南京金斯瑞生物科技有限公司测序。
2 结果与分析 2.1 不同芽胞杆菌间识别关系的差异许多性状优质的芽胞杆菌接种到特定的生境中后, 对原有土著微生物的生态性影响还不明确。研究发现, 利用芽胞杆菌群集运动表型能探索它们之间的识别关系[10]。从图 1可知:解淀粉芽胞杆菌SQR9(以下简称SQR9)与同种解淀粉芽胞杆菌(农业生物肥料菌株FZB42和T-5及工业生产菌株DSM7)形成边界表型, 说明这些功能良好的微生物菌种(FZB42和T-5)与菌株SQR9相互间不倾向于接触式合作。
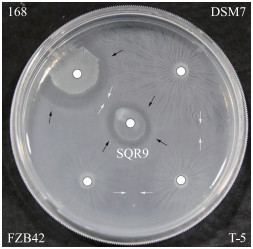
|
图 1 解淀粉芽胞杆菌菌株SQR9与同源芽胞杆菌FZB42、T-5、DSM7、168形成边界表型 Fig. 1 Boundary formation between swarms of Bacillus amyloliquefaciens SQR9 and strain FZB42, T-5, DSM7, 168 黑色箭头表示边界表型; 白色箭头表示融合表型; 白色圆圈代表接种位置。下同。 Black arrows indicate boundary phenotype; white arrows indicate merge phenotype; white circle indicate the inoculation location. The same as follows. |
为了探究菌株SQR9中参与其亲缘识别和辨别的基因, 通过2种方式进行研究。先利用已构建好的突变体库进行筛选, 然后构建菌株SQR9的转座子突变体库进行筛选。
2.2.1 利用SQR9已有突变体库的筛选将菌株SQR9的32株突变体分别与野生菌株SQR9和FZB42进行Swarming对峙试验, 结果见表 2和图 2。前期试验表明, 野生型菌株SQR9自己相互对峙时为融合表型, 菌株SQR9与FZB42对峙时为边界表型。突变体ΔsinR和ΔfliD与菌株SQR9对峙为边界表型, 而突变体Δspo0A与菌株FZB42对峙为融合表型, 说明调控基因sinR和鞭毛合成相关基因fliD可能参与菌株SQR9的亲缘识别过程, 而调控基因spo0A可能参与其亲缘辨别过程(图 2)。
| 功能Function | 突变体(Swarming表型)Mutants(Phenotype of Swarming) | |||
| 生物膜Biofilm | ΔbamD(+, -) ΔepsD(+, -) |
ΔdegQ(+, -) ΔftsE(+, -) |
ΔsinI(+, -) ΔcwlO(+, -) |
ΔsinR(-, -) Δspo0A(+, +) |
| 运动性Mobility | ΔcheA(+, -) | ΔfliD(-, -) | ΔluxS(+, -) | ΔykvO(+, -) |
| 趋化性Chemotaxis | Δ5mcp(+, -) | Δ6mcp(+, -) | Δ7mcp(+, -) | Δ8mcp(+, -) |
| 植物促生Plant growth promoting | ΔyfhA(+, -) ΔyclC(+, -) |
ΔfhuB(+, -) ΔdhaS(+, -) |
ΔyfmD(+, -) ΔyhcX(+, -) |
ΔywbL(+, -) ΔysnE(+, -) |
| 抗生素合成Antibiotic synthesis | ΔfenA(+, -) | Δdfn(+, -) | Δbae(+, -) | ΔsrfΔdfn(+, -) |
| 其他功能Others functions | Δsfp(+, -) | ΔresE(+, -) | Δ64(+, -) | ΔGI(+, -) |
| 注:“+”为融合表型; “-”为边界表型。括号内第1个符号表示与菌株SQR9的Swarming表型, 第2个符号表示与菌株FZB42的Swarming表型。“+”is merge phenotype; “-”is boundary phenotype. The first and second symbols in brackets indicate the Swarming phenotype with strain SQR9 and the Swarming phenotype with strain FZB42. | ||||
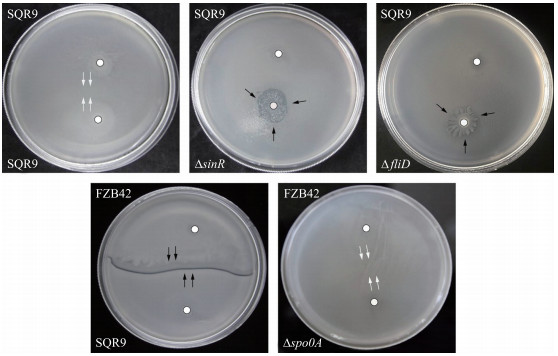
|
图 2 菌株SQR9、FZB42与突变体ΔsinR、ΔfliD、Δspo0A的游动对峙表型 Fig. 2 Swarm interaction phenotypes between strains SQR9, FZB42 and ΔsinR, ΔfliD, Δspo0A mutants |
利用转座子TnYLB-1成功构建SQR9的3 000株突变体库。将突变体与野生菌株SQR9和FZB42进行Swarming对峙试验, 挑出表型发生变化的菌株, 利用反向PCR扩增测序的方法, 进行突变位点分析。结果表明:7株突变体与菌株SQR9的Swarming对峙表型由融合变为边界(图 3); 突变体的插入位点分别为srfAA、patB、gerAA、gerAB、flhO、yugK和degU(表 3), 这7个基因可能与菌株SQR9的亲缘识别过程相关。
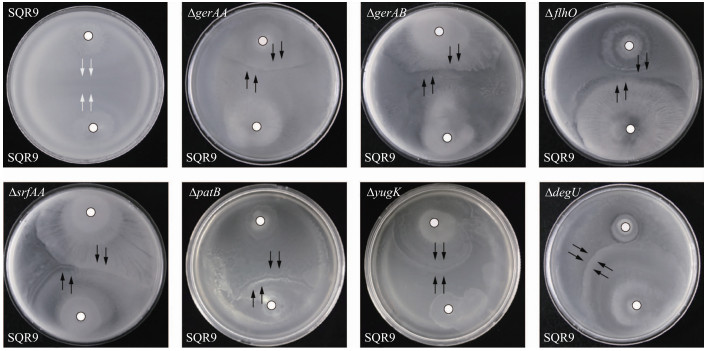
|
图 3 7株突变体与菌株SQR9的游动对峙表型 Fig. 3 Swarm interaction phenotypes between seven mutants and strain SQR9 |
| 插入位点 Insertion site |
突变的基因 Mutant gene |
功能 Function |
与菌株SQR9对峙表型 Swarming phenotypes with SQR9 |
| 331372—331577 | srfAA | Surfactin合成基因Surfactin synthetic gene | 边界Boundary |
| 3179038—3179476 | patB | 氨基转移酶Aminotransferase | 边界Boundary |
| 3336792—3337180 | gerAA | 孢子萌发受体蛋白Spore germination receptor protein | 边界Boundary |
| 3336792—3337180 | gerAB | 孢子萌发受体蛋白Spore germination receptor protein | 边界Boundary |
| 3679690—3680230 | flhO | 鞭毛合成相关蛋白Flagella synthesis related protein | 边界Boundary |
| 3171662—3171849 | yugK | 丁醇脱氢酶Butanol dehydrogenase | 边界Boundary |
| 3582906—583058 | degU | 全局性调控蛋白Global regulatory protein | 边界Boundary |
| 注:参考基因组NCBI登录号为No.CP006890。NCBI database accession No. of reference genome is CP006890. | |||
为了明确鞭毛合成相关基因flhO转座突变体Swarming表型变化是否由该基因突变引起的, 利用反筛标记pheS的解淀粉芽胞杆菌基因敲除方法(无痕敲除)敲除该基因进行进一步的验证。利用融合PCR方法构建4个融合片段FFS(4 446 bp, 包含上游重复片段Up、相邻基因片段Back、反筛标记pheS+壮观霉素抗性片段PS和下游重复片段Down)(图 4-A), 将FFS片段转入菌株SQR9中后, PCR筛选正确的转化子。由图 4-B可以看出, 删除500 bp的突变体B为正确的转化子, 突变体A为假阳性转化子, 正确转化子命名为ΔflhO, 并保存。
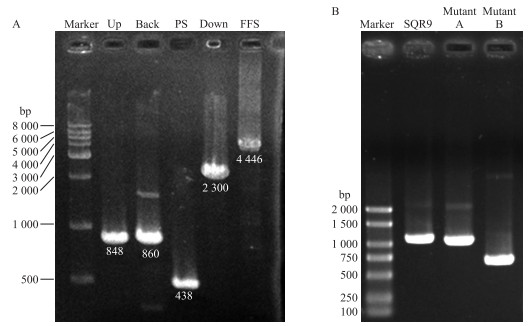
|
图 4 菌株SQR9中flhO基因的敲除以及突变体验证 Fig. 4 Deletion of the flhO gene in strain SQR9 and verification of the mutant A. FFS融合片段构建Construction of the FFS fusion fragment; B.突变体验证Verification of the mutant. |
全局性调控基因(spo0A、sinR和degU)、鞭毛合成相关基因(fliD和flhO)、芽胞萌发基因(gerAA和gerAB)、抗生素合成基因(srfAA)和其他基因(patB和yugK)参与菌株SQR9的亲缘识别和辨别过程。全局性调控蛋白DegU能调控芽胞杆菌的鞭毛形成[20], 所以猜测degU-flhO是菌株SQR9亲缘识别的主要调控通路之一。2种对峙试验表明, 突变体ΔflhO、ΔdegU[21]与野生型菌株SQR9的对峙表型由原本的融合表型变成边界表型, 生物膜对峙同样发现突变体与野生型形成类似边界表型, 相互之间不共享生物膜的公共物质(胞外基质), 而其基因回补菌株恢复了野生型的表型, 说明这2个基因的突变, 影响菌株间的亲缘识别过程(图 5)。
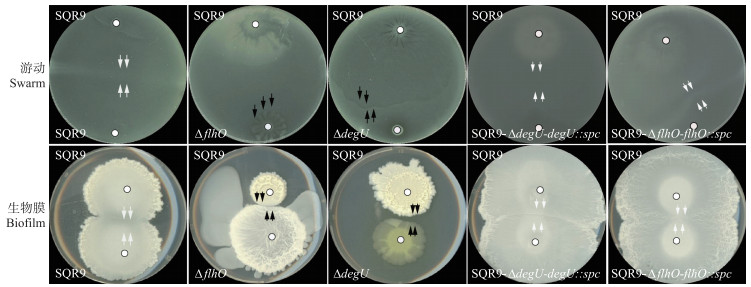
|
图 5 菌株SQR9与突变体ΔflhO、ΔdegU以及回补菌株SQR9-ΔflhO-flhO::spc、SQR9-ΔdegU-degU::spc的游动/生物膜对峙表型 Fig. 5 Swarm and biofilm interaction phenotypes between strains SQR9 and its mutants ΔflhO, ΔdegU and refeeding strain SQR9-ΔflhO-flhO::spc, SQR9-ΔdegU-degU::spc |
植物根际微生物被认为是植物的第二基因组, 对宿主的营养吸收以及协助植物应对各类生物和非生物胁迫中起到关键作用, 被寄望在农业“绿色革命”时代发挥重要作用, 因此应用植物根际促生菌(plant-growth-promoting microorganisms, PGPM)提高作物产量和品质是现代绿色农业发展的重要趋势之一[22]。近20年来, PGPM菌株在单菌水平上的研究揭示了其活化养分(溶磷、解钾、固氮等)、植物促生激素(吲哚乙酸、赤霉素、细胞分裂素等)、挥发性促生物质(乙偶姻和2, 3-丁二醇等)等的促生机制, 以及通过抗生作用、竞争作用、诱导植物系统抗性等防控土传病害的生物学机制[23-24]。提高物种多样性可以增强群落功能多样性和稳定性, 生产实践中多种功能微生物组合使用的效果也更好[25]。然而, 功能微生物的组合不是多种功能菌株的简单混合, 缺乏生态学理论指导的组合达不到预期的效果。解淀粉芽胞杆菌SQR9在盆栽和大田试验中表现出较好的促生和生防效果, 其促生抗病的分子机制已初步阐明, 但其施入土壤后与其他土著芽胞杆菌的关系未知。本研究通过分子生物学手段, 筛选出菌株SQR9中参与亲缘识别与辨别的基因, 为进一步阐明其生态学效应提供理论基础。
枯草芽胞杆菌的亲缘识别主要依赖非亲缘排斥, 而非亲缘协作[14]。芽胞杆菌通过分泌大量多样的抗生素和毒素进而创造一个屏障, 只有关系相近的种才能够共存, 并通过直接针对并杀死非亲缘种来进行间接识别[26]。变形菌门中部分细菌的接触依赖性抑制(contact-dependent inhibition, CDI)系统是典型的非亲缘排斥。同样, 该系统需要细胞间的黏附蛋白CdiA进行特异性的识别, 细胞相互接触后毒素物质能通过五型分泌系统注入对方细胞, 如有相关解毒蛋白即为近亲, 可继续合作; 反之, 毒素物质会在胞内积累影响细胞的正常代谢[27]。本研究通过大范围筛选突变体, 发现解淀粉芽胞杆菌SQR9中的全局性调控蛋白DegU和鞭毛合成相关蛋白FlhO参与其亲缘识别的过程。研究表明, 低磷酸化水平的DegU蛋白也调控芽胞杆菌鞭毛的合成[20]。鞭毛是微生物表面特殊的附属结构, 前期的研究表明其主要与细菌运动能力相关[28]。近年来, 科学家逐步发现鞭毛与宿主黏附、生物膜形成和免疫调节等作用有关, 其抗原多态性可用于细菌分类[29]。本研究结果推测, 芽胞杆菌的鞭毛蛋白可能与菌株间的亲缘识别相关, 因为定点突变flhO基因后其与野生型菌株的Swarming表型由融合变为半融合, 失去部分识别能力。研究发现, 鞭毛蛋白中存在非常保守的N末端和相对可变的C末端组成, 其暴露在外的中央区具有一定的可变性, 猜测与其亲缘菌株的识别有关, 但还需要进一步的证明[29]。
亲缘识别过程是芽胞杆菌社会性行为的基础。植物根表定殖试验表明, 亲缘关系近的菌株组合能共同定殖于植物根表, 而亲缘关系远的菌株组合只有一类芽胞杆菌能成功定殖, 并通过非亲缘排斥过程阻止其他芽胞杆菌定殖[10]。例如, 生物有机肥料中常用的功能菌株SQR9与FZB42的Swarming形成边界表型, 菌株关系不利于接触式合作。所以, 深入研究芽胞杆菌的亲缘识别分子机制, 能为科学组装复合型微生物肥料提供理论依据。
| [1] |
Parsek M R, Greenberg E P. Sociomicrobiology:the connections between quorum sensing and biofilms[J]. Trends in Microbiology, 2005, 13(1): 27-33. DOI:10.1016/j.tim.2004.11.007 |
| [2] |
Cornforth D M, Foster K R. Competition sensing:the social side of bacterial stress responses[J]. Nature Reviews:Microbiology, 2013, 11(4): 285-293. DOI:10.1038/nrmicro2977 |
| [3] |
Spoering A L, Lewis K. Biofilms and planktonic cells of Pseudomonas aeruginosa have similar resistance to killing by antimicrobials[J]. Journal of Bacteriology, 2001, 183(23): 6746-6751. DOI:10.1128/JB.183.23.6746-6751.2001 |
| [4] |
Queller D C. Expanded social fitness and Hamilton's rule for kin, kith, and kind[J]. Proc Natl Acad Sci USA, 2011, 108(Suppl 2): 10792-10799. |
| [5] |
Queller D C, Ponte E, Bozzaro S, et al. Single-gene greenbeard effects in the social amoeba Dictyostelium discoideum[J]. Science, 2003, 299(5603): 105-106. DOI:10.1126/science.1077742 |
| [6] |
Wall D. Kin recognition in bacteria[J]. Annual Review of Microbiology, 2016, 70(1): 143-160. DOI:10.1146/annurev-micro-102215-095325 |
| [7] |
Nudleman E. Cell-to-cell transfer of bacterial outer membrane lipoproteins[J]. Science, 2005, 309(5731): 125-127. DOI:10.1126/science.1112440 |
| [8] |
Cascales E, Buchanan S K, Duche Ae'1 D, et al. Colicin biology[J]. Microbiology and Molecular Biology Reviews, 2007, 71(1): 158-229. DOI:10.1128/MMBR.00036-06 |
| [9] |
Rendueles O, Zee P C, Dinkelacker I, et al. Rapid and widespread de novo evolution of kin discrimination[J]. Proc Natl Acad Sci USA, 2015, 112(29): 9076-9081. DOI:10.1073/pnas.1502251112 |
| [10] |
Stefanic P, Kraigher B, Lyons N A, et al. Kin discrimination between sympatric Bacillus subtilis isolates[J]. Proc Natl Acad Sci USA, 2015, 112(45): 14042-14047. DOI:10.1073/pnas.1512671112 |
| [11] |
Cao Y, Zhang Z H, Ling N, et al. Bacillus subtilis SQR9 can control Fusarium wilt in cucumber by colonizing plant roots[J]. Biology and Fertility of Soils, 2011, 47(5): 495-506. DOI:10.1007/s00374-011-0556-2 |
| [12] |
Xu Z H, Xie J Y, Zhang H H, et al. Enhanced control of plant wilt disease by a xylose-inducible degQ gene engineered into Bacillus velezensis strain SQR9XYQ[J]. Phytopathology, 2019, 109(1): 36-43. DOI:10.1094/PHYTO-02-18-0048-R |
| [13] |
Xu Z H, Mandic-Mulec I, Zhang H H, et al. Antibiotic bacillomycin D affects iron acquisition and biofilm formation in Bacillus velezensis through a btr-mediated FeuABC-dependent pathway[J]. Cell Reports, 2019, 29(5): 1192-1202. DOI:10.1016/j.celrep.2019.09.061 |
| [14] |
Chen X T, Ji J B, Liu Y C, et al. Artificial induction of genetic competence in Bacillus amyloliquefaciens isolates[J]. Biotechnology Letters, 2016, 38(12): 2109-2117. |
| [15] |
Le Breton Y, Mohapatra N P, Haldenwang W G. In vivo random mutagenesis of Bacillus subtilis by use of TnYLB-1, a mariner-based transposon[J]. Applied and Environmental Microbiology, 2006, 72(1): 327-333. DOI:10.1128/AEM.72.1.327-333.2006 |
| [16] |
邵佳慧.解淀粉芽孢杆菌SQR9吲哚乙酸合成途径及促生效应的研究[D].南京: 南京农业大学, 2014. Shao J H. Indole-3-acetic acid synthesis pathways and plant-growth-promotion effects of Bacillus amyloliquefaciens SQR9[D]. Nanjing: Nanjing Agricultural University, 2014(in Chinese with English abstract). |
| [17] |
徐志辉.解淀粉芽孢杆菌SQR9生物膜形成和根际定殖分子机理研究[D].南京: 南京农业大学, 2014. Xu Z H. The molecular mechanisms of biofilm formation and cucumber root colonization of Bacillus amyloliquefaciens SQR9[D]. Nanjing: Nanjing Agricultural University, 2014(in Chinese with English abstract). |
| [18] |
徐志辉, 黄蓉, 张钰婷, 等. 黄瓜根际芽孢杆菌亲缘辨识特性研究[J]. 南京农业大学学报, 2017, 40(6): 1023-1030. Xu Z H, Huang R, Zhang Y T, et al. Kin discrimination between Bacillus spp. strains from cucumber rhizosphere[J]. Journal of Nanjing Agricultural University, 2017, 40(6): 1023-1030 (in Chinese with English abstract). DOI:10.7685/jnau.201704025 |
| [19] |
Zhou C Y, Shi L L, Ye B, et al. pheS*, an effective host-genotype-independent counter-selectable marker for marker-free chromosome deletion in Bacillus amyloliquefaciens[J]. Applied Microbiology and Biotechnology, 2017, 101(1): 217-227. DOI:10.1007/s00253-016-7906-9 |
| [20] |
Verhamme D T, Kiley T B, Stanley-Wall N R. DegU co-ordinates multicellular behaviour exhibited by Bacillus subtilis[J]. Molecular Microbiology, 2007, 65(2): 554-568. DOI:10.1111/j.1365-2958.2007.05810.x |
| [21] |
Xu Z H, Zhang R F, Wang D D, et al. Enhanced control of cucumber wilt disease by Bacillus amyloliquefaciens SQR9 by altering the regulation of its DegU phosphorylation[J]. Applied and Environmental Microbiology, 2014, 80(9): 2941-2950. DOI:10.1128/AEM.03943-13 |
| [22] |
Lugtenberg B, Kamilova F. Plant-growth-promoting rhizobacteria[J]. Annual Review of Microbiology, 2009, 63(1): 541-556. DOI:10.1146/annurev.micro.62.081307.162918 |
| [23] |
Gamalero E, Glick B R. Mechanisms used by plant growth-promoting bacteria[M]. Berlin: Springer, 2011: 17-46.
|
| [24] |
Borriss R. Use of plant-associated Bacillus strains as biofertilizers and biocontrol agents in agriculture[M]. Berlin: Springer, 2011: 41-76.
|
| [25] |
de Roy K, Marzorati M, van den Abbeele P, et al. Synthetic microbial ecosystems:an exciting tool to understand and apply microbial communities[J]. Environmental Microbiology, 2014, 16(6): 1472-1481. |
| [26] |
Lyons N A, Kraigher B, Stefanic P, et al. A combinatorial kin discrimination system in Bacillus subtilis[J]. Current Biology, 2016, 26(6): 733-742. DOI:10.1016/j.cub.2016.01.032 |
| [27] |
Aoki S K, Malinverni J C, Jacoby K, et al. Contact-dependent growth inhibition requires the essential outer membrane protein BamA(YaeT)as the receptor and the inner membrane transport protein AcrB[J]. Molecular Microbiology, 2008, 70(2): 323-340. DOI:10.1111/j.1365-2958.2008.06404.x |
| [28] |
Yang Y, Yang Y, Ou B M, et al. The flagellin hypervariable region is a potential flagella display domain in probiotic Escherichia coli strain Nissle 1917[J]. Archives of Microbiology, 2016, 198(7): 603-610. DOI:10.1007/s00203-016-1219-3 |
| [29] |
Samatey F A, Imada K, Nagashima S, et al. Structure of the bacterial flagellar protofilament and implications for a switch for supercoiling[J]. Nature, 2001, 410(6826): 331-337. |




