文章信息
- 王小松, 马磊, 刘志颖, 李托, 朱瀚, 刘东阳, 沈其荣
- WANG Xiaosong, MA Lei, LIU Zhiying, LI Tuo, ZHU Han, LIU Dongyang, SHEN Qirong
- 贵州木霉NJAU4742几丁质酶基因chi8的克隆、表达和酶学特性研究
- Cloning, expression and enzymology characteristics of an endo-chitinase gene chi8 from Trichoderma guizhouense NJAU4742
- 南京农业大学学报, 2021, 44(1): 111-118
- Journal of Nanjing Agricultural University, 2021, 44(1): 111-118.
- http://dx.doi.org/10.7685/jnau.202003042
-
文章历史
- 收稿日期: 2020-03-20
几丁质又称为甲壳素, 是由β-1, 4-糖苷键将N-乙酰-D-氨基葡萄糖(N-acetyl-D-glucosamine, GlcNAc)连接而成的高分子多聚物, 广泛分布在真菌细胞壁及昆虫、甲壳类动物、线虫的外骨骼中, 是自然界中分布最广泛的氨基多糖[1]。几丁质脱乙酰化产生的壳聚糖, 是一种含有GlcNAc和D-葡萄糖胺(GlcN)的聚合物, D-葡萄糖胺通常占残留物的80%[2]。壳寡糖的生物活性主要取决其分子质量及所带电荷量[3-6], 它通常使用化学降解法来制备, 而酶促转化是一种更环保、可控的方法。几丁质酶以随机的方式水解几丁质糖链上的糖苷键, 水解产物为可溶性低分子质量混合物。几丁二糖酶, 可从非还原端裂解几丁质和几丁质聚合物, 最终产物为二乙酰几丁二糖[(GlcNAc)2][7]。N-乙酰氨基葡萄糖苷酶, 同样从非还原端裂解几丁质和几丁质聚合物, 最终产物为GlcNAc[8]。产生几丁质酶的微生物主要为细菌、放线菌、真菌, 不同微生物产生的几丁质酶并不相同, 如酿酒酵母能产生2种几丁质酶cts1和cts2[9]。木霉属(Trichoderma spp.)真菌广泛存在于各种生态系统, 共包含8个集合种[10], 它是一类生长繁殖迅速、适应环境能力强、高效利用养分和空间的丝状真菌, 寄生在其他真菌细胞表面后通过分泌几丁质酶、蛋白酶等细胞壁降解酶来实现对真菌细胞壁的溶解, 被广泛应用于生物防治[11-14]。本试验利用本实验室保存的一株木霉菌株Trichoderma guizhouense NJAU4742, 通过克隆、表达获得几丁质酶Chi8, 以胶体几丁质为底物, 在不同的温度、pH值、金属离子条件下测定其酶活性及其热稳定性, 再利用质谱鉴定其水解产物, 旨在为几丁质的资源化利用提供坚实的理论和物质基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒菌株Trichoderma guizhouense NJAU4742、质粒pET-29a、克隆菌株Escherichia coli DH5α以及表达宿主E.coli BL21(DE3)均由本实验室保存。
1.1.2 培养基液体LB培养基:蛋白胨10 g、酵母提取物5 g、NaCl 10 g、去离子水1 L, pH7.0。固体LB培养基:在液体LB培养基中加15 g·L-1的琼脂。
1.1.3 主要试剂限制性内切酶EcoRⅠ、KpnⅠ(TaKaRa公司); 硫酸卡那霉素、氨苄青霉素(GENVIEW公司); IPTG(北京索莱宝科技有限公司); 细胞裂解液(50 mL体系):2.5 mL 1 mol·L-1 Tris(pH8.0)、4 mL 5 mol·L-1 NaCl、50 μL 0.1 mol·L-1 PMSF(苯甲基磺酰氟)、5 mL 50%甘油、38.45 mL ddH2O; 甲壳素、壳聚糖、羧甲基壳聚糖(上海源叶生物科技有限公司)。
1.2 方法 1.2.1 基因的克隆基于NJAU4742的基因组数据库(http://bioinfo.njau.edu.cn/tgn4742/)设计引物, 正向引物:AGCCCCCTGGCTACAGAAGAG; 反向引物:TTAGTTCAGACCGTTCCTGATGTT。提取NJAU4742菌株总RNA后反转录得到cDNA, 以cDNA为模板扩增chi8(A1A104090.1)。PCR反应体系(50 μL):5×Prime STARⓇ Buffer(Mg2+ plus)10 μL, dNTP Mixture(2.5 mmol·L-1)4 μL, 正、反向引物(10 μmol·L-1)各1 μL, cDNA 1 μL, DNA聚合酶(2.5 U·μL-1)0.5 μL, 灭菌蒸馏水32.5 μL。每个样品设2个重复。反应条件:98 ℃ 2 min; 98 ℃ 10 s, 55 ℃ 15 s, 72 ℃ 1 min, 循环30次; 72 ℃ 10 min, 10 ℃ 10 min。
1.2.2 系统发育树及Motif分析利用BLAST(blast.ncbi.nlm.nih.gov)筛选其他相似序列, 在Non-Redundant Protein Sequence数据库中筛选相似度高的31条氨基酸序列, 利用1 000个重复的bootstrap值构建邻接树, 检测序列间的距离, 分析Chi8与其他几丁质酶的同源关系[15]。利用MEME(http://meme-suite.org/index.html)的Motif Discover工具预测不同蛋白序列的Motif信息。
1.2.3 载体构建及热激转化将20 μL反应体系(2 μL EcoRⅠ, 2 μL KpnⅠ, 14 μL pET-29a质粒与2 μL 10×M Buffer)混合均匀, 在37 ℃水浴锅中进行双酶切20 h后回收酶切产物。用T4连接酶在16 ℃条件下反应16 h。将酶连产物与DH5α感受态细胞混合, 放置冰上30 min, 转移至42 ℃水浴锅中热激90 s, 再置于冰上2 min, 转移至装有900 μL LB培养基的灭菌离心管中, 于37 ℃、100 r·min-1摇床上复苏1 h, 4 ℃、3 000~5 000 r·min-1离心3 min, 沉淀重悬后取100 μL涂布在LA(LB培养基中添加50.0 mg·L-1氨苄青霉素)固体培养基上, 37 ℃培养14~16 h。挑选转化子进行验证。
1.2.4 几丁质酶的异源表达、纯化和验证将阳性克隆转接至添加30 mg·L-1硫酸卡那霉素的LB液体培养基中, 在37 ℃、200 r·min-1的摇床上培养, 当D600达到0.5时, 每升培养基中加入0.5 mmol IPTG进行诱导, 同时将培养条件改为16 ℃、100 r·min-1培养12 h。培养结束后在4 ℃、10 000 r·min-1离心5 min, 收集菌体, 利用1×PBS溶液进行重悬, 将重悬液转移至50 mL新离心管中, 在4 ℃、8 000 r·min-1离心5 min, 弃上清液后称重。将菌体与细胞裂解液以1:10(质量体积比)的比例混合后进行重悬, 在冰水混合物中超声破碎菌体后, 4 ℃、8 000 r·min-1离心1 h, 利用SDS-PAGE分析验证。
利用蛋白纯化仪(NGC10, 伯乐)纯化上述制备的胞内总蛋白。平衡液(pH8.0)为50 mmol·L-1 NaH2PO4、300 mmol·L-1 NaCl、10 mmol·L-1咪唑, 0.22或0.45 μm滤膜过滤除菌; 洗脱液(pH8.0)为50 mmol·L-1 NaH2PO4、300 mmol·L-1 NaCl、500 mmol·L-1咪唑, 0.22 μm滤膜过滤除菌。梯度洗脱后得到Chi8纯化蛋白, 用Bradford方法测定蛋白浓度, 利用SDS-PAGE分析纯化的Chi8蛋白。
蛋白鉴定主要仪器:冷冻干燥仪(上海卢湘)、EASY-nLC1000系统(Thermo Scientific)、LTQ Orbitrap Velos Pro(Thermo Scientific); 主要试剂:脱色液[25 mmol·L-1 NH4HCO3、50%(体积分数)乙腈水溶液]、脱水液1(50%乙腈水溶液)、脱水液2(100%乙腈)、还原液1(含10 mmol·L-1 DTT和25 mmol·L-1 NH4HCO3的水溶液)、还原液2(含50 mmol·L-1碘乙酰胺和25 mmol·L-1 NH4HCO3水溶液)、吸胀液(25 mmol·L-1 NH4HCO3水溶液)、酶解覆盖液(25 mmol·L-1 NH4HCO3水溶液)、酶解工作液(含0.02 μg·μL-1胰蛋白酶的酶解覆盖液)、肽段萃取液(含5%甲酸、67%乙腈的水溶液)。
1.2.5 Chi8酶学特性及其对胶体几丁质的水解动力学参数测定胶体几丁质的制备和标准曲线的制作:称取5 g几丁质缓慢加入100 mL浓盐酸中, 并用磁力搅拌器混匀, 4 ℃环境下放置24 h后加入300 mL 50%(体积分数)的乙醇溶液, 然后用去离子水洗至溶液呈中性。N-乙酰氨基葡萄糖标准曲线配制方法:用1 mg·mL-1 N-乙酰-D-氨基葡萄糖标准溶液制备不同浓度的溶液, 并与DNS反应后在酶标仪520 nm波长下测定吸光值, 将N-乙酰-D-氨基葡萄糖含量作为纵轴, 吸光值作为横轴绘制标准曲线(y=0.147 7x-0.003 7, R2=0.993 7)。
不同条件下几丁质酶活性的测定:测定方法根据文献[13, 16]的方法进行适当调整。1 mL反应体系包括20 μL几丁质酶液、400 μL胶体几丁质、580 μL醋酸缓冲液(pH6.0);对照(CK)加入20 μL的灭活酶液(反应体系中蛋白量为0.5 μmol)。37 ℃、200 r·min-1条件下反应1 h后转移至冰上, 离心后取上清液与等体积DNS混匀, 沸水浴10 min, 冷却后520 nm波长下利用酶标仪测定吸光值。以1 μmol酶溶液1 h产生的还原糖定义为1个酶活性单位(U), 每个处理3个重复。测定不同pH值对酶活性的影响时, 分别加入pH值为4.0、5.0的磷酸缓冲液, pH值为5.0、6.0、7.0的醋酸缓冲液和pH值为7.0、8.0、9.0的Tris-HCl缓冲液。测定温度对酶活性的影响时, 温度梯度设定为20、30、40、50、60、70和80 ℃。测定金属离子对酶活性的影响时, 分别加入100 μL 50 mmol·L-1的NaCl、CaCl2、FeCl3、CoCl2、CuCl2溶液, 同时加入480 μL醋酸缓冲液。测定Chi8热稳定性时, 分别将蛋白在30、37、40、43、47和50 ℃条件下水浴处理20、40、60、80、100和120 min。测定Chi8底物特异性时, 底物分别为胶体几丁质、几丁质、羧甲基壳聚糖、壳聚糖。
1.2.6 降解产物分析在50 mL反应体系中分别加入20 mL胶体几丁质、29 mL醋酸缓冲液(pH6.0)、1 mL几丁质酶液, 37 ℃、200 r·min-1反应12 h, 得到水解产物。将上述产物11 000 r·min-1离心去除剩余底物, 上清液与氯仿、正丁醇按体积比25:4:1混匀, 剧烈摇晃20 min, 高速离心去除变性蛋白质, 利用考马斯亮蓝法检测残留蛋白。利用碳柱(SupelcleanTM ENVI-CarbTM SPE管)洗脱去除盐离子, 用不同浓度乙腈+三氟乙酸混合溶液梯度洗脱, 旋转蒸发浓缩至原体积的1/5后, 利用MALDI-TOF/MS检测产物的成分。
1.2.7 Chi8的同源结构建模及底物模拟对接利用SMART(smart.embl-heidelberg.de)在线预测Chi8的结构域, 选中genomic模式, 对Chi8的氨基酸序列进行比对; 利用SWISS-MODEL(swissmodel.expasy.org)将Chi8的氨基酸序列与PDB数据库已知结构的蛋白质进行比对来预测模型, 并利用PyMOL软件进行优化, 得到蛋白同源结构[17]。利用AutoDock(http://autodock.scripps.edu/)对Chi8进行半柔性对接(其中受体蛋白设为刚性, 配体小分子设为柔性), 利用分子软件提取配体(几丁六糖), 并利用Autodock toolkit加氢, 计算电荷, 确认质子化状态后保存为ligand.pdb, 作为分子对接的配体结构。将Chi8的同源结构蛋白删掉多余的水分子, 保存为protein.pdb, 作为分子对接的受体结构, 最后用AutoDock Tools将配体和受体进行对接。
2 结果与分析 2.1 基因的克隆及其生物信息学分析将PCR扩增获得的chi8完整片段进行琼脂糖凝胶电泳分析, 获得约为1 200 bp条带, 与理论片段长度1 224 bp相符(图 1-A)。目的基因与pET-29a融合后获得重组质粒pET-29a-chi8, 其电泳条带约为4 600 bp, 也与理论片段相符(图 1-B)。选取31种来源于不同微生物的几丁质酶基因序列进行分析, 根据相似性高低可将其分为3个主要的聚类, 第Ⅰ类全部属于木霉属真菌, 其中哈茨木霉的几丁质酶(ACM47359)与chi8的序列相似度高达93.38%。第Ⅱ、Ⅲ类中主要包括青霉属(Purpureocillium lilacinum)、Escovopsis属(Escovopsis weberi)和葡萄穗霉属(Stachybotrys elegans)等, 这些真菌具有一些相同的生物特性, 如对外界条件适应性强, 代谢能力强, 可以利用纤维素、几丁质等高聚合物作为碳源。chi8具有7个典型的Motif, 与哈茨木霉和土孢木霉的几丁质酶完全一致, 因此可以推测chi8与两者功能一致, 具有良好的几丁质水解活性(图 1-C)。
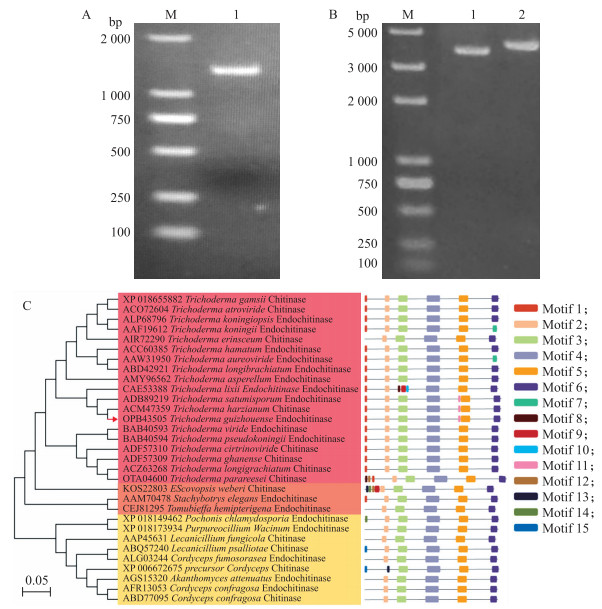
|
图 1 几丁质酶chi8基因的克隆及其信息学分析 Fig. 1 Cloning and informatics analysis of chitinase chi8 gene A. chi8完整片段的电泳图; B.重组质粒的电泳图(M. DNA标准品; 泳道1为pET-29a空载质粒线性化片段, 泳道2为pET-29a-chi8重组质粒线性化片段); C. chi8基因信息分析(左图为基因序列比对结果, 右图为对应基因的功能模块分析)。 A. The electrophoretic image of complete chi8 fragment; B. The electrophoretic image of the linearized recombinant plasmid(M is the marker, lane 1 is the linearized fragment of pET-29a, lane 2 is the linearized fragment of PET-29A-chi8 recombinant plasmid); C. The gene information analysis of chi8(The left is the gene sequence alignment result, and the right one is the functional module analysis of the corresponding gene). |
利用大肠杆菌原核表达系统对chi8进行诱导表达, 每升培养基加入0.5 mmol·L-1 IPTG培养一段时间后, 可以成功诱导目的基因表达, 表达蛋白的电泳分析结果如图 2所示。以空载菌株表达的蛋白条带1作为对照, 在2、3蛋白条带相对分子质量约46×103的位置清晰可见1条新的蛋白条带, 而且蛋白条带比较浓。该条带的相对分子质量与理论相对分子质量(46.365×103)相近, 说明NJAU4742菌株的几丁质酶基因chi8已在大肠杆菌BL21中成功表达。为了进一步验证表达的蛋白为目的蛋白, 将该蛋白进行特征性肽段的质谱鉴定。根据LC-MS蛋白肽段鉴定结果(结果未展示), 共检测到17条用于蛋白质鉴定分析的肽段, 其中4个特征性的肽段(AGATIQYDSVAK、ALGG-LDTTQNLLSYPNSK、ANGYANSVYFTNWGIYDR、ANG-YASVYFTNWGIYDR)与NJAU4742菌株中几丁质酶Chi8肽段的覆盖率高达51.16%。因此, 蛋白质电泳结果和特征性肽段质谱检测结果表明, 纯化蛋白是来源于贵州木霉NJAU4742菌株的几丁质酶Chi8。
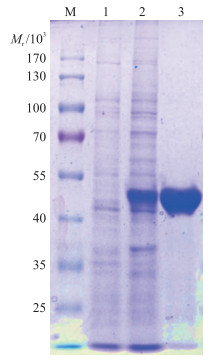
|
图 2 Chi8的SDS-PAGE分析 Fig. 2 SDS-PAGE analysis of Chi8 M.标准蛋白; 1. pET-29a空载转化菌株胞内总蛋白; 2. pET-29a-chi8转化菌株胞内总蛋白; 3.纯化后的蛋白。 M. The standard protein marker; 1. Total intracellular protein of the pET-29a transformed strain; 2. Total intracellular protein of pET-29a-chi8 transformed strain; 3. Purified protein. |
从图 3-A可知:几丁质酶Chi8在30~40 ℃的酶活性比较高, 而在低温下(20 ℃左右)依旧能保持较高活性, 温度达到50 ℃时酶活性显著降低, 温度继续升高后则基本失活, 可能是在高温条件下蛋白质结构发生改变, 不能形成与底物结合的空间结构, 从而失去降解底物的能力。从图 3-B可知:5 mmol·L-1 Ca2+可以显著提高Chi8的酶活性, 能够达到原酶活性的110.62%, 而5 mmol·L-1Co2+、Cu2+对Chi8具有显著抑制作用, 与酶作用后分别下降到原酶活性的89.70%和60.17%。尤其是添加5 mmol·L-1 Fe3+时, 酶活性下降到原来的5.93%。从图 3-C可知:Chi8酶活性在pH6.0时达到最高, 其中偏酸性条件抑制效应更明显, 在pH4.0时基本失去活性, 而在pH9.0时仍保留了部分酶活性, 可达到最高酶活性的50%。

|
图 3 不同条件下Chi8酶活性的变化 Fig. 3 The changes of enzyme activity of Chi8 in different conditions 不同小写字母表示处理间差异显著(P < 0.05)。下同。 The different small letters indicated significant difference in treatments at 0.05 level. The same as follows. |
从图 3-D可知:在相同的热处理时间(40 min)内, 随着热处理温度的升高, 酶活性显著降低。在30、37、40和43 ℃热处理条件下, 相对于热处理温度升高对酶活性造成的显著影响, 热处理时间增加对酶活性影响相对较小, 随着处理时间增加, 酶活性基本不变; 47 ℃热处理条件下, 随处理时间增加, 酶活性下降明显; 在50 ℃热处理条件下, 在初始的20 min内酶活性基本丧失。因此, 几丁质酶Chi8在30~43 ℃可以保持较好的热稳定性; 当温度高于47 ℃后蛋白迅速失活, 酶活性受抑制, 并且随着时间延长而更明显。
从图 4可知:Chi8对胶体几丁质具有良好的分解效果, 也能分解羧甲基壳聚糖, 而基本没有分解几丁质和壳聚糖的能力; 延长反应时间对于底物降解效果并不显著。因此, Chi8并不能直接降解几丁质, 需要对几丁质做预处理或者与其他几丁质酶协同作用才能实现对几丁质的分解与转化。
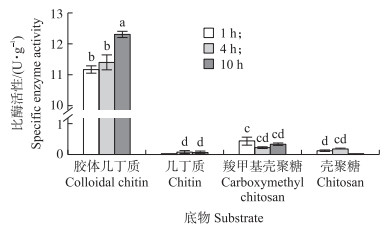
|
图 4 不同底物对Chi8比酶活性的影响 Fig. 4 The effect of different substrates on specific enzyme activity of Chi8 |
为了阐明Chi8对胶体几丁质的催化特性, 利用MALDI-TOF/MS法对水解产物进行分析(图 5)。Chi8水解胶体几丁质后形成了不同的多糖, 降解产物中以几丁二糖[(GlcNAc)2]丰度最高, 其丰度大于1 800;除此之外, 还检测到单糖。这表明Chi8降解胶体几丁质的最终产物以低聚糖为主, 尤其以几丁二糖为主。
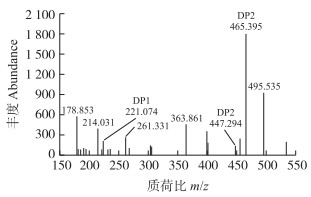
|
图 5 Chi8降解胶体几丁质产物 Fig. 5 Degradation of colloidal chitin products by Chi8 |
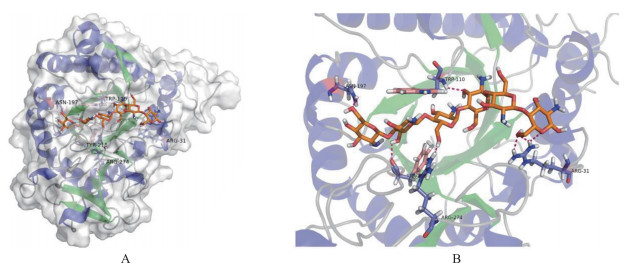
Chi8的结构域和同源蛋白结构分析结果表明, 其与GH18家族关系密切。Chi8的结构具有较低的复杂性, 只含有1个Glyco-18结构域(17~368), E值为5.53 e-143, 其主要功能为结合胶体几丁质。由图 6-A可知, Chi8蛋白同源结构模型由18个螺旋和15个β-折叠组成, 末端的1个免疫缺乏因子(immune deficiency, IMD)残基为咪唑环, 残基相对分子质量为69.08, 广泛存在于748种蛋白结构中, 包括5ZBJ、3QK1、4P8T等蛋白。蛋白与底物对接结果表明, 有4个氨基酸残基与配体相互作用, 同时还存在5个蛋白质配体相互作用通道, 主要以氢键和π堆垛的形式相互作用(图 6-B)。将几丁六糖模拟成活性物质与模拟的Chi8三维结构进行对接, 对接结果展示了该蛋白与几丁六糖结合的位点Asn197、Trp110、Tyr218、Arg274、Arg31, 这些位点可能为该蛋白的活性中心。
|
图 6 Chi8的同源蛋白结构模型(A)及底物模拟对接(B) Fig. 6 The homologous protein structure model(A)and substrate simulation docking of Chi8(B) |
几丁质及其衍生物在工业、农业, 尤其是生物医药等方面具有巨大的应用前景, 因此研究利用高活性几丁质酶产生壳聚糖具有重要意义。GH18家族中大多数几丁质酶的结构域均包含1个碳水化合物结合模块(CBM54), 同时还具备1个或多个几丁质酶催化域, 如ChiW[18]和Tc-ChiD[19]的结构域中均包含1个SLH结构域。TIM-barrel是GH18家族几丁质酶中常见的蛋白质结构, 这一类蛋白是由1个半桶进化而来, 每1个半桶自身不具备生物学功能, 2个半桶合在一起共同作用才能发挥蛋白质的功能。Chi8蛋白中同样含有TIM-barrel结构, 但是Chi8只有1个Glyco-18结构域用于结合底物, 因此催化能力稍微偏低且不能直接分解几丁质和壳聚糖。
不同种类和来源的几丁质酶对酸碱适应性差异较大, 通常最佳pH值范围为3.0~10.0[20-22], 本研究中Chi8的最佳pH值约为6.0。Wang等[23]从Pseudoalteromonas sp. DL-6获得1个非隐性内源性几丁质酶ChiA, 其最佳pH值为9.0。不同几丁质酶的最适温度也有较大的差异, 大部分几丁质酶在40~50 ℃酶活性保持稳定, 温度继续升高将会完全失去酶活性[7-9]。本研究中, Chi8在30~40 ℃具有很高的酶活性, 且具有较好的热稳定性, 当温度达到50 ℃时基本失去活性, 说明来源于NJAU4742的几丁质酶Chi8的热稳定性并不理想。不同金属离子对几丁质酶Chi8的酶活性影响比较大, Co2+、Fe2+、Fe3+及Hg2+对大部分几丁质酶活性具有抑制作用, 而Mg2+、Ca2+及Mn2+等具有增强作用[24]。一般情况下, 金属离子可能会影响蛋白分子结构的稳定性和多种酶的催化活性, 几丁质酶和底物结合过程需要盐离子形成盐桥加以稳定; 另外一些盐可以减轻蛋白表面的静电力, 从而使蛋白不容易形成紊乱结构, 降低蛋白不可逆失活的概率[25]。几丁质酶水解胶体几丁质后可产生不同的壳寡糖, Suginta等[26]检测到水解产物中从GlcNAc到(GlcNAc)6都有分布, 其中以(GlcNAc)2和(GlcNAc)4为主, 并且随反应时间延长, 水解产物呈现(GlcNAc)2增加的趋势, 说明(GlcNAc)4可以进一步被降解。本研究中, Chi8水解胶体几丁质的产物只检测到(GlcNAc)2和单糖, Ueda等[27]也获得了相似的结果。这主要是因为几丁质酶在水解几丁质多聚物时随着反应时间延长, 水解产物逐渐由几丁多糖转化为几丁二糖并不断累积。
目前, 关于几丁质酶的研究较多, 但由于几丁质酶的多样性和复杂性, 再加上自然界分泌的几丁质酶活性低等因素, 导致几丁质酶的工业化生产还存在较多的问题。另外, 国内对于几丁质酶在农业、医药等领域的研究起步晚, 对于几丁质酶在抗虫、抗菌中的具体机制至今尚未明确, 亟需更深入的研究。随着几丁质酶理论基础和应用研究日益受到重视, 几丁质酶的研究已成为糖化学催化领域中不可忽视的分支, 在环境、医药、农业等领域具有十分广阔的应用前景。
| [1] |
姜竹峪, 陈羽, 魏锦兴, 等. 几丁质酶的研究进展[J]. 沈阳药科大学学报, 2016, 33(5): 414-418. Jiang Z Y, Chen Y, Wei J X, et al. Recent advances in research on chitinase[J]. Journal of Shenyang Pharmaceutical University, 2016, 33(5): 414-418 (in Chinese with English abstract). |
| [2] |
Thadathil N, Velappan S P. Recent developments in chitosanase research and its biotechnological applications:a review[J]. Food Chemistry, 2014, 150: 392-399. DOI:10.1016/j.foodchem.2013.10.083 |
| [3] |
Rinaudo M. Chitin and chitosan:properties and applications[J]. Progress in Polymer Science, 2006, 31(7): 603-632. DOI:10.1016/j.progpolymsci.2006.06.001 |
| [4] |
李美玉, 曹洪玉, 张庆芳, 等. 几丁质结合蛋白基因克隆、表达与纯化[J]. 中国酿造, 2015, 34(11): 41-46. Li M Y, Cao H Y, Zhang Q F, et al. Cloning, expression and purification of chitin-binding protein gene[J]. China Brewing, 2015, 34(11): 41-46 (in Chinese with English abstract). DOI:10.11882/j.issn.0254-5071.2015.11.010 |
| [5] |
焦思明, 程功, 张毓宸, 等. 里氏木霉几丁质酶表达及其水解产物组成与结构分析[J]. 中国生物工程杂志, 2018, 38(10): 30-37. Jiao S M, Cheng G, Zhang Y C, et al. Expression of chitinase from Trichoderma reesei and analysis the composition and structure of its hydrolysates[J]. China Biotechnology, 2018, 38(10): 30-37 (in Chinese with English abstract). |
| [6] |
Langner T, Göhre V. Fungal chitinases:function, regulation, and potential roles in plant/pathogen interactions[J]. Current Genetics, 2016, 62(2): 243-254. DOI:10.1007/s00294-015-0530-x |
| [7] |
任立世, 程功, 焦思明, 等. 热紫链霉菌几丁质酶表达及低脱乙酰度壳寡糖制备[J]. 应用与环境生物学报, 2019, 25(2): 445-450. Ren L S, Cheng G, Jiao S M, et al. Expression of chitinase from Streptomyces thermoviolaceus and preparation of chitooligosaccharides with a low degree of deacetylation[J]. Chinese Journal of Applied & Environmental Biology, 2019, 25(2): 445-450 (in Chinese). |
| [8] |
张圆, 周国旺, 李海涛, 等. 苏云金芽胞杆菌chiA73基因的克隆、表达和酶活性分析[J]. 生物技术通报, 2015, 31(8): 147-152. Zhang Y, Zhou G W, Li H T, et al. Cloning, expression and enzymatic activities of chiA73 gene from Bacillus thuringiensis[J]. Biotechnology Bulletin, 2015, 31(8): 147-152 (in Chinese with English abstract). |
| [9] |
李丽, 杨雪松, 刘红全. 微生物几丁质酶的特性及其应用的研究进展[J]. 广西民族大学学报(自然科学版), 2011, 17(1): 92-96. Li L, Yang X S, Liu H Q. Research progress about characteristic and application of chitinase produced by microorganism[J]. Journal of Guangxi University for Nationalities(Natural Science Edition), 2011, 17(1): 92-96 (in Chinese with English abstract). DOI:10.3969/j.issn.1673-8462.2011.01.018 |
| [10] |
孟俊俊, 王斌, 程伟. 土孢木霉Trichoderma saturnisporum的次生代谢产物研究[J]. 中国海洋药物, 2017, 36(6): 27-31. Meng J J, Wang B, Cheng W. Study on the secondary metabolites of Trichoderma saturnisporum[J]. Chinese Journal of Marine Drugs, 2017, 36(6): 27-31 (in Chinese with English abstract). |
| [11] |
范海娟.哈茨木霉ACCC30371菌株转录组构建及生防相关基因功能研究[D].哈尔滨: 东北林业大学, 2013. Fan H J. Transcriptome construction and study on biocontrol functional genes of Trichoderma harzianum ACCC30371 strain[D]. Harbin: Northeast Forestry University, 2013(in Chinese with English abstract). |
| [12] |
张建.生防木霉(Trichoderma guizhouense NJAU 4742)重寄生分子机理研究.Ⅰ.中性金属肽酶NMP1和活性氧的功能分析[D].南京: 南京农业大学, 2015. Zhang J. The molecular mechanism of mycoparasitism of the biocontrol strain Trichoderma guizhouense NJAU 4742.Ⅰ. Function research of the neutral metalloprotease NMP1 and reactive oxygen species[D]. Nanjing: Nanjing Agricultural University, 2015(in Chinese with English abstract). |
| [13] |
咸洪泉.菌寄生真菌几丁质酶基因的克隆及功能研究[D].泰安: 山东农业大学, 2011. Xian H Q. Cloning, expression and function analysis of chitinase gene from mycoparasitic fungus[D]. Tai'an: Shandong Agricultural University, 2011(in Chinese with English abstract). |
| [14] |
王晓辉.海洋细菌DL-6几丁质酶和几丁质结合蛋白的生化性质与功能研究[D].大连: 大连理工大学, 2016. Wang X H. Biochemical properties and functions of chitinases and chitin-binding protein from Pseudoalteromonas sp. DL-6[D]. Dalian: Dalian University of Technology, 2016(in Chinese with English abstract). |
| [15] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. |
| [16] |
Liu D Y, Zhang R F, Yang X M, et al. Expression, purification and characterization of two thermostable endoglucanases cloned from a lignocellulosic decomposing fungi Aspergillus fumigatus Z5 isolated from compost[J]. Protein Expression and Purification, 2011, 79(2): 176-186. DOI:10.1016/j.pep.2011.06.008 |
| [17] |
Biasini M, Bienert S, Waterhouse A, et al. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research, 2014, 42: W252-W258. DOI:10.1093/nar/gku340 |
| [18] |
Itoh T, Hibi T, Suzuki F, et al. Crystal structure of chitinase ChiW from Paenibacillus sp. str. FPU-7 reveals a novel type of bacterial cell-surface-expressed multi-modular enzyme machinery[J]. PLoS One, 2016, 11(12): e0167310. DOI:10.1371/journal.pone.0167310 |
| [19] |
Horiuchi A, Aslam M, Kanai T, et al. a structurally novel chitinase from the chitin-degrading hyperthermophilic archaeon Thermococcus chitonophagus[J]. Applied and Environmental Microbiology, 2016, 82(12): 3554-3562. DOI:10.1128/AEM.00319-16 |
| [20] |
Howard M B, Ekborg N A, Weiner R M, et al. Detection and characterization of chitinases and other chitin-modifying enzymes[J]. Journal of Industrial Microbiology and Biotechnology, 2003, 30(11): 627-635. DOI:10.1007/s10295-003-0096-3 |
| [21] |
Sharma V, Salwan R, Sharma P N, et al. Molecular cloning and characterization of ech46 endochitinase from Trichoderma harzianum[J]. International Journal of Biological Macromolecules, 2016, 92: 615-624. DOI:10.1016/j.ijbiomac.2016.07.067 |
| [22] |
Farag A M, Abd-Elnabey H M, Ibrahim H A H, et al. Purification, characterization and antimicrobial activity of chitinase from marine-derived Aspergillus terrors[J]. The Egyptian Journal of Aquatic Research, 2016, 42(2): 185-192. |
| [23] |
Wang X H, Chi N Y, Bai F W, et al. Characterization of a cold-adapted and salt-tolerant exo-chitinase(ChiC)from Pseudoalteromonas sp. DL-6[J]. Extremophiles, 2016, 20(2): 167-176. DOI:10.1007/s00792-016-0810-5 |
| [24] |
Sosnowska M E, Jankiewicz U, Kutwin M, et al. Influence of salts and metal nanoparticles on the activity and thermal stability of a recombinant chitinase from Stenotrophomonas maltophilia N4[J]. Enzyme and Microbial Technology, 2018, 116: 6-15. DOI:10.1016/j.enzmictec.2018.05.003 |
| [25] |
Cherian E, Dharmendirakumar M, Baskar G. Immobilization of cellulase onto MnO2 nanoparticles for bioethanol production by enhanced hydrolysis of agricultural waste[J]. Chinese Journal of Catalysis, 2015, 36(8): 1223-1229. DOI:10.1016/S1872-2067(15)60906-8 |
| [26] |
Suginta W, Vongsuwan A, Songsiriritthigul C, et al. Enzymatic properties of wild-type and active site mutants of chitinase A from Vibrio carchariae, as revealed by HPLC-MS[J]. The FEBS Journal, 2005, 272(13): 3376-3386. |
| [27] |
Ueda M, Shioyama T, Nakadoi K, et al. Cloning and expression of a chitinase gene from Eisenia fetida[J]. International Journal of Biological Macromolecules, 2017, 104: 1648-1655. |




