文章信息
- 李婷, 朱文姣, 陈敏, 杨清
- LI Ting, ZHU Wenjiao, CHEN Min, YANG Qing
- 茄子SmNTF3基因的克隆及其表达模式分析
- Cloning and expression pattern analysis of SmNTF3 gene in eggplant
- 南京农业大学学报, 2021, 44(1): 103-110
- Journal of Nanjing Agricultural University, 2021, 44(1): 103-110.
- http://dx.doi.org/10.7685/jnau.202004037
-
文章历史
- 收稿日期: 2020-04-20
茄子(Solanum melongena L.)是世界范围内广泛种植的一种农作物, 含多种维生素和生物碱, 营养价值丰富并兼具药用作用。我国茄子种质资源丰富, 栽培普遍, 但其产量和质量极易受各种不良环境的影响[1]。近年来, 由于世界水资源的严重短缺以及保护地栽培造成的土壤盐碱化, 干旱和盐胁迫已成为影响农作物生长的重要胁迫源[2]。因此, 发掘抗旱、耐盐基因对于培育抗盐、抗旱茄子品种具有重要意义。
植物面对不利胁迫时会通过多种信号传导途径进行防卫, 促分裂原活化蛋白激酶(MAPK)级联途径是真核生物主要存在的信号传导模块[3]。在植物中, MAPK通路由MAPKKK(MAPKK激酶)、MAPKK(MAPK激酶)和MAPK组成, 通过磷酸传递机制将上游受体与下游靶点连接起来, 参与生长发育、程序性细胞死亡以及各种环境刺激的响应[4-5]。已有相关研究报道YODA(a MAPKKK)-MKK4/MKK5-MPK3/MPK6能够有效调控花序结构[6]; MKK9-MPK6对叶片衰老过程有调节作用[7]; GhMEKK14-GhMKK11-GhMPK31通路调节棉花的干旱胁迫耐受性等[8]。
MAPK位于级联途径的下游, 催化区域一般由300左右的氨基酸残基组成, 该区域包含11个保守亚结构域(Ⅰ—Ⅺ)和TXY双磷酸化位点[9]。根据TXY基序, 植物MAPK可被分为4组(A—D), 不同的序列结构参与多种生物学过程。研究表明机械损伤、干旱、低温、高盐及外源信号分子等胁迫都会诱导MAPK家族基因选择性表达[10-12]; 转MAPK基因的植株能够显著增强环境抗逆性[13], 表明该家族基因广泛参与植物的胁迫响应过程。
目前, MAPK家族已在拟南芥[14]、辣椒[15]、棉花[16]、水稻[17]等多种高等植物中被鉴定和研究。烟草NTF3基因已被报道在盐和干旱胁迫诱导下表达水平显著上调[18], 但关于茄子NTF3基因的研究还未见报道。本研究从茄子‘苏崎1号’中克隆得到SmNTF3基因, 对其系统发育关系、亚细胞定位及盐和干旱胁迫下的表达模式进行分析, 为进一步研究SmNTF3的生物学功能奠定基础。
1 材料与方法 1.1 植物材料与处理茄子‘苏崎1号’为江苏普遍种植品种, 购于江苏省农业科学院。将种子55 ℃恒温水浴15 min以打破休眠, 消毒漂洗, 平铺于MS培养基, 置于光照/黑暗时间为16 h/8 h、昼/夜温度为(28±1)℃/(25±1)℃、光照强度200 μmol·m-2·s-1培养箱中培育, 发芽后正常光照培养。待长出4~5片真叶, 取出幼苗水培1周, 选取长势健康且一致的幼苗作为试验材料。每个试验重复3次。
组织样品取样:取茄子‘苏崎1号’植株的主根、茎段和幼嫩叶片迅速置于液氮中冷冻保存, 用于SmNTF3基因组织表达模式检测。
盐胁迫处理:将茄子幼苗浸泡在0、50、100、150、200和250 mmol·L-1的NaCl培养液中12 h, 取叶片进行基因表达水平检测; 选择响应最显著的NaCl浓度进行不同时间处理, 分别于0、3、6、12、24和48 h时取样, 以去离子水作为对照。
干旱胁迫处理:将茄子幼苗分别浸泡在0、5%、10%、15%、20%和25%(体积分数)的PEG-6000培养液中处理12 h, 取叶片进行基因表达水平检测; 选择响应最显著的PEG-6000水平进行不同时间处理, 具体方法同盐胁迫。
1.2 SmNTF3基因的克隆采用Triozl法提取茄子总RNA, 测定核酸浓度, 使用GoldenstarTM RT6 cDNA Synthesis Kit(擎科)的两步法反转录获得cDNA。
将烟草NTF3(NtMPK14)基因序列在茄子基因组数据库(https://solgenomics.net/)中进行BLAST比对, 获得茄子同源序列Sme2.5_02990.1_g00003.1, 命名为SmNTF3。以茄子叶片cDNA为模板进行扩增, 引物见表 1。将PCR产物连接至pMD19-T载体, 转化大肠杆菌, 阳性菌落送上海生工生物工程有限公司测序。
| 引物名称Name of primer | 引物序列Sequence of primer(5′→3′) | 引物用途Usage of primer |
| SmNTF3-F SmNTF3-R |
ATGGCAACTCCAGTTGAGCCAC TCACATAACATCTCCCATTCCAGC |
编码区扩增Amplification of coding region |
| GFP-F GFP-R |
GGACTAGTATGGCAACTCCAGTTGAGCC CGGGATCCCATAACATCTCCCATTCCAGC |
亚细胞定位载体Subcellular localization vector |
| qPCR-F qPCR-R |
AAGCTCATCCGTTGGCCATT CCCAAGTCCTCGTCTATGTCAAG |
实时荧光定量PCR RT-qPCR |
| SmEF-1α-F SmEF-1α-R |
CCACACTTCTCATATTGCTGTCA ACCAGCATCACCATTCTTCAAAA |
茄子内参基因Reference gene of eggplant |
利用ORF finder(http://www.ncbi.nih.gov/gorf/gorf.html)进行SmNTF3基因的开放阅读框预测, 用ExPASy的ProtParam程序(http://web.expasy.org/protparam/)对蛋白理化性质进行分析, 用NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)进行磷酸化位点预测, 通过SMART(http://smart.emblheidel-berg.de/)网站进行保守结构域分析, 用Predict Protein(https://open.predictprotein.org/)和SWISS-MODEL(https://swissmodel.expasy.org/)进行蛋白二级和三级结构预测。
SmNTF3同源序列由NCBI网站下载, 利用DNAMAN 6.0软件进行氨基酸序列比对; MEGA 7.0软件用于构建进化树, 采用邻接法(Neighbor-joining method)绘图, bootstrap设置为1 000。
1.4 SmNTF3-GFP载体的构建与亚细胞定位将SmNTF3基因序列去除终止密码子, 设计用于亚细胞定位的特异性引物(表 1), 扩增目的片段并构建克隆载体, 经SpeⅠ和BamHⅠ双酶切连接至载体pCAMBIA1305, 构成重组质粒SmNTF3-GFP。利用农杆菌介导法将构建好的融合载体与Marker共同转化烟草叶片细胞, 正常培养2~3 d后, 选择长势良好的叶片进行制片, 置于激光共聚焦显微镜下观察。
1.5 实时荧光定量PCR根据SmNTF3基因序列设计特异引物, 以茄子EF-1α为内参基因, 引物序列见表 1。RT-qPCR具体步骤按照2×TSINGKE Master qPCR Mix(SYBRGreenI)(擎科)说明书进行。一次平行试验设3组重复。通过熔解曲线结果分析引物的可用性, 相对定量结果以2-ΔΔCT计算。采用Excel 2007和SPSS 19.0软件进行数据统计与分析。
2 结果与分析 2.1 茄子SmNTF3基因的克隆及测序以‘苏崎1号’叶片cDNA为模版, 扩增得到大小约1 100 bp的目的片段(图 1)。经测序及ORF FINDER分析, SmNTF3长度为1 119 bp, 编码1个含有372个氨基酸残基的蛋白, 该序列与茄子基因组数据库Sme2.5_02990.1_g00003.1基因序列完全一致。
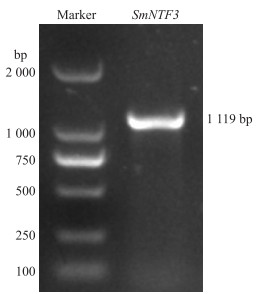
|
图 1 SmNTF3基因的PCR扩增产物 Fig. 1 The PCR product of SmNTF3 gene |
采用ProtParam工具对SmNTF3蛋白的生化特性进行分析, 结果显示SmNTF3蛋白的相对分子质量为42 699.36, 等电点为6.32, 总平均亲水系数(GRAVY)为-0.260, 说明它是1个亲水蛋白。
利用NetPhos网站对SmNTF3蛋白序列进行磷酸化位点预测。结果(图 2)显示:SmNTF3蛋白有潜在磷酸化位点32个, 丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)位点分别为15、9和8个。其中109、188、256和313位的Ser, 195位的Thr和197位的Tyr磷酸化概率较高, 推测是SmNTF3激酶的磷酸化位点。
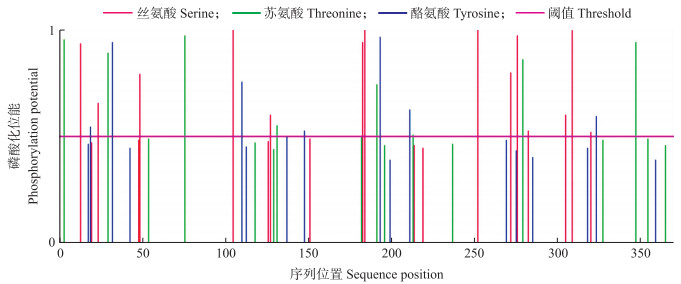
|
图 2 SmNTF3磷酸化位点预测 Fig. 2 Predicted phosphorylation sites in SmNTF3 |
对SmNTF3蛋白结构域进行分析, 发现在第32~319氨基酸残基处有1个丝氨酸/苏氨酸激酶催化结构域(S_TKc)。S_TKc是MAPK家族的保守结构域[19], 提示SmNTF3属于茄子MAPK家族。
蛋白二级结构分析结果(图 3)显示, SmNTF3蛋白中无规则卷曲占44.35%, α-螺旋占35.75%, β-折叠占19.89%, 并具有17个蛋白结合位点。进一步对其三级结构进行分析(图 4), 结果显示SmNTF3折叠交错呈复杂的三级结构, 在C端多为α-螺旋, 而N端β-折叠较多。
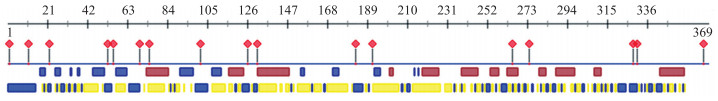
|
图 3 SmNTF3蛋白二级结构预测 Fig. 3 The predicted secondary structure of SmNTF3 protein |
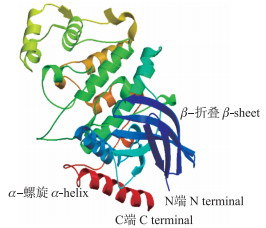
|
图 4 SmNTF3三级结构模型 Fig. 4 Tertiary structure model of SmNTF3 |
利用NCBI数据库检索SmNTF3同源序列, 对茄科及拟南芥中的NTF3同源序列进行比对, 结果(图 5)显示, 整体序列相似度高达87.06%, 6条氨基酸序列均具有MAPK家族的11个保守亚结构域, 第Ⅶ和第Ⅷ亚域之间存在TEY激活位点。SmNTF3与番茄SlMPK9(C组MAPK)及马铃薯StNTF3的同源性最高(98.39%), 与辣椒、烟草和拟南芥的序列同源性分别为96.77%、96.24%和84.95%。以上结果表明SmNTF3属于MAPK家族C组成员, 茄科NTF3序列保守性较高。
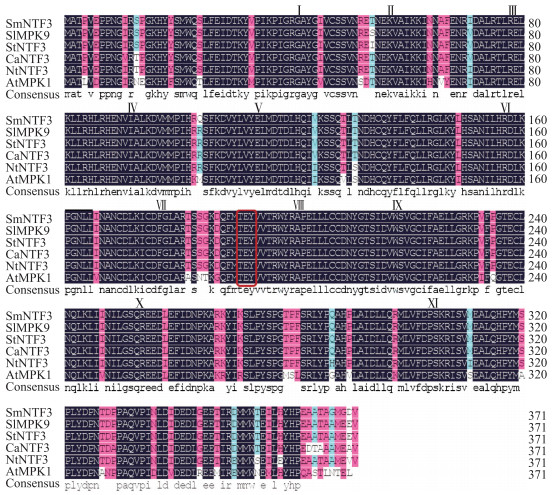
|
图 5 茄子与其他物种的NTF3氨基酸序列比对
Fig. 5 Alignment of amino acid sequence of NTF3 protein in eggplant and other species
Sm:茄子Solanum melongena; Sl:番茄Solanum lycopersicum(NP_001233761.1);St:马铃薯Solanum tuberosum(XP_006341972.1);Ca:辣椒Capsicum annuum(XP_016568515.1);Nt:烟草Nicotiana tabacum(Q40517.1);At:拟南芥Arabidopsis thaliana(NP 172492.1).下同。The same as follows. Ⅰ—Ⅺ分别表示MAPK家族11个保守亚结构域, 红色方框标注出TXY双磷酸化位点。Ⅰ-Ⅺ indicate the 11 conserved subdomains of the MAPK family, and the red box indicates the TXY double phosphorylation site. |
利用MEGA 7对14个物种的NTF3蛋白序列构建系统进化树, 结果(图 6)显示茄科的5种植物聚为同一分支, 茄子SmNTF3与来自番茄和马铃薯的同源序列亲缘关系最近。除茄科外SmNTF3与大花牵牛及油橄榄等进化关系相对较近, 而与拟南芥、西葫芦、狭叶羽扇豆等亲缘关系较远。
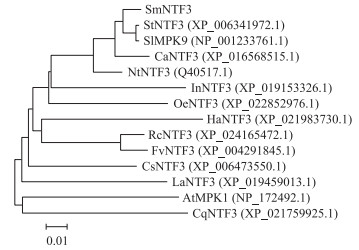
|
图 6 不同物种NTF3氨基酸序列的系统发育分析 Fig. 6 Phylogenetic analysis of amino acid sequences of NTF3 from different species In:大花牵牛Ipomoea nil; Oe:油橄榄Olea europaea var. sylvestris; Ha:向日葵Helianthus annuus; Rc:月季Rosa chinensis; Fv:野草莓Fragaria vesca subsp. vesca; Cs:甜橙Citrus sinensis; La:狭叶羽扇豆Lupinus angustifolius; Cq:西葫芦Cucurbita pepo. |
在SubLoc v1.0 database数据库中对SmNTF3进行分析, 预测结果显示其定位于细胞核和细胞质膜中。将SmNTF3-GFP与Marker载体转化烟草叶片进行瞬时表达, 采用激光共聚焦显微镜进行观察, 结果(图 7)显示, 在细胞核和细胞质膜上均能看到SmNTF3-GFP融合蛋白绿色荧光, 且与Marker红色荧光蛋白完全重叠后呈黄色, 说明SmNTF3可能定位于细胞核和细胞质膜。
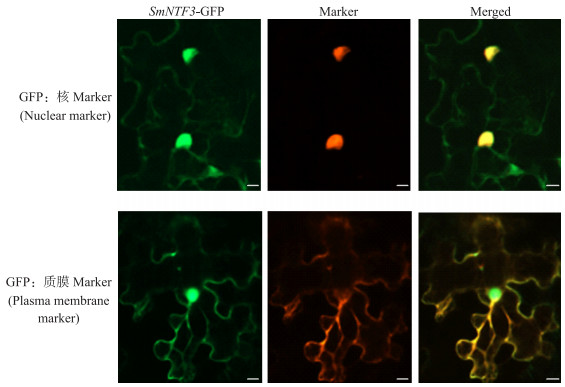
|
图 7 SmNTF3亚细胞定位 Fig. 7 Subcellular localization of SmNTF3 Bar=20 μm. |
对SmNTF3基因在茄子发育过程中的组织表达模式进行分析, 结果(图 8)显示:SmNTF3在茎中表达量最高, 显著高于根和叶, 在根和叶中表达量无明显差异, 说明SmNTF3在茄子中呈现组织特异性表达。
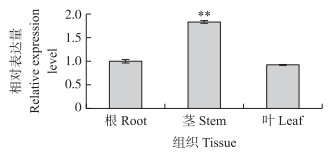
|
图 8 SmNTF3的组织表达分析 Fig. 8 Expression profiles of SmNTF3 in eggplant *P < 0.05, **P < 0.01. The same as follows. |
从图 9可知:SmNTF3的表达随NaCl处理浓度的增加呈先上升后下降的趋势, 在100 mmol·L-1 NaCl浓度胁迫下表达量最大, 约为对照的2倍(图 9-A)。选择100 mmol·L-1 NaCl进行不同时间处理试验, 结果(图 9-B)显示, 盐胁迫处理3 h, SmNTF3表达量显著上调, 而后逐步下降, 胁迫处理48 h, SmNTF3的表达量与对照差异不显著。以上结果说明SmNTF3基因响应盐胁迫。
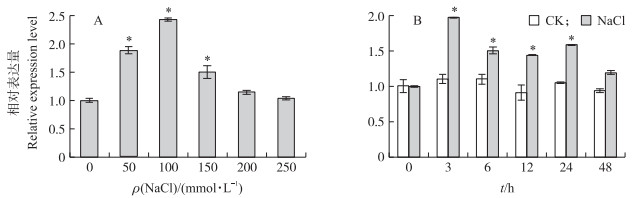
|
图 9 盐胁迫下SmNTF3基因的表达量分析 Fig. 9 Analysis of SmNTF3 expression level under salt stress A.不同浓度的NaCl处理12 h时SmNTF3基因表达量变化Changes of SmNTF3 gene expression level after treatment with different concentrations of NaCl for 12 h; B. 100 mmol·L-1 NaCl处理不同时间后SmNTF3基因表达量变化Changes of SmNTF3 gene expression level after treatment with 100 mmol·L-1 NaCl at different time. |
从图 10可知:PEG-6000胁迫处理显著诱导SmNTF3基因的表达, 在PEG-6000含量为5%、10%和15%时SmNTF3表达量持续上调, 在15% PEG-6000时表达量最高, 是对照的6.5倍(图 10-A)。选择15% PEG-6000进行时间处理试验, 结果(图 10-B)显示:随处理时间的延长, SmNTF3表达量呈先上升后下降趋势, 在12 h达到最高。这些结果说明SmNTF3基因响应干旱胁迫。
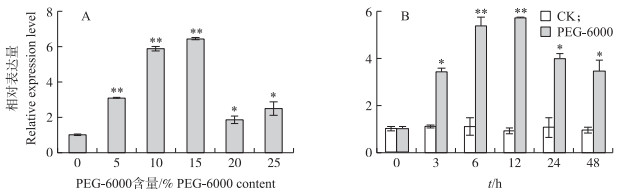
|
图 10 PEG-6000模拟干旱胁迫下SmNTF3基因的表达量分析 Fig. 10 Expression level analysis of SmNTF3 gene under PEG-6000 simulated drought stress A.不同含量的PEG-6000处理12 h时SmNTF3基因表达量变化Changes of SmNTF3 gene expression level after treatment with different contents of PEG-6000 for 12 h; B. 15% PEG-6000处理不同时间后SmNTF3基因表达量变化Changes of SmNTF3 gene expression level after treatment with 15% PEG-6000 at different time. |
MAPK是一类丝氨酸/苏氨酸(Ser/Thr)蛋白激酶, 在真核生物中具有高度的进化保守性[5]。NTF3基因最早作为MAPK同源体在烟草中被克隆, 基因鉴定发现其属于MAPK家族C组成员[18, 20]。本研究从茄子‘苏崎1号’中克隆得到SmNTF3基因, 其编码蛋白具有保守的S_TKc结构域。S_TKc结构域包含TXY双磷酸化位点, 这是上游基因进行磷酸化的部位。通过磷酸化位点预测, 我们寻找到SmNTF3蛋白可能发生磷酸化的氨基酸残基位置, 这对茄子MAPK级联途径的研究具有重要意义。氨基酸序列分析表明SmNTF3包含MAPK基因的保守结构域和TEY基序, 与MAPK家族C组成员序列高度同源, 认为SmNTF3属于C组。系统发育分析显示SmNTF3与来自番茄及马铃薯的同源序列亲缘关系最近, 同源性明显高于其他物种, 说明茄科NTF3同源序列高度保守。此外, SmNTF3与拟南芥、西葫芦等同源性也较高, 说明NTF3在不同物种中均具有保守性, 这在其他物种MAPK基因的研究中有相似结论[21]。
MAPK作为将信号分子从细胞表面传递到细胞核的重要因子, 主要在细胞核上表达, 部分MAPK也可能在细胞质或细胞膜等部位磷酸化特定的蛋白[4]。已有研究报道番茄SlMPK7定位于细胞核和细胞膜[22]; 玉米ZmMPK3-1主要定位于细胞膜和细胞核[23]; 玉米ZmMPK5定位于细胞核、细胞质和细胞膜[24]。本研究通过SmNTF3在烟草叶片中的瞬时表达发现其定位于细胞核和细胞质膜, 与上述研究结果一致, 也与其功能相符。
在植物MAPK的研究中, 尽管C组研究相对较少, 但已有的文献能够表明不同物种的MAPK家族C组成员广泛参与植物生长发育与外界刺激响应过程。目前研究较充分的是拟南芥中的4个C组MAPK基因: AtMPK1、AtMPK2、AtMPK7和AtMPK14。机械损伤和茉莉酸(JA)诱导拟南芥AtMPK1和AtMPK2的表达[25]; AtMPK1/2参与的级联途径能够有效调控拟南芥对盐胁迫的耐受性以及植物的衰老过程[26-27]; AtMPK7和AtMPK14在植物响应病原菌的防卫过程中起关键作用[28]。此外, 豌豆PsMPK2对机械损伤、脱落酸(ABA)和过氧化氢等信号分子的诱导产生响应[29]; 番茄SlMPK8和SlMPK9在热胁迫下表达量显著上调[30]; 烟草NtNTF3在JA和干旱处理时上调表达[18]。在本研究中, SmNTF3基因在盐和PEG-6000干旱处理下响应显著, 说明SmNTF3可能参与植物对盐及干旱胁迫的应答过程, 这些结果为进一步研究该基因的生物学功能提供理论依据。
| [1] |
刘丹, 崔彦玲, 潜宗伟. 茄子种业现状及遗传育种研究进展[J]. 北方园艺, 2019(1): 165-170. Liu D, Cui Y L, Qian Z W. Research advances in the seed industry and breeding of eggplant[J]. Northern Horticulture, 2019(1): 165-170 (in Chinese with English abstract). |
| [2] |
Golldack D, Li C, Mohan H, et al. Tolerance to drought and salt stress in plants:unraveling the signaling networks[J]. Frontiers in Plant Science, 2014, 5: 1-10. |
| [3] |
Xu J, Zhang S. Mitogen-activated protein kinase cascades in signaling plant growth and development[J]. Trends in Plant Science, 2015, 20(1): 56-64. DOI:10.1016/j.tplants.2014.10.001 |
| [4] |
Goyal R K, Dan T, Chomistek N, et al. Analysis of MAPK and MAPKK gene families in wheat and related Triticeae species[J]. BMC Genomics, 2018, 19(1): 2-26. DOI:10.1186/s12864-017-4249-6 |
| [5] |
Zhang M, Su J, Zhang Y, et al. Conveying endogenous and exogenous signals:MAPK cascades in plant growth and defense[J]. Current Opinion in Plant Biology, 2018, 45: 1-10. |
| [6] |
Smekalova V. Involvement of YODA and mitogen activated protein kinase 6 in Arabidopsis post-embryogenic root development through auxin up-regulation and cell division plane orientation[J]. New Phytologist, 2014, 203(4): 1175-1193. DOI:10.1111/nph.12880 |
| [7] |
Zhou C, Cai Z, Guo Y, et al. An Arabidopsis mitogen-activated protein kinase cascade, MKK9-MPK6, plays a role in leaf senescence[J]. Plant Physiology, 2009, 150(1): 167-177. DOI:10.1104/pp.108.133439 |
| [8] |
Chen L, Sun H, Wang F, et al. Genome-wide identification of MAPK cascade genes reveals the GhMAP3K14-GhMKK11-GhMPK31 pathway is involved in the drought response in cotton[J]. Plant Molecular Biology, 2020, 103: 211-223. DOI:10.1007/s11103-020-00986-0 |
| [9] |
Lewis T, Shapiro P, Ahn N. Signal transduction through MAP kinase cascades[J]. Advances in Cancer Research, 1998, 74: 49-139. |
| [10] |
Cai G, Wang G, Wang L, et al. A maize mitogen-activated protein kinase kinase, ZmMKK1, positively regulated the salt and drought tolerance in transgenic Arabidopsis[J]. Journal of Plant Physiology, 2014, 171(12): 1003-1016. DOI:10.1016/j.jplph.2014.02.012 |
| [11] |
Jalmi S K, Sinha A K. Functional involvement of a mitogen activated protein kinase module, OsMKK3-OsMPK7-OsWRK30 in mediating resistance against Xanthomonas oryzae in rice[J]. Scientific Reports, 2016, 6: 1-14. DOI:10.1038/s41598-016-0001-8 |
| [12] |
叶文武, 张萌, 曹明娜, 等. 大豆疫霉PsMPK1沉默突变体的基因表达谱分析[J]. 南京农业大学学报, 2016, 39(3): 386-393. Ye W W, Zhang M, Cao M N, et al. Gene expression profilling of a PSMPK1-silenced mutant of Phytophthora sojae[J]. Journal of Nanjing Agricultural University, 2016, 39(3): 386-393 (in Chinese with English abstract). DOI:10.7685/jnau.201509026 |
| [13] |
Ding H, He J, Wu Y, et al. The tomato mitogen-activated protein kinase SlMPK1 is as a negative regulator of the high temperature stress response[J]. Plant Physiology, 2018, 177(2): 633-651. DOI:10.1104/pp.18.00067 |
| [14] |
Ichimura K. Mitogen-activated protein kinase cascades in plants:a new nomenclature[J]. Trends in Plant Science, 2002, 7(7): 301-308. DOI:10.1016/S1360-1385(02)02302-6 |
| [15] |
朱彤彤, 杨磊, 孙文秀. 辣椒疫霉MAPK基因家族的鉴定及生物信息学分析[J]. 生物技术, 2018, 28(5): 460-466. Zhu T T, Yang L, Sun W X. Identification and bioinformatics analysis of the MAPK family in Phytophthora capsici[J]. Biotechnology, 2018, 28(5): 460-466 (in Chinese with English abstract). |
| [16] |
Zhang X, Xu X, Yu Y, et al. Integration analysis of MKK and MAPK family members highlights potential MAPK signaling modules in cotton[J]. Scientific Reports, 2016, 6(1): 1-13. DOI:10.1038/s41598-016-0001-8 |
| [17] |
Liu Q, Xue Q. Computational identification and phylogenetic analysis of the MAPK gene family in Oryza sativa[J]. Plant Physiology & Biochemistry, 2017, 45(1): 6-14. |
| [18] |
Zhang X, Cheng T, Wang G, et al. Cloning and evolutionary analysis of tobacco MAPK gene family[J]. Molecular Biology Reports, 2013, 40(2): 1407-1415. DOI:10.1007/s11033-012-2184-9 |
| [19] |
Mohanta T, Arora P, Mohanta N, et al. Identification of new members of the MAPK gene family in plants shows diverse conserved domains and novel activation loop variants[J]. BMC Genomics, 2015, 16(1): 58. DOI:10.1186/s12864-015-1244-7 |
| [20] |
Wilson C, Anglmayer R, Vicente O, et al. Molecular cloning, functional expression in Escherichia coli, and characterization of multiple mitogen-activated-protein kinases from tobacco[J]. European Journal of Biochemistry, 2004, 233(1): 249-257. |
| [21] |
Wu P, Wang W, Li Y, et al. Divergent evolutionary patterns of the MAPK cascade genes in Brassica rapa and plant phylogenetics[J]. Horticulture Research, 2017, 4(1): 170-179. |
| [22] |
关小燕, 陈丽妃, 何艳军, 等. 番茄SlMAPK7基因的亚细胞定位与组织表达特性[J]. 浙江大学学报(农业与生命科学版), 2014, 40(6): 598-604. Guan X Y, Chen L F, He Y J, et al. Subcellular localization and tissue expression pattern of SlMAPK7 gene in tomato[J]. Journal of Zhejiang University(Agriculture and Life Sciences Edition), 2014, 40(6): 598-604 (in Chinese with English abstract). |
| [23] |
Liu Y, Wang L, Zhang D, et al. Expression analysis of segmentally duplicated ZmMPK3-1 and ZmMPK3-2 genes in maize[J]. Plant Molecular Biology Reporter, 2013, 31(2): 457-463. DOI:10.1007/s11105-012-0489-4 |
| [24] |
Ding H, Zhang A, Wang J, et al. Identity of an ABA-activated 46 kDa mitogen-activated protein kinase from Zea mays leaves:partial purification, identification and characterization[J]. Planta, 2009, 230(2): 239-251. DOI:10.1007/s00425-009-0938-y |
| [25] |
Dolores O M, Amador P, Miguel A, et al. Diverse stress signals activate the C1 subgroup MAP kinases of Arabidopsis[J]. FEBS Letters, 2007, 581(9): 1834-1840. DOI:10.1016/j.febslet.2007.03.075 |
| [26] |
Zhang J, Gao J, Zhu Z, et al. MKK4/MKK5-MPK1/MPK2 cascade mediates SA-activated leaf senescence via phosphorylation of NPR1 in Arabidopsis[J]. Plant Molecular Biology, 2020, 102: 463-475. DOI:10.1007/s11103-019-00958-z |
| [27] |
Hwa C M, Yang X C. The AtMKK3 pathway mediates ABA and salt signaling in Arabidopsis[J]. Acta Physiologiae Plantarum, 2008, 30(3): 277-286. DOI:10.1007/s11738-007-0117-3 |
| [28] |
Dóczi R, Brader G, Pettkó Szandtner A, et al. The Arabidopsis mitogen-activated protein kinase kinase MKK3 is upstream of group C mitogen-activated protein kinases and participates in pathogen signaling[J]. The Plant Cell, 2007, 19(10): 3266-3279. DOI:10.1105/tpc.106.050039 |
| [29] |
Dolores O M, Miguel A, Amador P, et al. Characterization of PsMPK2, the first C1 subgroup MAP kinase from pea(Pisum sativum L.)[J]. Planta, 2008, 227(6): 1333-1342. DOI:10.1007/s00425-008-0705-5 |
| [30] |
Kong F, Wang J, Cheng L, et al. Genome-wide analysis of the mitogen-activated protein kinase gene family in Solanum lycopersicum[J]. Gene, 2011, 499(1): 108-120. |




