文章信息
- 计青青, 冯治洋
- JI Qingqing, FENG Zhiyang
- 基于序列宏基因组学技术的土壤Ⅱ型聚酮合酶KSα基因的挖掘
- Discovery of novel eDNA-derived KSα genes of type Ⅱ polyketide synthase using sequence-based metagenomic approach
- 南京农业大学学报, 2020, 43(2): 356-363
- Journal of Nanjing Agricultural University, 2020, 43(2): 356-363.
- http://dx.doi.org/10.7685/jnau.201903058
-
文章历史
- 收稿日期: 2019-03-26
微生物天然产物是小分子药物的重要来源, 已经发现和鉴定了大量结构与功能多样的微生物天然产物, 并成功应用于医药、畜牧和植物保护等领域[1-2]。近年来, 越来越多的微生物已经在实验室进行了培养发酵研究, 从微生物中发现新结构天然产物的概率越来越低[3-4]。Lloyd等[5]通过对26个环境样品中微生物的分析, 发现可培养微生物仅占样品中微生物总量的0.5%。绝大部分环境微生物不能在现有实验室条件下培养, 而这些在实验室条件下还不能被培养的微生物, 已被认为是天然产物发现的新资源[6]。应对绝大部分环境微生物的不可培养性, 科学家提出了宏基因组学策略, 即直接从环境样品中提取微生物总基因组DNA(eDNA), 并将含天然产物生物合成基因的eDNA克隆到可培养微生物中进行功能性表达, 为发掘非培养微生物来源的天然产物提供了可行性[7]。
芳香聚酮化合物(Ⅱ型聚酮)是一类具有多种结构类型和多样生物活性的微生物次级代谢产物, 有着广泛的应用, 如应用于疾病治疗的抗肿瘤药物乌达霉素(urdamycin A)和抗白血病药物柔红霉素(daunorubicin), 应用于畜牧生产的四环素(tetracycline)等。对芳香聚酮的生物合成研究发现, 芳香聚酮的聚酮链骨架均由最小PKS(mini-PKS)负责合成。mini-PKS包括酮基合成酶(KSα)、链长因子(KSβ)和酰基载体蛋白(ACP)。KSα负责起始底物的识别, 并完成随后的克莱森缩合; KSβ虽然缺少酮基合成酶的催化活性位点半胱氨酸残基, 但与合成的聚酮链长度相关; ACP(acyl-carrier-protein)连接碳链延长所用底物丙二酰辅酶A。Ⅱ型聚酮化合物生物合成通常先将起始单元加载到ACP上, 然后将其转移到KSα活性位点半胱氨酸残基上, 以丙二酰辅酶A进行迭代延伸, 形成碳链骨架, 再经环化酶/芳香化酶(aromatase/cyclase, ARO/CYC)催化形成芳香主环结构, 最后经后修饰(甲基化、糖基化、氧化还原等)形成结构多样的芳香聚酮化合物[8-10]。
在芳香聚酮的生物合成中, KSα和KSβ是高度保守的基因, 并决定后期合成的聚酮化合物类型[11-12]。因此, 对环境土壤中存在的KSα和KSβ序列进行分析可以预测相关土壤微生物生产芳香聚酮的潜力, 为发现新芳香聚酮化合物提供指导[13-14]。本研究利用KSα保守序列引物, 筛选峨眉山和珠穆朗玛峰土壤微生物宏基因组文库, 将获得的KSα基因片段与已知KSα基因进行比较分析, 并预测KSα基因生物合成所对应的聚酮化合物种类, 旨在为将来在相应宏基因组文库中挖掘新型聚酮化合物提供指导。
1 材料与方法 1.1 试验材料 1.1.1 土壤宏基因组文库已构建的珠峰土壤宏基因组文库的土壤采集于珠穆朗玛峰(北纬28°21′, 东经86°56′, 海拔5 500 m), 峨眉山土壤宏基因组文库的土壤采集于四川峨眉山森林土(北纬29°34′, 东经103°20′, 海拔900 m)。珠峰文库按照Brady[15]和Gu等[16]的方法使用pWEB-TNC Cosmid Cloning KitTM(Epicentre公司)在大肠杆菌EPI100-T1R(Epicentre公司)中构建, 文库菌具有氨苄青霉素与氯霉素抗性; 四川峨眉山文库在大肠杆菌JTU007[17]中用pJTU2554载体[18]构建, 文库菌具有安普霉素与卡那霉素抗性。
1.1.2 试剂与仪器2×Taq Master mix、氨苄青霉素、pTG19-T载体、DNA marker均购于上海捷瑞生物有限公司; BamHⅠ及T4-DNA Ligase购于赛默飞世尔科技(中国)有限公司; 质粒提取试剂盒(TIAN pure Mini Plasmid Kit)和琼脂糖凝胶回收试剂盒(TIAN gel Mini Purification Kit)购于天根生化科技(北京)有限公司。Hema9600基因扩增仪购于珠海黑马医学仪器有限公司; DYY-6C稳压稳流电泳仪购于北京六一生物科技有限公司; MicroPulser电穿孔仪购于美国BIO-RAD公司; DNP-9162电热恒温培养箱购于上海申安医疗器械有限公司; Gel X1520凝胶成像仪购于上海欧翔科学仪器有限公司; SW-CJ-1FD超净工作台购于苏州苏洁净化设备有限公司。
1.2 Ⅱ型聚酮KSα基因的筛选利用基于Ⅱ型PKS的KSα保守域设计的简并引物[19]dp540F(5′-GGNTGCACSTCNGGNMTSGAC-3′)和dp1100R(5′-CCGATSGCNCCSAGNGAGTG-3′)筛选珠峰文库质粒与四川峨眉山文库质粒。20 μL反应体系:2 μL模板, 10 μL 2×Taq Master mix, 上、下游引物各1 μL, 二甲基亚砜(DMSO)1 μL, ddH2O 5 μL。反应条件:95 ℃ 5 min; 95 ℃ 1 min, 59.7 ℃ 1 min, 72 ℃ 1 min, 共35个循环; 72 ℃ 10 min。扩增产物经10 g · L-1琼脂糖凝胶电泳检测, 将约554 bp大小的DNA产物用胶回收试剂盒回收。
1.3 Ⅱ型聚酮KSα基因片段的克隆与测序将Ⅱ型聚酮KSα片段的回收产物与pTG19-T载体16 ℃条件下连接4 h, 10 μL连接体系包含10×T4 DNA Ligase Buffer 1 μL, pTG19-T载体0.5 μL, 回收产物5.5 μL, ddH2O 3 μL。连接反应结束后, 将连接体系置于65 ℃水浴温育20 min, 使连接酶失活。连接产物电转化(电压为1.5 kV)至大肠杆菌EPI100-T1R, 与IPTG(异丙基-β-D-半乳糖苷, 终质量浓度为4 mg · mL-1)和X-gal(5-溴-4-氯-3-吲哚-β-D-半乳糖苷, 终质量浓度为0.8 mg · mL-1)混合, 涂布于含氨苄青霉素(终质量浓度为100 μg · mL-1)的LB琼脂平板, 37 ℃培养过夜。
次日挑取白色单菌落, 在含氨苄青霉素抗性的液体LB培养基中37 ℃振荡培养16~18 h, 提取质粒后BamHⅠ限制性酶切验证, 挑取酶切片段大小正确的质粒送南京金斯瑞生物科技有限公司测序。测序结果去除重复序列、载体及引物序列后, 用NCBI BLASTx(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行比对。获得的KSα基因序列以登录号MK569765—MK569797和MK569799—MK569804保存在NCBI(SRA)数据库中。
1.4 KSα基因的系统发生树构建使用MEGA 7.0 ClustalX的BLOSUM62距离矩阵构建多重比对。采用Nei等[20]的邻接法, 设定步长检验次数1 000次, cut-off值50%构建系统发生树。将与脂肪酸生物合成途径不相关的大肠杆菌fabB基因[21]作为系统发生树的外群。
2 结果与分析 2.1 土壤文库中Ⅱ型聚酮KSα基因的筛选以Ⅱ型聚酮KSα基因的简并引物dp540F/dp1100R分别对珠峰土壤文库与峨眉山土壤文库进行PCR筛选(图 1), 将具有明亮条带的PCR扩增产物(554 bp)割胶回收, 经TA克隆后测序。测序结果除去重复的序列, 获得珠峰文库KSα基因28个, 峨眉山文库KSα基因11个。
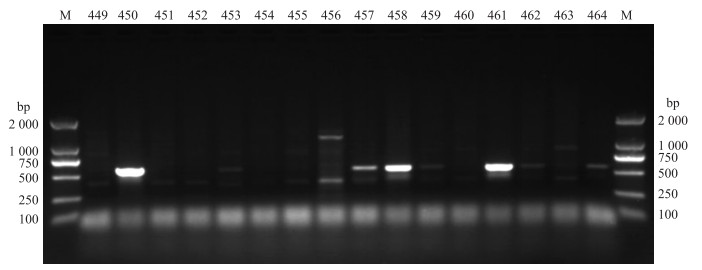
|
图 1 利用简并引物从土壤宏基因组文库中筛选KSα基因 Fig. 1 Screening KSα genes from soil metagenomic library using degenerated primers M. DNA marker; 449~464. PCR产物PCR products. |
将获得的39个Ⅱ型聚酮KSα基因与GenBank上已知的KS序列进行同源性比对, 结果(表 1)显示这些基因片段与已知的KS序列一致性为63%~96%(平均值75%)。其中EM86与simocyclinone生物合成基因簇中的ketosynthase SimA1具有73%的一致性, simocyclinones属于角环类芳香聚酮化合物, 具有抗肿瘤活性。EM129与普拉迪霉素(pradimicin)生物合成基因簇的酮基合成酶具有75%的一致性, 普拉迪霉素属于五元角环酚类化合物, 具有抗真菌活性。EM130和EM202分别与格瑞克霉素(grincamycin)的酮基合成酶具有85%和88%的一致性, 格瑞克霉素是带有糖取代基的角环类芳香聚酮, 具有抗肿瘤活性。ZF418、ZF439、ZF475、ZF648和ZF491与赫达霉素(hedamycin)的酮基合成酶具有71%的一致性。赫达霉素属于多色霉素(pluramycin)家族的一种具有抗肿瘤活性的角环类芳香聚酮, 其独特的生物合成起始单元(己烯酸酯)的生成和转移由Ⅰ型和Ⅱ型PKS协同完成。ZF508与黄酯菌素(xantholipin)的酮基合成酶有76%的一致性, 黄酯菌素属于氧杂蒽酮类五角多酚聚酮化合物。ZF518、ZF585与naphthocyclinone的酮基合成酶有82%的一致性, naphthocyclinone属于萘醌类芳香聚酮, 具有抑制革兰氏阳性细菌活性。
| 基因/蛋白 Gene/Protein |
同源蛋白(来源) Homologous protein(origin) |
一致性/% Identity |
序列号 Accession No. |
| EM33/EM33 | β-酮酰合成酶(玫瑰产色链霉菌) Beta-ketoacyl-synthase(Streptomyces roseochromogenus subsp. oscitans DS 12.976) |
72 | EST27264.1 |
| EM79/EM79 | β-酮酰合成酶(弗兰克氏菌) Beta-ketoacyl synthase(Frankia sp. EAN1pec) |
82 | ABW11825.1 |
| EM86/EM86 | 角环类抗生素simocyclinone生物合成中的酮基合成酶SimA1(链霉菌) Ketosynthase SimA1 in angucycline antibiotic simocyclinone biosynthesis(Streptomyces sp.) |
73 | AAK06784.1 |
| EM129/EM129 | 五角多酚类抗生素普那米星生物合成中的酮酰合成酶(马杜拉放线菌) Ketoacyl synthase in pentangular polyphenol antibiotic pradimicin biosynthesis (Actinomadura hibisca) |
75 | BAA23144.1 |
| EM130/EM130 | 角环类抗生素格瑞克霉素生物合成中的酮酰合成酶(葡萄牙链霉菌) Ketoacyl synthase in angucycline antibiotic grincamycin biosynthesis(Streptomyces lusitanus) |
85 | AGO50610.1 |
| EM167/EM167 | 酮基合成酶(海南阿萨诺菌) Ketosynthase(Asanoa hainanensis) |
73 | SNT54706.1 |
| EM169/EM169 | β-酰基载体蛋白合成酶(Blastocatellia bacterium) Beta-ACP synthase(Blastocatellia bacterium) |
82 | PWT80873.1 |
| EM202/EM202 | 角环类抗生素格瑞克霉素生物合成中的酮酰合成酶(葡萄牙链霉菌) Ketoacyl synthase in angucycline antibiotic grincamycin biosynthesis(Streptomyces lusitanus) |
88 | AGO50610.1 |
| EM221/EM221 | β-酰基载体蛋白合成酶(Blastocatellia bacterium) Beta-ACP synthase(Blastocatellia bacterium) |
81 | PWT80873.1 |
| EM278/EM278 | 酮基合成酶(弗兰克氏菌) Ketosynthase(Frankia sp. EI5c) |
75 | OAA20009.1 |
| EM324/EM324 | 假定的Ⅱ型聚酮合酶酮基合成酶α亚基(不可培养的细菌) Putative type Ⅱ PKS ketosynthase alpha subunit(uncultured bacterium) |
96 | AGG42678.1 |
| ZF418/ZF418 | 角环类抗生素赫达霉素生物合成中的酮基合成酶α亚基(灰红链霉菌) Keto synthase alpha in angucycline antibiotic hedamycin biosynthesis(Streptomyces griseoruber) |
71 | AAP85362.1 |
| ZF439/ZF439 | 角环类抗生素赫达霉素生物合成中的酮基合成酶α亚基(灰红链霉菌) Keto synthase alpha in angucycline antibiotic hedamycin biosynthesis(Streptomyces griseoruber) |
71 | AAP85362.1 |
| ZF475/ZF475 | 角环类抗生素赫达霉素生物合成中的酮基合成酶α亚基(灰红链霉菌) Keto synthase alpha in angucycline antibiotic hedamycin biosynthesis(Streptomyces griseoruber) |
71 | AAP85362.1 |
| ZF648/ZF648 | 角环类抗生素赫达霉素生物合成中的酮基合成酶α亚基(灰红链霉菌) Keto synthase alpha in angucycline antibiotic hedamycin biosynthesis(Streptomyces griseoruber) |
71 | AAP85362.1 |
| ZF461/ZF461 | Ⅱ型聚酮合酶酮基合成酶(链霉菌) Type Ⅱ PKS ketosynthase(Streptomyces sp. SCSIO 11527) |
63 | AHB18633.1 |
| ZF471/ZF471 | 角环类抗生素Sch 47554和Sch 47555生物合成中的酮酰合成酶(链霉菌) Ketoacyl synthase in angucycline antibiotic Sch 47554 and Sch 47555 biosynthesis (Streptomyces sp. SCC 2136) |
74 | CAH10117.1 |
| ZF478/ZF478 | Ⅱ型聚酮合酶(链霉菌) Type Ⅱ polyketide synthase(Streptomyces sp.) |
93 | APD71579.1 |
| ZF491/ZF491 | 角环类抗生素赫达霉素生物合成中的酮基合成酶α亚基(灰红链霉菌) Keto synthase alpha in angucycline antibiotic hedamycin biosynthesis(Streptomyces griseoruber) |
71 | AAP85362.1 |
| ZF495/ZF495 | β-酰基载体蛋白合成酶(链霉菌) Beta-ACP synthase(Streptomyces sp. PBH53) |
80 | AKN74861.1 |
| ZF496/ZF496 | β-酰基载体蛋白合成酶(贫养杆菌) Beta-ACP synthase(Modestobacter sp. VKM Ac-2676) |
88 | OMQ15373.1 |
| ZF506/ZF506 | 酮基合成酶(弗兰克氏菌) Ketosynthase(Frankia sp. EI5c) |
73 | OAA20009.1 |
| ZF508/ZF508 | 五角多酚类抗生素黄酯菌素生物合成中的酮基合成酶α亚基(东方拟无枝酸菌) Ketosynthase alpha subunit in pentangular polyphenol antibiotic xantholipin biosynthesis (Amycolatopsis orientalis subsp. vinearia) |
76 | ADE22315.1 |
| ZF518/ZF518 | 萘醌类抗生素naphthocyclinone生物合成中的β-酮酰合成酶(沙场链霉菌) Beta-ketoacyl synthase in naphthaquinone antibiotic naphthocyclinone biosynthesis (Streptomyces arenae) |
82 | AAD20267.1 |
| ZF585/ZF585 | 萘醌类抗生素naphthocyclinone生物合成中的β-酮酰合成酶(沙场链霉菌) Beta-ketoacyl synthase in naphthaquinone antibiotic naphthocyclinone biosynthesis (Streptomyces arenae) |
82 | AAD20267.1 |
| ZF539/ZF539 | 酮酰合成酶α亚基(链霉菌) Keto-acyl synthase alpha(Streptomyces sp.) |
92 | ARK36155.1 |
| ZF605/ZF605 | 酮基合成酶(弗兰克氏菌) Ketosynthase(Frankia sp. EI5c) |
78 | OAA20009.1 |
| ZF665/ZF665 | β-酰基载体蛋白合成酶(浅紫灰链霉菌) Beta-ACP synthase(Streptomyces lavendulae subsp. lavendulae) |
71 | AAK61718 |
| ZF672/ZF672 | 假定的聚酮β-酮酰合成酶(弗兰克氏菌) Putative polyketide beta-ketoacyl synthase(Frankia sp.) |
71 | SBW18691.1 |
| ZF1556/ZF1556 | β-酰基载体蛋白合成酶(链霉菌) Beta-ACP synthase(Streptomyces sp. PBH53) |
80 | AKN74861.1 |
| ZF1045/ZF1045 | 3-氧酰-酰基载体蛋白合成酶(筑波链霉菌) 3-oxoacyl-ACP synthase(Streptomyces tsukubensis NRRL18488) |
82 | EIF92240.1 |
| ZF1107/ZF1107 | 酮酰合成酶(不可培养的细菌) Ketoacyl synthase(uncultured bacterium) |
76 | AAW59142.1 |
| ZF1146/ZF1146 | 酮基合成酶(阿萨诺氏菌) Ketosynthase(Asanoa ishikariensis) |
83 | SDZ60853.1 |
| ZF1240/ZF1240 | β-酮酰合成酶(古月颓子弗兰克氏菌) Beta-ketoacyl synthase(Frankia elaeagni) |
74 | WP_084692218.1 |
| ZF1288/ZF1288 | 酮基合成酶(弗兰克氏菌) Ketosynthase(Frankia sp. EI5c) |
71 | OAA20009.1 |
| ZF1558/ZF1558 | β-酰基载体蛋白合成酶(马桑弗兰克氏菌) Beta-ACP synthase(Frankia coriariae) |
76 | KLL10717.1 |
| ZF1559/ZF1559 | β-酰基载体蛋白合成酶(链霉菌) Beta-ketoacylsynthase(Streptomyces sp. KIB-H033) |
71 | AOZ61212.1 |
| ZF1660/ZF1660 | β-酰基载体蛋白合成酶(贫养杆菌) Beta-ACP synthase(Modestobacter sp. VKM Ac-2676) |
73 | OMQ15373.1 |
| ZF1655/ZF1655 | 角环类抗生素BE-7585A生物合成中的酮基合成酶α亚基(东方拟无枝酸菌) Ketosynthase alpha subunit in angucycline antibiotic BE-7585A biosynthesis (Amycolatopsis orientalis subsp. vinearia) |
76 | ADI71443.1 |
以34种编码已知的Ⅱ型聚酮化合物KSα基因和5种聚酮化合物孢子色素KSα基因的氨基酸序列为基础, 与珠峰土壤文库、峨眉山土壤文库中筛选得到的39个KSα序列一起构建系统发生树, 大肠杆菌的fabB基因作为系统发生树的外群(图 2)。系统发生树结果显示, 从土壤宏基因组文库中筛选获得的39个KSα基因片段大多与已知聚酮化合物的KSα基因相似性不高, 尤其是一些酮基合成酶如EM33、ZF461、ZF495、ZF665等远离聚酮类抗生素与孢子色素分支, 形成独立的分支, 表明这些酮基合成酶编码基因所在的基因簇具有合成结构新颖化合物的潜力。
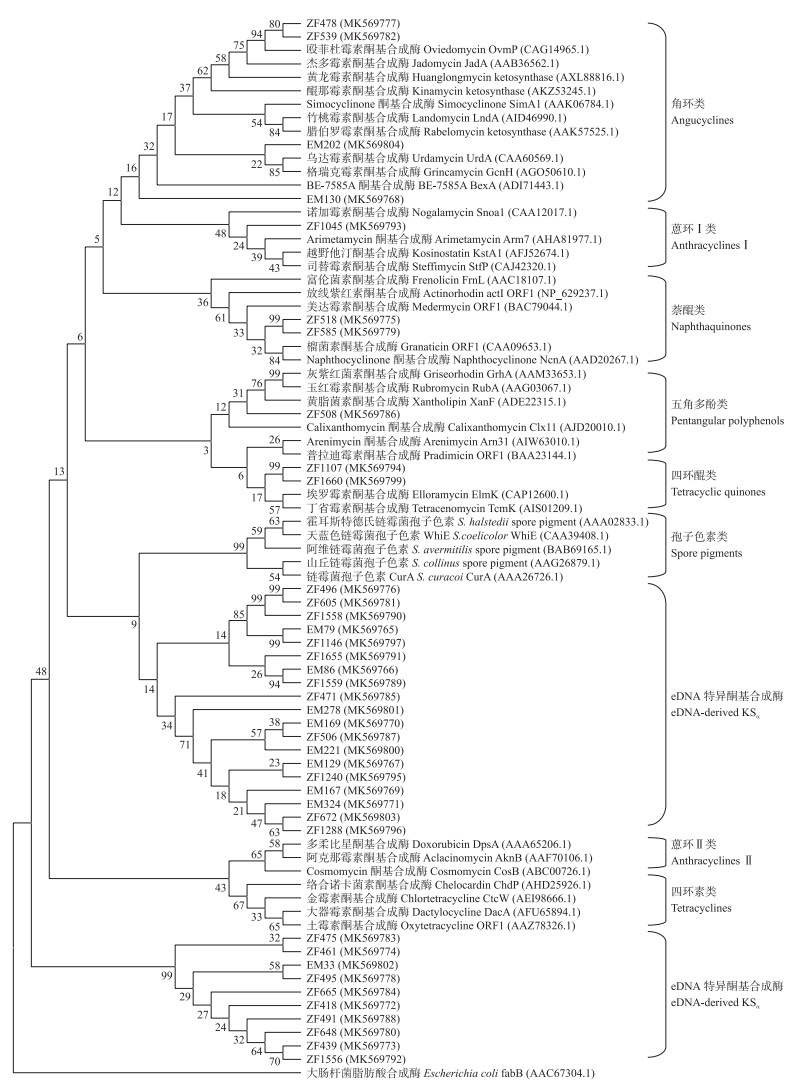
|
图 2 基于KSα基因片段所编码蛋白构建的系统发生树 Fig. 2 Phylogenetic tree constructed from protein encoded by KSα gene fragments |
此外, Metsä-Ketelä等[22]报道, 利用KSα序列构建的系统发生树中各类芳香族化合物与孢子色素能形成明显的分支, 由此可对未知的芳香族化合物进行初步分类。其中ZF478、ZF539、EM130与EM202在发生树中与角环类Ⅱ型聚酮化合物(图 3-A)处在同一分支, 推测这些酮基合成酶编码基因所在的基因簇可能合成角环类聚酮化合物; ZF518、ZF585与萘醌类化合物(图 3-B)处在同一分支, 且两者又形成独立的进化亚支, 自展值达99, 推测这2种酮基合成酶编码基因所在的基因簇可能合成结构非常相似的萘醌类聚酮化合物; ZF508与五角多酚类化合物(图 3-C)聚为一支, 推测该酮基合成酶编码基因所在的基因簇可能合成五角多酚类芳香聚酮化合物; 蒽环类Ⅱ型聚酮化合物(图 3-D)在树中形成2个不同的分支, ZF1045与蒽环类化合物nogalamycin、arimetamycin在同一分支, 推测该酮基合成酶编码基因所在的基因簇可能合成与其结构相似的蒽环类化合物; ZF1107、ZF1660在树中与四环醌类(图 3-F)处在同一分支, 推测这2种酮基合成酶编码基因所在的基因簇可能合成四环醌类化合物, 这与KSα序列一致性分析结果基本一致。
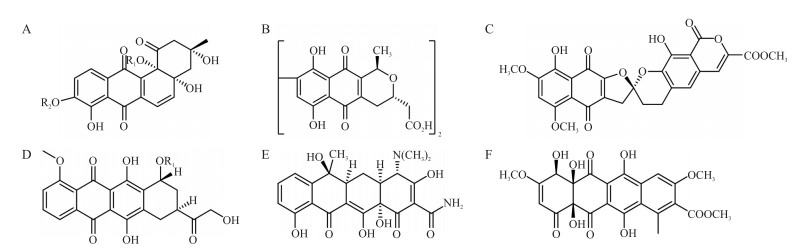
|
图 3 不同种类芳香聚酮化合物的结构 Fig. 3 Structures of different classes of aromatic polyketides compounds A.角环类抗生素乌达霉素A Angucycline antibiotic urdamycin A; B.萘醌类抗生素放线紫红素Naphthaquinone antibiotic actinorhodin; C.五角多酚类抗生素玉红霉素β Pentangular polyphenols antibiotic rubromycin β; D.蒽环类抗生素阿霉素Anthracycline antibiotic doxorubicin; E.四环素类抗生素四环素Tetracycline antibiotic tetracycline; F.四环醌类抗生素特曲霉素Tetracyclic quinones antibiotic tetracenomycin C. R1、R2为糖取代基部分。R1 and R2 refer to sugar moieties. |
在过去的几十年中, 抗生素耐药菌的出现及广泛传播已经成为严重的公共卫生威胁, 临床中可供选择的抗生素越来越有限[23-24]。微生物是新抗生素发现的重要来源, 但传统获得天然活性化合物的方法依赖于纯种微生物的分离培养, 而仅有很小一部分微生物能在实验室培养, 极大地限制了微生物来源抗生素的发掘。20世纪90年代, 利用传统方法发现新抗生素的速率明显减慢, 不以微生物培养性为前提的宏基因组学的快速发展给活性天然产物的发现带来了新的生机[25-26]。
利用宏基因组学策略, 在新化合物的发现上获得了很大的进展。在挖掘聚酮化合物方面, 近年来也有成果报道, 如Feng等[14]通过对土壤宏基因组文库中筛选获得的KSβ序列的系统发生树分析, 预测不同分支中的KSβ可能对应不同的聚酮结构类型, 并通过异源表达获得了不同结构类型的新颖聚酮化合物, 验证了化合物结构的可预测性; Kang等[27]利用同样的KSβ序列标签, 利用宏基因组学方法获得了对结肠(HCT-116和WiDr)和乳腺(MDA-MB-231)癌细胞系具有显著抗增殖活性的五角多酚化合物Arixanthomycin A。以上这些研究, 均从克隆KS基因着手, 再根据所获得的基因序列, 对克隆获得的KS基因所对应的产物进行预测, 以此为基础, 选择特殊类型的基因进行表达, 最终获得新的聚酮化合物。本研究利用Ⅱ型聚酮合酶的KSα基因保守域设计探针, 筛选珠峰土壤文库与峨眉山土壤文库, 共得到39个KSα序列, 将这些KSα序列与已知Ⅱ型聚酮化合物KSα基因及孢子色素KSα基因的氨基酸序列构建系统发生树, 初步预测这些KSα基因所在的基因簇可能编码合成的聚酮化合物种类。在此基础上, 可以对新的KSα基因克隆进行进一步功能性表达, 获得新的聚酮化合物。
本研究从土壤宏基因组文库中发现的KSα基因序列与已知的Ⅱ型聚酮化合物的KSα基因相似性均不高, 除在不同结构类型的Ⅱ型聚酮对应的KSα基因分支中有分布外, 有些酮基合成酶如EM33、ZF461、ZF495、ZF665等远离聚酮类抗生素与孢子色素分支形成独立的分支, 这表明土壤中可能含有未被挖掘的生物合成基因类型。进一步的研究将揭示这些基因簇所编码的聚酮化合物结构, 获得新的生物合成基因与化合物结构的对应关系, 为获得新型生物活性分子奠定基础。
| [1] |
Vásquez-Bochm L X, Velázquez-Paniagua M, Castro-Vázquez S S, et al. Transcriptome-based identification of lovastatin as a breast cancer stem cell-targeting drug[J]. Pharmacological Reports, 2019, 71(3): 535-544. DOI:10.1016/j.pharep.2019.02.011 |
| [2] |
Alekseev A, Tyurin M, Khairov K, et al. Characterization and biological action of avermectin granules on the Moroccan locust, Dociostaurus maroccanus(Orthoptera:Acrididae)[J]. Journal of Economic Entomology, 2019, 112(6): 2663-2669. |
| [3] |
Banik J J, Brady S F. Cloning and characterization of new glycopeptide gene clusters found in an environmental DNA megalibrary[J]. Proc Natl Acad Sci USA, 2008, 105(45): 17273-17277. DOI:10.1073/pnas.0807564105 |
| [4] |
Banik J J, Craig J W, Calle P Y, et al. Tailoring enzyme-rich environmental DNA clones:a source of enzymes for generating libraries of unnatural natural products[J]. Journal of the American Chemical Society, 2010, 132(44): 15661-15670. DOI:10.1021/ja105825a |
| [5] |
Lloyd K G, Ladau J, Steen A D, et al. Phylogenetically novel uncultured microbial cells dominate Earth microbiomes[J]. mSystems, 2018, 3(5). DOI:10.1128/mSystems.00055-18 |
| [6] |
Rappé M S, Giovannoni S J. The uncultured microbial majority[J]. Annual Review of Microbiology, 2003, 57(1): 369-394. DOI:10.1146/annurev.micro.57.030502.090759 |
| [7] |
Handelsman J, Rondon M R, Brady S F, et al. Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J]. Chemistry and Biology, 1998, 5(10): R245-R249. DOI:10.1016/S1074-5521(98)90108-9 |
| [8] |
Das A, Khosla C. Biosynthesis of aromatic polyketides in bacteria[J]. Accounts of Chemical Research, 2009, 42(5): 631-639. DOI:10.1021/ar8002249 |
| [9] |
Zhou H, Li Y R, Tang Y. Cyclization of aromatic polyketides from bacteria and fungi[J]. Natural Product Reports, 2010, 27(6): 839-868. DOI:10.1039/b911518h |
| [10] |
Zhang Z, Pan H X, Tang G L. New insights into bacterial type Ⅱ polyketide biosynthesis[J]. F1000Research, 2017, 6: 172. DOI:10.12688/f1000research.10466.1 |
| [11] |
Gullón S, Olano C, Abdelfattah M S, et al. Isolation, characterization, and heterologous expression of the biosynthesis gene cluster for the antitumor anthracycline steffimycin[J]. Applied and Environmental Microbiology, 2006, 72(6): 4172-4183. DOI:10.1128/AEM.00734-06 |
| [12] |
Basnet D B, Oh T J, Vu T T, et al. Angucyclines Sch 47554 and Sch 47555 from Streptomyces sp. SCC-2136:cloning, sequencing, and characterization[J]. Molecules and Cells, 2006, 22(2): 154-162. |
| [13] |
Metsä-Ketelä M, Salo V, Halo L, et al. An efficient approach for screening minimal PKS genes from Streptomyces[J]. FEMS Microbiology Letters, 1999, 180(1): 1-6. DOI:10.1111/j.1574-6968.1999.tb08770.x |
| [14] |
Feng Z Y, Chakraborty D, Dewell S B, et al. Environmental DNA-encoded antibiotics fasamycins A and B inhibit FabF in type Ⅱ fatty acid biosynthesis[J]. Journal of the American Chemical Society, 2012, 134(6): 2981-2987. DOI:10.1021/ja207662w |
| [15] |
Brady S F. Construction of soil environmental DNA cosmid libraries and screening for clones that produce biologically active small molecules[J]. Nature Protocols, 2007, 2(5): 1297-1305. DOI:10.1038/nprot.2007.195 |
| [16] |
Gu X Q, Wang S L, Wang S C, et al. Identification and characterization of two novel esterases from a metagenomic library[J]. Food Science and Technology Research, 2015, 21(5): 649-657. DOI:10.3136/fstr.21.649 |
| [17] |
Zhou H, Wang Y, Yu Y, et al. A non-restricting and non-methylating Escherichia coli strain for DNA cloning and high-throughput conjugation to Streptomyces coelicolor[J]. Current Microbiology, 2012, 64(2): 185-190. DOI:10.1007/s00284-011-0048-5 |
| [18] |
Li L, Xu Z N, Xu X Y, et al. The mildiomycin biosynthesis:initial steps for sequential generation of 5'-hydroxymethylcytidine 5'-monophosphate and 5-hydroxymethylcytosine in Streptoverticillium rimofaciens ZJU5119[J]. ChemBioChem, 2008, 9(8): 1286-1294. DOI:10.1002/cbic.200800008 |
| [19] |
Wawrik B, Kerkhof L, Zylstra G J, et al. Identification of unique type Ⅱ polyketide synthase genes in soil[J]. Applied and Environmental Microbiology, 2005, 71(5): 2232-2238. DOI:10.1128/AEM.71.5.2232-2238.2005 |
| [20] |
Nei M, Saitou N. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [21] |
Kauppinen S, Siggaard-Andersen M, von Wettstein-Knowles P. β-ketoacyl-ACP synthase Ⅰ of Escherichia coli:nucleotide sequence of the fabB gene and identification of the cerulenin binding residue[J]. Carlsberg Research Communications, 1988, 53(6): 357-370. DOI:10.1007/BF02983311 |
| [22] |
Metsä-Ketelä M, Halo L, Munukka E, et al. Molecular evolution of aromatic polyketides and comparative sequence analysis of polyketide ketosynthase and 16S ribosomal DNA genes from various Streptomyces species[J]. Applied and Environmental Microbiology, 2002, 68(9): 4472-4479. DOI:10.1128/AEM.68.9.4472-4479.2002 |
| [23] |
Allsop A, Illingworth R. The impact of genomics and related technologies on the search for new antibiotics[J]. Journal of Applied Microbiology, 2002, 92(1): 7-12. |
| [24] |
Witte W. BIOMEDICINE:medical consequences of antibiotic use in agriculture[J]. Science, 1998, 279(5353): 996-997. DOI:10.1126/science.279.5353.996 |
| [25] |
Mardis E R. DNA sequencing technologies:2006-2016[J]. Nature Protocols, 2017, 12(2): 213-218. DOI:10.1038/nprot.2016.182 |
| [26] |
Harvey A L, Edrada-Ebel R, Quinn R J. The re-emergence of natural products for drug discovery in the genomics era[J]. Nature Reviews Drug Discovery, 2015, 14(2): 111-129. DOI:10.1038/nrd4510 |
| [27] |
Kang H S, Brady S F. Arixanthomycins A-C:phylogeny-guided discovery of biologically active eDNA-derived pentangular polyphenols[J]. ACS Chemical Biology, 2014, 9(6): 1267-1272. DOI:10.1021/cb500141b |




