文章信息
- 王钦, 李建林, 唐永凯, 李红霞, 张磊, 沈泽恩, 俞菊华
- WANG Qin, LI Jianlin, TANG Yongkai, LI Hongxia, ZHANG Lei, SHEN Ze'en, YU Juhua
- 鲤鱼PLA2g3a1催化活性区的原核表达及酶活性分析
- Prokaryotic expression and activity analysis of catalytic domain of Cyprinus carpio PLA2g3a1
- 南京农业大学学报, 2020, 43(2): 339-346
- Journal of Nanjing Agricultural University, 2020, 43(2): 339-346.
- http://dx.doi.org/10.7685/jnau.201904011
-
文章历史
- 收稿日期: 2019-04-08
2. 中国水产科学研究院淡水渔业研究中心, 江苏 无锡 214128
2. Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214128, China
磷脂酶A2(phospholipase A2, PLA2)能水解磷脂二位酰基(sn-2)释放脂肪酸和溶血磷脂[1]。根据蛋白序列、对Ca2+的需求、细胞定位和生理功能等特点, 磷脂酶被分为5个亚型:分泌型PLA2(sPLA2)、胞浆型PLA2(cPLA2)、非Ca2+依赖型PLA2(iPLA2)、血小板活化因子乙酰水解酶(PAF-AH)和溶酶体型PLA2(LPLA2)[1-2]。其中, sPLA2是PLA2中最大的一个亚族, 是花生四烯酸、前列腺素及血小板活化因子等生物活性物质生成的限速酶[3]。sPLA2又被分为Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅹ和Ⅻ等11个亚型[4], 与sPLA2亚家族中其他亚型相比, 脊椎动物的Group Ⅲ sPLA2(PLA2g3)的N-和C-端较长, 相对分子质量55×103左右, 具有保守的PLA2蜂毒样功能区(PLA2 bee venom like region, PBVR)。PBVR是磷脂酶催化活性区, 内有10个半胱氨酸构成的5个二硫键起着稳定空间结构作用[1, 4], 及His-Asp构成的催化二联体。
对哺乳动物的研究表明, PLA2g3在多种生理、病理过程中起作用, 包括磷脂代谢[2]、炎症反应[5]、神经系统疾病[6]、细胞增殖[7]和动脉粥样硬化[8]等。在鱼类, 已有科学家从Salmo gairdneri微粒体分离了PLA2并测定了其酶活特性[9-10], 在Pollachius virens[11]肌肉组织分离到了PLA, 并对其酶活特性进行了分析, 但这些都是PLA的混合物。随着基因组挖掘的不断深入, 在斑马鱼、青鳉、鲑鳟鱼类、罗非鱼等鱼类, 均能找到PLA2g3基因。本实验室已对鲤鱼PLA2g3亚型基因进行了比较全面的挖掘, 对鱼类PLA2g3 亚型基因进行了系统进化分析[12]。体外获得具有活性的该基因表达产物, 特别是明确PLA2g3较长的N-和C-端对酶活性的作用特征将为研究基因的作用机制奠定基础[3]。通过构建原核重组表达质粒, 在细菌中诱导重组蛋白表达, 是研究蛋白特性较好的方法。相比真核表达, 原核表达可获得的蛋白量多, 所需时间短、成本低, 但由于细菌缺乏真核蛋白准确折叠、修饰等功能, 在细菌体内获得真核表达的可溶蛋白是主要瓶颈。除了改造宿主菌[13-15]、优化诱导培养条件[16-17]外, 使用合适的标签蛋白是获得可溶蛋白的较常用策略[18]。
为了分析鲤鱼PLA2g3a1催化活性区(PBVR)的酶活特性, 本试验比较了促溶标签蛋白SUMO、TrxA、NusA和MBP对该蛋白的促溶作用, 获得了有活性的原核表达蛋白, 确定了该基因功能区的磷脂酶酶活特性, 为获得其他原核表达活性蛋白提供参考。
1 材料与方法 1.1 载体的构建标签蛋白TrxA、SUMO、NusA和MBP的原核表达质粒pET32a、pCold、pET43.1a和pMAL-c2X均购自杭州研真生物科技有限公司; 选择NcoⅠ和XhoⅠ为插入位点, 根据已有鲤鱼PLA 2g3a1 基因(GenBank登录号KF793833)序列, 经过BLAST比对分析, 确定该基因的催化活性区PBVR, 长423 bp, 编码141个氨基酸。设计扩增该区域的引物, 在正、反向引物的5′端分别添加NcoⅠ和XhoⅠ酶切位点(下划线处)及保护碱基(F:5′-CATGCCATGGGCATGGATGATTCCAGGGAC-3′, R:5′-CCGCTCGAGCACCTCCATGTCTTGGTCTGG-3′)。在使用pMAL-c2X构建原核表达载体时, 在反向引物加上编码6个His的碱基(R1:5′-CCGCTCGAGGTGGTGGTGGTGGTGGTGCACCTCCATGTCTTGGTCTGG-3′)。以本课题组构建的鲤鱼PLA2g3a1 全长cDNA克隆载体为模板, 使用上述引物进行PCR扩增, PCR产物经15 g · L-1琼脂糖电泳, 割胶, 使用DNA胶回收试剂盒(Omega, 美国), 37 ℃酶切3 h, 使用DNA回收试剂盒回收目的DNA后, 用T4 DNA连接酶(TaKaRa, 大连)连接与同样使用双酶切线性化的pET32a、pCold、pET43.1a和pMAL-c2X质粒, 16 ℃连接过夜, 然后转化入E.coli DH5α敏感态细胞(全式金生物技术科技有限公司)中, 涂布于含氨苄青霉素的LB固体平板, 37 ℃培养过夜。挑取单菌落, 使用菌液PCR和双酶切进行阳性检测, 并送金唯智生物科技有限公司测序。测序准确的为构建好的原核重组表达质粒, 分别记为pET32a-PBVR、pCold-PBVR、pET43.1a-PBVR和pMAL-c2X-PBVR。
1.2 表达蛋白的诱导和纯化将上述构建好的重组表达质粒, 转化入大肠杆菌Transetta(DE3)敏感态细胞, 涂布于含氨苄青霉素的LB固体培养基, 37 ℃培养过夜。挑取单菌落于10 mL含氨苄青霉素的LB培养液中, 37 ℃、220 r · min-1培养过夜。次日, 以1 : 100在氨苄青霉素的LB液体培养基中扩大培养, 37 ℃、200 r · min-1培养至D600值为0.5~0.6, 加0.1 mmol · L-1 IPTG继续培养。离心收集细菌, 加入裂解液(50 mmol · L-1 NaH2PO4、300 mmol · L-1 NaCl), 超声裂解细菌后, 取总菌和上清液进行SDS-PAGE, 检测重组蛋白表达情况。
取上清液, 使用镍柱(无锡天演生物科技有限公司)亲和层析分离重组蛋白, 在SCGTM UV-VIS Detector(苏州赛谱仪器有限公司)上纯化, 用含200 mmol · L-1咪唑的裂解液洗脱目的蛋白, 通过SDS-PAGE电泳检测目的蛋白的纯度与质量。纯化后的蛋白使用超滤管(截留相对分子质量1×104的蛋白, Millipore公司)超滤浓缩, 并更换蛋白缓冲液为PBS(pH7.4), 将浓缩后的蛋白与不同质量的BSA蛋白标准品进行电泳, 使用Tanon GIS imagine进行灰度分析, 计算纯化蛋白的质量。
1.3 重组蛋白PBVR的磷酯酶A2活性的测定采用偶联脂氧合酶法[19]测定重组蛋白PBVR的磷脂酶A2(PLA2)活性。以1, 2-二亚油酰基-sn-甘油-3-磷酸胆碱(DL-PC, Sigma)为PLA2的底物, 脂氧合酶为偶联酶(lipoxygenase, Sigma), 磷脂酶水解底物释放出的二亚油酸被脂氧合酶氧化, 产生过氧化氢, 引起A234值变化, 间接获得被磷脂酶水解生成二亚油酸的量, 从而确定磷脂酶活性。以1.3 mmol · L-1 DL-PC为底物, 先溶解在少量氯仿中, 用N2吹干, 然后溶解在10 mmol · L-1脱氧胆酸钠(deoxycholate, Sigma)(脱氧胆酸钠使用50 mmol · L-1 Tris-HCl配制, pH7.5)中, 所得到的底物溶液在25 ℃稳定10 min。反应总体积200 μL, 内含65 μmol · L-1 DL-PC、1.5 μg · mL-1脂氧合酶和2 mmol · L-1脱氧胆酸钠, 加入磷脂酶4 min后测定吸光值A234。每隔2 min测定1次, 共检测10次。以蜂毒磷脂酶A2(bee venom PLA2, Sigma)为阳性对照, 以对应的标签蛋白为阴性对照, 设置3个平行。在一定酶量下, 分别测定22、25、28和30 ℃下的磷脂酶活性, 确定最适温度; 在最适温度下, 比较不同pH值(6.0、7.0、7.5、8.0、8.5、9.0和10.0)的磷脂酶活性, 确定最适pH值; 在最适温度和最适pH值下, 测定在缓冲液中分别添加0.01、0.1、1和2 mmol · L-1 Ca2+的酶促反应速度, 确定MBP-PBVR磷脂酶的Kd值; 在最适温度和最适pH值下, 比较在不同浓度(10.8、16.3、21.7、32.5、65.0、94.5和195.0 μmol · L-1)底物的酶促反应速度, 确定MBP-PBVR的磷脂酶活性的米氏方程Km值。PLA2比活力(U · μmol-1)计算公式如下:

|
式中:t表示反应时间(min); v表示反应体积(L); ε为过氧化氢在A234时的消光系数(25 000 L · mol-1 · cm-1); h表示液体高度(cm); m表示蛋白的量(μmol)。
1.4 数据分析运用SPSS 20.0软件, 对数据进行单因素方差分析(One-way ANOVA)和差异显著性检验。
2 结果与分析 2.1 原核表达质粒的构建和重组蛋白的诱导表达与纯化 2.1.1 重组表达质粒的鉴定以本实验室已构建的鲤鱼PLA2g3a1全长cDNA克隆载体为模板, 使用带不同酶切位点的引物, 扩增PLA2g3a1中长度为423 bp的PBVR, 经双酶切后与同样双酶切的线性质粒pET32a、pCold、pET43.1a、pMAL-c2X连接, 转入DH5α敏感态细胞, 挑选单克隆使用双酶切检测(图 1), 再挑选阳性克隆测序, 获得重组原核表达质粒pET32a-PBVR、pCold-PBVR、pET43.1a-PBVR和pMAL-c2X-PBVR。
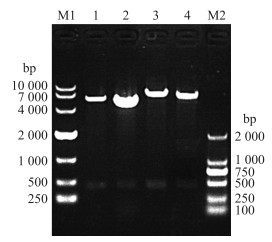
|
图 1 重组表达质粒阳性克隆的筛选 Fig. 1 Screening of positive clones of recombinant expression plasmids M1, M2. DNA marker; 1. pET32a-PBVR; 2. pCold-PBVR; 3. pET43.1a-PBVR; 4. pMAL-c2X-PBVR. |
预试验结果显示, 构建的4个重组质粒在IPTG添加终浓度分别为0.1、0.5和1.0 mmol · L-1时, 重组蛋白表达量无差异, 因此扩大诱导培养时IPTG添加终浓度为0.1 mmol · L-1。比较TrxA-PBVR、NusA-PBVR和MBP-PBVR在20 ℃和25 ℃下诱导的可溶目的蛋白表达量, 发现在诱导培养时间相同条件下, 25 ℃获得的可溶蛋白量多于20 ℃; 在相同浓度IPTG的诱导下, 培养8 h可溶目的蛋白表达量高于2、4和6 h。确定扩大诱导培养温度为25 ℃, 诱导培养时间8~10 h。SUMO-PBVR诱导培养温度设为15 ℃, 诱导培养时间为24 h。
对收集的菌体超声破碎、离心后, 取总菌和上清液分别进行SDS-PAGE电泳(图 2), 灰度分析结果表明:促溶作用从高到低依次为MBP、NusA和TrxA, 上清液目的蛋白占总目的蛋白的比例依次为98%、96%和71%, 而SUMO对PBVR蛋白的促溶作用不明显。但总菌重组蛋白表达量从高到低的标签蛋白为TrxA、SUMO、MBP和NusA, 1 g菌可溶目的蛋白获得量最高的为重组蛋白MBP-PBVR。
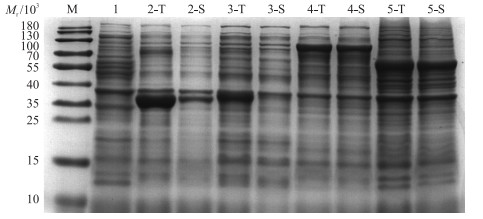
|
图 2 标签蛋白的促溶效果 Fig. 2 Soluble identification of expression proteins M.蛋白标准品; 1.无IPTG总菌蛋白; 2. TrxA-PBVR; 3. SUMO-PBVR; 4. NusA-PBVR; 5. MBP-PBVR; T.总菌蛋白; S.培养上清液。 M. Protein marker; 1. Total bacterial protein without IPTG; 2. TrxA-PBVR; 3. SUMO-PBVR; 4. NusA-PBVR; 5. MBP-PBVR; T. Total protein; S. Supernatant protein. |
经过镍柱亲和层析纯化的蛋白, 浓缩后进行SDS-PAGE电泳检测, 发现存在杂蛋白, 使用与BSA蛋白标准品进行灰度分析、比对, 确定纯化后目的蛋白的质量。结果表明:纯化的MBP-PBVR、NusA-PBVR和TrxA-PBVR中杂蛋白分别为13.6%、38.3%、8.4%(图 3)。最终1 g总菌获得MBP-PBVR、NusA-PBVR和TrxA-PBVR可溶蛋白的质量分别为0.43、0.38和0.21 mg, 蛋白摩尔量分别为7.2、4.9和6.0 nmol。
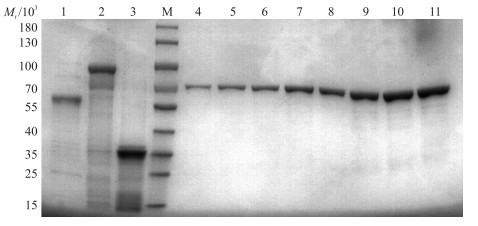
|
图 3 纯化后蛋白质量的测定 Fig. 3 The correction of the purified protein quality M.蛋白标准品; 1. MBP-PBVR; 2. NusA-PBVR; 3. TrxA-PBVR; 4~11.BSA标准品分别为0.250、0.375、0.50、0.75、1.00、1.50、2.00和2.50 μg。 M. Protein marker; 1. MBP-PBVR; 2. NusA-PBVR; 3. TrxA-PBVR; 4-11.0.250, 0.375, 0.50, 0.75, 1.00, 1.50, 2.00 and 2.50 μg BSA, inspectively. |
在25 ℃、pH 7.5条件下测定TrxA-PBVR、NusA- PBVR、MBP-PBVR蛋白的PLA2比活力。结果表明:MBP-PBVR的PLA2比活力最高, 为(13.68±0.12)U · μmol-1, TrxA-PBVR和NusA-PBVR分别是(0.94±0.01)和(0.93±0.04)U · μmol-1(图 4)。
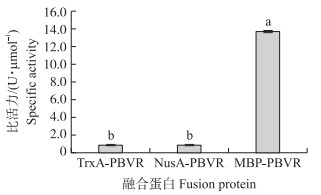
|
图 4 融合蛋白的磷脂酶A2(PLA2)活性 Fig. 4 PLA2 activity of different PBVR fusion proteins |
分别在22、25、28和30 ℃条件下, 测定MBP-PBVR的PLA2比活力。结果表明(图 5):这几个温度下酶活性无显著性差异(P>0.05), 25 ℃时酶活性最高。在25 ℃时, 分别测定不同pH值(6.0、7.0、7.5、8.0、8.5、9.0和10.0)对PLA2酶活性的影响, 发现pH 7.5时酶活性最高。
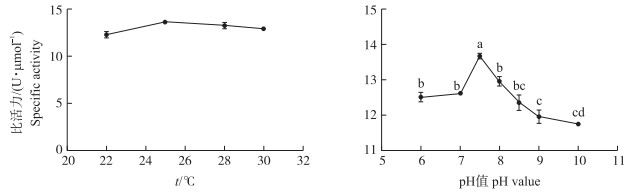
|
图 5 温度和pH值对MBP-PBVR的PLA2活性的影响 Fig. 5 The effect of temperature and pH on the PLA2 activity of MBP-PBVR |
在缓冲液中添加不同浓度的Ca2+, 结果表明:0.01 mmol · L-1 Ca2+条件下, MBP-PBVR的PLA2活性是对照的1.5倍, 在Ca2+浓度为0.1 mmol · L-1时, 酶活性达到最高值, 为对照的2.4倍, Ca2+浓度大于0.1 mmol · L-1时, 酶活性达到平台期, 不再持续提高。说明MBP-PBVR水解磷脂酰胆碱的速率依赖于Ca2+浓度, Kd为15.3 mol · L-1(图 6)。
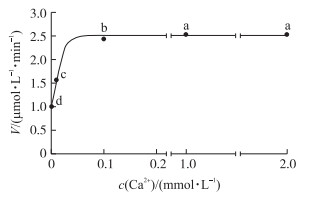
|
图 6 Ca2+浓度对MBP-PBVR PLA2酶促反应速度的影响 Fig. 6 Effect of Ca2+ concentration on PLA2 enzymatic reaction velocity of MBP-PBVR |
在25 ℃、pH 7.5条件下, 测定不同浓度底物时MBP-PBVR酶促反应的速度(V), 得到MBP-PBVR PLA2的米氏方程(曲线)参数:Vm为2.2 μmol · L-1 · min-1, Km为31.9 μmol · L-1, Vm/Km为0.069 min-1(图 7)。
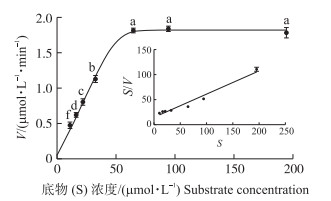
|
图 7 MBP-PBVR的酶活性与底物二亚油酸磷脂酰胆碱浓度的相关性 Fig. 7 Correlation of the PLA2 activity of MBP-PBVR and dilinoleoyl phosphatidylcholine(DL-PC) |
外源蛋白的原核表达系统, 特别是大肠杆菌蛋白表达系统, 具有表达量高、经济、操作简单等优点, 但大部分外源蛋白在大肠杆菌中以不可溶的包涵体形式表达[20], 要获得具有生物活性的蛋白, 必须提高外源蛋白在大肠杆菌中的可溶性表达。试验表明, 在目的蛋白的N-或C-端连接合适的标签蛋白, 把没有完全折叠的目的蛋白包在可溶的胶束状聚集物内[21], 标签蛋白作为伴侣分子[22]或伴侣分子吸附物[23]帮助目的蛋白正确折叠, 使包涵体表达的外源蛋白变为胞质表达的可溶蛋白。常用的促溶标签蛋白有来自大肠杆菌的硫氧还原蛋白TrxA[24]、大肠杆菌NusA[25]、MBP[25-26]以及人源的SUMO[27]与Fh8[28]等。但标签蛋白的促溶效果与目的蛋白的特性有关, 不容易找到对所有蛋白均具有促溶作用的标签蛋白。因此, 通过构建不同标签蛋白的表达载体, 筛选促溶效果好的标签蛋白是获得可溶表达的外源蛋白的较好方法[18]。为了测定鲤鱼PLA2g3a1催化活性区PBVR的酶活性, 本试验构建了标签蛋白为SUMO、TrxA、NusA和MBP的该基因催化活性区的原核表达质粒, 结果表明3个大肠杆菌来源的标签蛋白TrxA、NusA和MBP对该肽段均有促溶作用, 且相对分子质量较大的NusA(55×103)和MBP(43×103)比TrxA(12×103)的促溶作用明显, 和Lee等[29]的试验结果一致, 而人源的SUMO没有明显的促溶效果。
本试验用镍柱对原核表达蛋白进行了纯化, 结果发现纯化蛋白中或多或少存在杂蛋白, 这可能是镍柱纯化蛋白的一个普遍现象[30-32]。杂蛋白的存在使BCA法确定的蛋白质量比实际的高, 为此, 本试验通过比较目的蛋白与标准蛋白电泳条带灰度, 确定目的蛋白浓度和质量。
本试验使用脂氧合酶偶联法测定了原核重组蛋白的磷脂酶活性特征, 结果表明, MBP-PBVR的PLA2酶活性明显比TrxA-PBVR和NusA-PBVR的高, 说明尽管获得的都是可溶蛋白, 但可能蛋白结构存在差异, 导致蛋白活性不一致。Lee等[29]也发现MBP具有保持和稳定重组人肾素综合蛋白(renin binding protein)活性结构, 而NusA、TrxA等则不能。赵志文等[33]研究表明MBP-IBTX(Iberiotoxin)的活性比Trx-IBTX和GST-IBTX的高, 推测MBP标签的助准确折叠功能优于Trx及GST。MBP-PBVR的PLA2比活力为(13.68±0.12)U · μmol-1, 低于蜂毒PLA2的比活力[(30.30±1.03)U · μmol-1]。除了标签蛋白可能影响酶活性外, 鲤鱼PBVR与蜂毒PBVR序列相似性只有60%, 这可能是导致酶活性差异的主要原因。本试验结果表明, 在20~30 ℃时MBP-PBVR的PLA2活性没有明显变化, 很可能磷脂酶最适温度超过了鲤鱼生活温度。据报道猪骨骼肌的磷脂酶最适温度为42 ℃[34]、大西洋鳕鱼的磷脂酶最适温度为40 ℃[35]、蛇毒PLA2(sPLA2)最适温度为50 ℃[36]。MBP-PBVR的PLA2活性最适pH值为7.5, 偏碱性, 与报道的不同物种PLA2的酶活特性相似[3, 37-38]。此外, 本试验结果表明, 与人的PLA2g3 PLA2特性类似, 0.01 mmol · L-1 Ca2+即能明显增加MBP-PBVR的PLA2活性, 但其Kd值(0.015 3 mmol · L-1)大于人PLA2g3的Kd值(0.006 mmol · L-1)[3]。这种差异可能由于鲤鱼和人PLA2g3氨基酸序列差异(氨基酸序列相似性仅为31%)导致的, 也有可能是所使用的酶活性测定方法不同, 人PLA2g3的Kd值测定使用的是荧光分析法[3, 12]。PLA2能水解各种磷脂, 包括磷脂酰胆碱(PC)、磷脂酰乙醇胺(phosphatidylethanolamine, PE)、磷脂酰丝氨酸(phosphatidylserine, PS)、磷脂酰甘油(phosphatidylglycerol, PG)、磷脂酰肌醇(phosphatidylinositol, PI)等。Mansfeld[39]研究表明动植物膜磷脂主要含有PC和PE, 动植物PLA2主要作用底物是PC和PE。Murakami等[40]发现人的PLA2g3对不同种类的PC和PE底物有明显的选择性。Arai等[41]发现三叶黄杨(Polyandrocarpa misakiensis)PLA2重组蛋白首选PC和PE作为底物, 然后是PS和PI; Mazzucotelli等[42]研究表明硬粒小麦(Triticum durum) PLA2对PC有很强的选择性。测定猪胰腺和真鲷盲肠在不同底物(PC、PE、PS、PG)下PLA2的酶活性, 结果表明猪胰腺和真鲷盲肠PLA2酶活性在PG条件下最高[9]。但从猪胰腺和真鲷盲肠组织中提取出的是蛋白混合物, 因此不能确定猪和真鲷group Ⅲ PLA2的最适底物是PG。目前, 关于对底物选择性的报道大多集中在人的PLA2g3[3, 40]。本试验采用偶联脂氧合酶法, 使用相同的底物PC测定PLA2活性, 鲤鱼MBP-PBVR磷脂酶的米氏方程Km值明显比猪PLA2的高[20], 推测DL-PC可能不是其最适底物。PLA2测定方法需要使用含有不饱和双键的磷脂作为底物, 但市面上未见其他不饱和双键的PC, 因此选择DL-PC作为底物。
本试验在鲤鱼肝脏获得高表达PLA 2g3a1 基因的PLA2酶活性区的可溶重组蛋白PBVR, 并测定了该蛋白的酶活特性, 为进一步揭示鲤鱼PLA2g3特有的N-端和C-端序列对该蛋白酶活性的影响, 以及为比较鲤鱼不同PLA2蛋白的酶活特性奠定了基础。
| [1] |
Dennis E A, Cao J, Hsu Y H, et al. Phospholipase A2 enzymes:physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention[J]. Chemical Reviews, 2011, 111(10): 6130-6185. DOI:10.1021/cr200085w |
| [2] |
Murakami M, Taketomi Y, Miki Y, et al. Recent progress in phospholipase A research:from cells to animals to humans[J]. Progress in Lipid Research, 2011, 50(2): 152-192. DOI:10.1016/j.plipres.2010.12.001 |
| [3] |
Valentin E, Ghomashchi F, Gelb M H, et al. Novel human secreted phospholipase A2 with homology to the group Ⅲ bee venom enzyme[J]. Journal of Biological Chemistry, 2000, 275(11): 7492-7496. DOI:10.1074/jbc.275.11.7492 |
| [4] |
Hariprasad G, Srinivasan A, Singh R. Structural and phylogenetic basis for the classification of group Ⅲ phospholipase A2[J]. Journal of Molecular Modeling, 2013, 19(9): 3779-3791. DOI:10.1007/s00894-013-1913-x |
| [5] |
Sato H, Kato R, Isogai Y, et al. Analyses of group Ⅲ secreted phospholipase A2 transgenic mice reveal potential participation of this enzyme in plasma lipoprotein modification, macrophage foam cell formation, and atherosclerosis[J]. Journal of Biological Chemistry, 2008, 283(48): 33483-33497. DOI:10.1074/jbc.M804628200 |
| [6] |
Masuda S, Yamamoto K, Hirabayashi T, et al. Human group Ⅲ secreted phospholipase A2 promotes neuronal outgrowth and survival[J]. Biochemical Journal, 2008, 409(2): 429-438. DOI:10.1042/BJ20070844 |
| [7] |
Murakami M, Masuda S, Shimbara S, et al. Cellular distribution, post-translational modification, and tumorigenic potential of human group Ⅲ secreted phospholipase A2[J]. Journal of Biological Chemistry, 2005, 280(26): 24987-24998. DOI:10.1074/jbc.M502088200 |
| [8] |
Kimura M M, Ishikawa Y, Komiyama K, et al. Expression of secretory phospholipase A2s in human atherosclerosis development[J]. Atherosclerosis, 2008, 196(1): 81-91. |
| [9] |
Iijima N, Chosa S, Uematsu K, et al. Purification and characterization of phospholipase A2 from the pyloric caeca of red sea bream, Pagrus major[J]. Fish Physiology & Biochemistry, 1997, 16(6): 487-498. |
| [10] |
Tocher D R, Bendiksen E Å, Campbell P J, et al. The role of phospholipids in nutrition and metabolism of teleost fish[J]. Aquaculture, 2008, 280(1/2/3/4): 21-34. |
| [11] |
Audley M A, Shetty K J, Kinsella J E. Isolation and properties of phospholipase a from pollock muscle[J]. Journal of Food Science, 1978, 43(6): 1771-1775. DOI:10.1111/j.1365-2621.1978.tb07410.x |
| [12] |
徐迪辉, 李红霞, 俞菊华, 等. 鲤sPLA2-Ⅲ家族基因结构、系统发育和表达特征[J]. 水生生物学报, 2019, 43(6): 1172-1181. Xu D H, Li H X, Yu J H, et al. Structure, phylogeny and expression of sPLA2-Ⅲ gene family in Cyprinus carpio[J]. Acta Hydrobiologica Sinica, 2019, 43(6): 1172-1181 (in Chinese with English abstract). |
| [13] |
Premkumar L, Bageshwar U K, Gokhman I, et al. An unusual halotolerant α-type carbonic anhydrase from the alga Dunaliella salina functionally expressed in Escherichia coli[J]. Protein Expression & Purification, 2003, 28(1): 151-157. |
| [14] |
Lehmann K, Hoffmann S, Neudecker P, et al. High-yield expression in Escherichia coli, purification, and characterization of properly folded major peanut allergen Arah2[J]. Protein Expression and Purification, 2003, 31(2): 250-259. DOI:10.1016/S1046-5928(03)00190-6 |
| [15] |
Chen Y, Song J, Sui S F, et al. Dnak and DnaJ facilitated the folding process and reduced inclusion body formation of magnesium transporter CorA overexpressed in Escherichia coli[J]. Protein Expr Purif, 2013, 32(2): 221-231. |
| [16] |
Sim J, Sim T S. Amino acid substitutions affecting protein solubility:high level expression of Streptomyces clavuligerus isopenicillin N synthase in Escherichia coli[J]. Journal of Molecular Catalysis B Enzymatic, 1999, 6(3): 133-143. DOI:10.1016/S1381-1177(98)00072-1 |
| [17] |
Sørensen H P, Mortensen K K. Soluble expression of recombinant proteins in the cytoplasm of Escherichia coli[J]. Microbial Cell Factories, 2005, 4(1): 1-8. DOI:10.1186/1475-2859-4-1 |
| [18] |
Esposito D, Chatterjee D K. Enhancement of soluble protein expression through the use of fusion tags[J]. Curr Opin Biotechnol, 2006, 17(4): 353-358. DOI:10.1016/j.copbio.2006.06.003 |
| [19] |
Cavallaro A S, Mahony D, Commins M, et al. Endotoxin-free purification for the isolation of bovine viral diarrhoea virus E2 protein from insoluble inclusion body aggregates[J]. Microbial Cell Factories, 2011, 10(1): 57. DOI:10.1186/1475-2859-10-57 |
| [20] |
Jiménez M, Cabanes J, Gandía H F, et al. A continuous spectrophotometric assay for phospholipase A2 activity[J]. Analytical Biochemistry, 2003, 319(1): 131-137. |
| [21] |
Nominé Y, Ristriani T, Laurent C, et al. A strategy for optimizing the monodispersity of fusion proteins:application to purification of recombinant HPV E6 on coprotein[J]. Protein Engineering, 2001, 14(4): 297-305. DOI:10.1093/protein/14.4.297 |
| [22] |
Mamipour M, Yousefi M, Hasanzadeh M. An overview on molecular chaperones enhancing solubility of expressed recombinant proteins with correct folding[J]. International Journal of Biological Macromolecules, 2017, 102: 367-375. DOI:10.1016/j.ijbiomac.2017.04.025 |
| [23] |
Fox J D, Kapust R B, Waugh D S. Single amino acid substitutions on the surface of Escherichia coli maltose-binding protein can have a profound impact on the solubility of fusion proteins[J]. Protein Science, 2001, 10(3): 622-630. DOI:10.1110/ps.45201 |
| [24] |
Yasukawa T, Kaneiishii C, Maekawa T, et al. Increase of solubility of foreign proteins in Escherichia coli by coproduction of the bacterial thioredoxin[J]. Journal of Biological Chemistry, 1995, 270(43): 25328-25331. DOI:10.1074/jbc.270.43.25328 |
| [25] |
De M V, Stier G, Blandin S, et al. The solubility and stability of recombinant proteins are increased by their fusion to NusA[J]. Biochemical & Biophysical Research Communications, 2004, 322(3): 766-771. |
| [26] |
Nallamsetty S, Waugh D S. Solubility-enhancing proteins MBP and NusA play a passive role in the folding of their fusion partners[J]. Protein Expression & Purification, 2006, 45(1): 175-182. |
| [27] |
Marblestone J G, Edavettal S C, Lim Y, et al. Comparison of SUMO fusion technology with traditional gene fusion systems:enhanced expression and solubility with SUMO[J]. Protein Science, 2006, 15(1): 182-189. |
| [28] |
Costa S, Almeida A, Castro A, et al. Fusion tags for protein solubility, purification and immunogenicity in Escherichia coli:the novel Fh8 system[J]. Frontiers in Microbiology, 2014, 5(63): 1-20. |
| [29] |
Lee C, Lee S G, Takahashi S, et al. The soluble expression of the human renin binding protein using fusion partners:a comparison of ubiquitin, thioredoxin, maltose binding protein and NusA[J]. Biotechnology & Bioprocess Engineering, 2003, 8(2): 89-93. |
| [30] |
Gu J, Stephenson C G, Iadarola M J. Recombinant proteins attached to a nickel-NTA column:use in affinity purification of antibodies[J]. Bio Techniques, 1994, 17(2): 257-262. |
| [31] |
Joanne Crowe B S M, Ribbe J. One-step purification of recombinant proteins with the 6xHis tag and Ni-NTA resin[J]. Molecular Biotechnology, 1995, 4(3): 247-258. DOI:10.1007/BF02779018 |
| [32] |
Block H, Maertens B, Spriestersbach A, et al. Reprint of immobilized-metal affinity chromatography(IMAC):a review[J]. Protein Expression & Purification, 2009, 463(9): 439-473. |
| [33] |
赵志文, 张峥, 刘浩文, 等. 不同标签辅助的IBTX(Iberiotoxin)原核表达纯化及活性鉴定[J]. 现代生物医学进展, 2014, 14(7): 1207-1211. Zhao Z W, Zhang Z, Liu H W, et al. The expression, purification and identification of IBTX in E.coli with different tags[J]. Progress in Modern Biomedicine, 2014, 14(7): 1207-1211 (in Chinese with English abstract). |
| [34] |
周光宏, 徐幸莲. 应用响应曲面法研究金华火腿生产过程中磷脂酶的变化[J]. 食品与发酵工业, 2005, 31(2): 120-123. Zhou G H, Xu X L. Study on time-related changes of phospholipase during processing of Jinhua ham using response surface method[J]. Food and Fermentation Industries, 2005, 31(2): 120-123 (in Chinese with English abstract). DOI:10.3321/j.issn:0253-990X.2005.02.032 |
| [35] |
Aaen B, Jessen F, Jensen B. Partial purification and characterization of a cellular acidic phospholipase A2 from cod(Gadus morhua) muscle[J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 1995, 110(3): 547-554. DOI:10.1016/0305-0491(94)00185-W |
| [36] |
Nair B C, Nair C, Elliott W B. Temperature stability of phospholipase A activity:Ⅱ. variations in optimum temperature of phospholipases A2 from various snake venoms[J]. Toxicon, 1976, 14(1): 43-47. DOI:10.1016/0041-0101(76)90118-5 |
| [37] |
Morales R, Ramírez H S, Feliu V. Adaptive control based on fast online algebraic identification and GPI control for magnetic levitation systems with time-varying input gain[J]. International Journal of Control, 2014, 87(8): 3077-3097. |
| [38] |
Ono H, Iijima N. Purification and characterization of phospholipase A2 isoforms from the hepatopancreas of red sea bream, Pagrus major[J]. Fish Physiology & Biochemistry, 1998, 18(2): 135-147. |
| [39] |
Mansfeld J. Plant phospholipases A2:perspectives on biotechnological applications[J]. Biotechnology Letters, 2009, 31(9): 1373-1380. DOI:10.1007/s10529-009-0034-1 |
| [40] |
Murakami M, Masuda S, Shimbara S, et al. Cellular arachidonate-releasing function of novel classes of secretory phospholipase A2s(Groups Ⅲ and Ⅻ)[J]. Journal of Biological Chemistry, 2003, 278(12): 10657-10667. DOI:10.1074/jbc.M211325200 |
| [41] |
Arai K, Yoshida S, Nakatani M, et al. Phospholipids and their derivatives as mitogen and motogen of budding tunicates[J]. The Journal of Biochemistry, 2004, 135(1): 71-78. DOI:10.1093/jb/mvh008 |
| [42] |
Mazzucotelli E, Trono D. Cloning, expression analysis, and functional characterization of two secretory phospholipases A2 in durum wheat(Triticum durum Desf.)[J]. Plant Science, 2015, 241: 295-306. DOI:10.1016/j.plantsci.2015.10.009 |




