文章信息
- 王敏, 戴伟民, 强胜, 宋小玲
- WANG Min, DAI Weimin, QIANG Sheng, SONG Xiaoling
- 转基因水稻抗性基因漂移频率检测的抽样技术研究
- Research on sampling technique for verifying gene flow frequency of transgenic rice
- 南京农业大学学报, 2020, 43(2): 260-266
- Journal of Nanjing Agricultural University, 2020, 43(2): 260-266.
- http://dx.doi.org/10.7685/jnau.201904024
-
文章历史
- 收稿日期: 2019-04-13
水稻(Oryza sativa L.)是世界重要粮食作物之一, 在我国粮食生产中占有重要地位, 所以转基因水稻的培育一直备受关注[1]。目前已经培育出了抗虫、抗除草剂、抗病害、抗干旱、抗盐碱以及提高稻米品质的转基因水稻种质资源, 部分转基因水稻品种已进入环境释放阶段[2-3]。然而转基因水稻的抗性基因可通过花粉漂移到非转基因栽培水稻、普通野生稻(O. rufipogon Griff)和杂草稻(Oryza sativa f. spontanea)中, 产生携带抗性基因的杂交后代, 因此转基因水稻的环境释放具有潜在的生态风险。
尽管在自然条件下稻属的栽培水稻、普通野生稻以及水稻同属同种的杂草稻主要通过自花授粉繁殖后代, 但栽培水稻能接受其他品种栽培水稻的花粉, 普通野生稻及杂草稻也能够不同程度地接受栽培水稻的花粉。研究发现:转基因水稻通过花粉介导向非转基因水稻的抗性基因漂移频率为0.019%~0.919%[4-15], 最大漂移距离为19 m[8]; 如果花粉受体是不育系水稻, 则抗性基因漂移频率会提高至3.145%~46.250%[16-18]。转基因水稻向普通野生稻的抗性基因漂移频率为3.550%~18.000%[19-20]; 向杂草稻的抗性基因漂移频率为0.011%~0.666%[4-5, 12, 14], 最大漂移距离为7 m[14]。
转基因水稻的抗性基因漂移频率的检测均需要从收获的花粉受体种子中进行抽样。在检测标准《转基因植物及其产品环境安全检测》中, 只规定了每种花粉受体材料种子的检测量低于3 000粒时需要全部进行检测, 但对于收获种子数大于3 000粒时, 需要检测的花粉受体种子量并没有明确规定。
空间分布型决定了抽样技术[21-22], 因此转基因水稻向非转基因水稻、普通野生稻和杂草稻发生抗性基因漂移后形成的杂交种在花粉受体种子中的空间分布型决定了检测抗性基因漂移频率的抽样技术。种群空间分布型一般可分为随机分布、均匀分布和集群分布, 分别对应的抽样方法为简单随机抽样、系统抽样、分层抽样[21-24]。首先需要弄清转基因水稻向花粉受体发生基因漂移后产生的杂交种在花粉受体种子中的空间分布型; 其次抽样量的多少对抽样结果影响很大, 检测全部的花粉受体种子能够准确反映收获花粉受体种子中含有抗性基因的杂交种数量, 这对收获的种子数量较少时是可行的, 但当收获的种子数量过大时, 将所有的种子全部检测就会花费大量的人力、物力和时间。因此需要研究不同抗性基因漂移频率下的最佳抽样量, 以达到既能反映田间实际发生的抗性基因漂移频率, 又能省时省力。
为给检测转基因水稻的抗性基因漂移频率提供科学的抽样技术, 包括抽样方法和最佳检测量, 本文模拟研究转基因水稻向非转基因水稻、普通野生稻和杂草稻基因漂移后的杂交种混杂在花粉受体种子中的空间分布型, 以分布型作为基础, 确定合适的抽样方法, 并研究不同抗性基因漂移频率下的最佳抽样量, 为转基因水稻抗性基因漂移频率的检测提供科学依据。
1 材料与方法 1.1 材料常规水稻(Oryza sativa L.)‘明恢63’种子代表花粉受体, 从‘明恢63’中挑选大小一致的水稻种子用红墨水染色, 代表转基因水稻向花粉受体发生抗性基因漂移后形成的杂交种。
1.2 模拟转基因水稻抗性基因漂移后杂交种在花粉受体种子中的空间分布型按照红墨水染色水稻种子与非红墨水染色水稻种子混杂比(质量比)为2.5%、1.25%、0.8%、0.625%、0.5%、0.4%、0.3%、0.2%、0.1%、0.05%、0.025%, 混杂比是指模拟的转基因水稻抗性基因漂移频率, 抗性基因漂移频率=含抗性基因种子数/检测种子总数×100%。各混杂比的总量均为10 kg, 模拟转基因水稻抗性基因漂移后产生的杂交种经过收获、运输和储藏等环节混杂在花粉受体种子中, 将混合水稻种子翻转30次后, 抽样, 样本量占总量的20%, 即2 kg。将抽取的水稻种子摊开铺平桌面上50格内, 每格长×宽为10 cm×10 cm, 统计每格内染色水稻种子数量。每个混杂比均设置4个重复, 采用聚集度指标(即平均拥挤度与平均密度之比、方差均值比、Morisita指数检验、χ2检验)检测模拟不同抗性基因漂移频率下杂交种的空间分布型。
平均拥挤度与平均密度之比:m*/m。式中:m为平均密度(即为每格中红色水稻种子粒数的平均值); m*为平均拥挤度, 是指每个个体在1个样方中的平均邻居数(表示种群密度)[25], m*=m+(S2/m-1), S2为方差(即为各区中红色水稻粒数的方差)。当m*/m < 1时为均匀分布; 当m*/m=1时为随机分布; 当m*/m>1时为聚集分布[26]。
方差均值比:
Morisita指数检验:
χ2检验:指通过检验不同个体样方频率的观测值和Poisson分布的预测值之间的差异性来检测空间分布型。即: χ2=(预测值-实际观测值)2/预测值; 自由度=组数-2。假设:实际观测值符合Poisson分布, 并给出概率P在0.05处的卡方值(预测值)。预测值高于实际观测值趋近于聚集分布, 小于实际观测值则趋近于随机分布[27]。
1.3 抽样技术研究将红墨水染色水稻种子与非红墨水染色水稻种子按照0.025%、0.05%、0.1%、0.2%、0.3%、0.4%、0.5%(质量比)混合后, 将混合水稻种子翻转30次, 模拟转基因水稻抗性基因漂移后产生的杂交种经过收获、运输和储藏等环节混杂在花粉受体种子中。根据测量的该水稻种子千粒质量(26.112 3±0.082 0)g, 随机从总量10 kg水稻种子中, 分别称取5 000、10 000、20 000、30 000、40 000、50 000粒种子的质量, 并统计染色水稻种子数量, 重复16次, 然后以染色水稻种子与非染色水稻种子模拟的混杂比和抽样得到的混杂比的偏离系数小于0.05为标准, 确定不同混杂比下的最佳抽样量。
偏离系数:
由表 1可知:在供试的11个染色水稻种子和非染色水稻种子混杂比例(即0.025%~2.5%)下, 平均拥挤度与平均密度之比(m*/m)为0.985~1.004, 均近似于1。根据m*/m=1为随机分布的原则, 可以判断染色水稻种子在非染色水稻种子中的空间分布型为随机分布。方差均值比(V/X)为0.951~1.201, Morisita指数(Iδ值)为0.948~1.004, 均近似于1。根据V/X=1和Iδ=1为随机分布的原则, 证明染色水稻种子在非染色水稻种子中的空间分布型为随机分布。另外, 卡方检测的实际观测值为46.067~57.398, 均小于预测值65.171。根据卡方检验的原则也可知染色水稻种子在非染色水稻种子中为随机分布。综上种群空间分布型聚集度指标, 可以推断转基因水稻抗性基因漂移后形成的杂交种混杂在收获的花粉受体种子中的空间分布型为随机分布。
| 混杂比/% Mixed ratio |
平均拥挤度与平均密度之比(m*/m) Ratio of average crowdingto average density |
方差均值比(V/X) Variance to mean ratio |
Morisita指数(Iδ) Morisita index |
χ2检验 χ2 test |
|
| 实际观测值 Actual observed value |
预测值 Predicted value |
||||
| 0.025 | 0.995 | 0.998 | 0.948 | 48.712 | 65.171 |
| 0.05 | 0.985 | 0.988 | 0.958 | 48.415 | 65.171 |
| 0.1 | 0.997 | 0.964 | 0.965 | 47.243 | 65.171 |
| 0.2 | 0.989 | 0.965 | 0.983 | 47.301 | 65.171 |
| 0.3 | 0.993 | 0.969 | 0.989 | 47.505 | 65.171 |
| 0.4 | 0.993 | 0.951 | 0.990 | 46.601 | 65.171 |
| 0.5 | 1.001 | 1.005 | 0.998 | 49.413 | 65.171 |
| 0.625 | 1.001 | 1.007 | 0.999 | 49.332 | 65.171 |
| 0.8 | 0.995 | 0.940 | 0.994 | 46.067 | 65.171 |
| 1.25 | 0.997 | 0.941 | 0.995 | 46.093 | 65.171 |
| 2.5 | 1.004 | 1.201 | 1.004 | 57.398 | 65.171 |
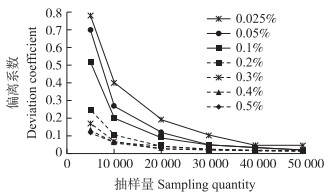
从表 2和图 1可知:在染色水稻种子和非染色水稻种子不同混杂比下, 随着抽样量的增加, 实际混杂比和抽样得到的混杂比的偏离系数变小, 抽样得到的混杂比也越接近实际混杂比。在实际混杂比为0.025%, 抽样量为5 000~30 000粒时, 抽样得到的混杂比为0.021 3%~0.024 8%, 与实际混杂比的偏离系数为0.100 8~0.799 0, 均大于0.05;抽样量为40 000~50 000粒时, 抽样得到的混杂比为0.024 5%~0.025 4%, 与实际混杂比的偏离系数为0.044 4~0.044 8, 均小于0.05, 因此推断转基因水稻抗性基因漂移频率为0.025%时, 最少需要抽取40 000粒种子才能检测出在田间发生的漂移频率。
| 实际混杂比/% Actualmixedratio |
抽样量Sampling size | ||||||||||||||||
| 5 000 | 10 000 | 20 000 | 30 000 | 40 000 | 50 000 | ||||||||||||
| 抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
抽样得到的混杂比/% Mixed ratioobtainedby sampling |
偏离系数 Deviationcoefficient |
||||||
| 0.025 | 0.021 3 | 0.779 0 | 0.022 5 | 0.400 8 | 0.024 7 | 0.190 3 | 0.024 8 | 0.100 8 | 0.024 5 | 0.044 8 | 0.025 4 | 0.044 4 | |||||
| 0.05 | 0.036 3 | 0.699 5 | 0.050 6 | 0.269 9 | 0.047 5 | 0.120 0 | 0.051 3 | 0.049 0 | 0.049 7 | 0.037 7 | 0.050 0 | 0.020 1 | |||||
| 0.1 | 0.086 3 | 0.519 3 | 0.010 0 | 0.204 0 | 0.094 1 | 0.089 2 | 0.099 8 | 0.048 0 | 0.099 5 | 0.033 0 | 0.098 3 | 0.005 9 | |||||
| 0.2 | 0.226 3 | 0.247 8 | 0.203 8 | 0.104 3 | 0.200 6 | 0.043 3 | 0.198 3 | 0.025 3 | 0.200 8 | 0.019 5 | 0.200 5 | 0.017 4 | |||||
| 0.3 | 0.315 0 | 0.169 6 | 0.306 9 | 0.067 3 | 0.298 8 | 0.040 5 | 0.301 9 | 0.017 2 | 0.301 1 | 0.012 9 | 0.300 6 | 0.012 9 | |||||
| 0.4 | 0.403 8 | 0.133 8 | 0.404 4 | 0.063 4 | 0.402 2 | 0.028 8 | 0.399 2 | 0.017 8 | 0.397 2 | 0.012 6 | 0.400 5 | 0.011 9 | |||||
| 0.5 | 0.472 5 | 0.115 3 | 0.470 6 | 0.061 8 | 0.488 1 | 0.026 9 | 0.493 1 | 0.020 8 | 0.503 3 | 0.012 7 | 0.500 4 | 0.007 2 | |||||
|
注:实际混杂比表示染色水稻数量在非染色水稻中所占的比例。 Note:Actual mixed ratio means proportion of dyed rice seed number in non-stained rice seed number. |
|||||||||||||||||
|
图 1 红墨水染色水稻种子与非染色水稻种子在不同混杂比时各抽样量下的偏离系数 Fig. 1 Deviation coefficient of red-ink dyed rice seed innon-dyed rice seed with different mixed ratiosunder different sampling size |
在染色水稻种子和非染色水稻种子混杂比为0.05%~0.1%, 抽样量为5 000~20 000粒时, 抽样得到的混杂比与实际混杂比的偏离系数为0.089 2~0.699 5, 均大于0.05;抽样量为30 000~50 000粒时, 抽样得到的混杂比与实际混杂比的偏离系数为0.005 9~0.049 0, 均小于0.05, 因此推断转基因水稻抗性基因漂移频率为0.05%~0.1%时, 最少需要抽取30 000粒种子才能检测出在田间发生的漂移频率。
在染色水稻种子和非染色水稻种子实际混杂比为0.2%~0.5%, 抽样量为5 000~10 000粒时, 抽样得到的混杂比与实际混杂比的偏离系数为0.061 8~0.247 8, 均大于0.05;抽样量为20 000~50 000粒, 抽样得到的混杂比与实际混杂比的偏离系数为0.007 2~0.043 3, 均小于0.05, 可以推断转基因水稻抗性基因漂移频率为0.2%~0.5%时, 最少需要抽取20 000粒种子才能检测出在田间发生的漂移频率。
3 讨论转基因水稻向花粉受体, 包括非转基因水稻、普通野生稻以及杂草稻的抗性基因漂移频率的检测方法主要有生物学检测和分子检测[28]。收获的种子萌发后根据筛选标记的生化特性确认水稻幼苗是否含有抗性基因, 如抗除草剂性状, 可以采用叶面喷洒除草剂或种子用一定浓度除草剂溶液浸泡的方法进行检测[29-30]。分子生物学方法需要检测花粉受体种子中是否含有特异的基因或蛋白。转基因水稻向非转基因栽培稻、普通野生稻及杂草稻发生抗性基因漂移后, 采用何种抽样方法以及抽取多少量进行检测才能最科学地反映转基因水稻在田间实际发生的基因漂移频率是值得研究的问题。
不同的空间分布型应该采用不同的抽样方法, 因此转基因水稻抗性基因漂移后形成的杂交种混杂在收获的花粉受体种子中的空间分布型是决定抽样技术的关键因子[21-22]。在本试验设置的11个模拟抗性基因漂移频率下, 转基因水稻抗性基因漂移后形成的杂交种混杂在收获的花粉受体种子中的空间分布型均近似于随机分布, 因此应采用随机抽样方法对收获的花粉受体种子进行抽样, 用于检测抗性基因漂移频率。由于本试验模拟花粉受体收获和装卸运输过程是将种子进行了多次混合, 因此在实际检测转基因水稻抗性基因漂移频率时也应对收获的花粉受体种子充分混合后再进行抽样检测。张炎等[27]模拟转基因大豆与常规大豆混杂比为1:40、1:60、1:80时, 转基因大豆混杂在常规大豆中的分布型均属于随机分布, 其结果与本试验结果一致。
除抽样方法外, 抽样检测量的大小对试验结果的准确性有重要影响。抽样检测量越大, 越能反应田间真实发生的基因漂移率, 但检测量过大, 也会花费更多的人力、物力和时间。本文在模拟不同抗性基因漂移频率下的抽样技术研究结果表明, 随着抽样量的增加, 偏离系数越来越小, 当模拟漂移频率为0.025%时检测40 000粒种子, 0.05%与0.1%时检测30 000粒种子, 0.2%、0.3%和0.5%时检测20 000粒种子, 其偏离系数均小于0.05。根据以上研究结果, 建议可先检测20 000粒花粉受体种子, 若得到的漂移频率大于0.2%, 则不需要增加花粉受体种子检测量; 若得到的漂移频率小于0.2%, 则需再检测10 000粒花粉受体种子, 这时若得到的漂移频率大于0.05%, 则不需要增加花粉受体种子检测量; 若漂移频率小于0.05%, 还需再检测10 000粒花粉受体种子。本研究结果可以为转基因水稻抗性基因漂移频率检测中的抽样量提供参考, 也可以为完善我国目前转基因水稻外源基因漂移的检测标准提供依据。
很多研究者在检测转基因水稻的抗性基因漂移频率时均是从收获的种子中随机抽取一部分种子进行抗性筛选[5, 11, 13]。在转基因水稻向杂草稻的抗性基因漂移相关研究中, 黄鹞等[5]随机抽取10 000粒种子进行抗性筛选; Chen[11]随机抽取了3 000粒种子进行抗性筛选; Sun等[13]随机抽取10 000~20 000粒种子进行抗性筛选。以上研究采用的是随机抽样方法, 与本文得到的用于研究转基因水稻抗性基因漂移频率检测的抽样方法一致。以上研究得到的抗性基因漂移频率为0.1%~0.3%[5, 11, 13]。在本研究中, 当漂移频率为0.1%~0.3%时, 最少需要检测的花粉受体种子数为20 000粒。由于这些文献[5, 11, 13]中抽取的种子数量少于20 000粒, 得到的抗性漂移频率并不能准确反映田间实际发生的转基因水稻向杂草稻的抗性基因漂移情况, 也不能正确评估该转基因水稻田间释放所带来的潜在风险。
部分研究者将收获的花粉受体种子全部进行抗性检测[4, 14], 虽然能够准确反映真实的抗性基因漂移频率, 但检测种子量过大, 所需成本高、时间长。崔荣荣等[4]在研究转基因水稻Ⅱ优86B向泰州杂草稻的抗性基因漂移频率时, 将收获的超过100 000粒的花粉受体种子全部进行检测, 得到的抗性基因漂移频率为0.136%。Zuo等[14]在研究抗草铵膦转基因水稻Y0003向杂草稻WRZJ003的抗性基因漂移频率时, 将收获的近100 000粒种子全部检测, 得到的漂移频率为0.131%。在本研究中, 当漂移频率为0.1%~0.2%时, 检测30 000粒种子就能够准确反映田间实际发生的抗性基因漂移频率。
多数文献报道表明转基因水稻向栽培稻的抗性基因漂移频率为0.019%~0.919%[4-15], 转基因水稻向杂草稻的抗性基因漂移频率为0.011%~0.666%[4-5, 12, 14], 转基因水稻向普通野生稻和不育系水稻的抗性基因漂移频率分别为3.550%~18.000%[19-20]和3.145%~46.250%[16-18]。本研究中, 当转基因水稻的抗性基因漂移频率为0.025%~0.5%可以采用本文建议的抽样策略, 即可先检测20 000粒花粉受体种子, 根据得到的抗性基因漂移频率确定是否需要增加检测量。如果抗性基因漂移频率不在本研究设定的范围内, 即低于0.025%和高于0.5%时, 还需要研究杂交种在花粉受体种子中的空间分布格局和最佳检测种子量。我们预测当抗性基因漂移频率高于0.5%时, 检测20 000粒花粉受体种子可以得到田间实际发生的真实抗性基因漂移频率。
本研究结果是在室内模拟的条件下得出的, 因此该结果尚需要通过转基因水稻抗性基因漂移频率的实际检测数据进行验证。
| [1] |
岳玉峰. 我国水稻常规育种居世界领先地位的形成与展望[J]. 北方水稻, 2014, 44(1): 1-4. Yue Y F. Formation and prospect of world leading on conventional breeding in our country[J]. North Rice, 2014, 44(1): 1-4 (in Chinese). DOI:10.3969/j.issn.1673-6737.2014.01.002 |
| [2] |
朱祯. 转基因水稻研发进展[J]. 中国农业科技导报, 2010, 12(2): 9-16. Zhu Z. Progress in research and development of transgenic rice[J]. Journal of Agricultural Science and Technology, 2010, 12(2): 9-16 (in Chinese with English abstract). DOI:10.3969/j.issn.1008-0864.2010.02.03 |
| [3] |
李黎红, 叶卫军, 郭龙彪. 我国转基因水稻研究进展和商业化前景分析[J]. 中国稻米, 2012, 18(6): 1-4. Li L H, Ye W J, Guo L B. Research progress and commercialization prospects of transgenic rice in China[J]. China Rice, 2012, 18(6): 1-4 (in Chinese). DOI:10.3969/j.issn.1006-8082.2012.06.001 |
| [4] |
崔荣荣, 戴伟民, 强胜, 等. 抗草铵膦转基因杂交水稻Ⅱ优86B及其恢复系86B向杂草稻基因漂移的研究[J]. 江苏农业学报, 2013, 29(4): 708-714. Cui R R, Dai W M, Qiang S, et al. Gene flow from transgenic glufosinate-resistant rice Ⅱ You 86B and its restorer line 86B to weedy rice[J]. Jiangsu Journal of Agricultural Sciences, 2013, 29(4): 708-714 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-4440.2013.04.003 |
| [5] |
黄鹞, 李继坤, 强胜, 等. 复合性状转基因水稻T1c-19向受体水稻及杂草稻的基因漂移[J]. 应用与环境生物学报, 2015, 21(6): 1112-1119. Huang Y, Li J K, Qiang S, et al. Gene flow from transgenic rice T1c-19 with stacked cry1C*/bar genes to weedy and cultivated rice species[J]. Chinese Journal of Applied and Environmental Biology, 2015, 21(6): 1112-1119 (in Chinese with English abstract). |
| [6] |
Messeguer J, Marfà V, Català M M, et al. A field study of pollen-mediated gene flow from Mediterranean GM rice to conventional rice and the red rice weed[J]. Molecular Breeding, 2004, 13(1): 103-112. |
| [7] |
Zhang W Q, Linscombe S D, Webster E, et al. Risk assessment of the transfer of imazethapyr herbicide tolerance from Clearfield rice to red rice(Oryza sativa)[J]. Euphytica, 2006, 152(1): 75-86. DOI:10.1007/s10681-006-9180-x |
| [8] |
Yuan Q H, Shi L, Wang F, et al. Investigation of rice transgene flow in compass sectors by using male sterile line as a pollen detector[J]. Theoretical and Applied Genetics, 2007, 115(4): 549-560. DOI:10.1007/s00122-007-0588-z |
| [9] |
Shivrain V K, Burgos N R, Gealy D R, et al. Maximum outcrossing rate and genetic compatibility between red rice(Oryza sativa) biotypes and ClearfieldTM rice[J]. Weed Science, 2008, 56(6): 807-813. DOI:10.1614/WS-08-026.1 |
| [10] |
Zhang W Q, Linscombe S D, Oard J H. Genetic and agronomic analyses of red rice-Clearfield hybrids and their progeny produced from natural and controlled crosses[J]. Euphytica, 2008, 164(3): 659-668. DOI:10.1007/s10681-008-9661-1 |
| [11] |
Chen L J. Gene flow from cultivated rice(Oryza sativa) to its weedy and wild relatives[J]. Annals of Botany, 2004, 93(1): 67-73. DOI:10.1093/aob/mch006 |
| [12] |
Chun Y J, Kim D I, Park K W, et al. Gene flow from herbicide-tolerant GM rice and the heterosis of GM rice-weed F2 progeny[J]. Planta, 2011, 233(4): 807-815. DOI:10.1007/s00425-010-1339-y |
| [13] |
Sun G H, Dai W M, Cui R R, et al. Gene flow from glufosinate-resistant transgenic hybrid rice Xiang 125S/Bar68-1 to weedy rice and cultivated rice under different experimental designs[J]. Euphytica, 2015, 204(1): 211-227. DOI:10.1007/s10681-015-1370-y |
| [14] |
Zuo J, Zhang L J, Song X L, et al. Innate factors causing differences in gene flow frequency from transgenic rice to different weedy rice biotypes[J]. Pest Management Science, 2011, 67(6): 677-690. DOI:10.1002/ps.2108 |
| [15] |
Han S M, Lee B, Won O J, et al. Gene flow from herbicide resistant genetically modified rice to conventional riceu(Oryza sativa L.) cultivars[J]. Journal of Ecology and Environment, 2015, 38(4): 397-403. DOI:10.5141/ecoenv.2015.042 |
| [16] |
Jia S R, Wang F, Shi L, et al. Transgene flow to hybrid rice and its male-sterile lines[J]. Transgenic Research, 2007, 16(4): 491-501. DOI:10.1007/s11248-006-9037-z |
| [17] |
贾士荣, 袁潜华, 王丰, 等. 转基因水稻基因飘流研究十年回顾[J]. 中国农业科学, 2014, 47(1): 1-10. Jia S R, Yuan Q H, Wang F, et al. What we have learnt in ten years' study of rice transgene flow[J]. Scientia Agricultura Sinica, 2014, 47(1): 1-10 (in Chinese with English abstract). DOI:10.3864/j.issn.0578-1752.2014.01.001 |
| [18] |
姜大刚, 梁奕涛, 黄靖, 等. 转基因水稻外源基因的漂移研究[J]. 生物技术通报, 2010(6): 95-99. Jiang D G, Liang Y T, Huang J, et al. Study on gene flow of transgenic rice[J]. Biotechnology Bulletin, 2010(6): 95-99 (in Chinese with English abstract). |
| [19] |
余昌花.转bar基因水稻向普通野生稻的基因漂流及与开花习性的关系[D].海口: 海南大学, 2011. Yu C H. Relationship between flowering characteristics and gene flow frequency from bar transgenic rice to common wild rice[D]. Haikou: Hainan University, 2011(in Chinese with English abstract). |
| [20] |
Wang F, Yuan Q H, Shi L, et al. A large-scale field study of transgene flow from cultivated rice(Oryza sativa) to common wild rice(O. rufipogon) and barnyard grass(Echinochloa crusgalli)[J]. Plant Biotechnology Journal, 2006, 4(6): 667-676. DOI:10.1111/j.1467-7652.2006.00210.x |
| [21] |
夏基康. 昆虫种群空间分布型与抽样调查[J]. 南京农业大学学报, 1982, 5(1): 42-49. Xia J K. Spatial distribution pattern and sampling investigation of insect population[J]. Journal of Nanjing Agricultural University, 1982, 5(1): 42-49 (in Chinese with English abstract). DOI:10.7685/j.issn.1000-2030.1982.01.006 |
| [22] |
余泽高, 汤百高, 顾正清, 等. 农作物三种基本抽样调查方法的比较[J]. 种子, 2006, 25(1): 57-59. Yu Z G, Tang B G, Gu Z Q, et al. Comparison of three basic sampling methods for crops[J]. Seed, 2006, 25(1): 57-59 (in Chinese). |
| [23] |
李海涛. 植物种群分布格局研究概况[J]. 植物学通报, 1995, 30(2): 19-26. Li H T. Introduction to studies of the pattern of plant population[J]. Chinese Bulletin of Botany, 1995, 30(2): 19-26 (in Chinese with English abstract). |
| [24] |
张文馨.钉螺空间分布格局和抽样方法优化研究[D].南昌: 江西师范大学, 2015. Zhang W X. An study on spatial distribution pattern and sampling optimization of oncomlania snail[D]. Nanchang: Jiangxi Normal University, 2015(in Chinese with English abstract). |
| [25] |
周洪华, 陈亚宁, 李卫红. 塔里木河下游绿洲-荒漠过渡带植物多样性特征及优势种群分布格局[J]. 中国沙漠, 2009, 29(4): 688-696. Zhou H H, Chen Y N, Li W H. Species diversity and dominance population distribution pattern in oasis-desert ecotone[J]. Journal of Desert Research, 2009, 29(4): 688-696 (in Chinese with English abstract). |
| [26] |
张传根. 稻茬油菜田稻槎菜的空间分布型及抽样技术[J]. 中国植保导刊, 2017, 37(9): 40-43. Zhang C G. Spatial distribution patterns and sampling techniques of Lapsana apogonoides Maxim. in rapeseed field with rice stubble[J]. China Plant Protection, 2017, 37(9): 40-43 (in Chinese). DOI:10.3969/j.issn.1672-6820.2017.09.007 |
| [27] |
张炎, 强胜, 宋小玲. 模拟转基因大豆混杂在常规大豆中的空间分布格局研究[J]. 杂草学报, 2010, 28(4): 32-34. Zhang Y, Qiang S, Song X L. Study on spatial distribution pattern of simulated transgenic soybean mixed in conventional soybean[J]. Journal of Weed Science, 2010, 28(4): 32-34 (in Chinese). DOI:10.3969/j.issn.1003-935X.2010.04.009 |
| [28] |
王莹, 袁英. 抗除草剂转基因玉米的快速鉴定方法[J]. 江苏农业科学, 2014, 42(4): 54-56. Wang Y, Yuan Y. Rapid identification of herbicide-resistant transgenic maize[J]. Jiangsu Agricultural Sciences, 2014, 42(4): 54-56 (in Chinese with English abstract). DOI:10.3969/j.issn.1002-1302.2014.04.020 |
| [29] |
Rong J, Lu B R, Song Z P, et al. Dramatic reduction of crop-to-crop gene flow within a short distance from transgenic rice fields[J]. New Phytologist, 2007, 173(2): 346-353. DOI:10.1111/j.1469-8137.2006.01906.x |
| [30] |
陆永良, 彭于发, 王渭霞, 等. 抗除草剂转基因水稻基因漂移至常规栽培稻的频率研究初报[J]. 中国水稻科学, 2010, 24(6): 663-666. Lu Y L, Peng Y F, Wang W X, et al. Frequency of gene flow from herbicide-resistant transgenic rice to conventional rice varieties[J]. Chinese Journal of Rice Science, 2010, 24(6): 663-666 (in Chinese with English abstract). |




