文章信息
- 夏磊, 王团团, 段莉莉, 李季, 陈劲枫
- XIA Lei, WANG Tuantuan, DUAN Lili, LI Ji, CHEN Jinfeng
- 黄瓜亚硝酸还原酶基因(CsNiR)的克隆及对外植体分化的影响
- Cloning of nitrite reductase gene(CsNiR) in cucumber and its effect on explant differentiation
- 南京农业大学学报, 2020, 43(2): 231-237
- Journal of Nanjing Agricultural University, 2020, 43(2): 231-237.
- http://dx.doi.org/10.7685/jnau.201906021
-
文章历史
- 收稿日期: 2019-06-14
随着黄瓜基因组测序的完成与植物基因工程的快速发展, 转基因技术成为黄瓜品种改良的重要方法之一[1-2]。高效的离体再生体系能够显著提高黄瓜遗传转化效率[3-5]。目前, 黄瓜离体再生培养研究主要集中在基因型、苗龄、外植体、激素等方面, 关于其分子机制鲜有研究[6-8]。Wang等[9]通过QTL定位与GWAS关联分析, 预测了1个与黄瓜离体再生相关的基因Csa1G642540, 且过表达该基因能显著提高外植体分化率。
氮是植物生长发育必需的营养元素, 植物吸收的主要氮源为硝态氮和铵态氮, 不同植物对不同氮源的吸收具有偏好性[10]。不同氮源对植物营养元素的吸收、根系发育、光合生理、内源激素的调节等植物生长发育过程具有重要影响[11]。植物体内氮的同化主要受到硝酸还原酶(NR)和亚硝酸还原酶(NiR)的调控, NR降解NO3-为NO2-, NiR催化NO2-还原为NH4+[12]。研究发现, NiR基因与植物再生过程密切相关。Nishimura等[13-14]发现控制水稻再生主效基因为亚硝酸还原酶编码基因(OsNiR), 人工过量表达OsNiR可以显著提高水稻再生能力, 并建立了一个高效的水稻遗传转化体系。研究发现, 小麦TaNiR通过促进其愈伤组织的光合代谢, 从而提高其再生效率[15]。杜光玲等[16]对玉米不同类型愈伤组织分化中硝态氮代谢的差异进行分析, 发现高活性的NiR可以诱导产生优良愈伤组织, 从而提高植株再生效率。在香蕉中, 也发现NiR基因可能是影响外植体再生的正调控因子[17]。虽然NiR基因对植物外植体分化过程具有重要影响, 但目前在黄瓜中尚未见报道。
本试验克隆了黄瓜亚硝酸还原酶基因(CsNiR), 研究其在黄瓜各个组织器官和亚细胞层面的表达特征, 并探讨CsNIR对黄瓜外植体分化的影响, 旨在为进一步优化黄瓜遗传转化再生体系提供一定的参考。
1 材料与方法 1.1 试验材料选取6个不同基因型黄瓜自交系作为试验材料:CCMC(华北型)、XTMC(华北型)、Amir(欧洲温室型)、‘二早子’(华南型)、NJMC(美国生态型)、7011A(加工型), 由南京农业大学葫芦科作物遗传与种质创新重点实验室创制和保存, 在南京农业大学白马基地温室和本实验室的组织培养室进行种植培养。
外植体培养:将黄瓜种子在温水中浸泡2~3 h, 然后将种子用无水乙醇表面灭菌1 min, 并将2%次氯酸钠表面灭菌15~20 min, 然后用无菌水冲洗3遍。将消毒好的种子种在MS培养基中[4.43 g·L-1 MS粉+30 g·L-1蔗糖+2.8 g·L-1结冷胶, pH(5.8±0.03)], 28 ℃黑暗条件下萌发3~6 d。待子叶直立刚刚展开, 却未完全展开时, 纵向切割每个子叶, 剔除生长点, 获得2个外植体。将来自不同基因型的外植体平铺在MS培养基、MS+培养基(MS培养基+1.5 mg·L-1 6-BA)及不同氮素比例培养基, 并转移至26 ℃的正常培养条件, 16 h光照/8 h黑暗, 光照度为2 000 lx。4个不同氮素比例的培养基基本成分包括NH4Cl、KNO3、铁盐等营养成分。其中, 铵盐浓度分别为10、20、30、40 mmol·L-1, 硝酸盐浓度分别为50、40、30、20 mmol·L-1, 即NH4+/NO3-比例分别为1:5、1:2、1:1、2:1, pH值平均为5.8±0.03。
1.2 CsNiR基因的克隆利用RNA simple total RNA Kit(北京Tiangen公司)提取CCMC叶片总RNA, 利用Prime Script RT Reagent Kit(大连TaKaRa公司)将RNA反转录成cDNA。通过黄瓜全基因组数据库(http://cucurbitgenomics.org)检索NiR基因序列, 获得黄瓜CsNiR基因全长序列。利用Primer Premier 5.0软件获得CsNiR基因正、反向引物(表 1)。利用Prime STARⓇ GXL DNA Polymerase(大连TaKaRa公司)扩增目标基因片段, 对PCR产物进行切胶回收, 连接T载体, 委托南京金斯瑞生物科技有限公司测序。
| 引物名称 Primer name |
引物序列 Primer sequence(5′→3′) |
用途 Usage |
| CsNiR-PCR-F/R | TTTCTACATCCAAGCACACTAGCTATC/ TTTGCCCTTTTTCTTCACTAATCAG |
CsNiR基因克隆 Cloning of CsNiR gene |
| Csa2M301530.1-qPCR-F/R | ATTGTTCTCAGTGGTGGTTCTAC/ CCTTTGAGATCCACATCTGCT |
RT-qPCR中内参基因引物 Primer of reference gene for RT-qPCR |
| CsNiR-qPCR-F/R | GAGGACATCGTCCCTCTCTG/ AAATCCTCCGACGATGCTCT |
RT-qPCR中CsNiR特异性引物 Special primer of CsNiR for RT-qPCR |
| CsNiR-C-F/R | ATCCCCATGTCCTCCTCTT/ CCCATCCTCTTCTTCCTCCT |
亚细胞定位 Subcellular localization |
利用DNAMAN 6.0软件获得CsNiR基因编码的氨基酸序列; 通过InterProScan sequence search(http://www.ebi.ac.uk/interpro/search/sequence-search)进行结构域预测; 利用Prosite软件(http://prosite.expasy.org/)找到酶活位点; 利用NCBI数据库BLAST工具, 获得不同物种CsNiR基因及氨基酸序列; 利用MEGA 7.0软件构建系统进化树; 利用SignalP 4.1软件进行信号肽分析预测; 利用ProtComp v.9.0、PSORT和TargetP1.1软件对CsNiR蛋白亚细胞定位在线预测。
1.4 CsNiR的亚细胞定位将回收的目的片段与经SmaⅠ酶切后的载体pGreen0029进行T4连接, 构建融合表达载体pGreen0029-CsNiR∷GFP。表达载体和空载体通过注射法转化本氏烟草表皮细胞, 遮光培养72 h后, 用激光共聚焦显微镜观察细胞内GFP激发的绿色荧光信号和叶绿体激发的红色荧光信号。
1.5 CsNiR的荧光定量PCR分别取黄瓜CCMC的根、茎、成熟叶、幼叶、雄花、雌花、果实、茎尖、侧芽作为样品。其中, 当植株长至18~20节位时, 取成熟叶(中部第5、6片叶)、幼叶(顶部第2、3片叶)、果实(雌花开后3~4 d); 外植体取子叶节再生区域, 即MS和MS+培养基中培养0、7、14 d时不同基因型黄瓜子叶节外植体, 每个样品3个重复。以上述样品的cDNA为模板, 使用Real-Time PCR检测系统(Bio-rad, CFX96, USA)进行实时定量PCR反应。选用黄瓜看家基因Csa2M301530.1作为内参基因[18]。根据扩增的CsNiR序列设计定量PCR引物, 见表 1。实时定量PCR按照SYBR Premix Ex Taq试剂盒(大连TaKaRa公司)说明书进行。20 μL反应体系:cDNA 1.0 μL, 正、反向荧光定量引物各0.5 μL, SYBR Green Ⅰ mix 10 μL和ddH2O 8 μL。重复3次。使用Excel 2007软件分析不同组织中基因表达水平, 通过公式2-ΔΔCT计算基因相对表达量[19]。
1.6 黄瓜亚硝酸还原酶活性及子叶节再生能力的测定根据Nishimura等[13]提供的方法, 测定黄瓜子叶节再生区域内NiR的活性。将不同基因型黄瓜的子叶节外植体平铺在不同培养基中, 培养14 d左右。对分化出丛芽的外植体进行统计, 每次统计50个外植体, 3次重复。分化率=分化芽的外植体数/外植体总数×100%。
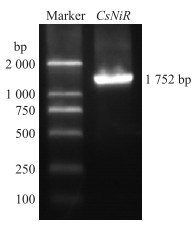
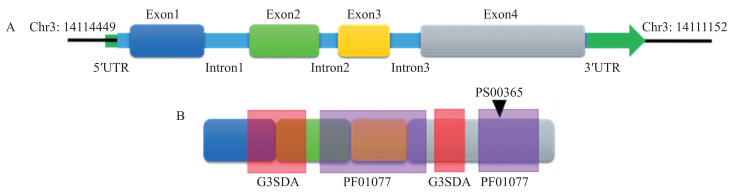
2 结果与分析 2.1 黄瓜CsNiR基因的克隆及序列分析以黄瓜CCMC叶片cDNA为模板, 用引物CsNiR-PCR-F/R进行PCR扩增, 获得1 752 bp扩增片段(图 1), 测序结果与黄瓜基因组数据中NiR基因(CsaV3_3G018610)序列一致。CsNiR基因的结构如图 2所示, 具有4个外显子和3个内含子, 编码583个氨基酸。通过InterProScan sequence search进行结构域预测, 发现CsNiR具有完整的NiR蛋白结构, 含血红素蛋白β-化合物区域(G3DSA)、4Fe-4S区域(PF0177), 属于NiR超级家族(图 2)。利用Prosite软件对黄瓜亚硝酸还原酶活性位点进行分析, 黄瓜NiR序列区域的499~515的TGCPNTCGQVQVADIGF为CsNiR蛋白的主要活性位点(PSOO365)。
|
图 1 黄瓜CsNiR基因克隆电泳图 Fig. 1 The electrophoresis diagram ofthe clone of CsNiR in cucumber |
|
图 2 黄瓜CsNiR的基因(A)和蛋白结构(B) Fig. 2 Genomic(A)and protein structure(B)of CsNiR in cucumber Exon:外显子; Intron:内含子; G3SDA:血红素蛋白β-化合物Heme protein β-compound; PF0177:4Fe-4S. |
将DNAMAN 6.0软件翻译获得的CsNiR编码的氨基酸序列在NCBI蛋白数据库中比对, 发现该蛋白与甜瓜、印度南瓜、西瓜等18个物种NiR蛋白有76%~96%的相似性, 其中与甜瓜的蛋白相似性最高为96%。系统进化树分析结果(图 3)显示, 这些蛋白序列聚为2类, 一类为黄瓜、烟草和拟南芥等双子叶植物, 一类为水稻、小麦等单子叶植物, 且葫芦科作物中NiR高度保守。

|
图 3 黄瓜与其他植物CsNiR蛋白系统进化树分析 Fig. 3 The phylogenetic tree analysis of CsNiR in cucumber and other plants |
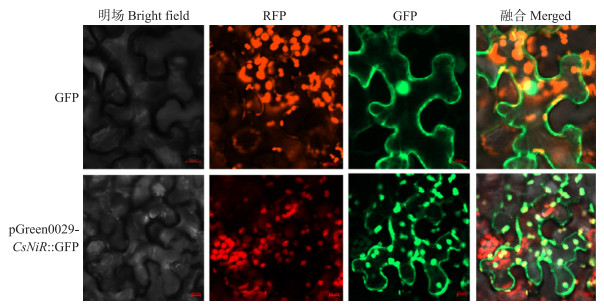
利用SignalP 4.1软件进行信号肽预测, 结果显示CsNiR没有明显的信号肽序列。利用ProtComp v.9.0、PSORT和TargetP1.1软件对CsNiR蛋白进行亚细胞定位预测, 结果发现其定位于叶绿体的得分最高, 分别为9.28、0.762、0.863。共聚焦显微镜观察结果(图 4)显示, 融合载体pGreen0029-CsNiR∷GFP在细胞膜中表达, 在细胞质中沿膜成点状分布, 与叶绿体荧光完全重叠, 表明CsNiR蛋白定位于细胞膜和叶绿体上。
|
图 4 CsNiR蛋白的亚细胞定位 Fig. 4 Subcellular localization analysis of CsNiR protein |
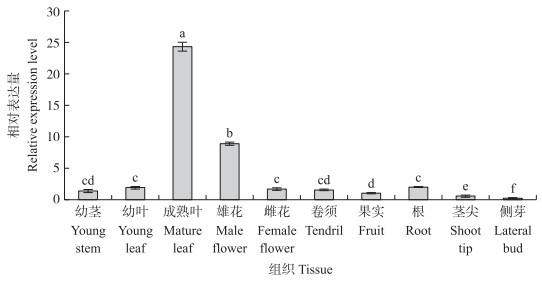
如图 5所示:CsNiR在成熟叶中的表达量最高, 雄花中次之, 而在茎尖和侧芽的表达量最低, 说明叶片是CsNiR参与NO2-催化反应的主要场所。
|
图 5 黄瓜CsNiR基因在不同组织中的表达量 Fig. 5 The expression levels of CsNiR gene in different tissues of cucumber 不同小写字母表示差异显著(P < 0.05)。下同。 Different small letters indicate significant difference at 0.05 level. The same as follows. |
由图 6可见:在Amir中NiR活性最低, 分化率最高, 而‘二早子’中NiR活性最高, 分化率最低。进一步对NiR活性和外植体分化率进行相关性分析表明, 两者呈显著负相关, 相关系数为-0.876。
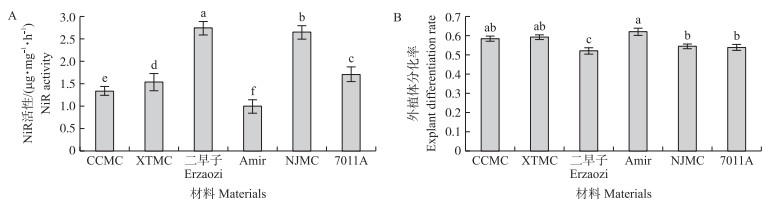
|
图 6 不同基因型黄瓜子叶节的NiR活性和外植体分化率 Fig. 6 NiR activity and explant differentiation rate of cotyledon nodes in different genotypes cucumber |
由图 7可见:不同基因型黄瓜外植体分化过程中, 在含有外源激素的MS+培养基上外植体分化能力显著提高。在MS+培养基上外植体再生区域内CsNiR基因表达量显著低于MS培养基, 表明外源激素的添加可以提高外植体分化率, 同时降低CsNiR基因的表达量。
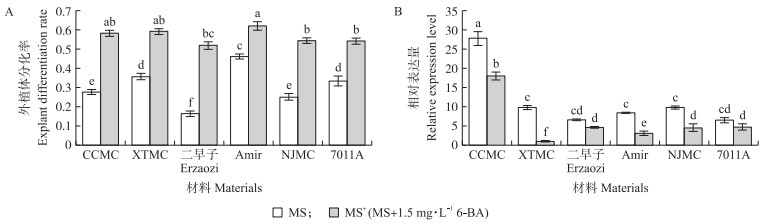
|
图 7 不同处理条件下黄瓜外植体分化率及CsNiR基因的相对表达量 Fig. 7 Differentiation rate of explants and relative expression level of CsNiR genein cucumber under different treatment conditions |
在不同氮素比例的分化培养基上, 外植体分化率随NH4+/NO3-的增加而下降。即当NH4+/NO3-分别为1:5、1:2、1:1、2:1时, 外植体平均分化率分别为0.355 2、0.337 0、0.255 5和0.132 5。差异显著性比较结果显示, NH4+/NO3-为1:5时外植体分化率极显著高于2:1(P < 0.01)。因此, 降低NH4+/NO3-比例有助于外植体分化。
3 讨论本试验以黄瓜CCMC为材料, 克隆得到了亚硝酸还原酶基因CsNiR, 其具有完整的血红素蛋白β-化合物区域、4Fe-4S区域结构[20]。系统进化树分析发现植物NiR高度保守, 黄瓜与甜瓜氨基酸序列相似性高达96%, 与水稻、烟草等物种的NiR蛋白相似性均大于78%。但同时发现NiR蛋白在进化过程中分化为单子叶和双子叶两个方向的分支, 这有可能是单子叶植物和双子叶植物对不同氮源偏好性差异的原因之一[10]。植物亚硝酸还原酶的主要催化反应场所在叶绿体中。本研究亚细胞定位结果显示黄瓜NiR蛋白存在于细胞膜和叶绿体中, 这与Mitra等[21]研究发现NiR蛋白定位在叶片的叶绿体、非绿色组织的质体及细胞膜中的结果相符合。荧光定量PCR结果显示在成熟叶片中CsNiR的表达量最高, 这间接说明黄瓜亚硝酸盐代谢的场所主要分布于叶片中。
研究发现NiR与植物组培再生活力密切相关, 在水稻和小麦中, 提高NiR转录水平显著提高了其外植体分化效率[13-15]。但本研究却发现在茎尖、侧芽等细胞增殖分化旺盛的组织中CsNiR基因表达量最低, 不同基因型黄瓜NiR活性与外植体再生率呈显著负相关, 这与在水稻中的研究结果恰好相反[13]。硝态氮有利于器官的分化, 而铵态氮有利于细胞的生长[22], 合适的NH4+/NO3-比例有助于植物外植体再生。本研究发现外植体分化率随NH4+/NO3-的增加而下降, 这与在小麦[23]、草莓[24]上的研究结果一致。植物激素参与植物体内氮循环调控, 如细胞分裂素诱导小麦中硝酸还原酶的形成[25]; 桑树谷氨酸脱氢酶基因的表达受到细胞分裂素的调控[26]; 水杨酸通过促进细胞分裂素的形成, 提高水稻幼苗硝酸还原酶活性[27]。我们研究发现, 在黄瓜中添加外源细胞分裂素对外植体分化具有促进作用, 但也显著抑制了CsNiR的表达。因此, 我们推测NiR与细胞分裂素可能存在拮抗作用。黄瓜CsNiR基因如何调控外植体分化活力, 是否通过参与黄瓜内源细胞分裂素的合成, 或者与其他植物激素相关基因存在互作, 仍需进一步研究。
| [1] |
Huang S W, Li R Q, Zhang Z H, et al. The genome of the cucumber, Cucumis sativus L.[J]. Nature Genetics, 2009, 41: 1275-1281. DOI:10.1038/ng.475 |
| [2] |
Qi J J, Liu X, Shen D, et al. A genomic variation map provides insights into the genetic basis of cucumber domestication and diversity[J]. Nature Genetics, 2013, 45(12): 1510-1515. DOI:10.1038/ng.2801 |
| [3] |
Kose E, Koç N K. Agrobacterium-mediated transformation of cucumber(Cucumis sativus L.) and plant regeneration[J]. Biotechnology & Biotechnological Equipment, 2003, 17(2): 56-62. DOI:10.1080/13102818.2003.10817059 |
| [4] |
王艳蓉, 陈丽梅, 潘俊松, 等. 黄瓜子叶高效再生体系的建立与遗传转化[J]. 上海交通大学学报(农业科学版), 2006, 24(2): 152-156, 164. Wang Y R, Chen L M, Pan J S, et al. Establishment of high effective regeneration system in cucumber(Cucumis sativus L.) and Agrobacterium tumefaciens mediated genetic transformation[J]. Journal of Shanghai Jiao Tong University(Agricultural Science), 2006, 24(2): 152-156, 164 (in Chinese with English abstract). DOI:10.3969/j.issn.1671-9964.2006.02.008 |
| [5] |
Zhang Z X, Li X, Ma S, et al. A protocol for Agrobacterium-mediated transformation of cucumber(Cucumis sativus L.) from cotyledon explants[J/OL]. Protocol Exchange, 2017. DOI: 10.1038/protex.2017.107.
|
| [6] |
侯爱菊, 朱延明, 杨爱馥, 等. 诱导黄瓜直接器官发生主要影响因素的研究[J]. 园艺学报, 2003, 30(1): 101-103. Hou A J, Zhu Y M, Yang A F, et al. Main factors influencing the frequency of direct organogenesis of cucumber in vitro[J]. Acta Horticulturae Sinica, 2003, 30(1): 101-103 (in Chinese with English abstract). DOI:10.3321/j.issn:0513-353X.2003.01.027 |
| [7] |
Shukla P S, Das A K, Jha B, et al. High-frequency in vitro shoot regeneration in Cucumis sativus by inhibition of endogenous auxin[J]. In Vitro Cellular & Developmental Biology-Plant, 2014, 50(6): 729-737. |
| [8] |
唐莉, 周晏起, 王毅, 等. 黄瓜离体再生影响因素研究进展[J]. 农业科技通讯, 2017(7): 232-236. Tang L, Zhou Y Q, Wang Y, et al. Research progress on influencing factors of cucumber in vitro regeneration[J]. Bulletin of Agricultural Science and Technology, 2017(7): 232-236 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-6400.2017.07.078 |
| [9] |
Wang Y, Zhou Q, Zhu G T, et al. Genetic analysis and identification of a candidate gene associated with in vitro regeneration ability of cucumber[J]. Theoretical and Applied Genetics, 2018, 131(12): 2663-2675. DOI:10.1007/s00122-018-3182-7 |
| [10] |
Patterson K, Cakmak T, Cooper A, et al. Distinct signalling pathways and transcriptome response signatures differentiate ammonium- and nitrate-supplied plants[J]. Plant, Cell & Environment, 2010, 33(9): 1486-1501. |
| [11] |
邢瑶, 马兴华. 氮素形态对植物生长影响的研究进展[J]. 中国农业科技导报, 2015, 17(2): 109-117. Xing Y, Ma X H. Research progress on effect of nitrogen form on plant growth[J]. Journal of Agricultural Science and Technology, 2015, 17(2): 109-117 (in Chinese with English abstract). |
| [12] |
Miflin B J, Lea P J. The pathway of nitrogen assimilation in plants[J]. Phytochemistry, 1976, 15(6): 873-885. DOI:10.1016/S0031-9422(00)84362-9 |
| [13] |
Nishimura A, Ashikari M, Lin S, et al. Isolation of a rice regeneration quantitative trait loci gene and its application to transformation systems[J]. Proc Natl Acad Sci USA, 2005, 102(33): 11940-11944. DOI:10.1073/pnas.0504220102 |
| [14] |
Nishimura A, Aichi I, Matsuoka M. A protocol for Agrobacterium-mediated transformation in rice[J]. Nature Protocols, 2006, 1(6): 2796-2802. DOI:10.1038/nprot.2006.469 |
| [15] |
佘茂云, 陈朵朵, 冯晨, 等. 小麦亚硝酸还原酶基因及调控序列克隆、定位和表达分析[J]. 作物学报, 2011, 37(1): 28-39. She M Y, Chen D D, Feng C, et al. Isolation, chromosome assignment, and expression assay of nitrite reductase gene and regulatory sequence in wheat[J]. Acta Agronomica Sinica, 2011, 37(1): 28-39 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-2561.2011.01.006 |
| [16] |
杜光玲, 秦文娟, 邓士政, 等. 玉米不同类型愈伤组织分化中硝态氮代谢的差异分析[J]. 河南农业科学, 2010, 39(6): 18-21. Du G L, Qin W J, Deng S Z, et al. The variation analysis of nitrate-nitrogen metabolism of different types of maize callus in the process of callus differentiation[J]. Journal of Henan Agricultural Sciences, 2010, 39(6): 18-21 (in Chinese with English abstract). DOI:10.3969/j.issn.1004-3268.2010.06.005 |
| [17] |
毛宇源.福建香蕉离体培养条件的优化及其再生能力研究[D].福州: 福建农林大学, 2016. Mao Y Y. Study on the optimization of the in vitro culture conditions and the regeneration capacity of some Fujian banana germplasm resources in Fujian[D]. Fuzhou: Fujian Agriculture and Forestry University, 2016(in Chinese with English abstract). |
| [18] |
Wan H J, Zhao Z G, Qian C T, et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry, 2010, 399(2): 257-261. DOI:10.1016/j.ab.2009.12.008 |
| [19] |
Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research, 2001, 29(9): e45. DOI:10.1093/nar/29.9.e45 |
| [20] |
Swamy U, Wang M T, Tripathy J N, et al. Structure of spinach nitrite reductase:implications for multi-Electron reactions by the iron-sulfur:siroheme cofactor[J]. Biochemistry, 2005, 44(49): 16054-16063. DOI:10.1021/bi050981y |
| [21] |
Mitra S K, Gantt J A, Ruby J F, et al. Membrane proteomic analysis of Arabidopsis thaliana using alternative solubilization techniques[J]. Journal of Proteome Research, 2007, 6(5): 1933-1950. DOI:10.1021/pr060525b |
| [22] |
Hinnen M G J, Pierik R L M, Bronsema F B F. The influence of macronutrients and some other factors on growth of Phalaenopsis hybrid seedlings in vitro[J]. Scientia Horticulturae, 1989, 41(1/2): 105-116. DOI:10.1016/0304-4238(89)90054-X |
| [23] |
王海波, 魏景芳, 葛亚新, 等. 小麦愈伤组织状态调控与原生质体培养[J]. 中国农业科学, 1996, 29(6): 8-14. Wang H B, Wei J F, Ge Y X, et al. Regulation of callus status and culture of protoplast in wheat[J]. Scientia Agricutura Sinica, 1996, 29(6): 8-14 (in Chinese with English abstract). DOI:10.3321/j.issn:0578-1752.1996.06.002 |
| [24] |
张洁.草莓(Fragaria ananassa Duch.)悬浮细胞培养与植株再生的研究[D].保定: 河北农业大学, 2002. Zhang J. The studies on suspension cell culture and plant regeneration in strawberry[D]. Baoding: Hebei Agricultural University, 2002(in Chinese with English abstract). |
| [25] |
陆嘉陵, 简志英, 汤玉玮. 细胞分裂素促进小麦硝酸还原酶的诱导[J]. 植物学报(英文版), 1989, 31(2): 123-127. Lu J L, Jian Z Y, Tang Y W. The accelerative effects of cytokinin on nitrate reductase induction[J]. Journal of Integrative Plant Biology, 1989, 31(2): 123-127 (in Chinese with English abstract). |
| [26] |
植爽, 任艳红, 唐星, 等. 桑树谷氨酸脱氢酶基因MaGDHs的克隆及表达分析[J]. 中国农业科学, 2018, 51(4): 758-769. Zhi S, Ren Y H, Tang X, et al. Molecular cloning and expression analysis of genes MaGDHs encoding glutamate dehydrogenase in mulberry[J]. Scientia Agricultura Sinica, 2018, 51(4): 758-769 (in Chinese with English abstract). |
| [27] |
高夕全, 刘爱荣, 叶梅荣, 等. 水杨酸对水稻幼苗硝酸还原酶活性和根系生长的影响[J]. 安徽农业技术师范学院学报, 2000, 14(1): 13-15. Gao X Q, Liu A R, Ye M R, et al. Effects of salicylic acid on roots growth and NRA in rice[J]. Journal of Anhui Agrotechnical Teachers College, 2000, 14(1): 13-15 (in Chinese with English abstract). |




