文章信息
- 朱静, 岳思宁, 陈琛, 宋书琪, 连玲丹, 赵明文
- ZHU Jing, YUE Sining, CHEN Chen, SONG Shuqi, LIAN Lingdan, ZHAO Mingwen
- 谷氨酸合酶在灵芝中生物学功能的研究
- Study on the biological function of glutamate synthase in Ganoderma lucidum
- 南京农业大学学报, 2019, 42(6): 1073-1079
- Journal of Nanjing Agricultural University, 2019, 42(6): 1073-1079.
- http://dx.doi.org/10.7685/jnau.201907058
-
文章历史
- 收稿日期: 2019-07-27
氮源是参与合成蛋白、核酸以及其他含氮化合物的重要元素之一, 其不仅能够影响真菌的生长发育, 还能调控真菌的次级代谢和侵染能力等[1-3]。谷氨酸和谷氨酰胺作为细胞中其他含氮化合物的前体, 其含量可以作为衡量胞内氮源水平的标志。谷氨酰胺可以为合成碱基提供氮元素。谷氨酸可以在转氨酶的催化下合成大多数氨基酸, 也可以通过谷氨酸脱氢酶分解生成α-酮戊二酸, 为TCA循环提供中间产物。此外谷氨酸也是合成胞内重要还原物质谷胱甘肽(glutathione, GSH)的重要底物, GSH能参与维持胞内氧化还原水平[4]。
真菌中谷氨酸的合成有2条途径:一种是在谷氨酸脱氢酶(gutamate dehydrogenase, GDH)的催化下, 将铵盐和α-酮戊二酸合成为谷氨酸, 但是这个反应是可逆的; 另一种是谷氨酰胺和α-酮戊二酸在谷氨酸合酶(glutamate synthase, GOGAT)作用下生成两分子的谷氨酸[5]。在酿酒酵母中已报道了谷氨酸合酶基因的转录调控, 即在谷氨酸含量丰富的氮源情况下, 谷氨酸合酶的转录水平是下降的; 而在氮源缺乏的情况下, AreA和GCN4转录因子能够激活谷氨酸合酶基因的转录[6]。
灵芝是一种重要的中药材, 能产生大量的具有生物活性的小分子化合物, 具有极高的营养保健价值和经济发展前景[7]。灵芝培养常用的氮源有酵母粉、蛋白胨、豆饼、麸皮、硫酸铵、硝酸铵等, 一般认为有机氮源更利于灵芝的生长[8]。冯杰等[9]研究了不同浓度的酵母自溶粉对灵芝菌丝体发酵合成灵芝三萜中的生物量和灵芝三萜产量的影响, 确定了酵母自溶粉的最佳添加量。相比高浓度的氮源, 当使用低浓度的谷氨酰胺培养灵芝菌丝时, 能够显著提高灵芝三萜的含量; 并且发现1个重要的氮代谢转录因子AreA的转录水平也显著升高[10]。尽管越来越多的研究表明氮源能影响灵芝的生长及次级代谢, 但是具体的分子机制并不清楚。
本文通过生物信息学分析比对获得了灵芝的谷氨酸合酶基因, 通过检测其酶活性以及构建谷氨酸合酶基因的沉默菌株, 验证GOGAT在不同氮源条件下对灵芝生长及次级代谢的重要作用。这些结果有利于从核心氮代谢中重要基因功能的角度研究氮源对灵芝生理生化的影响, 为研究氮源调控灵芝三萜的生物合成提供理论基础。
1 材料与方法 1.1 菌株和培养基灵芝(Ganoderma lucidum)菌株由中国农业微生物菌种保藏管理中心提供, 并由本实验室保存。灵芝菌株在CYM培养基(葡萄糖20 g·L-1, 蛋白胨2 g·L-1, 酵母膏2 g·L-1, 七水硫酸镁0.5 g·L-1, 磷酸二氢钾0.46 g·L-1, 琼脂20 g·L-1)中培养, 培养温度为28 ℃, 摇床培养转速为150 r·min-1。氮源的培养基配方为:葡萄糖20 g·L-1, 30 mmol·L-1对应的氮源(谷氨酰胺、铵盐、脯氨酸或硝酸盐), 七水硫酸镁0.5 g·L-1, 磷酸二氢钾0.46 g·L-1。
1.2 灵芝谷氨酸合酶(GOGAT)基因的生物信息学分析根据酿酒酵母和构巢曲霉中谷氨酸合酶的基因信息, 在灵芝基因组库中搜索获得可能的GOGAT基因序列。将获得的序列在NCBI网站上进行BLAST序列分析。利用其他物种中的谷氨酸合酶蛋白序列, 使用MEGA 6.0软件中的Neighbor-joining方法聚类分析灵芝谷氨酸合酶与其他物种中的同源蛋白的系统发育关系。
1.3 NADH-GOGAT型的谷氨酸合酶比酶活测定取在不同氮源的CYM培养基上培养7 d的野生菌株菌丝, 液氮研磨后, 称取0.5 g粉末于EP管中, 加入0.5 mL提取液(含6.118 g·L-1 Tris、0.498 g·L-1硫酸镁、0.617 2 g·L-1二硫苏糖醇和137 g·L-1蔗糖, pH8.0)。野生菌株在每1种培养条件下培养3瓶, 为3次生物学重复。反应体系:150 μL 25 mmol·L-1 Tris, 60 μL粗酶液, 50 μL 20 mmol·L-1 α-酮戊二酸, 10 μL 10 mmol·L-1氯化钾, 30 μL 3 mmol·L-1 NADH以及40 μL 20 mmol·L-1谷氨酰胺。30 ℃水浴反应30 min后煮沸3 min。10 000 r·min-1离心, 于340 nm处检测上清液的吸光值。反应液中的蛋白浓度利用BCA试剂盒(碧云天生物技术公司)进行检测, 具体方法按照试剂盒方法进行。NADH-GOGAT型的谷氨酸合酶的比酶活定义为每微克粗酶液的酶活力单位数, 单位为IU·μg-1。
1.4 GOGAT基因转录水平检测取在不同氮源的CYM培养基上培养7 d的菌丝, 液氮研磨后, 加入RNAiso Plus(TaKaRa), 按照该公司操作手册的方法提取RNA, 并利用试剂盒(TaKaRa)反转录为cDNA。在SYBR Premix Ex TaqTM的作用下进行qPCR分析, 以18 S基因作为内参测定GOGAT基因的转录水平, 所用引物见表 1。采用2-ΔΔCT方法[11], 计算灵芝GOGAT基因在铵盐、脯氨酸或硝酸盐条件下相对于谷氨酰胺条件下的转录水平。每个菌株在每1种培养条件下培养3瓶, 为3次生物学重复。
| 用途Purpose | 正向引物序列Forward primer sequence(5′→3′) | 反向引物序列Reverse primer sequence(5′→3′) |
| 沉默引物Primers for silencing | ATCGGGTACCGGACCTCCTACTCCCTAAA | GCTAACTAGTGCCATCCAACCCATCT |
| 定量引物Primers for quantity | GCCGTCTTATTGTCTACCC | CAGTCATGTATTCGCAACC |
| 内参基因Reference gene | TATCGAGTTCTGACTGGGTTGT | ATCCGTTGCTGAAAGTTGTAT |
利用Primer Premier 5.0软件设计引物(表 1), 以灵芝cDNA为模板, 用高保真酶扩增获得GOGAT的沉默片段, 并测序验证。沉默片段回收以后与质粒pAN7-ura 3 -dual使用KpnⅠ和SpeⅠ双酶切后连接, 构建沉默载体pRNAi-GOGAT。
1.5.2 GOGAT沉默菌株的筛选将菌块接种于CYM培养基上培养7 d, 用打孔器在菌丝边缘打孔接种于CYM液体培养基中, 28 ℃培养6 d后用组织粉碎机粉碎培养物。取1 mL于100 mL CYM液体培养基中, 28 ℃静置培养3 d, 每天摇匀3次。收集菌丝, 用0.6 mmol·L-1甘露醇洗涤菌丝后4 000 g离心10 min。每500 mg菌丝中加入1 mL 15 g·L-1溶壁酶后, 于30 ℃酶解2 h。4 000 g离心10 min, 获得菌丝残片, 用电击缓冲液重悬, 加入上述构建好的pRNAi-GOGAT质粒以及没有连GOGAT片段的空质粒作为对照, 进行电击转化。涂布于CYM培养基(含卡那霉素50 μg·mL-1, 潮霉素100 μg·mL-1)上, 28 ℃静置培养。
1.5.3 转化子验证挑取50个在潮霉素抗性平板上能够稳定传代的转化子, 接种于CYM培养基上培养5 d。取少量菌丝液氮研磨后, 加入RNAiso Plus后, 按照操作手册提取转化子的RNA。利用试剂盒反转录为cDNA后, 按照1.4节中的方法对沉默效率进行验证, 引物见表 1。挑选沉默效率50%以上的菌株用于后续试验。
1.6 灵芝GOGAT对菌丝生长的影响分别将野生型(WT)、对照菌株(SiCtrl)、GOGAT沉默菌株-8和GOGAT沉默菌株-11接种于含有浓度均为30 mmol·L-1谷氨酰胺、铵盐、脯氨酸和硝酸盐的CYM培养基上, 28 ℃静置培养7 d。在菌丝边缘用打孔器打孔, 将菌块接种于含有不同氮源的CYM固体平板上, 28 ℃培养7 d后, 记录菌落生长直径并拍照。每个菌株在每一种培养条件下培养3瓶, 为3次生物学重复。
1.7 灵芝三萜含量测定将WT、SiCtrl、GOGAT沉默菌株-8及GOGAT沉默菌株-11在不同氮源培养基中培养7 d, 收集菌丝, 将菌丝上残留的培养基用蒸馏水清洗干净, 用无纺布过滤, 收集菌丝于60 ℃烘箱烘干至恒质量, 取出后研磨成粉。每个菌株在每1种培养条件下培养3瓶, 为3次生物学重复。总灵芝三萜含量测定方法参照文献[12]中报道的方法。称取烘干的菌丝粉末0.2 g于10 mL容量瓶中, 用95%(体积分数)乙醇定容, 超声破碎2 h, 每20 min摇匀1次。吸取1 mL样品于离心管中, 12 000 g离心10 min后取100 μL上清液于新的离心管中。依次加入200 μL 5%的香草醛和500 μL高氯酸, 混匀后于60 ℃水浴20 min。加入冰醋酸混匀后, 检测550 nm处的吸光值。以100 μL 95%乙醇为空白对照。
1.8 数据分析试验所有测量均设3个生物学重复。误差线表示3次重复的标准差。采用t测验及Duncan ’ s新复极差法进行差异显著性分析。
2 结果与分析 2.1 GOGAT蛋白进化树和结构域分析将酿酒酵母和构巢曲霉中的谷氨酸合酶(GOGAT)的蛋白序列在灵芝蛋白库中进行比对, 获得灵芝的GOGAT蛋白序列。将该序列在灵芝蛋白库中比对, 未得到其他同源蛋白, 说明这个蛋白没有其他家族成员。将GOGAT基因序列在灵芝的全基因组库中比对, 仅在染色体上获得1个拷贝基因信息。经生物信息学分析, 灵芝GOGAT蛋白含有保守的谷氨酸合酶α亚基(gltB)和谷氨酸合酶β亚基(gltD)。进一步分析灵芝GOGAT和其他物种中的GOGAT的亲缘关系发现, 灵芝GOGAT蛋白在担子菌中较为保守, 与污叉丝孔菌的谷氨酸合酶亲缘关系最近。

|
图 1 谷氨酸合酶蛋白结构域(A)及进化树(B)分析 Fig. 1 Domain(A)and phylogenetic(B)analysis of glutamate synthase(GOGAT) gltB表示谷氨酸合酶α亚基; gltD表示谷氨酸合酶β亚基。 gltB represents glutamate synthase domain alpha; gltD represents glutamate synthase domain beta. |
以谷氨酰胺、铵盐、脯氨酸和硝酸盐为唯一氮源培养条件, 检测灵芝谷氨酸合酶的比酶活(图 2), 在不同氮源条件下, 灵芝野生菌丝的谷氨酸合酶的比酶活无显著变化, 约为(3.12±0.33)IU·μg-1。进一步通过检测GOGAT的转录水平发现, 以谷氨酰胺培养作为对照, GOGAT的转录水平在其他氮源培养时也未有显著变化。
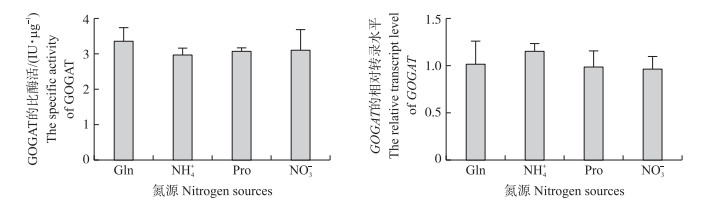
|
图 2 不同氮源条件下灵芝GOGAT的比酶活和转录水平 Fig. 2 Enzyme activity and transcript level of GOGAT of Ganoderma lucidum under different nitrogen sources Gln:谷氨酰胺Glutamine; NH4+:铵盐Ammonium salt; Pro:脯氨酸Proline; NO3-:硝酸盐Nitrate.下同。The same as follows. |
以灵芝cDNA为模板, 利用GOGAT的沉默引物PCR扩增获得沉默片段, 连入前期构建成功的pAN7-ura 3 -dual质粒中[13], 获得GOGAT的沉默载体(图 3-A)。将构建成功的载体电击转化入新鲜制备的灵芝原生质体中, 在含有潮霉素抗性的平板上挑取转化子, 通过RT-qPCR进行筛选, 获得沉默效率分别为(77.0±1.9)%和(77.5±2.0)%的GOGAT沉默菌株2株(图 3-B)。对筛选得到的沉默转化子进行比酶活检测发现, 与对照相比, GOGAT沉默转化子的比酶活下降了(55.4±4.4)%(图 3-C)。
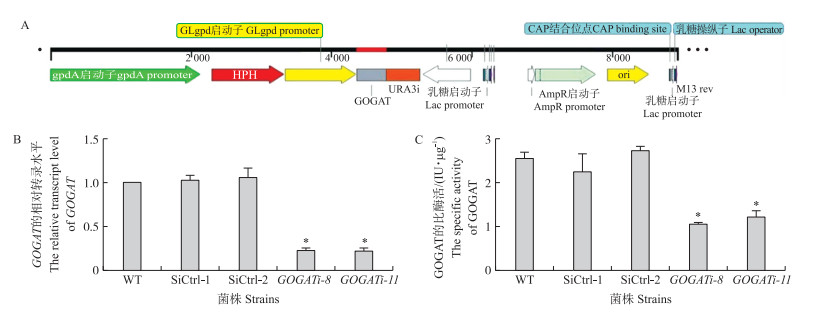
|
图 3 灵芝GOGAT沉默载体构建及沉默菌株的筛选 Fig. 3 Construction of GOGAT silenced vector and screening of GOGAT silenced strains of G. lucidum WT:野生菌株Wild type; SiCtrl-1:对照菌株1 Control strain 1;SiCtrl-2:对照菌株2 Control strain 2;GOGATi-8 :GOGAT沉默菌株-8 GOGAT-silenced strain-8;GOGATi-11 :GOGAT沉默菌株-11 GOGAT-silenced strain-11。*P < 0.05. The same as follows. |
从图 4可知:在以谷氨酰胺和铵盐为唯一氮源培养时, GOGAT沉默菌株(GOGATi-8和GOGATi-11)表现出与对照菌株相似的生长速率。在以脯氨酸为唯一氮源培养时, GOGAT沉默后菌丝的生长速率显著降低; 相比于野生菌株, 2株GOGAT沉默菌株的菌落生长直径分别下降了(16.0±1.9)%和(21.0±1.3)%。而在以硝酸盐为唯一氮源培养时发现, 对照菌株和沉默菌株菌丝的生长速率均极为缓慢, 尤其是沉默菌株的菌丝生长几乎停滞。
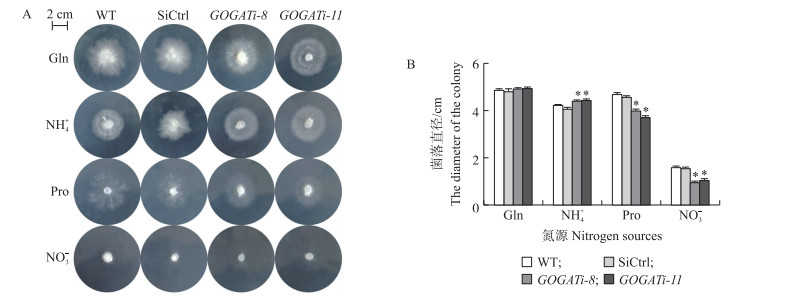
|
图 4 各菌株在不同氮源培养基上的生长(A)和菌落直径(B) Fig. 4 Growth(A)and the diameter(B)of the colony under different nitrogen sources |
由图 5可知:对于野生菌株而言, 在以谷氨酰胺或者铵盐为唯一氮源培养下, 灵芝三萜含量是最高的, 而以硝酸盐为唯一氮源培养时灵芝三萜含量最低。在以谷氨酰胺为唯一氮源培养时, 将GOGAT基因沉默后并不影响灵芝三萜的含量。而在以铵盐或者脯氨酸为唯一氮源时, 与野生菌株相比, GOGAT沉默菌株中灵芝三萜含量分别降低了(32±4.5)%和(11±1.1)%。而以硝酸盐为唯一氮源培养时, 可能由于野生菌株中灵芝三萜含量较低, 再将GOGAT沉默后也不会影响灵芝三萜的含量。这表明不同氮源培养条件能够影响灵芝三萜的生物合成, 并且GOGAT也参与了不同氮源对灵芝三萜生物合成的调控。
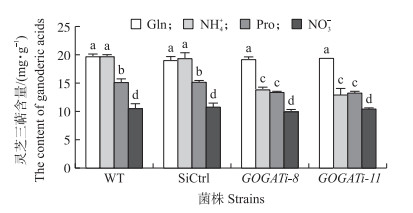
|
图 5 不同氮源培养条件下各菌株中灵芝三萜的含量 Fig. 5 The contents of ganoderic acid in the tested strains under different nitrogen sources 不同字母表示不同处理组之间在0.05水平显著差异。 Different letters indicate significant difference at 0.05 level among the treatments. |
氮源代谢能够显著影响真菌的次级代谢[2], 但是其具体的分子机制尚不清楚。作为氮源代谢中的一个重要酶——谷氨酸合酶, 其对氮源代谢的调控作用在酿酒酵母以及子囊菌中已有较多文献报道[14]。然而, 谷氨酸合酶在担子菌中的研究还未见报道, 尤其是对次级代谢的调控作用更少。本研究首次在大型担子菌灵芝中初步明确谷氨酸合酶基因在灵芝生长及次级代谢中的重要作用。这一研究结果能为今后进一步研究氮代谢如何调控真菌次级代谢提供一定的研究基础。
所有的氮源必须转变为谷氨酸和谷氨酰胺才能进一步发挥作用, 而谷氨酸和谷氨酰胺在氮代谢中发挥着重要作用。因此, 在酵母中通常将氨、谷氨酸和谷氨酰胺之间的相互转变称为核心氮代谢[5]。这3种化合物之间的相互转化有3个酶参与, 分别是谷氨酸脱氢酶、谷氨酰胺合成酶以及谷氨酸合酶。酿酒酵母和白色念珠菌的谷氨酸合酶能够催化一分子谷氨酰胺和一分子α-酮戊二酸生成两分子谷氨酸[14]。本研究在不同氮源培养条件下检测灵芝GOGAT比酶活时也发现, 灵芝GOGAT能够将谷氨酰胺和α-酮戊二酸催化生成谷氨酸。而将灵芝的GOGAT基因沉默后, 其比酶活也显著降低。硝酸盐和脯氨酸被认为是次要氮源。在酿酒酵母中研究发现当以脯氨酸为氮源培养时, 脯氨酸进入细胞后需要2条途径进行代谢[15-16]:首先是PUT途径, 包括脯氨酸氧化酶和吡咯啉-5-羧酸脱氢酶, 生成谷氨酸; 其次通过核心氮代谢途径实现胞内谷氨酸和谷氨酰胺2种物质的转换。这2条代谢途径被认为是脯氨酸代谢所必需的, 因此, 将其中GOGAT沉默后会影响次要氮源代谢的第2条途径, 从而影响胞内谷氨酸、谷氨酰胺以及α-酮戊二酸的含量, 进而可能影响菌丝的生长发育。
在酵母中的研究发现, 谷氨酸合酶基因能够被氮代谢中的2个关键转录因子激活。针对酿酒酵母GOGAT基因的启动子进行预测发现, 其启动子区域存在1个GCN4结合位点和3个Dal180结合位点[6]。当谷氨酸丰富的条件下, 谷氨酸合酶基因的转录水平较低; 而在氮源缺乏的条件下, 激活的GCN4和Gln3能够激活谷氨酸合酶基因的转录。本研究中, 在4种氮源条件下检测谷氨酸合酶的转录水平, 结果没有明显变化。这说明灵芝中的谷氨酸合酶基因的转录调控可能和酵母中的调控方式不同, 值得在今后的工作中进一步深入研究。
GOGAT是核心氮代谢的重要酶之一, 且在氮源利用过程中发挥重要功能, 本研究还发现灵芝谷氨酸合酶的缺失能够显著降低灵芝三萜的含量。作为GOGAT催化的产物, 谷氨酸是胞内谷胱甘肽的重要前体物质, 其含量的改变能够影响胞内氧化还原的稳态[4, 17]。在灵芝的前期研究中发现, 胞内氧化还原水平和灵芝三萜的含量密切相关[18], 由此推测灵芝GOGAT的缺失是通过影响胞内ROS的稳态从而影响灵芝三萜的生物合成。因此, 深入研究参与核心氮代谢的GOGAT的生物学功能, 能为研究氮源调控灵芝次级代谢的分子机制奠定基础。
| [1] |
Mihlan M, Homann V, Liu T W D, et al. AREA directly mediates nitrogen regulation of gibberellin biosynthesis in Gibberella fujikuroi, but its activity is not affected by NMR[J]. Molecular Microbiology, 2003, 47(4): 975-991. DOI:10.1046/j.1365-2958.2003.03326.x |
| [2] |
Tudzynski B. Nitrogen regulation of fungal secondary metabolism in fungi[J]. Frontiers in Microbiology, 2014, 5: 656. |
| [3] |
Wang B, Han X Y, Bai Y H, et al. Effects of nitrogen metabolism on growth and aflatoxin biosynthesis in Aspergillus flavus[J]. Journal of Hazardous Materials, 2017, 324: 691-700. DOI:10.1016/j.jhazmat.2016.11.043 |
| [4] |
Cooper A, Jeitner T. Central role of glutamate metabolism in the maintenance of nitrogen homeostasis in normal and hyperammonemic brain[J]. Biomolecules, 2016, 6(2): 16. DOI:10.3390/biom6020016 |
| [5] |
ter Schure E G, van Riel N A W, Verrips C T. The role of ammonia metabolism in nitrogen catabolite repression in Saccharomyces cerevisiae[J]. FEMS Microbiology Reviews, 2000, 24(1): 67-83. |
| [6] |
Valenzuela L, Ballario P, Aranda C, et al. Regulation of expression of GLT1, the gene encoding glutamate synthase in Saccharomyces cerevisiae[J]. Journal of Bacteriology, 1998, 180(14): 3533-3540. |
| [7] |
Cao F R, Feng L, Ye L H, et al. Ganoderic acid A metabolites and their metabolic kinetics[J]. Frontiers in Pharmacology, 2017, 8: 101. |
| [8] |
刘冬, 李世敏, 许柏球, 等. 灵芝菌丝体深层液体发酵培养基研究[J]. 微生物学杂志, 2001, 21(2): 15-17. Liu D, Li S M, Xu B Q, et al. A probe into medium formula for submerged culture of Ganoderma lucidum[J]. Journal of Microbiology, 2001, 21(2): 15-17 (in Chinese with English abstract). DOI:10.3969/j.issn.1005-7021.2001.02.007 |
| [9] |
冯杰, 冯娜, 贾薇, 等. 氮源对灵芝菌丝体液态深层发酵合成灵芝三萜的影响[J]. 菌物学报, 2016, 35(6): 722-733. Feng J, Feng N, Jia W, et al. Effects of nitrogen source on triterpene production by Ganoderma lingzhi G0119 based on liquid submerged fermentation[J]. Mycosystema, 2016, 35(6): 722-733 (in Chinese with English abstract). |
| [10] |
Zhao W, Xu J W, Zhong J J. Enhanced production of ganoderic acids in static liquid culture of Ganoderma lucidum under nitrogen-limiting conditions[J]. Bioresource Technology, 2011, 102(17): 8185-8190. DOI:10.1016/j.biortech.2011.06.043 |
| [11] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25: 402-408. DOI:10.1006/meth.2001.1262 |
| [12] |
Ren A, Qin L, Shi L, et al. Methyl jasmonate induces ganoderic acid biosynthesis in the basidiomycetous fungus Ganoderma lucidum[J]. Bioresource Technology, 2010, 101(17): 6785-6790. DOI:10.1016/j.biortech.2010.03.118 |
| [13] |
Mu D S, Shi L, Ren A, et al. The development and application of a multiple gene Co-silencing system using endogenous URA3 as a reporter gene in Ganoderma lucidum[J]. PLoS One, 2012, 7(8): e43737. DOI:10.1371/journal.pone.0043737 |
| [14] |
Holmes A R, Collings A, Farnden K J F, et al. Ammonium assimilation by Candida albicans and other yeasts:evidence for activity of glutamate synthase[J]. Microbiology, 1989, 135(6): 1423-1430. DOI:10.1099/00221287-135-6-1423 |
| [15] |
Wanduragala S, Sanyal N, Liang X W, et al. Purification and characterization of Put1p from Saccharomyces cerevisiae[J]. Archives of Biochemistry and Biophysics, 2010, 498(2): 136-142. DOI:10.1016/j.abb.2010.04.020 |
| [16] |
Sieg A G, Trotter P J. Differential contribution of the proline and glutamine pathways to glutamate biosynthesis and nitrogen assimilation in yeast lacking glutamate dehydrogenase[J]. Microbiological Research, 2014, 169(9/10): 709-716. |
| [17] |
Schmacht M, Lorenz E, Senz M. Microbial production of glutathione[J]. World Journal of Microbiology and Biotechnology, 2017, 33(6): 106. DOI:10.1007/s11274-017-2277-7 |
| [18] |
Ren A, Shi L, Zhu J, et al. Shedding light on the mechanisms underlying the environmental regulation of secondary metabolite ganoderic acid in Ganoderma lucidum using physiological and genetic methods[J]. Fungal Genetics and Biology, 2019, 128: 43-48. DOI:10.1016/j.fgb.2019.03.009 |




