文章信息
- 高志红, 侍婷, 倪照君, 潘振朋, 倪晓鹏
- GAO Zhihong, SHI Ting, NI Zhaojun, PAN Zhenpeng, NI Xiaopeng
- 梅种质资源与分子生物学研究进展
- Research progress on germplasm and molecular biology in Japanese apricot
- 南京农业大学学报, 2019, 42(6): 975-985
- Journal of Nanjing Agricultural University, 2019, 42(6): 975-985.
- http://dx.doi.org/10.7685/jnau.201811022
-
文章历史
- 收稿日期: 2018-11-15
梅(Prunus mume Sieb. et Zucc.)原产于中国, 为蔷薇科李属植物。梅种质资源丰富, 遗传多样性广泛, 与核果类果树杏、李、樱桃和桃均可进行种间杂交。梅果实具有较高的营养价值, 其经济栽培主要分布在东亚和东南亚的亚热带地区, 中国是目前梅种植面积最大的国家。根据用途, 梅可分为果梅和花梅2种类型。2011年国家果梅种质资源圃的建立和2012年基因组测序的完成, 促进了梅种质资源的收集、保存、评价利用以及分子生物学的发展。近年来, 国内外学者在梅资源鉴定和分子生物学研究等方面取得了较大的进展, 本文对近20年梅种质资源评价鉴定和分子生物学研究等方面进行全面综述, 为今后梅的科学研究提供借鉴和参考。
1 梅起源分布与资源研究 1.1 梅的起源和分布一般认为梅栽培历史有3 000多年[1], 但据近年在我国河南省发现的梅核化石分析认为, 在距今7 000~7 500年前新郑裴李岗文化时期就有梅的种植。梅的迁移和分布与地理气候的变迁有很大关系。据古书籍记载, 古代梅树曾分布于我国华北和西北一带, 后因气温降低而缩小了梅的适宜栽培区域, 目前主要分布在我国的亚热带地区及其与北温带的过渡地带, 而华北和西北地区仅有零星栽培[2]。据调查数据显示, 我国野生梅主要分布于四川、云南、西藏、湖北、贵州、湖南、广东、广西、江西、安徽、浙江、福建、江苏、陕西、台湾15个省(自治区)[3-4]。目前, 我国的四川、云南、西藏、贵州、湖北、湖南、广东、广西、福建、江西、安徽、浙江、江苏、河南、陕西、甘肃、台湾17个省(自治区)均有果梅栽培[5], 其中广东、福建、云南、浙江、江苏等省是中国果梅的传统产区, 全国栽培面积为80 000 hm2左右。梅主栽品种主要为‘龙眼梅’‘软枝大粒梅’‘照水梅’‘白粉梅’和‘长农17’等。相比而言, 观花用的梅花种植范围更加广泛, 长春、沈阳、延安、拉萨和北京等地均有梅花的栽培, 甚至海南省部分地区也有梅花的栽培[5]。此外, 老挝及越南北部也有梅的分布, 日本、朝鲜、韩国均有较多的梅花栽培, 新西兰、巴西、泰国、英国、法国、意大利、美国、德国等国家也有少量的梅花栽培[6]。
除了中国以外, 日本梅的栽培面积最大, 从日本中部的东京到南部的和歌山都有种植, 近20年种植区域和面积也比较稳定。据日本农林水产省统计数据, 2017年日本全国梅产量为15 000 hm2左右, 主要集中在和歌山、群马、奈良、长野、三重等地, 栽培品种主要有‘南高’‘古城’和‘小梅’等。
1.2 梅资源收集和鉴定评价2011年, 我国农业部批准建立国家级的果梅种质资源圃, 依托单位为南京农业大学, 经过后期的改建和扩建, 目前占地5.3 hm2, 共收集了国内外近200份梅野生资源、地方品种和近缘种。所收集的梅种质资源主要来自我国台湾、云南、广东、浙江、福建、广西、湖南、四川、安徽、江苏等省(自治区)以及日本等其他国家。果梅种质资源圃对现有资源的植物学性状、园艺学性状和抗逆性进行了全面的鉴定和分析[7], 建立了果梅种质资源核心种质20份(表 1)[8], 筛选出10多个花果兼用品种以及2个优质的抗寒果梅品种‘软条红梅’和‘细叶青’[9], 从47个果梅品种中鉴定出1个完全花比例较高的种质资源[10]。除此以外, 还通过田间鉴定和实验室鉴定相结合的方法鉴定出抗疮痂病的种质资源[11]以及自交亲和变异的种质资源‘早红’和‘四川白梅’[12-13]。通过多年连续的田间观测并结合实验室鉴定, 明确了69个果梅品种的需冷量[14]以及43个梅品种的S基因型[15], 为不同地区选择品种和生产配置授粉品种提供了依据。采用分子标记技术对梅不同种质资源及其与近缘种杏、桃、李、樱桃等核果类果树的亲缘关系进行了系统分析, 发现了梅演化的分子依据, 尤其是日本梅是由中国传播的事实[16], 进一步通过基因组序列和叶绿体基因序列分析发现杏和梅的亲缘关系比其他近缘种更近[17]。
| 品种或变种 Cultivar or variety |
主要特征 Characteristic |
| 丰产厚叶梅 Fengchanhouyemei |
白梅类, 叶厚 White Japanese apricot group, thick leaf |
| 果用照水梅 Guoyongzhaoshuimei |
丰产, 花柄下垂 High yield, pendulous petiole |
| 台湾大青梅 Taiwandaqingmei |
完全花比例高 High ratio of perfect flower |
| 鹅嗉 Esu |
大果形 Large fruit |
| 青佳 Qingjia |
青梅类, 丰产 Green Japanese apricot group, high yield |
| 之枝梅 Zhizhimei |
枝条呈Z字形 Z shape of branch |
| 浙江小梅 Zhejiangxiaomei |
果实小 Small fruit |
| 皱叶 Zhouye |
抗旱性强 Strong drought resistant |
| 品字梅 Pinzimei |
品字梅变型, 1花结3果 Forma of‘Pinzimei’, three fruits after one flower |
| 云南杏梅 Yunnanxingmei |
杏梅类 Hybrid of Japanese apricot and apricot |
| 诏安白梅 Zhaoanbaimei |
白梅类 White Japanese apricot group |
| 奉化桃梅 Fenghuataomei |
果实桃形 Peach shape of fruit |
| 绿萼 Lüe |
萼片绿色 Green sepal |
| 长梗梅 Changgengmei |
果梗特长 Long fruit stalk |
| 软枝大粒梅 Ruanzhidalimei |
抗逆性强 Strong stress resistance |
| 软条红梅 Ruantiaohongmei |
耐氟 Fluorine tolerance |
| 早花梅 Zaohuamei |
花期早 Earlier blooming date |
| 迟红梅 Chihongmei |
杂合体 Hybrid |
| 绿芯红梅 Lüxinhongmei |
维生素A含量高 High vitamin A content |
| 南红 Nanhong |
花粉量多 High quantity of pollen |
虽然木本植物的高度杂合性增加了基因组整合的难度, 但Zhang等[18]利用全基因组酶切图谱技术(WGM)克服了组装难度, 完成了梅全基因组测序和组装, 构建了首个长度为237 Mb的梅基因组图谱, 并预测梅基因组实际大小约为280 Mb; 结合已完成的苹果和草莓基因组序列, 重建了蔷薇科植物的祖先染色体, 并深入分析苹果属、草莓属和李属3个种属不同的染色体融合、断裂的过程。此外, Zhang等[18]还探索了与梅开花相关的基因以及在低温下打破芽休眠的分子机制, 共鉴定出6个与休眠相关的DAM基因, 呈串联重复分布, 在DAM基因上游有6个CBF基因的结合位点。Sasaki等[19]推测DAM基因和多个CBF结合位点是梅提早解除休眠的关键因子, 这些因子使得梅对温度变化非常敏感, 从而导致梅的休眠期短而开花较早。梅基因组测序的完成推动了梅分子生物学研究的深入开展, 提升了研究水平。
梅花具有特殊而独特的香气。Hao等[20]鉴定出梅花香气重要成分为乙酸苯甲酯, 同时分析了其所调控的基因苯甲醇乙酰基转移酶基因(BEAT), 该基因家族在梅基因组中为34个, 而苹果只有16个, 草莓只有14个, 据此推测这些BEAT基因可增加乙酸苯甲酯含量的剂量效应, 从而使梅具有独特的花香。此外, Zhang等[21]以梅花的近缘物种山杏、山桃和李作为参照, 构建了梅花品种的系统发育树, 从16个亚群中选择7个梅花品种和1个野生梅花, 与山杏、山桃、李进行重新测序和基因组组装, 并结合已经发表的桃和梅花基因组, 通过GWAS分析确定了多个数量性状基因座(QTL)和基因组区域, 并发现MYB108基因和花色有关。
2.2 叶绿体基因组植物细胞中包含3大基因组:核基因组、叶绿体基因组和线粒体基因组。与核基因组相比, 叶绿体基因组体积小, 拥有相对独立的遗传物质叶绿体DNA、细胞质遗传、核苷酸替换率低、单倍体性质和高度保守的基因组结构[22-23], 因此叶绿体基因组被认为是系统发育研究的理想系统。1984年, 烟草和地钱的叶绿体基因组测序完成[24], 这是最早报道的植物叶绿体基因组序列。截至2018年3月, NCBI上收录的完整的植物叶绿体基因组共有13 602条。
高等植物叶绿体基因组DNA一般为双链环状结构, 呈典型的四段式结构, 极少数为线性分子结构, 由1个大单拷贝区(LSC)、1个小单拷贝区(SSC)和1对反向互补重复区(IR)组成, 其中IR区将LSC和SSC区分隔, 大小一般为120~160 kb。梅叶绿体基因组为典型的闭合双链环状DNA分子结构。笔者所在的实验室测序获得果梅叶绿体基因组全长为157 815 bp, LSC区长度为86 113 bp, SSC区长度为18 916 bp; Ira和IRb区长度相等, 为26 393 bp。梅叶绿体基因组GC含量为36.74。其中, IR区GC含量为42.56, LSC和SSC区域的GC含量比IR区的分别低8%和12%。在梅的基因注释表中, 梅叶绿体基因组编码133个基因, 其中110个是unique基因。这110个unique基因包括80个蛋白编码基因, 26个tRNA基因, 4个rRNA基因。在IR区上有18个基因重复, 包括所有的8个rRNA基因, IR区域内还有13个基因。此外, 共12个基因含有内含子, 其中10个基因含有1个内含子, 2个基因含有2个内含子。这些内含子中有9个分布在LSC区, 2个分布在SSC区, 3个分布在IR区[17]。这与Wang等[25]发表的梅叶绿体序列(NC_023798)类似, 但在序列长度、基因组成和内含子数目上有所不同。
梅核基因组和叶绿体基因组序列图谱的测序完成, 有助于开发分子标记进行辅助育种和开展近缘物种的进化分析。
3 梅花的发育研究 3.1 雌蕊发育研究梅花花器官通常由外到内包括4轮花器官:萼片、花瓣、雄蕊和雌蕊, 呈半径不等的同心圆排列。梅花不但具有独特的香气而且花型繁多, 变异大, 是研究花器官发育的良好材料, 而果树雌蕊更是重要的生殖器官, 因此雌蕊发育的分子生物学也成为关注的热点。
梅完全花通常含有1枚正常发育的雌蕊, 也有少数品种含有3至数枚雌蕊并且能够正常授粉。李晓颖等[26]对南京梅花品种花性状进行评价, 结果发现梅花雌蕊有多种形态, 单瓣品种雌蕊多发达, 1枚雌蕊多数能正常发育并结实; 重瓣花雌蕊变化较大, 有完全正常的雌蕊, 也有完全退化的雌蕊, 雌蕊数量不定, 最多为15枚; 对所有梅花品种的统计表明:有136个品种(41.21%)的雌蕊数量为1~6个。侍婷等[7]通过石蜡切片观察了果梅品种‘龙眼’和‘大嵌蒂’雌蕊分化进程, 发现10月中下旬为雌蕊分化初期, 11月下旬至12月上旬为雌蕊分化期, 12月中旬以后为雌蕊分化末期, 雌蕊分化基本完成, 因此南京地区雌蕊分化的关键时期为10月上旬, 而雌蕊发育的关键时期为12月上旬。梅花中除了具有正常生殖能力的完全花外, 还包括雌蕊发育不正常的不完全花, 主要表现为雌蕊的缺失、畸形和褐化。Gao等[27]研究表明, 果梅品种‘大嵌蒂’的不完全花比例高达76.3%, 不完全花的存在严重影响到梅果实产量。因此, 雌蕊败育的分子机制也受到科研人员的关注, 希望寻求解决雌蕊发育异常问题的途径, 从而提高完全花比例, 提高果实产量和品质。
Gao等[27]利用高通量测序技术对与完全花和不完全花雌蕊败育相关的miRNA进行了分析, 鉴定出7个已知miRNA和6个新miRNA在2种花中表达差异显著。Wang等[28]发现Pm-miR319a影响梅花雌蕊的发育, 并通过负调控目标基因Pm-TCP4促进雌蕊败育。同时, Shi等[29]发现完全花和不完全花差异蛋白中咖啡酰-辅酶A-O-甲基转移酶(CCoAOMT)只存在于‘大嵌蒂’完全花中。Sun等[30]进一步克隆了CCoAOMT基因, 并通过洋葱瞬时表达试验证明CCoAOM蛋白定位在细胞质中; 同时发现PmCCoAOMT在雌蕊发育前期‘大嵌蒂’中的表达量高于‘龙眼’, 而在雌蕊发育后期则刚好相反, 并且完全花中表达量高于不完全花; PmCCoAOMT的过量表达导致花变大、花瓣变深以及雌蕊形态改变, 证明该基因对雌蕊的发育具有一定的作用。植物激素尤其是生长素是影响雌蕊发育的重要因素, Song等[31]研究发现植物生长素相应因子ARF基因对雌蕊发育起作用, 尤其是PmARF13和PmARF17可能是梅花雌蕊发育所必需的。
MADS-box基因除调控开花时间外, 对花器官形成具有重要调节作用。其中C类功能基因AG(AGAMOUS)主要负责调控雄蕊和心皮的发育, E类功能基因SEP 1/2/3/4(SEPALLATA 1/2/3/4)主要负责调控4轮花器官的形成。Hou等[32]克隆得到了梅花AG基因, 同时通过原位杂交发现PmAG在萼片、心皮和雄蕊中有高表达。Zhou等[33]克隆了4个PmSEP基因, 通过表达模式分析和酵母双杂证明这4个PmSEP涉及到花器官形成, 其中雌蕊发育受PmSEP2和PmSEP3调控。综上所述, 目前仅仅初步揭示了梅雌蕊败育的分子机制(图 1)。
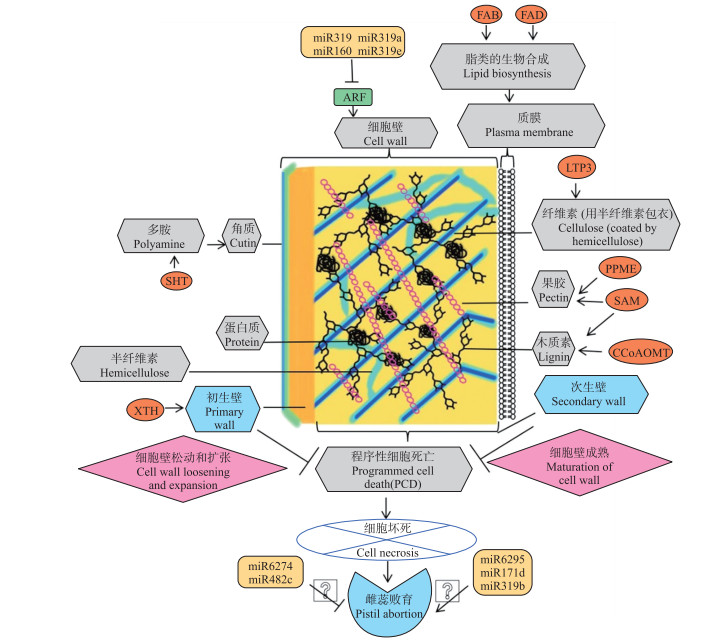
|
图 1 梅雌蕊败育机制示意图[34] Fig. 1 Pathway of mechanism of pistil abortion in Japanese apricot[34] FAB:脂肪酸合成Fatty acid biosynthesis; FAD:脂肪酸去饱和酶Fatty acid desaturase; ARF:生长素响应因子Auxin response factor; LTP3:脂质转移蛋白3 Lipid transfer protein 3;SHT:亚精胺羟基肉桂酰转移酶Spermidine hydroxycinnamoyl transferase; PPME:果胶裂解样超家族蛋白Pectin lyase-like superfamily protein; SAM:S-腺苷甲硫氨酸S-adenosylmethionine; CCoAOMT:咖啡酰-辅酶A-O-甲基转移酶Caffeoyl-CoA-O-methyl transferase; XHT:木葡聚糖内转葡糖基酶/水解酶Xyloglucan endo-transglucosylase/hydrolases. |
梅为蔷薇科李属典型的配子体自交不亲和(gametophytic self-incompatibility, GSI)树种[35-36]。自交不亲和现象指可育的雌雄同花的被子植物在自花授粉后不能产生合子的现象, 在蔷薇科植物中普遍存在。蔷薇科的配子体自交不亲和性受单一位点S-locus的至少2个多态性基因控制, 即花柱决定基因S-RNase基因(S-ribonucleases)和花粉决定基因SLF基因(S-locus F-box protein gene)或SFB基因(S haplotype-specific F-box gene)[37]。
3.2.1 梅花柱S-RNase基因鉴定自交不亲和现象首先在烟草属植物上证实存在, 并且通过杂交试验首次发现了S基因的存在[38]。梅S基因研究起步较晚, 直到2000年梅首个Sf-RNase(登录号:AB101437)等位基因在自交亲和品种中利用AS-PCR(allele-specific PCR)技术克隆, 并且开发分子标记来鉴别自交不亲和品种[35, 39]。Yaegaki等[36]运用RT-PCR技术扩增部分S-RNase基因编码区(MSRN1-3), 并确定了6个品种的S-基因型。Habu等[40]运用AS-PCR扩增技术克隆了S1至S11, 以及Sf-RNase并且更新了3种S-基因型。Entani等[41]克隆出与Habu等[40]登录序列不同的S9-RNase(登录号:AB092646.1)。Heng等[42]鉴定了S10至S16 (登录号:DQ011150、DQ201191、DQ201192、DQ345781、DQ768219、DQ903312和EF990750)。Xu等[43]克隆了S17至S26 (登录号:FJ602078—FJ602087), 并通过田间试验异花授粉验证了24个品种的S-基因型。Wang等[44]克隆得到S30至S33共4个S-RNase基因(登录号:JN232975—JN232978), 并鉴定了17个品种的S-基因型。Gao等[45]运用聚丙烯酰胺凝胶电泳分析克隆得到S34、S35、S36共3个S-RNase基因和6个SFB基因(PmSFB14、PmSFB18、PmSFB22、PmSFB24、PmSFB31和PmSFB34)。
虽然梅自交不亲和研究起步较晚, 但是从2000年到2013年, 果梅的研究取得了较大进展。迄今为止, 已经鉴定并登录的果梅S-RNase基因有S1—S26、S30—S36、Sf-RNase、S9-RNase、S10共36个S基因和40多个栽培品种的S-基因型(表 2), 同时发现了4个自交亲和的品种, 并进一步分析了其自交亲和变异的机制[46]。这些研究结果为生产配置授粉品种和育种组合的亲本选择提供了依据。
| S-基因型 S-genotypes |
品种 Cultivars |
| S1S5 | 莺宿Ousyuku |
| S1S6 | 月世界Gessekai |
| S1S7 | 南高Nanko |
| S2S6 | 玉英Gyokuei |
| S2S9 | 杭州白梅Hangzhoubaimei |
| S2S15 | 早红、福建白梅、东青、细叶青、青佳2、奉化李梅、胭脂梅Zaohong, Fujianbaimei, Dongqing, Xiyeqing, Qingjia 2, Fenghualimei, Yanzhimei |
| S2S18 | 小青、67号Xiaoqing, No. 67 |
| S2S24 | 大白梅Dabaimei |
| S2S26 | 晚红Wanhong |
| S3S4 | 改良内田梅Kairyouchidaume |
| S5S9 | 雪梅Xuemei |
| S6S10 | 加贺地藏、梅乡Kagajizo, Baigo |
| S6S20 | 烟花Yanhua |
| S7Sf | 红映Benisashi |
| S7S9b | 鹿畑Shikanohata-1 |
| S7S10 | 小粒南高Kotsubunanko |
| S8Sf | 竜峡小梅、八郎Ryukyokoume, Hachiro |
| S9Sf | 淋洲梅Rinsyu |
| S9S14 | 红农Hongnong |
| S9S15 | 大叶猪肝、卫山种Dayezhugan, Weishanzhong |
| S9S18 | 黄小大Huangxiaoda |
| S9S23 | 品字梅Pinzimei |
| S10Sf | 新平太夫、地藏梅Shinheidayu, Jizoume |
| S10S14 | 龙眼Longyan |
| S11Sf | 织姬Orihime |
| S15S17 | 东山李梅Dongshanlimei |
| S15S19 | 大羽Dayu |
| S21S22 | 桃形梅Taoxingmei |
| S24S25 | 铜绿Tonglü |
| SfSf | 剑先Kensaki |
| S3′Sf | 越之梅, 藤五郎, 八房Koshinoume, Tougoro, Yatsubusa |
与雌蕊S-RNase基因相比, 果梅花粉SFB/SLF基因研究要滞后一些, 但也取得了显著的进展。2003年Etani等[41]进一步克隆了SLF 1、SLF7和SLF9共3个基因(AB092621、AB092622和AB092645)。Ushijima等[47]利用RT-PCR克隆了自交亲和基因SFBf(AB280956.1、AB280957.1、AB280958.1)。Yamane等[48]克隆并登录了SFB3 (AB376968), 并获得SFB3的自交亲和突变基因SFB3′。Heng等[49]鉴定了PmSFB10至PmSFB16, 氨基酸序列相似度为73%~91%。Wang等[46]克隆并鉴定了7个新SFB基因, 分别为PmSFB2 (JQ356589)、PmSFB12 (JQ356586)、PmSFB15、PmSFB40 (JQ356585)、PmSFB41 (JQ356593)、PmSFB42 (JQ356581)和PmSFB43 (JQ356578), 其氨基酸序列相似度为67%~97%。Gao等[45]运用聚丙烯酰胺凝胶电泳分析克隆得到6个SFB基因:PmSFB14 (JQ356596)、PmSFB18 (JX065217)、PmSFB22 (JQ356584)、PmSFB24 (JQ356595)、PmSFB31 (JQ356588)和PmSFB34 (JQ356597)。
在果梅自交不亲和基因鉴定的研究工作中, 以PCR技术为基础的鉴定方法最为普遍。迄今为止, 已经鉴定并登录的果梅SFB基因有PmSFB1、PmSFB2、PmSFB3、PmSFB7、PmSFB12、PmSFB14、PmSFB18、PmSFB22、PmSFB24、PmSFB31、PmSFB34、PmSFB40、PmSFB41、PmSFB42、PmSFB43共15个, SLY基因有PmSLF 1、PmSLF7和PmSLF9共3个, 自交亲和基因PmSFB3 ′和PmSFBf共2个。这足以证明了以基因序列的特异性来鉴定花粉基因型的可行性, 但花粉S基因及其在自交亲和性变异中的作用还需进一步深入研究。
3.3 梅季节性休眠分子机制多年生木本植物季节性休眠是对外界不良环境条件适应的机制。根据季节性休眠的不同时期, 一般分为3种类型:抑制性休眠、自然休眠和生态休眠[50]。虽然种子休眠和木本植物的季节性休眠机制相似, 但也存在差异。梅树具有休眠期短、春季萌芽早的特点, 是研究休眠过程, 尤其是木本植物解除休眠的良好材料。植物休眠解除过程涉及到许多信号分子, 其中植物激素是最重要的一种信号物质[51]。脱落酸(ABA)、生长素(IAA)和赤霉素(GA)被认为是休眠的形成与解除过程中非常关键的信号分子, 起着至关重要的作用[52-53]。Zheng等[54]研究发现, 拟南芥ABA不敏感突变体(abi1—abi5)种子萌发对ABA诱导的生长抑制不敏感。
近年来, 随着拟南芥等模式植物以及种子休眠机制的逐渐揭示, 国内外学者对梅休眠机制的研究也取得了较大进展。Zhuang等[55]研究发现, 用GA4处理不同休眠时期的果梅枝条后, 许多与能量代谢和氧化还原相关的蛋白活性都被激活了, 因此GA4处理能够代替部分需冷量, 有效解除果梅季节性休眠。此外, Zhuang等[55]通过组学研究还提出了GA4提早解除果梅季节性休眠的模式图(图 2)。Wen等[56]进一步用外源GA4处理低需冷量果梅品种‘桃形梅’的叶芽后, 其抗氧化酶的活性显著高于需冷量高的品种‘丰后’, 表明低需冷量果梅品种比高需冷量品种具有更高的抗氧化酶活性。在模式植物拟南芥和水稻中, 赤霉素信号传导和生物合成途径已基本明确。在拟南芥中, GA4(主要的生物活性形式)和赤霉素受体[GIBBERELLIN INSENSITIVE DWARF(GID)1a-c]结合导致构象变化, 使DELLA蛋白与GA4-GID1复合物结合形成三聚体, 随后被26S蛋白酶体泛素化, 从而发生降解, 解除植株的生长抑制[57-58]。Lv等[59]在梅中的研究和拟南芥一致, 梅DELLA蛋白(RGL2)能够影响转基因杨树中与GA代谢相关基因(GA20ox2、GA3ox1和GID1b)的表达, 从而抑制转基因杨树的休眠。除了植物激素在梅休眠诱导、保持和解除中发挥作用外, 日本学者首先发现休眠相关基因(DAMs)参与休眠调控, DAM6基因被认为和休眠保持关系最为密切[60]。Zhao等[61]证实了位于细胞核中的PmDAM影响了花芽的生长和休眠, 在此过程中, 主要由PmDAM1和PmDAM2形成的蛋白质复合物变为由PmDAM1、PmDAM2、PmDAM3和PmDAM6组成的更复杂的结构, 其随后演变为PmDAM6蛋白质占优势的复合物; 同时还发现在休眠期开始时, PmCBF负向调控PmDAM6的表达, PmCBF5能与PmDAM1和PmDAM6形成异质复合物。PmCBF1、PmCBF3和PmDAM4通过替代结合位点识别PmDAM6的启动子, 从而参与调控打破休眠[61-62]。Kitamura等[63]利用2个F1分离群体鉴定了一系列与需冷量相关的QTL, 初步定位在李属植物通用遗传图谱的LG4连锁群; 此外还定位了PmDAM6下游基因的QTL区域。Yamane等[64]发现通过在苹果上异位过量表达PmDAM6诱导了芽的休眠而抑制了休眠的解除。
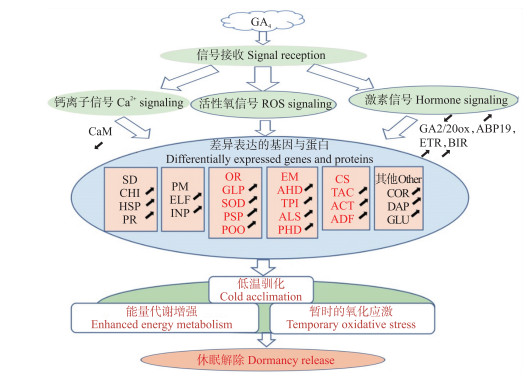
|
图 2 梅休眠解除机制示意图[55] Fig. 2 Pathway of the mechanism of bud dormancy release in Japanese apricot[55] GA4:赤霉素4 Gibberellin 4;CaM:钙调素Calmodulin; GA2/20ox:赤霉素2/20氧化酶GA2/20-oxidases; ABP19:生长素结合蛋白19 Auxin-binding protein 19;ETR:乙烯受体Ethylene receptor; BIR:油菜素类固醇不敏感1-相关受体激酶Brassnosteroid insensitive 1-associated receptor kinase; SD:抗逆和防卫Stress and defence; CHI:几丁质酶Chitinase; HSP:热激蛋白Heat shock protein; PR:病程相关蛋白Pathogenesis-related protein; PM:蛋白质代谢Protein metabolism; ELF:延伸因子Elongation factor; INP:胰岛素酶蛋白Insulinase protein; OR:氧化还原Oxidation reduction; GLP:谷胱甘肽过氧化物酶Glutathione peroxidase; SOD:超氧化物歧化酶Superoxide dismutase; PSP:过氧化物酶超家族蛋白Peroxidase superfamily protein; POO:多酚氧化酶Polyphenol oxidase; EM:能量代谢Energy metabolism; AHD:醇脱氢酶Alcohol dehydrogenase; TPI:磷酸丙糖异构酶Triosephosphate isomerase; ALS:醛缩酶Aldolase; PHD:D-3-磷酸甘油酸脱氢酶D-3-phosphoglycerate dehydrogenase; CS:细胞结构Cell structure; TAC:微管蛋白α-2链Tubulin α-2 chain; ACT:肌动蛋白Actin; ADF:肌动蛋白解聚因子Actin-depolymerizing factor; COR:冷响应蛋白Cold regulated protein; DAP:休眠相关蛋白Dormancy-associated protein; GLU:β-1, 3-葡聚糖酶β-1, 3-glucanase. |
综上所述, 梅作为落叶果树中休眠期最短的果树, 被认为是研究休眠解除机制的良好材料, 近年来也取得了令人瞩目的结果, 但并未全面揭示其机制, 还需进一步深入研究。
4 梅分子标记开发根据开发的方法梅分子标记分为2大类。第1类是以电泳技术和PCR技术为核心的分子标记, 包括随机扩增多态性DNA(RAPD)、简单重复序列(SSR)、扩增片段多态性长度(AFLP)以及逆转座子分子标记; 第2类是以DNA序列为核心的分子标记, 典型的有单核苷酸多态性(SNP)和表达序列标签(EST)标记[65]。
Yamane等[66]利用RFLP技术使用Pm-SFB探针来分辨果梅自交不亲和S1至S8单倍型以及自交亲和单倍型, 这是比较早的梅分子标记应用的报道。在基因组测序完成之前, 分子标记作为梅品种鉴定和亲缘关系分析的重要手段被广泛应用。Li等[67]利用RAPD标记技术随机扩增基因组不同区域, 并采用聚丙烯凝胶(PAGE)电泳检测扩增引物, 提高了检测多态性数量, 能够客观反映所测果梅品种间的遗传关系和遗传差异。Fang等[68]应用AFLP分子标记技术将中国和日本的14个品种分成2类, 利用2对AFLP引物组合的多态性分别为57.92%和63.04%。侍婷等[7]和高志红[69]利用SSR分子标记技术对果梅种质资源的遗传多样性进行了分析, 并筛选了花果兼用的优异梅种质, 建立了果梅的核心种质。
Li等[70]利用SNP标记分析梅的遗传多样性和亲缘关系, 研究表明果梅SNP的出现频率高于花梅, 说明果梅品种间的遗传差异较大, 而花梅品种的育种亲本相似或者地理起源相近, 但试验证明果梅与花梅在遗传上具有很高的相似性, 是同一树种中某些性状(主要是花器官)存在一定差异的不同品种类型。2012年梅全基因组测序的公布为基于参考基因组发掘不同品种间基因SNP差异分析提供了方便[18]。Du等[71]利用EST数据库在全基因组上鉴定30个胚胎发育晚期丰富蛋白, 这类蛋白除3号染色体外, 在其他7条染色体上均有分布。Sun等[72]结合SSR和SNP分子标记构建了高密度连锁图谱来进行梅童期性状和树体结构等数量性状的QTL定位。Zhang等[73]利用简化基因组测序SLAF-Seq技术开发SLAF分子标记, 用来构建高密度连锁图谱, 其中SLAF分子标记包含8 007个标记, 平均标记距离为0.195 cM, 使其成为李属植物最密集的遗传图谱, 并且将垂枝性状定位在7号连锁群上, 为应用SLAF标记构建的高密度遗传图谱探究木本植物的重要特征提供参考。
虽然分子标记在梅品种分类、遗传变异、基因标记、亲缘关系分析中得到了广泛的应用, 但由于梅杂交坐果率低等原因, 可用于育种实践的分子标记还很缺乏, 如在其他果树上已经获得的与果实性状、物候期、抗性相关的分子标记还有待进一步开发。
5 组培与遗传转化研究目前, 用于果梅组培与遗传转化的外植体类型主要包括叶片[74-75]、叶柄[76]、一年生枝条[74-77]、未成熟合子胚[75-79]等4大类, 外植体灭菌主要使用75%乙醇和0.1%氯化汞消毒, 基本培养基主要用MS、WPM、QL等, 使用的细胞分裂素和生长素主要包括6-苄氨基腺嘌呤(6-BA)、萘乙酸(NAA)、6-糖基氨基嘌呤(KT)、2, 4-二氯苯氧乙酸(2, 4-D)、苯基噻二唑基脲(TDZ)、吲哚丁酸(IBA)等。朱丹红等[74]研究表明, 一年生枝条基部的叶片, 背面朝上放于培养基上诱导愈伤组织效果更佳。杨丽青等[78]研究表明, 果梅的一年生枝条不宜在刚抽生的时候采样, 枝条太幼嫩, 容易受灭菌剂影响而褐化死亡, 最好在新稍生长旺盛期采样, 一般在4至6月。杨洁等[79]研究表明, 未成熟合子胚在长3.5~5.0 mm的体胚诱导率和体胚诱导效果最佳。
Ning等[80]研究表明, 未成熟胚在以1/2 MS为基本培养基, 加入13.2 μmol·L-1 6-BA和2.7 μmol·L-1 NAA的培养基上诱导效果最好, 诱导率达到89.5%。Gao等[81]研究表明, 未成熟胚在以MS为基本培养基, 加入1 μmol·L-1 2, 4-D和1 μmol·L-1 6-BA的培养基上诱导效果最好。在组培和遗传转化过程中, 不论是外植体的取样时间、灭菌方法的交叉组合, 还是不同基本培养基以及不同的激素配比, 都是影响外植体生长和增殖的重要因素。
综上所述, 虽然梅组培和再生研究有些零星的报道, 也取得了一些经验, 初步获得了组培快繁的技术体系, 但组培苗的伸长生长和再生效率还存在很大问题, 尤其是梅遗传转化体系至今仍未建立。
6 问题与展望梅作为原产于中国的特色树种, 主要分布在中国和日本等东亚和东南亚地区。虽然果实具有较高的保健价值[82-83], 但因其为小水果, 投入的科研力量与大宗水果相比还相对比较薄弱。针对资源挖掘和分子生物学研究, 需重点开展以下研究工作。
1) 深度鉴定种质资源和挖掘优异基因。随着城镇化的进程加快, 越来越多栽植在城郊的梅园被毁, 随之而来的是资源的流失, 因此加快收集和保存资源应作为梅资源研究的重点工作。同时进一步深度鉴定已经收集的种质资源, 明确其演化和进化规律, 挖掘优异基因, 为生产和科研服务。例如梅具有一些优异性状, 如果实高酸低糖、休眠期短以及流胶病抗性等, 其相关基因资源都有待进一步的研究和挖掘利用。
2) 建立梅稳定高效的遗传转化体系。作为核果类果树的共性问题, 梅遗传转化体系一直未能建立, 这也是阻碍梅分子生物学研究水平进一步提升的瓶颈问题。只有利用高效的遗传转化体系才能进一步明确梅特有基因的功能。目前虽然已建立了梅的组培快繁体系, 但也存在繁殖系数不高、生根困难等问题。因此, 建立高效的遗传转化体系是今后梅研究的重点课题。
3) 自交亲和变异机制应深入研究。虽然梅是典型的自交不亲和树种, 如果授粉树配置不当, 会造成产量低和效益差的问题。但也存在自交亲和变异的类型和资源, 进一步揭示自交亲和的变异机制及演化关系, 并开发分子标记加快自交亲和专用品种的选育, 对于提高生产效率具有重要的意义。
4) 揭示梅短休眠的分子机制。梅为落叶果树休眠期最短的树种, 利用这一特点进行木本果树休眠解除机制的研究有利于揭示木本果树的休眠机制, 为果树短休眠分子标记的开发和进一步在生产中应用提供依据。
| [1] |
褚孟嫄. 中国果树志:梅卷[M]. 北京: 中国林业出版社, 1999. Chu M Y. China Fruit Tree:Mei[M]. Beijing: China Forestry Publishing House, 1999 (in Chinese). |
| [2] |
舒迎澜. 我国古代梅的分布利用与种植[J]. 中国农史, 1986, 5(3): 60-72. Shu Y L. Distribution and planting of ancient Prunus mume in China[J]. Agricultural History of China, 1986, 5(3): 60-72 (in Chinese). |
| [3] |
陈俊愉. 中国梅花研究的几个方面[J]. 北京林业大学学报, 1995, 17(S1): 1-7. Chen J Y. Some aspects on Chinese mei flowers research[J]. Journal of Beijing Forestry University, 1995, 17(S1): 1-7 (in Chinese with English abstract). |
| [4] |
张孝岳. 梅与梅文化[M]. 北京: 中国农业出版社, 2005. Zhang X Y. Mei and Mei Culture[M]. Beijing: China Agriculture Press, 2005 (in Chinese). |
| [5] |
陈红, 谭志刚. 中国果梅种质资源研究与利用[J]. 天津农业科学, 2011, 17(6): 108-110. Chen H, Tan Z G. Research and utilization of Prunus mume germplasm resources in China[J]. Tianjin Agricultural Sciences, 2011, 17(6): 108-110 (in Chinese with English abstract). DOI:10.3969/j.issn.1006-6500.2011.06.029 |
| [6] |
朱世阳, 应晓亮, 王晓东. 浅谈中国梅:梅花的野生资源与栽培分布[J]. 华东森林经理, 2012, 26(1): 52-55, 62. Zhu S Y, Ying X L, Wang X D. Discussion on the wild resources and cultivation distribution of Prunus mume in China[J]. East China Forest Management, 2012, 26(1): 52-55, 62 (in Chinese). |
| [7] |
侍婷, 张其林, 高志红, 等. 2个果梅品种雌蕊分化进程及相关生化指标分析[J]. 植物资源与环境学报, 2011, 20(4): 35-41. Shi T, Zhang Q L, Gao Z H, et al. Analyses on pistil differentiation process and related biochemical indexes of two cultivars of Prunus mume[J]. Journal of Plant Resources and Environment, 2011, 20(4): 35-41 (in Chinese with English abstract). DOI:10.3969/j.issn.1674-7895.2011.04.007 |
| [8] |
高志红, 章镇, 韩振海, 等. 中国果梅核心种质的构建与检测[J]. 中国农业科学, 2005, 38(2): 363-368. Gao Z H, Zhang Z, Han Z H, et al. Development and evaluation of core collection of Japanese apricot germplasms in China[J]. Scientia Agricultura Sinica, 2005, 38(2): 363-368 (in Chinese with English abstract). DOI:10.3321/j.issn:0578-1752.2005.02.023 |
| [9] |
高志红, 章镇, 韩振海. 果梅种质枝条抗寒性鉴定[J]. 果树学报, 2005, 22(6): 709-711. Gao Z H, Zhang Z, Han Z H. Identification of freezing resistance in Japanese apricot germplasm[J]. Journal of Fruit Science, 2005, 22(6): 709-711 (in Chinese with English abstract). |
| [10] |
侍婷, 高志红, 章镇, 等. 47个果梅品种开花生物学特性和花粉萌发率比较[J]. 中国农学通报, 2011, 27(4): 227-232. Shi T, Gao Z H, Zhang Z, et al. Comparison of biological traits of flowers and the rate of pollen germination among 47Prunus mume cultivars[J]. Chinese Agricultural Science Bulletin, 2011, 27(4): 227-232 (in Chinese with English abstract). |
| [11] |
邵静.果梅抑菌、抗癌活性及其有效成分的研究[D].南京: 南京农业大学, 2013. Shao J. Evaluation of the antibacterial and anticancer effect of Japanese apricot and the active substance[D]. Nanjing: Nanjing Agricultural University, 2013(in Chinese with English abstract). |
| [12] |
王培培, 侍婷, 高志红, 等. 中国原产果梅自交亲和变异品种花粉决定基因SFB的插入突变[J]. 园艺学报, 2012, 39(3): 453-460. Wang P P, Shi T, Gao Z H, et al. Insertion mutation of pollen SFB gene in self-compatibility of Japanese apricot cultivars native to China[J]. Acta Horticulturae Sinica, 2012, 39(3): 453-460 (in Chinese with English abstract). |
| [13] |
Wang P P, Gao Z H, Ni Z J, et al. Self-compatibility in 'Zaohong' Japanese apricot is associated with the loss of function of pollen S genes[J]. Molecular Biology Reports, 2013, 40(11): 6485-6493. DOI:10.1007/s11033-013-2765-2 |
| [14] |
Zhuang W B, Cai B H, Gao Z H, et al. Determination of chilling and heat requirements of 69 Japanese apricot cultivars[J]. European Journal of Agronomy, 2016, 74: 68-74. DOI:10.1016/j.eja.2015.10.006 |
| [15] |
王培培, 高志红, 章镇. 果梅自交不亲和相关基因型(S、SFB/SLF)研究进展[J]. 中国农学通报, 2012, 28(22): 119-123. Wang P P, Gao Z H, Zhang Z. Recent research advances of self-incompatibility genotypes (S and SFB/SLF) in Japanese apricot[J]. Chinese Agricultural Science Bulletin, 2012, 28(22): 119-123 (in Chinese with English abstract). |
| [16] |
Shen Y Y, Ding X J, Wang F, et al. Analysis of genetic diversity in Japanese apricot (Prunus mume Sieb. et Zucc.) based on REMAP and IRAP molecular markers[J]. Scientia Horticulturae, 2011, 132: 50-58. DOI:10.1016/j.scienta.2011.10.005 |
| [17] |
Xue S, Shi T, Luo W J, et al. Comparative analysis of the complete chloroplast genome among Prunus mume, P.armeniaca, and P.salicina[J]. Horticulture Research, 2019, 6: 89. DOI:10.1038/s41438-019-0171-1 |
| [18] |
Zhang Q X, Chen W B, Sun L D, et al. The genome of Prunus mume[J]. Nature Communications, 2012, 3: 1318. DOI:10.1038/ncomms2290 |
| [19] |
Sasaki R, Yamane H, Ooka T, et al. Functional and expressional analyses of PmDAM genes associated with endodormancy in Japanese apricot[J]. Plant Physiology, 2011, 157(1): 485-497. DOI:10.1104/pp.111.181982 |
| [20] |
Hao R J, Zhang Q, Yang W R, et al. Emitted and endogenous floral scent compounds of Prunus mume and hybrids[J]. Biochemical Systematics and Ecology, 2014, 54: 23-30. DOI:10.1016/j.bse.2013.12.007 |
| [21] |
Zhang Q X, Zhang H, Sun L D, et al. The genetic architecture of floral traits in the woody plant Prunus mume[J]. Nature Communications, 2018, 9: 1702. DOI:10.1038/s41467-018-04093-z |
| [22] |
Yang J B, Yang S X, Li H T, et al. Comparative chloroplast genomes of Camellia species[J]. PLoS One, 2013, 8(8): e73053. DOI:10.1371/journal.pone.0073053 |
| [23] |
Dyall S D. Ancient invasions:from endosymbionts to organelles[J]. Science, 2004, 304(5668): 253-257. DOI:10.1126/science.1094884 |
| [24] |
Umesono K, Inokuchi H, Ohyama K, et al. Nucleotide sequence of Marchantia polymorphachloroplast DNA:a region possibly encoding three tRNAs and three proteins including a homologue of E.coli ribosomal protein S14[J]. Nucleic Acids Research, 1984, 12(24): 9551-9565. DOI:10.1093/nar/12.24.9551 |
| [25] |
Wang S, Gao C W, Gao L Z. Plastid genome sequence of an ornamental and editable fruit tree of Rosaceae, Prunus mume[J]. Mitochondrial DNA Part A, 2016, 27(6): 4407-4408. DOI:10.3109/19401736.2015.1089546 |
| [26] |
李晓颖, 余金保, 王保根, 等. 南京梅花品种资源若干花性状的数量分布及评价[J]. 北京林业大学学报, 2012, 34(S1): 45-49. Li X Y, Yu J B, Wang B G, et al. Quantitative distribution and evaluation of some flower characteristics of Prunus mume cultivars[J]. Journal of Beijing Forestry University, 2012, 34(S1): 45-49 (in Chinese with English abstract). |
| [27] |
Gao Z H, Shi T, Luo X Y, et al. High-throughput sequencing of small RNAs and analysis of differentially expressed microRNAs associated with pistil development in Japanese apricot[J]. BMC Genomics, 2012, 13(1): 371. DOI:10.1186/1471-2164-13-371 |
| [28] |
Wang W X, Shi T, Ni X P, et al. The role of miR319a and its target gene TCP4 in the regulation of pistil development in Prunus mume[J]. Genome, 2018, 61(1): 43-48. DOI:10.1139/gen-2017-0118 |
| [29] |
Shi T, Zhuang W B, Zhang Z, et al. Comparative proteomic analysis of pistil abortion in Japanese apricot (Prunus mume Sieb. et Zucc)[J]. Journal of Plant Physiology, 2012, 169(13): 1301-1310. DOI:10.1016/j.jplph.2012.05.009 |
| [30] |
Sun H L, Shi T, Song J, et al. Pistil abortion in Japanese apricot (Prunus mume Sieb. et Zucc.):isolation and functional analysis of PmCCoAOMT gene[J]. Acta Physiologiae Plantarum, 2016, 38(5): 114. DOI:10.1007/s11738-016-2131-9 |
| [31] |
Song J, Gao Z H, Huo X M, et al. Genome-wide identification of the auxin response factor(ARF)gene family and expression analysis of its role associated with pistil development in Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Acta Physiologiae Plantarum, 2015, 37(8): 1-13. |
| [32] |
Hou J H, Gao Z H, Zhang Z, et al. Isolation and characterization of an AGAMOUS homologue PmAG from the Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Plant Molecular Biology Reporter, 2011, 29(2): 473-480. DOI:10.1007/s11105-010-0248-3 |
| [33] |
Zhou Y Z, Xu Z D, Yong X, et al. SEP-class genes in Prunus mume and their likely role in floral organ development[J]. BMC Plant Biology, 2017, 17: 10. DOI:10.1186/s12870-016-0954-6 |
| [34] |
侍婷.基于组学的果梅雌蕊败育分子机理[D].南京: 南京农业大学, 2014. Shi T. The molecular mechanism of pistil abortion based on omics in Japanese apricot[D]. Nanjing: Nanjing Agricultural University, 2014(in Chinese with English abstract). http://cdmd.cnki.com.cn/Article/CDMD-10307-1016037585.htm |
| [35] |
Tao R, Habu T, Yamane H, et al. Molecular markers for self-compatibility in Japanese apricot (Prunus mume)[J]. HortScience, 2000, 35(6): 1121-1123. DOI:10.21273/HORTSCI.35.6.1121 |
| [36] |
Yaegaki H, Shimada T, Moriguchi T, et al. Molecular characterization of S-RNase genes and S-genotypes in the Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Sexual Plant Reproduction, 2001, 13(5): 251-257. DOI:10.1007/s004970100064 |
| [37] |
Matsumoto D, Tao R. Recognition of a wide-range of S-RNases by S locus F-box like 2, a general-inhibitor candidate in the Prunus-specific S-RNase-based self-incompatibility system[J]. Plant Molecular Biology, 2016, 91(4/5): 459-469. |
| [38] |
East E M, Mangelsdorf A J. A new interpretation of the hereditary behavior of self-sterile plants[J]. Proc Natl Acad Sci USA, 1925, 11(2): 166-171. DOI:10.1073/pnas.11.2.166 |
| [39] |
Tao R, Habu T, Yamane H, et al. Characterization and cDNA cloning for Sf-RNase, a molecular marker for self-compatibility, in Japanese apricot (Prunus mume)[J]. Engei Gakkai Zasshi, 2002, 71(5): 595-600. DOI:10.2503/jjshs.71.595 |
| [40] |
Habu T, Matsumoto D, Fukuta K, et al. Cloning and characterization of twelve S-RNase alleles in Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Journal of the Japanese Society for Horticultural Science, 2008, 77(4): 374-381. DOI:10.2503/jjshs1.77.374 |
| [41] |
Entani T, Iwano M, Shiba H, et al. Comparative analysis of the self-incompatibility(S-)locus region of Prunus mume:identification of a pollen-expressed F-box gene with allelic diversity[J]. Genes to Cells, 2003, 8(3): 203-213. DOI:10.1046/j.1365-2443.2003.00626.x |
| [42] |
Heng W, Wu H Q, Chen Q X, et al. Identification of S-genotypes and novel S-RNase alleles in Prunus mume[J]. The Journal of Horticultural Science and Biotechnology, 2008, 83(6): 689-694. DOI:10.1080/14620316.2008.11512445 |
| [43] |
Xu J X, Gao Z H, Zhang Z. Identification of S-genotypes and novel S-RNase alleles in Japanese apricot cultivars native to China[J]. Scientia Horticulturae, 2010, 123(4): 459-463. DOI:10.1016/j.scienta.2009.11.005 |
| [44] |
Wang P P, Shi T, Zhuang W B, et al. Determination of S-RNase genotypes and isolation of four novel S-RNase genes in Japanese apricot (Prunus mume Sieb. et Zucc.)native to China[J]. The Journal of Horticultural Science and Biotechnology, 2012, 87(3): 266-270. DOI:10.1080/14620316.2012.11512863 |
| [45] |
Gao Z H, Wang P P, Zhuang W B, et al. Sequence analysis of new S-RNase and SFB alleles in Japanese apricot (Prunus mume)[J]. Plant Molecular Biology Reporter, 2013, 31(3): 751-762. DOI:10.1007/s11105-012-0535-2 |
| [46] |
Wang P P, Gao Z H, Ni Z J, et al. Isolation and identification of new pollen-specific SFB genes in Japanese apricot (Prunus mume)[J]. Genetics and Molecular Research:GMR, 2013, 12(3): 3286-3295. DOI:10.4238/2013.September.3.5 |
| [47] |
Ushijima K, Yamane H, Watari A, et al. The S haplotype-specific F-box protein gene, SFB, is defective in self-compatible haplotypes of Prunus avium and P.mume[J]. The Plant Journal, 2004, 39(4): 573-586. DOI:10.1111/j.1365-313X.2004.02154.x |
| [48] |
Yamane H, Fukuta K, Matsumoto D, et al. Characterization of a novel self-compatible S3' haplotype leads to the development of a universal PCR marker for two distinctly originated self-compatible S haplotypes in Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Journal of the Japanese Society for Horticultural Science, 2009, 78(1): 40-48. DOI:10.2503/jjshs1.78.40 |
| [49] |
Heng W, Wu J, Wu H Q, et al. Identification and characterisation of SFBs in Prunus mume[J]. Plant Molecular Biology Reporter, 2012, 30(4): 878-884. DOI:10.1007/s11105-011-0392-4 |
| [50] |
Lang G. Endo-, para-, and eco-dormancy:physiological terminology and classification for dormancy research[J]. HortScience, 1987, 22: 371-377. |
| [51] |
Zhang Z Y, Zhuo X K, Zhao K, et al. Transcriptome profiles reveal the crucial roles of hormone and sugar in the bud dormancy of Prunus mume[J]. Scientific Reports, 2018, 8: 5090. DOI:10.1038/s41598-018-23108-9 |
| [52] |
da Silveira Falavigna V, Miotto Y E, Porto D D, et al. Functional diversification of the dehydrin gene family in apple and its contribution to cold acclimation during dormancy[J]. Physiologia Plantarum, 2015, 155(3): 315-329. DOI:10.1111/ppl.12338 |
| [53] |
Koornneef M, Léon-Kloosterziel K M, Schwartz S H, et al. The genetic and molecular dissection of abscisic acid biosynthesis and signal transduction in Arabidopsis[J]. Plant Physiology and Biochemistry, 1998, 36(1/2): 83-89. |
| [54] |
Zheng C L, Halaly T, Acheampong A K, et al. Abscisic acid(ABA)regulates grape bud dormancy, and dormancy release stimuli may act through modification of ABA metabolism[J]. Journal of Experimental Botany, 2015, 66(5): 1527-1542. DOI:10.1093/jxb/eru519 |
| [55] |
Zhuang W B, Gao Z H, Wang L J, et al. Comparative proteomic and transcriptomic approaches to address the active role of GA4 in Japanese apricot flower bud dormancy release[J]. Journal of Experimental Botany, 2013, 64(16): 4953-4966. DOI:10.1093/jxb/ert284 |
| [56] |
Wen L H, Zhong W J, Huo X M, et al. Expression analysis of ABA-and GA-related genes during four stages of bud dormancy in Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. The Journal of Horticultural Science and Biotechnology, 2016, 91(4): 362-369. DOI:10.1080/14620316.2016.1160546 |
| [57] |
Griffiths J, Murase K, Rieu I, et al. Genetic characterization and functional analysis of the GID1 gibberellin receptors in Arabidopsis[J]. The Plant Cell, 2006, 18(12): 3399-3414. DOI:10.1105/tpc.106.047415 |
| [58] |
Dill A, Thomas S G, Hu J H, et al. The Arabidopsis F-box protein SLEEPY1 targets gibberellin signaling repressors for gibberellin-induced degradation[J]. The Plant Cell, 2004, 16(6): 1392-1405. DOI:10.1105/tpc.020958 |
| [59] |
Lv L, Huo X M, Wen L H, et al. Isolation and role of PmRGL2 in GA-mediated floral bud dormancy release in Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. Frontiers in Plant Science, 2018, 9: 27. DOI:10.3389/fpls.2018.00027 |
| [60] |
Zhao K, Zhou Y Z, Ahmad S, et al. Comprehensive cloning of Prunus mume dormancy associated MADS-box genes and their response in flower bud development and dormancy[J]. Frontiers in Plant Science, 2018, 9: 17. DOI:10.3389/fpls.2018.00017 |
| [61] |
Zhao K, Zhou Y Z, Ahmad S, et al. PmCBFs synthetically affect PmDAM6 by alternative promoter binding and protein complexes towards the dormancy of bud for Prunus mume[J]. Scientific Reports, 2018, 8: 4527. DOI:10.1038/s41598-018-22537-w |
| [62] |
Zhao K, Zhou Y Z, Li Y S, et al. Crosstalk of PmCBFs and PmDAMs based on the changes of phytohormones under seasonal cold stress in the stem of Prunus mume[J]. International Journal of Molecular Sciences, 2018, 19(2): 15. DOI:10.3390/ijms19020015 |
| [63] |
Kitamura Y, Habu T, Yamane H, et al. Identification of QTLs controlling chilling and heat requirements for dormancy release and bud break in Japanese apricot (Prunus mume)[J]. Tree Genetics & Genomes, 2018, 14(2): 33. |
| [64] |
Yamane I H, Wada M, Honda C, et al. Overexpression of Prunus DAM6 inhibits growth, represses bud break competency of dormant buds and delays bud outgrowth in apple plants[J]. PLoS One, 2019, 14(4): e0214788. DOI:10.1371/journal.pone.0214788 |
| [65] |
张学宁, 郭保林, 张开春. 果树分子标记研究进展[J]. 河北农业大学学报, 2003, 26(S1): 75-78. Zhang X N, Guo B L, Zhang K C. Research advances on molecular markers in fruit trees[J]. Journal of Hebei Agricultural University, 2003, 26(S1): 75-78 (in Chinese with English abstract). |
| [66] |
Yamane H, Ushijima K, Sassa H, et al. The use of the S haplotype-specific F-box protein gene, SFB, as a molecular marker for S-haplotypes and self-compatibility in Japanese apricot (Prunus mume)[J]. Theoretical and Applied Genetics, 2003, 107(8): 1357-1361. DOI:10.1007/s00122-003-1389-7 |
| [67] |
Li X Y, Wang C, Yang G, et al. Employment of a new strategy for identification of Prunus mume cultivars using random amplified polymorphic deoxyribonucleic acid(RAPD)markers[J]. African Journal of Plant Science, 2011, 5(9): 500-509. |
| [68] |
Fang J G, Qiao Y S, Zhang Z, et al. Genotyping fruiting mei (Prunus mume Sieb. et Zucc.)cultivars using amplified fragment-length polymorphism markers[J]. HortScience, 2005, 40(2): 325-328. DOI:10.21273/HORTSCI.40.2.325 |
| [69] |
高志红.果梅核心种质的构建与分子标记的研究[D].北京: 中国农业大学, 2003. Gao Z H. Core collection and molecular marker of Japanese apricot (Prunus mume Sieb. et Zucc.)[D]. Beijing: China Agricultural University, 2003(in Chinese with English abstract). |
| [70] |
Li X Y, Wang B, Wang C, et al. Genetic relationships between fruiting and flowering mei (Prunus mume) cultivars using SNP markers[J]. The Journal of Horticultural Science and Biotechnology, 2010, 85(4): 329-334. DOI:10.1080/14620316.2010.11512676 |
| [71] |
Du D L, Zhang Q X, Cheng T R, et al. Genome-wide identification and analysis of late embryogenesis abundant (LEA) genes in Prunus mume[J]. Molecular Biology Reports, 2013, 40(2): 1937-1946. DOI:10.1007/s11033-012-2250-3 |
| [72] |
Sun L D, Wang Y Q, Yan X L, et al. Genetic control of juvenile growth and botanical architecture in an ornamental woody plant, Prunus mume Sieb. et Zucc. as revealed by a high-density linkage map[J]. BMC Genetics, 2014, 15(Suppl 1): S1. DOI:10.1186/1471-2156-15-S1-S1 |
| [73] |
Zhang J, Zhang Q X, Cheng T R, et al. High-density genetic map construction and identification of a locus controlling weeping trait in an ornamental woody plant (Prunus mume Sieb. et Zucc.)[J]. DNA Research, 2015, 22(3): 183-191. DOI:10.1093/dnares/dsv003 |
| [74] |
朱丹红, 杨炜茹, 李媛, 等. 真梅系梅花'三轮玉蝶'叶片再生体系的初步建立[J]. 西北农林科技大学学报(自然科学版), 2013, 41(7): 209-216. Zhu D H, Yang W R, Li Y, et al. Preliminary establishment of the regeneration systems for Prunus mume 'Sanlun Yudie' leaf-explants[J]. Journal of Northwest A & F University(Natural Science Edition), 2013, 41(7): 209-216 (in Chinese with English abstract). |
| [75] |
宁国贵, 吕海燕, 张俊卫, 等. 梅花不同外植体离体培养及体细胞胚诱导植株再生[J]. 园艺学报, 2010, 37(1): 114-120. Ning G G, Lü H Y, Zhang J W, et al. In vitro culture of different explants and plant regeneration via embryogenesis from immature cotyledons of Prunus mume[J]. Acta Horticulturae Sinica, 2010, 37(1): 114-120 (in Chinese with English abstract). |
| [76] |
胡淑英, 张春红, 王小敏, 等. 滨梅组培苗玻璃化成因及其克服技术研究[J]. 中国南方果树, 2013, 42(5): 37-41. Hu S Y, Zhang C H, Wang X M, et al. Study on the causes and prevention methods of vitrification in tissue culture of beach plum (Prunus maritima Marshall)[J]. South China Fruits, 2013, 42(5): 37-41 (in Chinese with English abstract). |
| [77] |
燕志, 张焕仕, 王猛, 等. 滨梅茎段组培污染率控制方法的研究[J]. 中国南方果树, 2013, 42(6): 9-13. Yan Z, Zhang H S, Wang M, et al. Studies on the methods for control of contamination in tissue culture of Prunus maritima stems[J]. South China Fruits, 2013, 42(6): 9-13 (in Chinese with English abstract). |
| [78] |
杨丽青, 杨洁, 毛庆山, 等. '小绿萼'梅离体快繁体系的建立[J]. 江苏农业科学, 2015, 43(4): 62-64. Yang L Q, Yang J, Mao Q S, et al. Establishment of rapid propagation system in vitro of Prunus mume 'Xiaolüe'[J]. Jiangsu Agricultural Sciences, 2015, 43(4): 62-64 (in Chinese). |
| [79] |
杨洁, 闻娟, 晏晓兰, 等. '雪梅'未成熟合子胚体胚发生与植株再生[J]. 北京林业大学学报, 2013, 35(S1): 21-24. Yang J, Wen J, Yan X L, et al. Somatic embryogenesis and plant regeneration from immature zygotic embryos of Prunus mume 'Xuemei'[J]. Journal of Beijing Forestry University, 2013, 35(S1): 21-24 (in Chinese with English abstract). |
| [80] |
Ning G G, Bai S P, Bao M Z, et al. Factors affecting plantlet regeneration from in vitro cultured immature embryos and cotyledons of Prunus mume 'Xuemei'[J]. In Vitro Cellular & Developmental Biology-Plant, 2007, 43(3): 225-230. |
| [81] |
Gao M, Kawabe M, Tsukamoto T, et al. Somatic embryogenesis and Agrobacterium-mediated transformation of Japanese apricot (Prunus mume) using immature cotyledons[J]. Scientia Horticulturae, 2010, 124(3): 360-367. DOI:10.1016/j.scienta.2010.01.021 |
| [82] |
Go M R, Kim H J, Yu J, et al. Toxicity and toxicokinetics of amygdalin in maesil (Prunus mume) syrup:protective effect of maesil against amygdalin toxicity[J]. Journal of Agricultural and Food Chemistry, 2018, 66(43): 11432-11440. DOI:10.1021/acs.jafc.8b03686 |
| [83] |
Jang A J, Lee J H, Yotsu-Yamashita M, et al. A novel compound, FA-1 isolated from Prunus mume, protects human bronchial epithelial cells and keratinocytes from cigarette smoke extract-induced damage[J]. Science Reports, 2018, 8: 11504. DOI:10.1038/s41598-018-29701-2 |




