文章信息
- 刘金定, 王权, 黄水清, 沈其荣
- LIU Jinding, WANG Quan, HUANG Shuiqing, SHEN Qirong
- 基于RNA-seq的木霉长链非编码RNA的生物信息学预测及其重寄生相关性分析
- Bioinformatics prediction of long noncoding RNA of Trichoderma guizhouense based on strand-specific RNA-seq and their potential association with mycoparasitism
- 南京农业大学学报, 2019, 42(5): 877-886
- Journal of Nanjing Agricultural University, 2019, 42(5): 877-886.
- http://dx.doi.org/10.7685/jnau.201812054
-
文章历史
- 收稿日期: 2018-12-30
2. 南京农业大学信息科学技术学院/领域知识关联研究中心, 江苏 南京 210095
2. College of Information Science and Technology/Research Center for Correlation of Domain Knowledge, Nanjing Agricultural University, Nanjing 210095, China
真核生物大部分基因组序列都可以被转录为能够翻译成蛋白序列的信使RNA(message RNA, mRNA)和不能翻译成蛋白的非编码RNA(noncoding RNA, ncRNA)[1]。长链非编码RNA(long noncoding RNA, lncRNA)是转录本长度超过200 nt且不具有蛋白编码潜能的一类非编码RNA分子。lncRNA广泛存在于包括低等生物酵母和高等哺乳动物在内的各种真核生物体内, 与多种生物过程有关。lncRNA可以作为互作分子的招募者、系结者、诱捕者和信号分子, 通过表观遗传、转录和翻译等形式发挥其调控功能[2]。lncRNA对靶基因调控可分为顺式(cis-)和反式(trans-)2种作用模式。lncRNA对靶基因的调控既可以是正向调控也可以是负向调控, 这与非编码微小RNA(micro RNA, miRNA)(以负调控为主的调控方式)是有明显区别的。lncRNA还可作为miRNA的靶标, 通过竞争结合miRNA从而影响miRNA对基因的沉默[3]。目前, 对动物lncRNA研究表明, lncRNA能够辅助蛋白质转运、参与等位基因和染色体的失活调控, 在免疫应答、胚胎发育、脂肪沉积以及肌肉生长等生物过程中发挥重要作用[4]。在植物中许多lncRNA对植物春化、根瘤(固氮豆科)形成、花粉发育、侧根发育、抗病性、非生物胁迫反应等方面具有调控功能[5]。真菌lncRNA研究刚刚起步, 其功能研究主要集中于酵母菌, lncRNA能参与丝氨酸、半乳糖以及核酸合成以应对营养匮乏等胁迫性环境[6-7]。在全基因组范围内对灵芝(Ganoderma lucidum)[8]和禾谷镰刀菌(Fusarium graminearum)[9]的lncRNA研究结果表明, lncRNA对真菌生长发育具有重要的调控作用。在里氏木霉(Trichoderma reesei)中发现1个名为HAX1的lncRNA[10], 该lncRNA能有效调控纤维素酶的表达, 这对工业生产具有十分重要的意义。
在农业生产中, 木霉菌能够促进作物生长且资源丰富, 对发展可持续农业具有重要作用。现有研究表明, 多种碳水化合物活性酶、蛋白酶以及次生代谢物在生防木霉菌对病原菌重寄生过程中发挥了重要调控作用[11]。lncRNA在重寄生过程中是否发挥调控作用, 目前尚未见报道。木霉菌(Trichoderma guizhouense)NJAU 4742(NJAU 4742)对多种病原菌有很强的寄生能力, 是重寄生分子机制研究的理想材料[12]。本研究利用链特异性RNA-seq技术对重寄生互作前、后以及独立培养的木霉菌进行转录组深度测序, 利用生物信息学方法在全基因组范围内识别lncRNA并分析其靶标和表达情况, 为重寄生分子机制研究提供理论依据和参考。
1 材料与方法 1.1 菌株及试剂木霉NJAU 4742和尖孢镰刀菌古巴专化型4号生理小种(Fusarium oxysporum f. sp. cubense 4, Foc4)均保存于本实验室。将菌落直径约为4 mm的木霉菌株和尖孢镰刀菌株分别接种在PDA平板的两侧(距平板边缘1 cm), 以2株菌独立培养为对照, 置于28 ℃恒温培养。分别收集对峙接触前(相距1 cm)木霉生长前沿1 cm区域内的菌丝(样品设为BC)、对峙接触后1 cm互作区内的混合菌丝(AC)以及独立培养木霉菌生长前沿1 cm区域内的菌丝(IC), 迅速用液氮冷冻, 放入-80 ℃冰箱保存备用。
本试验用RNeasy Plant Mini Kit(Tiangen)提取样品菌丝总RNA, 用DNase I(Qiagen, UK)对DNA进行消化处理, 用生物分析仪(Agilent)检测RNA质量和完整性。质检合格后用Ribo-Zero rRNA Removal Kit(Epicentre, Madison, USA)去除核糖体rRNA, 然后用NEBNextⓇ UltraTm Directional RNA Library Prep Kit(NEB, USA)构建链特异性文库, 最后用Illumina HiSeq 4000测序仪对构建的文库执行双端150 bp测序。
1.2 识别长链非编码RNA采用Trimmomatic软件[13]对每个文库的测序数据进行处理, 剔除低质量或者包含测序接头的读段(reads), 确保每个文库测序数据干净, 质量可靠。用Hisat2软件[14]将每个文库中有效读段(clean reads)与NJAU 4742参考基因组进行比对, 参考基因组来自NCBI基因组数据库, 编号为LVVK00000000.1。
为了识别菌株NJAU 4742的lncRNA, 设计生物信息学流程(图 1):1)用StringTie软件[15]将每个文库比对结果组装起来, 获得转录本结构; 2)用Cufflinks软件包[16]中的Cuffcompare将6个文库组装结果合并, 剔除与蛋白编码基因重叠的转录本; 3)剔除序列长度小于200 nt或含有大于300 nt ORF框的转录本; 4)将剩余的转录本在NCBI-NR数据库中进行BLASTx比对, 剔除与数据库中蛋白序列同源的转录本; 5)将剩余的转录本在Pfam数据库[17]中进行比对, 剔除包含保守结构域(domain)或者模体(motif)的转录本; 6)用编码潜能计算器CPC[18]评估剩余转录本编码潜能, 剔除具有较高编码潜能的转录本(CPC < 0);7)用Rfam数据库[19]剔除rRNA、tRNA、snoRNA和snRNA等持家非编码RNA; 8)剔除4个以上样本中表达丰度都低于0.5 FPKM的转录本。
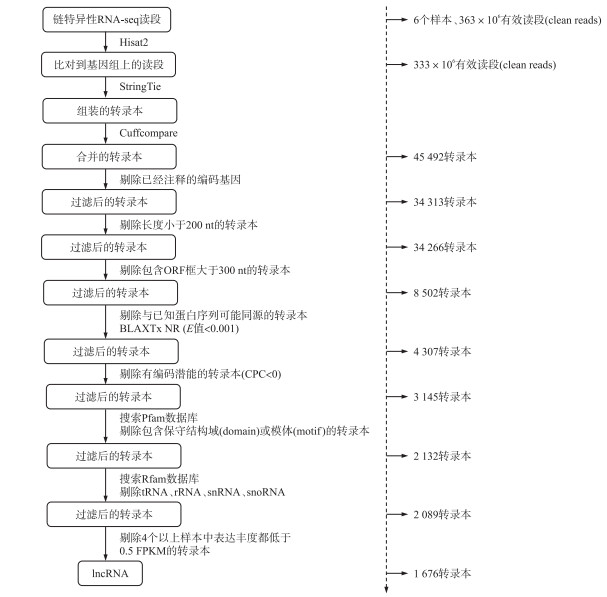
|
图 1 lncRNA识别流程 Fig. 1 The flowchart for identifying lncRNA |
根据RNA-seq序列比对结果, 利用RSEM软件[20]评估转录本表达丰度, 表达丰度单位为FPKM(fragments per kilobase of transcript per million fragments mapped)。DESeq2软件[21]用于分析不同样本中转录本差异表达情况, 将P < 0.05且差异倍数不低于2倍的转录本确定为差异表达转录本。转录本之间表达相关性用皮尔逊相关系数评估。
1.4 碳水化合物活性酶、蛋白酶以及次级代谢产物注释利用在线dbCAN2数据库[22]注释木霉中的碳水化合物活性酶基因, 过滤参数E值和Coverage分别设置为1×10-15和0.35。用BLASTp比对MEROPS数据库[23]中的蛋白酶序列, 放弃一致率小于35%、E值大于1×10-5、得分小于30的比对结果。用SMURF软件[24]识别木霉中的次生代谢合成相关的非核糖体多肽合成酶和聚酮合成酶基因。
1.5 靶标预测lncRNA的靶标分为顺式作用(cis-acting)靶标和反式作用(trans-acting)靶标两大类。顺式作用靶标指在DNA水平上lncRNA上、下游5 kb范围内的编码基因。在RNA水平上筛选lncRNA的反式作用靶标, 利用BLASTx分析lncRNA与mRNA序列互补性, 然后用RNA plex软件[25]评估两者结合的稳定性。
2 结果与分析 2.1 木霉链特异性RNA-seq测序与评估分别收集对峙接触前木霉菌丝(BC)、对峙接触后互作区内混合菌丝(AC)以及独立培养木霉菌丝(IC)用于链特异性转录组测序。每个样品2个重复, 分别标注为BC(1/2)、AC(1/2)和IC(1/2)。6个文库测序后总测序序列数量为363×106, 质控处理后有效序列总数为333×106(表 1)。在IC(1/2)和BC(1/2)文库中, 每个文库均有超过97%的有效序列能够与木霉参考基因组进行比对(表 1)。在AC1和AC2文库中, 只有43%和47%的有效序列能够与木霉参考基因组比对, 并且没有发现能同时与NJAU 4742和Foc4基因组比对的测序序列, 这说明通过与参考基因组比对可以将2个物种转录组测序序列分开。尽管AC1和AC2中与木霉基因组上的比对序列比例少于50%, 但是比对上的序列在基因组上的覆盖度都超过了100层(X)。
| 指标 Index |
IC1 | IC2 | BC1 | BC2 | AC1 | AC2 |
| 原始读段数 No. of raw reads |
49 562 212 | 44 009 108 | 63 639 198 | 76 265 382 | 66 112 358 | 63 856 866 |
| 有效读段数 No. of clean reads |
47 475 706 | 42 406 903 | 57 363 987 | 68 665 617 | 59 661 339 | 57 443 964 |
| 比对比例/% Mapping rate | 98 | 98 | 97 | 97 | 43 | 47 |
| 测序覆盖度(X) Sequencing coverage | 183 | 163 | 219 | 262 | 101 | 107 |
| 注:IC1、IC2分别表示独立培养木霉菌丝样品; BC1、BC2分别表示对峙接触前木霉菌丝样品; AC1、AC2分别表示对峙接触后木霉和镰刀菌混合菌丝样品。下同。 Note:IC1 and IC2 indicate 2 Trichodema hypha samples independently cultured; BC1 and BC2 indicate 2 Trichodema hypha samples before contacting with Fusarium; AC1 and AC2 indicate 2 hypha samples from the zone of interaction of Trichodema after contacting with Fusarium. The same as follows. |
||||||
根据所有编码基因的表达丰度, 对生物学重复进行相关性分析。样品的2个生物重复间的皮尔逊相关系数分别为0.99(IC)、0.97(AC)和0.98(BC), 这表明样品的生物重复间具有很好的一致性。
2.2 全基因组范围的lncRNA鉴定全基因组范围内共识别了1 676个lncRNA。根据lncRNA相对于编码基因的位置, 可将lncRNA分为4类:1)1 049个基因间型lncRNA(intergenic lncRNA), 约占总数62.6%, 位于2个编码基因之间; 2)590个反义型lncRNA(antisense lncRNA), 约占总数35.2%, 位于编码基因反义链上且和编码基因部分或者全部外显子重叠; 3)32个正义型lncRNA(sense lncRNA), 约占总数的1.9%, 位于编码基因同义链上, 与部分外显子重叠; 4)5个内含子型lncRNA(intronic lncRNA), 低于总数的1%, 位于编码基因内含子内。
2.3 木霉lncRNA的特征分析与动植物lncRNA相似, 木霉lncRNA的结构和编码基因也显著不同。木霉lncRNA外显子数量明显低于编码基因, 单外显子lncRNA占总数的78%, 而单外显子编码基因只占总数的23%(图 2-A)。lncRNA平均外显子长度比编码基因短, 前者为422 bp, 后者为734 bp(图 2-B)。lncRNA的外显子数量少而且序列短的特征决定其转录本序列长度也偏短。此外, lncRNA在基因组上跨度明显短于蛋白编码基因(图 2-C)。与编码基因相比, lncRNA的表达水平也明显偏低, 在IC、BC和AC样品中lncRNA平均表达水平分别为6.12、5.81和6.06 FPKM, 而编码基因平均表达水平分别为46.3、49.1和49.2 FPKM(图 2-D)。
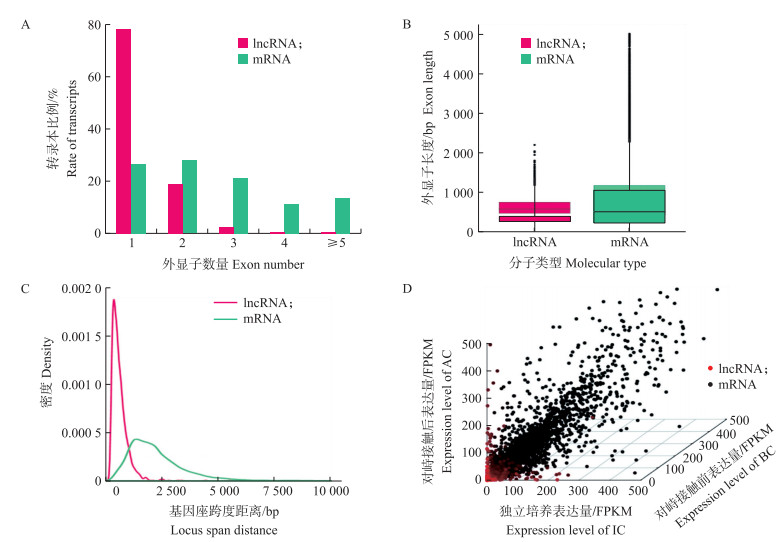
|
图 2 木霉lncRNA和mRNA特征比较 Fig. 2 Comparison of feature between lncRNA of Trichoderma guizhouense and mRNA |
lncRNA能够通过染色体重塑与编码基因互作, 倾向与临近编码基因共表达[26]。在1 492个lncRNA的上、下游5 kb范围内共发现了2 262个编码基因, 构成2 765对顺式作用调控关系。lncRNA和顺式作用靶标共表达的平均皮尔逊相关系数为0.128 021, 显著高于2个随机选择lncRNA间的平均皮尔逊相关系数(0.002 223 59), 也显著高于lncRNA和不相邻编码基因间的平均皮尔逊相关系数(0.005 478 77)。在这2 765对顺式作用调控关系中, 285个lncRNA调控其同义链上游靶标(targets on upstream sense strand, TUSS), 592个lncRNA调控其同义链下游靶标(targets on downstream sense strand, TDSS), 865个lncRNA调控其上游反义链靶标(targets on upstream antisense strand, TUAS), 371个lncRNA调控其下游反义链靶标(targets on downstream antisense strand, TDAS), 23个lncRNA调控其同义链上有重叠的靶标(targets overlapped on sense strand, TOSS), 629个lncRNA调控其反义链上有重叠的靶标(targets overlapped on antisense strand, TOAS)(图 3)。在6类调控关系中, TUAS和TOAS的数量最多, 并且lncRNA与靶标共表达的相关性最高, 对应的平均皮尔逊相关系数分别为0.170 588和0.151 358。另外, 有4个lncRNA预测到7个反式作用靶标基因, 这些lncRNA和反式作用靶标基因都不在同一条参考基因组序列上。
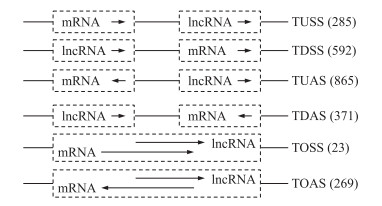
|
图 3 lncRNA和相邻靶标基因共表达的皮尔逊相关性系数分布 Fig. 3 The distribution of Pearson product-moment correlation coefficient between lncRNA and target genes TUSS:同义链上游靶标Targets on upstream sense strand; TDSS:同义链下游靶标Targets on downstream sense strand; TUAS:上游反义链靶标Targets on upstream antisense strand; TDAS:下游反义链靶标Targets on downstream antisense strand; TOSS:同义链重叠靶标Targets overlapped on sense strand; TOAS:反义链重叠靶标Targets overlapped on antisense strand. |
分别从生物学功能(biological process)、细胞组成(cellular component)和分子功能(molecular function)方面对lncRNA靶标基因进行功能分类, 共计有1 713个靶标基因。分析结果表明, lncRNA靶标基因在GO(gene ontology)第2层可分为29个类别, 其中代谢过程(metabolic process)、催化活性(catalytic activity)和细胞过程(cellular process)分布的靶标基因数量最多(图 4)。
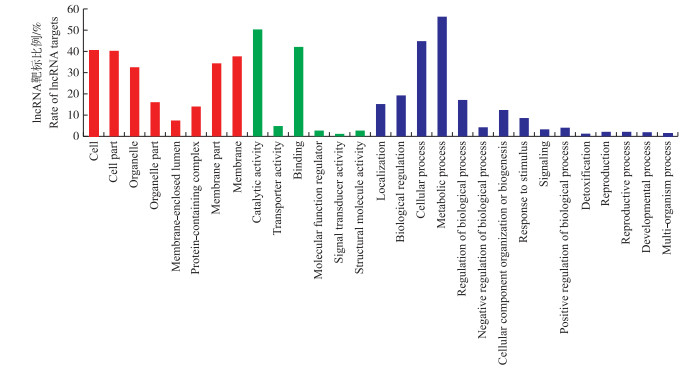
|
图 4 lncRNA靶标基因GO分类 Fig. 4 Gene ontology(GO)classifications for lncRNA targets Cell:细胞; Cell part:细胞部件; Organelle:细胞器; Organelle part:细胞器部件; Membrane-enclosed lumen:膜封闭腔; Protein-containing complex:含蛋白复合物; Membrane part:膜部件; Membrane:膜; Catalytic activity:催化活性; Transporter activity:转运活性; Binding:绑定; Molecular function regulator:分子功能调控; Signal transducer activity:信号转导活性; Structural molecule activity:结构分子活性; Localization:定位; Biological regulation:生物调控; Cellular process:细胞过程; Metabolic process:代谢过程; Regulation of biological process:生物过程调控; Negative regulation of biological process:生物过程负调控; Cellular component organization or biogenesis:细胞成分组织或生物发生; Response to stimulus:刺激响应; Signaling:信号; Positive regulation of biological process:生物过程正调控; Detoxification:解毒; Reproduction:生殖; Reproductive process:生殖过程; Developmental process:发育过程; Multi-organism process:多生物过程。 |
进一步用KEGG信号通路对lncRNA靶标基因进行注释分类, 共发现768个靶标基因在KEGG信号通路上。注释的靶标基因数量最多的前3类通路为信号转导(signal transduction)、转运与分解代谢(transport and catabolism)和碳水化合物代谢(carbohydrate metabolism)(图 5)。
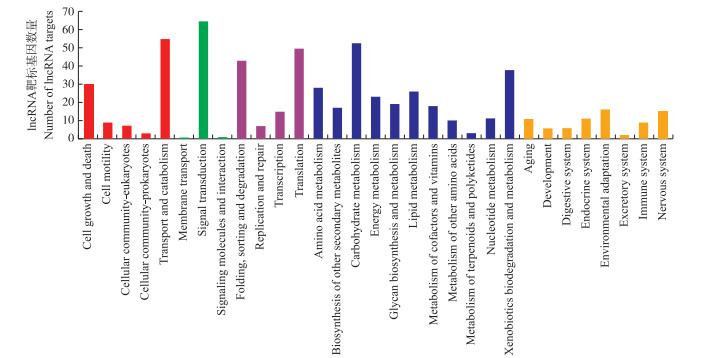
|
图 5 lncRNA靶标基因的KEGG功能注释分类 Fig. 5 The categories of KEGG function annotation of lncRNA targets Cell growth and death:细胞生长和死亡; Cell motility:细胞运动; Cellular community-eukaryotes:真核细胞群落; Cellular community-prokaryotes:原核细胞群落; Transport and catabolism:转运和代谢; Membrane transport:膜运输; Signal transduction:信号转导; Signaling molecules and interaction:信号分子和互作; Folding, sorting and degradation:折叠、整理和降解; Replication and repair:复制和修复; Transcription:转录; Translation:翻译; Amino acid metabolism:氨基酸代谢; Biosynthesis of other secondary metabolites:其他次生代谢产物合成; Carbohydrate metabolism:碳水化合物代谢; Energy metabolism:能量代谢; Glycan biosynthesis and metabolism:聚糖生物合成与代谢; Lipid metabolism:脂代谢; Metabolism of cofactors and vitamins:辅助因子和维生素代谢; Metabolism of other amino acids:其他氨基酸代谢; Metabolism of terpenoids and polyketides:萜类化合物和聚酮类化合物的代谢; Nucleotide metabolism:核苷酸代谢; Xenobiotics biodegradation and metabolism:异种生物的生物降解和代谢; Aging:老化; Development:发育; Digestive system:消化系统; Endocrine system:内分泌系统; Environmental adaptation:环境适应; Excretory system:排泄系统; Immune system:免疫系统; Nervous system:神经系统。 |
碳水化合物活性酶、蛋白酶及次生代谢物在重寄生过程中发挥了重要的作用[11]。顺式靶标预测结果显示:147个lncRNA能够顺式靶向编码90个碳水化合物活性酶、58个蛋白酶和1个与次生代谢物合成相关基因(表 2)。
| 靶标类别 Class of targets |
靶标数 Number of targets |
| 碳水化合物活性酶 Carbohydrate active enzymes(CAZy) |
|
| 辅助酶 Auxiliary activities(Aa) |
13 |
| 碳水化合物结合模块 Carbohydrate-binding modules(CBM) |
1 |
| 糖类酯酶 Carbohydrate esterases(Ce) |
3 |
| 糖苷水解酶 Glycoside hydrolases(GH) |
47 |
| 糖基转移酶 Glycosyl transferases(GT) |
24 |
| 多糖裂解酶 Polysaccharide lyases(PL) |
2 |
| 蛋白酶 Proteases |
|
| 天冬氨酸 Aspartic |
5 |
| 半胱氨酸 Cysteine |
10 |
| 谷氨酸 Glutamic |
2 |
| 金属肽酶 Metallo |
13 |
| 混合酶 Mixed |
1 |
| 丝氨酸 Serine |
25 |
| 苏氨酸 Threonine |
2 |
| 次生代谢 Secondary metabolism |
|
| 聚酮合成酶 Polyketide synthases(PKS) |
1 |
将147个lncRNA在BC和AC样品中的表达量分别与IC样品中的表达量进行比较, 发现30个lncRNA在重寄生过程中表达水平发生显著改变(|log2FC|>1, P < 0.05), 其中包含17个lncRNA靶向编码碳水化合物活性酶基因、12个靶向编码蛋白酶基因、1个靶向与次生代谢物合成相关基因(表 3)。在30个差异表达的lncRNA中, 有10个lncRNA在互作接触前表达水平发生显著改变, 有24个lncRNA在互作接触后表达水平发生显著改变, 有5个lncRNA在互作接触前、后表达水平都发生改变(表 3)。
| lncRNA编号 ID of lncRNA |
BC/IC | AC/IC | 靶标基因 Target gene |
|||||||
| 差异倍数 log2FC |
P值 P-value |
差异倍数 log2FC |
P值 P-value |
基因编号 ID of gene |
家族 Family |
皮尔逊相关 系数R |
P值 P-value |
|||
| TCONS_00004984 | -2.93 | 1.20×10-3 | — | — | A1A201029 | GT34 | 0.83 | 2.11×10-2 | ||
| TCONS_00034390 | — | — | -9.13 | 4.19×10-8 | A1A208299 | GT2 | 0.87 | 1.27×10-2 | ||
| TCONS_00026794 | — | — | -1.57 | 3.19×10-2 | A1A206186 | GT1 | 0.74 | 4.75×10-2 | ||
| TCONS_00008139 | — | — | 1.34 | 5.02×10-6 | A1A201842 | GT2 | — | — | ||
| TCONS_00042046 | — | — | 4.47 | 5.77×10-3 | A1A209465 | GH75 | 0.92 | 6.80×10-3 | ||
| TCONS_00007597 | — | — | -2.34 | 2.11×10-7 | A1A202012 | GH71 | — | — | ||
| TCONS_00007598 | — | — | 1.86 | 4.66×10-2 | A1A202182 | GH27 | — | — | ||
| TCONS_00007596 | -2.30 | 7.10×10-5 | -3.93 | 7.93×10-9 | A1A201901 | GH54 | — | — | ||
| TCONS_00024279 | 3.55 | 2.46×10-3 | 3.25 | 6.98×10-4 | A1A205117 | GH5 | — | — | ||
| TCONS_00008144 | — | — | 3.59 | 7.83×10-5 | A1A201855 | GH47 | -0.82 | 2.26×10-2 | ||
| TCONS_00006786 | — | — | -3.39 | 1.54×10-2 | A1A201955 | GH47 | — | — | ||
| TCONS_00031932 | 1.95 | 4.64×10-2 | 2.36 | 1.15×10-2 | A1A207729 | GH30 | — | — | ||
| TCONS_00008221 | — | — | -6.27 | 1.08×10-2 | A1A202056 | GH18 | — | — | ||
| TCONS_00049958 | — | — | -6.74 | 1.15×10-3 | A1A211822 | GH1 | -0.77 | 3.64×10-2 | ||
| TCONS_00001868 | — | — | 7.24 | 7.42×10-4 | A1A200217 | AA7 | 0.87 | 1.22×10-2 | ||
| TCONS_00004027 | — | — | 2.90 | 2.92×10-2 | A1A200902 | AA7 | 0.82 | 1.44×10-2 | ||
| TCONS_00019280 | 1.14 | 6.98×10-3 | — | — | A1A204632 | AA5 | — | — | ||
| TCONS_00038941 | -3.78 | 4.59×10-2 | — | — | A1A209068 | 天冬氨酸 Aspartic |
— | — | ||
| TCONS_00046908 | — | — | -5.07 | 9.01×10-3 | A1A210822 | 天冬氨酸 Aspartic |
— | — | ||
| TCONS_00000365 | — | — | 7.34 | 7.48×10-4 | A1A200350 | 金属肽酶 Metallo |
— | — | ||
| TCONS_00002682 | 4.02 | 2.96×10-3 | 5.01 | 4.31×10-7 | A1A200295 | 金属肽酶 Metallo |
— | — | ||
| TCONS_00012731 | — | — | — | — | A1A203305 | 金属肽酶 Metallo |
— | — | ||
| TCONS_00035336 | — | — | 5.43 | 2.23×10-2 | A1A208482 | 丝氨酸 Serine |
-0.93 | 3.43×10-3 | ||
| TCONS_00036884 | — | — | -9.30 | 1.43×10-8 | A1A208999 | 丝氨酸 Serine |
— | — | ||
| TCONS_00010210 | -1.35 | 4.97×10-2 | — | — | A1A202550 | 丝氨酸 Serine |
— | — | ||
| TCONS_00030624 | — | — | -1.93 | 1.08×10-5 | A1A207374 | 丝氨酸 Serine |
— | — | ||
| TCONS_00031032 | — | — | -1.19 | 3.42×10-2 | A1A207893 | 丝氨酸 Serine |
— | — | ||
| TCONS_00032054 | — | — | 1.90 | 2.68×10-2 | A1A206034 | 丝氨酸 Serine |
— | — | ||
| TCONS_00029086 | 1.18 | 3.73×10-3 | 1.20 | 6.12×10-3 | A1A207066 | 丝氨酸 Serine |
0.97 | 6.53×10-4 | ||
| TCONS_00013724 | 2.21 | 4.99×10-2 | — | — | A1A203005 | 聚酮合成酶 PKS |
— | — | ||
| Note:FC:Fold change. | ||||||||||
lncRNA与互作靶标基因之间的表达模式具有较高的相关性。在30个差异表达的lncRNA中, 发现10个lncRNA的表达模式和靶标基因显著相关(皮尔逊相关系数|R|>0.7, P < 0.05)(表 3)。有8个lncRNA其表达模式和编码碳水化合物活性酶基因显著相关, 其中6个为正相关, 2个为负相关。有2个lncRNA表达模式和编码蛋白酶基因显著相关, 1个为正相关, 1个为负相关。
3 讨论长链非编码RNA广泛存在于真核生物体内, 是一类能够调节基因表达、影响酶活性和染色体构象的多功能核酸分子。lncRNA序列、结构、基因表达及与靶标互作等信息的获取, 是深入研究其功能的基础。木霉菌对病原菌重寄生的分子机制研究一直受到科学界关注, 但研究主要集中于编码基因。lncRNA是否在重寄生过程中发挥调控作用尚不清楚。因此, 本研究采用链特异性RNA-seq技术, 对木霉NJAU 4742与病原菌Foc4重寄生互作接触前、后的菌株以及独立培养菌株进行转录组测序, 并且在全基因组范围内鉴定木霉菌lncRNA。
本研究在木霉中共识别了1 676个lncRNA, 其数量远比动植物少, 但与禾谷镰刀菌的lncRNA数量[9]相近。与动物lncRNA相似, 木霉lncRNA也具有外显子数量偏少、序列偏短、表达水平偏低等特征[27]。由于lncRNA不能像编码基因那样利用蛋白同源性确定方向, 因此采用链特异性RNA-seq技术对转录组进行测序, 保证lncRNA方向的正确性。lncRNA表达丰度偏低, 本研究采用深度测序策略, 使每个样品有效测序覆盖度都达100X以上, 部分样品覆盖度甚至达到200层以上, 尽可能保证lncRNA目录完整性。另外, 采用去核糖体方式构建RNA-seq文库, 缺少polyA尾巴的lncRNA也可以被检测到[28]。
在动植物中, lncRNA能与其上、下游100 kb范围内的编码基因互作, 因此常将lncRNA的上、下游编码基因预测为顺式作用靶标对其进行功能分析[26]。与动植物相比, 木霉菌NJAU 4742基因组紧凑, 将lncRNA上、下游5 kb范围的编码基因视作其顺式作用靶标。相关性分析显示:lncRNA与5 kb范围内的靶标基因间的表达相关性显著高于不相邻编码基因间的相关性, 也高于随机选择的2个lncRNA间的相关性, 这暗示lncRNA与5 kb范围内编码基因互作是存在的。在木霉菌中发现lncRNA的表达和上游反义链上的靶标基因(TUAS)最相关, 类似现象在猪睾丸中也被发现[27]。虽然共表达分析常用于反式作用靶标预测, 但需要更多不同处理类型的样品[29]。本研究只有3种类型样品, 采用非常保守的序列互补方式对lncRNA的反式作用靶标进行预测, 因此反式作用靶标数量可能被严重低估了。
本研究对重寄生过程中的lncRNA进行差异表达和靶标基因功能分析, 共识别30个可能与重寄生相关的lncRNA, 其中10个lncRNA的表达与靶标基因显著相关。高通量测序分析结果表明:生防木霉菌(T.atroviride)中38%的编码基因在重寄生互作接触前就能够被诱导差异表达。本研究也发现了10个在重寄生互作接触前就发生差异表达的lncRNA, 这表明lncRNA在非直接接触的情况下也能够受寄主病原菌诱导。几丁质酶是裂解病原菌细胞壁的关键因子, 而蛋白酶能直接破坏病原菌生理结构达到抑制病原菌的作用[30]。本研究发现了2个lncRNA(TCONS_00042046和TCONS_00029086)分别靶向编码几丁质酶GH75和丝氨酸蛋白酶的基因, 其表达和靶标基因呈显著正相关(皮尔逊相关系数分别为0.92和0.97)。在葡萄糖饥饿情况下, Saccharomyces pombe长链非编码mlonRNA表达能够使相邻基因fbp1启动子处的染色质重塑, 从而使其结构更加利于转录因子的结合, 这样mlonRNA的表达正相关地促进了靶标fbp1基因的表达[31]。本研究还发现另一个lncRNA(TCONS_00035336)靶向编码丝氨酸蛋白酶的基因, 其表达模式和靶标基因呈显著负相关(皮尔逊相关系数为-0.93)。这种负相关的调控机制在酵母(Saccharomy cerevisiae)中也有报道, 长链非编码SRG1表达会激活复合物, 这些复合物使SER3启动子区域的核小体占据量增加, 从而抑制SER3的表达[6]。值得注意的是, 尽管本研究的结果表明lncRNA可能参与重寄生过程的调控, 但这些都来自生物信息学分析, 还需要在重寄生过程中对lncRNA进行功能分析和验证。
| [1] |
Berretta J, Morillon A. Pervasive transcription constitutes a new level of eukaryotic genome regulation[J]. EMBO Rep, 2009, 10(9): 973-982. DOI:10.1038/embor.2009.181 |
| [2] |
Gomes A Q, Nolasco S, Soares H. Non-coding RNAs:multi-tasking molecules in the cell[J]. Int J Mol Sci, 2013, 14(8): 16010-16039. DOI:10.3390/ijms140816010 |
| [3] |
Wu H J, Wang Z M, Wang M, et al. Widespread long noncoding RNAs as endogenous target mimics for microRNAs in plants[J]. Plant Physiol, 2013, 161(4): 1875-1884. |
| [4] |
谭玉荣, 王丹, 高璇, 等. 植物长链非编码RNA研究进展[J]. 生物技术通报, 2018, 34(10): 1-10. Tan Y R, Wang D, Gao X, et al. Research advance on plant long noncoding RNAs[J]. Biotechnology Bulletin, 2018, 34(10): 1-10 (in Chinese with English abstract). |
| [5] |
路畅, 黄银花. 动物长链非编码RNA研究进展[J]. 遗传, 2017, 39(11): 1054-1065. Lu C, Hang Y H. Progress in long non-coding RNAs in animals[J]. Hereditas, 2017, 39(11): 1054-1065 (in Chinese with English abstract). |
| [6] |
Hainer S J, Pruneski J A, Mitchell R D, et al. Intergenic transcription causes repression by directing nucleosome assembly[J]. Gene Dev, 2011, 25(1): 29-40. DOI:10.1101/gad.1975011 |
| [7] |
Pinskaya M, Gourvennec S, Morillon A. H3 lysine 4 di-and tri-methylation deposited by cryptic transcription attenuates promoter activation[J]. The EMBO Journal, 2009, 28(12): 1697-1707. DOI:10.1038/emboj.2009.108 |
| [8] |
Shao J J, Chen H M, Yang D, et al. Genome-wide identification and characterization of natural antisense transcripts by strand specific RNA sequencing in Ganoderma lucidum[J]. Scientific Reports, 2017, 7(1): 5711. DOI:10.1038/s41598-017-04303-6 |
| [9] |
Kim W, Miguel-Rojas C, Wang J, et al. Developmental dynamics of long noncoding RNA expression during sexual fruiting body formation in Fusarium graminearum[J]. eMbio, 2018, 9(4): e01292-18. |
| [10] |
Till P, Pucher M E, Mach R L, et al. A long noncoding RNA promotes cellulase expression in Trichoderma reesei[J]. Biotechnol Biofuels, 2018, 11: 78. DOI:10.1186/s13068-018-1081-4 |
| [11] |
Steindorff A S, Ramada M H, Coelho A S, et al. Identification of mycoparasitism-related genes against the phytopathogen sclerotinia sclerotiorum through transcriptome and expression profile analysis in Trichoderma harzianum[J]. BMC Genomics, 2014, 15: 204. DOI:10.1186/1471-2164-15-204 |
| [12] |
Druzhinina I S, Chenthamara K, Zhang J, et al. Massive lateral transfer of genes encoding plant cell wall-degrading enzymes to the mycoparasitic fungus Trichoderma from its plant-associated hosts[J]. PLoS Genetics, 2018, 14(4): e1007322. DOI:10.1371/journal.pgen.1007322 |
| [13] |
Bolger A M, Lohse M, Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [14] |
Kim D, Landmead B, Salzberg S L. HISAT:a fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. |
| [15] |
Pertea M, Pertea G M, Antonescu C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290-295. |
| [16] |
Trapnell C, Williams B A, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nat Biotechnol, 2010, 28(5): 511-515. DOI:10.1038/nbt.1621 |
| [17] |
Finn R D, Coggill P, Eberhardt R Y, et al. The Pfam protein families database:towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44(D1): D279-D285. DOI:10.1093/nar/gkv1344 |
| [18] |
Kong L, Zhang Y, Ye Z Q, et al. CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine[J]. Nucleic Acids Research, 2007, 35: W345-W349. DOI:10.1093/nar/gkm391 |
| [19] |
Kalvari I, Argasinska J, Quinones-Olvera N, et al. Rfam 13.0:shifting to a genome-centric resource for non-coding RNA families[J]. Nucleic Acids Research, 2018, 46(D1): D335-D342. DOI:10.1093/nar/gkx1038 |
| [20] |
Li B, Dewey C N. RSEM:accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12: 323. DOI:10.1186/1471-2105-12-323 |
| [21] |
Love M I, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 |
| [22] |
Zhang H, Yohe T, Huang L, et al. dbCAN2:a meta server for automated carbohydrate-active enzyme annotation[J]. Nucleic Acids Research, 2018, 46: W95-W101. DOI:10.1093/nar/gky418 |
| [23] |
Rawlings N D, Waller M, Barrett A J, et al. MEROPS:the database of proteolytic enzymes, their substrates and inhibitors[J]. Nucleic Acids Research, 2014, 42: D503-D509. DOI:10.1093/nar/gkt953 |
| [24] |
Khaldi N, Seifuddin F T, Turner G, et al. SMURF:genomic mapping of fungal secondary metabolite clusters[J]. Fungal Genet Biol, 2010, 47(9): 736-741. DOI:10.1016/j.fgb.2010.06.003 |
| [25] |
Tafer H, Hofacker I L. RNA plex:a fast tool for RNARNA interaction search[J]. Bioinformatics, 2008, 24(22): 2657-2663. |
| [26] |
Liao Q, Liu C N, Yuan X Y, et al. Large-scale prediction of long non-coding RNA functions in a coding-non-coding gene co-expression network[J]. Nucleic Acids Research, 2011, 39(9): 3864-3878. DOI:10.1093/nar/gkq1348 |
| [27] |
Weng B, Ran M L, Chen B, et al. Genome-wide analysis of long non-coding RNAs and their role in postnatal porcine testis development[J]. Genomics, 2017, 109(5/6): 446-456. |
| [28] |
Ariel F, Romero-Barrios N, Jegu T, et al. Battles and hijacks:noncoding transcription in plants[J]. Trends in Plant Science, 2015, 20(6): 362-371. DOI:10.1016/j.tplants.2015.03.003 |
| [29] |
Zhou J J, Zhang S Y, Wang H T, et al. LncFunNet:an integrated computational framework for identification of functional long noncoding RNAs in mouse skeletal muscle cells[J]. Nucleic Acids Research, 2017, 45(12): e108. DOI:10.1093/nar/gkx232 |
| [30] |
Atanasova L, Crom L S, Gruber S, et al. Comparative transcriptomics reveals different strategies of Trichoderma mycoparasitism[J]. BMC Genomics, 2013, 14(1): 121. DOI:10.1186/1471-2164-14-121 |
| [31] |
Hirota K, Miyoshi T, Kugou K, et al. Stepwise chromatin remodelling by a cascade of transcription initiation of non-coding RNAs[J]. Nature, 2008, 456(7218): 130-134. DOI:10.1038/nature07348 |




