文章信息
- 童月霞, 胡晓璇, 程宗明, 仲岩
- TONG Yuexia, HU Xiaoxuan, CHENG Zongming, ZHONG Yan
- 苹果NBS-encoding基因对斑点落叶病菌侵染的表达响应
- Expression of apple NBS-encoding genes in response to the infection of Alternaria blotch pathogen
- 南京农业大学学报, 2019, 42(2): 253-260
- Journal of Nanjing Agricultural University, 2019, 42(2): 253-260.
- http://dx.doi.org/10.7685/jnau.201806006
-
文章历史
- 收稿日期: 2018-06-08
苹果(Malus domestica)是世界上栽培最广、产量最大的温带水果, 其果实具有丰富的营养成分。但在苹果生长过程中容易受到各种病原微生物(真菌、细菌、病毒等)及害虫的侵害, 造成树势减弱、果实减产等诸多问题, 从而给生产上带来巨大的经济损失[1]。
苹果斑点落叶病(apple Alternaria blotch)又称褐纹病, 是由苹果链格孢菌(Alternaria alternaria f. sp. mali)侵染而引起的一种世界性分布的气传病害, 同时也是危害亚洲苹果产区最为严重的病害之一[2]。苹果斑点落叶病主要危害叶片、新梢及果实。20世纪70年代后期, 我国开始有关于苹果斑点落叶病的报道, 目前该病已成为我国苹果主产区三大病害之一[3]。此外, 国内外对于该病的研究集中于发病症状、防治方法、病原菌鉴定和种质资源评价等方面[4]。
植物在抵御病原物侵染过程中进化出一系列防卫机制, 其中最关键的是抗病基因介导的抗病性。DeYoung等[5]研究认为最重要的抗病基因是具有核苷酸结合位点结构域(nucleotide-binding site, NBS)和富含亮氨酸重复结构域(leucine rich repeat, LRR)的一类基因, 即NBS-LRR类抗病基因, 它们通过编码抗病蛋白从而抵抗病原物的侵害。NBS-LRR类基因是分布最广和数量最大的一类抗性基因家族。根据N端结构域的不同, NBS-LRR蛋白可分为2个亚类:一类是TIR-NBS-LRR(TNL)蛋白, 其N末端具有白细胞介素受体(Toll/Interleukin-1 receptor)同源结构域; 另一类是non-TIR-NBS-LRR(non-TNL)蛋白, 其N端具有螺旋卷曲结构域(CC)或其他结构域[6]。近年来, 已经有相关研究报道NBS基因家族, 如拟南芥[7]等; NBS-encoding基因与植物抗病性间的关系, 如马铃薯晚疫病[8]和水稻稻瘟病[9]等; 果树NBS基因, 如刺梨抗白粉病相关NBS基因[10]等。但是, 由于苹果复杂的遗传背景[11], 在抗病基因方面的研究尚未取得实质性的进展。自从Baldi等[12]初次在苹果基因组中鉴定了30个NBS-LRR类的同源序列, 苹果抗病性与NBS-LRR基因的相关研究才逐渐展开。Calenge等[13]根据苹果NBS基因家族序列鉴定出了23个与抗病性相关的NBS基因。目前, 已发现苹果火疫病[14]和白粉病[15]的抗性基因位点均包含有NBS-LRR基因。
本研究根据苹果斑点落叶病菌侵染‘红星’后的转录组数据(RNA-seq), 筛选得出差异表达的NBS-encoding基因并对其进行分析, 包括基因的理化性质、分类、表达模式及共表达网络的构建, 并根据苹果NBS-encoding基因家族系统进化树计算差异表达的NBS-encoding基因的Ka/Ks值, 旨在为苹果NBS-encoding基因的抗病功能研究和苹果抗病分子育种工作提供理论依据。
1 材料与方法 1.1 数据来源数据来源为苹果品种‘红星’接种苹果斑点落叶病菌后的RNA-seq数据[16]。
1.2 苹果NBS-encoding差异表达基因的筛选及其理化性质分析根据RNA-seq数据筛选差异表达的NBS-encoding基因。分别以时间点0、12、18、36和72 h为对照, 以错误发现率(FDR)≤0.001为阈值[17](FDR值越低说明基因表达差异越显著), 筛选出各个样本间的差异表达基因(differentially expressed genes, DEG), 并对差异倍数(fold change, FC)(log2 FC≥2)进行评估。
用ProtParam(https://web.expasy.org/tools/protparam.html)分析各个差异表达的NBS-encoding蛋白的理化性质, 包括氨基酸数目、预测分子质量、等电点、对脂溶指数和总平均亲水性。利用Pfam(http://pfam.janelia.org/)分析验证所有NBS-encoding基因的NB-ARC结构域与LRR基序的存在, 并采用SMART(http://smart.embl-heidelberg.de/)蛋白基序分析提高LRR鉴定的准确性。最后使用Pfam和COILS(http://embnet.vital-it.ch/software/COILS_form.html)验证NBS-encoding基因是否编码TIR和CC等基序, 从而对差异表达的NBS-encoding基因进行分类。
1.3 差异表达NBS-encoding基因的表达分析及其基因家族系统进化树的构建差异表达NBS-encoding基因的表达水平用log2RPKM值来衡量[16](reads per kilobase of exon model per million mapped reads, RPKM, 即每百万reads中来自于某基因每千碱基长度的reads数)。利用R studio软件绘制表达量热图(heatmap)。
利用FastTree 2软件对苹果NBS-encoding基因家族[18]的NBS结构域序列构建系统进化树。在系统进化树中, 找到差异表达NBS-encoding基因所在的系统进化支, 并使用MEGA 6.0对各个进化支重新构建系统进化树。
1.4 同义突变率(Ks)和非同义突变率(Ka)的计算使用MEGA 6.0软件计算系统进化支中各个基因的同义突变率(Ks)、非同义突变率(Ka)和Ka/Ks值[19]。若Ka/Ks < 1, 说明基因受到负选择压力的作用; 若Ka/Ks=1, 说明基因受到中性选择的作用; 若Ka/Ks>1, 说明基因受到正选择压力的作用[20]。
1.5 差异表达NBS-encoding基因的共表达网络构建根据RPKM值, 使用IBM SPSS Statistics 19软件计算差异表达NBS-encoding基因的PCC(Pearson correlation coefficient, 皮尔森相关系数)值, 选取PCC>0.8且P < 0.05的基因对, 并使用Cytoscape 3.0软件构建其共表达网络图[21]。
2 结果与分析 2.1 苹果NBS-encoding差异表达基因的确定及其理化性质分析由表 1可见:根据RNA-seq数据共筛选出19个差异表达的NBS-encoding基因, 分为5种类型, 分别为TIR-NBS-LRR、CC-NBS-LRR、X-NBS-LRR、CC-NBS和X-NBS。蛋白质预测结果显示, 基因登录号为MDP0000751628所编码的氨基酸数量最少, 只有181个, 其蛋白相对分子质量为20 084.25;基因登录号为MDP0000291205所编码的氨基酸数量最多, 为1 216个, 其蛋白相对分子质量为137 737.88。结构域预测表明除了登录号为MDP0000259862、MDP0000295195、MDP0000751628和MDP0000640906的基因外, 其他NBS-encoding基因都属于NBS-LRR基因家族。NBS-encoding基因所编码的蛋白理论等电点为5.11~9.02, 其中有3个NBS-encoding蛋白(基因登录号为MDP0000259862、MDP0000751628和MDP0000240537所编码的蛋白)为碱性蛋白, 其他均为酸性蛋白。NBS-encoding蛋白的对脂溶指数为86.02~105.26。NBS-encoding蛋白的总平均亲水性为-0.456~0.445, 其中只有基因登录号为MDP0000372663所编码的蛋白为疏水蛋白, 其他均为亲水蛋白。
| 基因登录号 Gene accession No. |
类型 Type |
氨基酸数量 Number ofamino acids |
相对分子质量 Relative molecularweight |
理论等电点 Theoretical pI |
对脂溶指数 Aliphatic index |
总平均亲水性 Grand average ofhydropathicity(GRAVY) |
| MDP0000304378 | X-NBS-LRR | 1 038 | 118 167.72 | 6.43 | 98.66 | -0.221 |
| MDP0000292810* | X-NBS-LRR | 947 | 106 956.85 | 5.91 | 102.98 | -0.056 |
| MDP0000259862* | CC-NBS | 429 | 48 363.98 | 7.66 | 101.72 | -0.157 |
| MDP0000710999* | TIR-NBS-LRR | 782 | 88 704.00 | 5.72 | 103.18 | -0.172 |
| MDP0000127009* | X-NBS-LRR | 1 154 | 130 039.94 | 5.78 | 100.39 | -0.189 |
| MDP0000641906* | CC-NBS-LRR | 1 163 | 132 061.39 | 6.24 | 98.99 | -0.208 |
| MDP0000144730 | X-NBS-LRR | 908 | 101 846.30 | 5.78 | 97.48 | -0.145 |
| MDP0000174796 | CC-NBS-LRR | 864 | 97 729.09 | 5.81 | 96.46 | -0.256 |
| MDP0000310092 | CC-NBS-LRR | 831 | 94 025.90 | 6.10 | 97.65 | -0.128 |
| MDP0000295195 | CC-NBS | 298 | 34 225.59 | 5.73 | 95.87 | -0.456 |
| MDP0000751628 | X-NBS | 181 | 20 084.25 | 9.02 | 93.20 | -0.322 |
| MDP0000306942 | CC-NBS-LRR | 1 132 | 128 950.90 | 6.31 | 94.15 | -0.269 |
| MDP0000210357 | X-NBS-LRR | 444 | 50 535.41 | 5.11 | 104.71 | -0.017 |
| MDP0000640906 | X-NBS | 330 | 37 048.29 | 5.30 | 95.18 | -0.098 |
| MDP0000372663 | TIR-NBS-LRR | 943 | 107 776.08 | 5.99 | 86.02 | 0.445 |
| MDP0000147704 | X-NBS-LRR | 723 | 81 957.09 | 6.07 | 92.57 | -0.386 |
| MDP0000291205 | X-NBS-LRR | 1 216 | 137 737.88 | 5.98 | 105.26 | -0.036 |
| MDP0000240537 | X-NBS-LRR | 572 | 64 654.48 | 7.10 | 102.94 | -0.128 |
| MDP0000162375 | X-NBS-LRR | 808 | 91 595.03 | 6.71 | 103.42 | -0.093 |
| 注:*数据来源于文献[22]。Data are derived from reference [22]. | ||||||
由图 1可见:根据表达模式的变化情况, 可将这19个基因分为3种表达模式:第1种, 在侵染初期的表达量均处于较高水平且一直保持上升趋势, 包括7个基因(登录号为MDP0000259862、MDP0000304378、MDP0000292810、MDP0000710999、MDP0000127009、MDP0000295195和MDP0000751628), 其中登录号为MDP0000259862、MDP0000304378和MDP0000292810的基因在后期持续高表达; 第2种, 表达量持续上调, 但一直处于低表达量水平, 包括8个基因(登录号为MDP0000210357、MDP0000640906、MDP0000162375、MDP0000291205、MDP0000147704、MDP0000240537、MDP0000306942和MDP0000372663);第3种, 表达量处于中间水平, 包括4个基因(登录号为MDP0000144730、MDP0000641906、MDP0000174796和MDP0000310092), 其中除了登录号为MDP0000641906的基因, 其他3个基因在前期表达量上调, 后期均有下调。
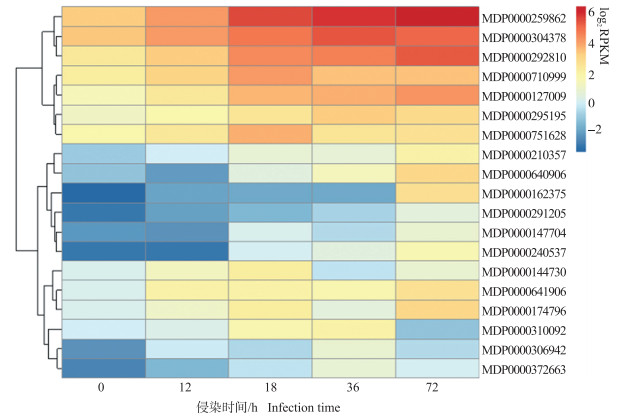
|
图 1 差异表达NBS-encoding基因在‘红星’接种苹果斑点落叶病菌过程中的表达热图 Fig. 1 Expression heatmap of differentially expressed NBS-encoding genes during the infection in'Starking'with apple Alternaria blotch pathogen RPKM:每百万reads中来自于某基因每千碱基长度的reads数Reads per kilobase of exon model per million mapped reads. |
根据苹果NBS-encoding基因家族系统进化树, 将差异表达的NBS-encoding基因定位到了15个进化支中, 并对各进化支重新构建系统进化树(图 2), 最后计算出每个进化支中NBS-encoding基因的Ka/Ks值。进化支10、12、14和15的平均Ka/Ks值均大于1, 其中包括登录号为MDP0000210357、MDP0000751628、MDP0000147704和MDP0000372663的基因, 说明这些基因在进化过程中受到正选择压力的作用。进化支15的平均Ka/Ks值最大, 达到了2.24。而其余11个进化支的平均Ka/Ks值均小于1, 其中包括其他15个NBS-encoding基因, 表明这些进化支中的基因在进化过程中受到纯化选择的作用。
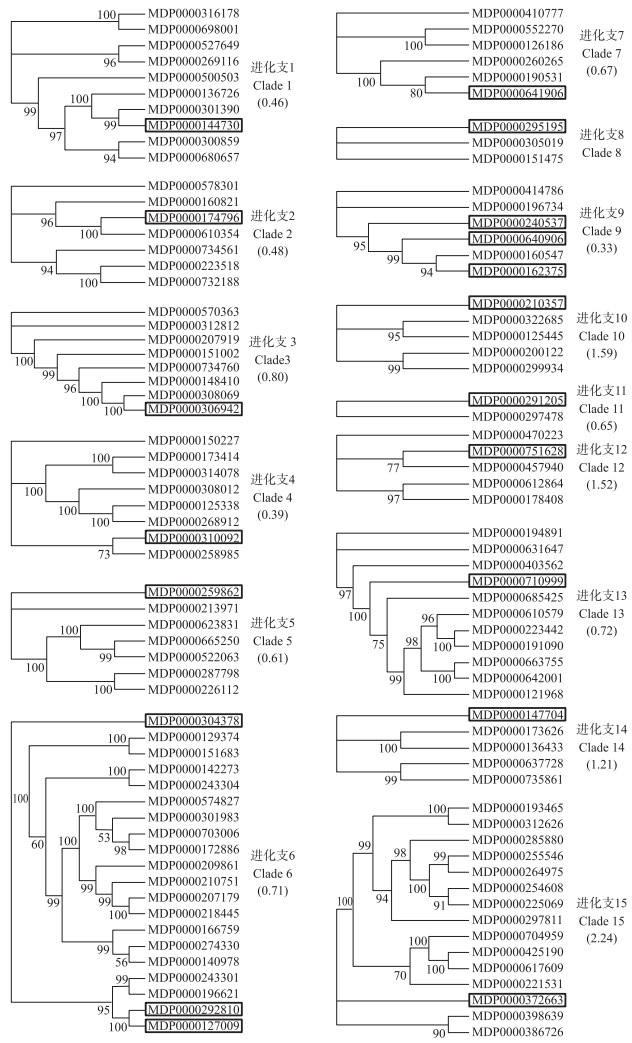
|
图 2 19个差异表达NBS-encoding基因的系统进化树 Fig. 2 Clades of 19 differentially expressed NBS-encoding genes 1)方框内基因为19个差异表达NBS-encoding基因; 2)括号内数值表示各进化支中基因的平均Ka/Ks值; 3)由于系统进化支8中Ka/Ks值无效, 因此没有标注。 1)The genes in the box are 19 differentially expressed NBS-encoding genes; 2)Bracketed values are average Ka/Ks ratios between the genes in each clade; 3)The Ka/Ks ratio of clade 8 is not shown because of the invalid ratio. |
由图 3可见:除了登录号为MDP0000310092、MDP0000306942和MDP0000640906的基因外, 其余16个基因之间存在一定的关联性。共表达网络图由3个不同的模块构成。第1个模块里共包含10个节点(登录号为MDP0000174796、MDP0000162375、MDP0000147704、MDP0000641906、MDP0000240537、MDP0000259862、MDP0000291205、MDP0000127009、MDP0000292810和MDP0000210357的基因)和34条边。第2个模块里包含3个节点(登录号为MDP0000372663、MDP0000295195和MDP0000304378的基因)和3条边。第3个模块中含有3个节点(登录号为MDP0000144730、MDP0000751628和MDP0000710999的基因)和2条边。每个节点都有不同数量的边, 从1个(登录号为MDP0000144730和MDP0000710999的基因)到9个(登录号为MDP0000210357的基因)不等。此外, 登录号为MDP0000210357的基因所在节点最大; 登录号为MDP0000240537和MDP0000291205的基因所在节点次之; 而登录号为MDP0000144730和MDP0000710999的基因所在节点最小。
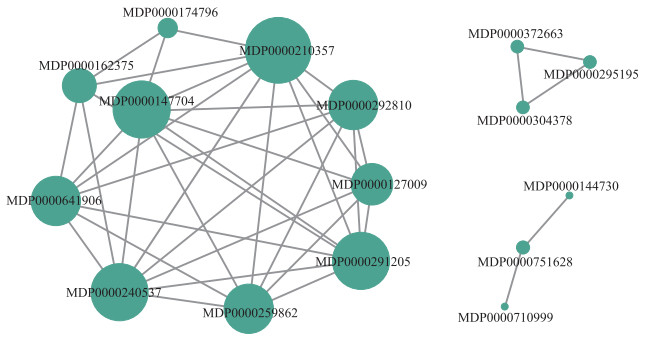
|
图 3 19个差异表达NBS-encoding基因的共表达网络 Fig. 3 Co-expression network of 19 differentially expressed NBS-encoding genes 节点代表基因; 节点大小代表渡值的大小; 节点之间的边缘线代表基因共表达间的相关性。 Nodes represent genes, and node sizes represent the magnitude of the degree value. Edge lines among nodes represent the correlation between gene co-expression. |
NBS类基因是植物中最普遍存在的抗病基因, 在植物抵御各种病原物侵染过程中发挥着重要作用。虽然关于NBS基因在抗病方面的研究已有不少报道, 如白粉病[15]、火疫病[14]等, 但对于苹果斑点落叶病的研究还鲜有报道。本研究根据RNA-seq数据共筛选出19个苹果差异表达NBS-encoding基因, 并对其理化性质以及表达模式进行分析, 同时构建共表达网络, 可为进一步探究苹果NBS-encoding基因家族在苹果斑点落叶病方面的功能提供理论依据, 同时为苹果的分子育种提供一定参考。
本研究根据苹果差异表达NBS-encoding基因在不同时间的表达热图可将这些基因分为3个表达模式。其中第1种表达模式中有3个基因(登录号为MDP0000259862、MDP0000304378和MDP0000292810), 其表达量在侵染后期一直保持高水平且不断上调, 可以在今后进行NBS-encoding基因响应苹果斑点落叶病的功能验证时, 对这3个基因予以重视。部分NBS-encoding基因在侵染过程中表达量发生不同程度变化, 说明它们可能参与响应苹果斑点落叶病的过程。根据宋霄等[1]的研究结果, MdNBS基因在抗病机制方面可能与SA和MeJA信号途径有关, 苹果轮纹病病原菌可诱导MdNBS-LRR基因的上调表达, 进一步说明MdNBS-LRR参与了苹果抗病防御反应。同样地, Arya等[23]的研究发现, 无论是苹果花叶病毒还是苹草缢管蚜(Rhopalosiphum insertum)侵染苹果叶片后, MdNBS70、MdNBS236和MdNBS275等基因的表达量均显著上调, 表明这些基因在植物抗病响应过程中具有一定的作用。此外, 在抗黄曲霉花生的果实组织中也发现了高表达的NBS-LRR基因(PnAG3)[24]。
我们对选择压力研究后发现, 登录号为MDP0000210357、MDP0000751628、MDP0000147704和MDP0000372663的基因在进化过程中受到了正选择压力的作用, 而其余15个NBS-encoding基因受到纯化选择的作用。Li等[25]研究表明猕猴桃NBS-encoding基因在进化过程中大多数受到了纯化选择的作用, 但仍有部分基因受到正选择压力的作用; 这意味着纯化选择对于NBS-encoding基因的进化起着重要的作用。Zhong等[6, 26]发现, 5个蔷薇科物种(草莓、苹果、梨、桃和梅)及6个不同草莓品种中大部分的NBS-LRR基因都受到了纯化选择的作用。此外, Mondragón-Palomino等[27]也已证明拟南芥中植物抗病基因(NBS-LRR)基本都受到正选择的作用。由此说明, 正选择作用在植物NBS-encoding基因进化的过程中起着重要的作用。
共表达分析能识别和发现一些新的调节因子或未知的相互关系。处于共表达网络里同一个模块的基因对, 其表达模式相似, 如登录号为MDP0000259862和MDP0000292810的基因。Yin等[28]研究发现, 在不同组织以及不同年龄和不同基因型的根中, 吉林人参的NBS-encoding基因都是共同表达的, 并形成一个单一的共表达网络, 其中归类为应激和细胞死亡功能的NBS-encoding基因可能在植物对病原体的防御中发挥作用。Kugler等[29]研究表明, 处于共表达网络同一模块中的小麦枯萎病抗性基因具有相似的表达模式, 包括NBS-LRR基因、WRKY基因和UGT基因。此外, 也有研究通过对霜霉病菌(Pseudoperonospora cubensis)侵染黄瓜[30]后的表达谱分析表明, 处于同一个共表达网络中的基因对病原体的反应是协同作用的。由此我们推测, 本文中处于共表达网络同一模块的NBS-encoding基因, 在一定程度上会共同表达以响应苹果斑点落叶病菌的侵染。
| [1] |
宋霄.苹果NBS基因的鉴定及表达分析[D].青岛: 青岛农业大学, 2013. Song X. Identification and expression analysis of NBS genes in the apple[D]. Qingdao: Qingdao Agricultural University, 2013(in Chinese with English abstract). |
| [2] |
Abe K, Iwanami H, Kotoda N, et al. Evaluation of apple genotypes and Malus species for resistance to Alternaria blotch caused by Alternaria alternata apple pathotype using detached-leaf method[J]. Plant Breeding, 2010, 129(2): 208-218. |
| [3] |
胡同乐, 曹克强, 王树桐, 等. 生长季苹果斑点落叶病流行主导因素的确定[J]. 植物病理学报, 2005, 35(4): 374-377. Hu T L, Cao K Q, Wang S T, et al. Study on the main factor for the epidemic of alternaria blotch on apple[J]. Acta Phytopathologica Sinica, 2005, 35(4): 374-377 (in Chinese). DOI:10.3321/j.issn:0412-0914.2005.04.017 |
| [4] |
Harteveld D O C, Akinsanmi O A, Drenth A. Pathogenic variation of Alternaria species associated with leaf blotch and fruit spot of apple in Australia[J]. European Journal of Plant Pathology, 2014, 139(4): 789-799. DOI:10.1007/s10658-014-0433-6 |
| [5] |
DeYoung B J, Innes R W. Plant NBS-LRR proteins in pathogen sensing and host defense[J]. Nature Immunology, 2006, 7(12): 1243-1249. DOI:10.1038/ni1410 |
| [6] |
Zhong Y, Yin H, Sargent D, et al. Species-specific duplications driving the recent expansion of NBS-LRR genes in five Rosaceae species[J]. BMC Genomics, 2015, 16(1): 77. DOI:10.1186/s12864-015-1291-0 |
| [7] |
Meyers B C, Kozik A, Griego A, et al. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis[J]. The Plant Cell, 2003, 15(4): 809-834. DOI:10.1105/tpc.009308 |
| [8] |
Ballvora A, Ercolano M R, Weiss J, et al. The R1 gene for potato resistance to late blight(Phytophthora infestans)belongs to the leucine zipper/NBS/LRR class of plant resistance genes[J]. The Plant Journal, 2002, 30(3): 361-371. DOI:10.1046/j.1365-313X.2001.01292.x |
| [9] |
Jeung J U, Kim B R, Cho Y C, et al. A novel gene, Pi40(t), linked to the DNA markers derived from NBS-LRR motifs confers broad spectrum of blast resistance in rice[J]. Theoretical and Applied Genetics, 2007, 115(8): 1163-1177. DOI:10.1007/s00122-007-0642-x |
| [10] |
Xu Q, Wen X P, Deng X X. Isolation of TIR and non-TIR NBS-LRR resistance gene analogues and identification of molecular markers linked to a powdery mildew resistance locus in chestnut rose(Rosa roxburghii Tratt)[J]. Theoretical and Applied Genetics, 2005, 111(5): 819-830. DOI:10.1007/s00122-005-0002-7 |
| [11] |
Jensen P J, Fazio G, Altman N, et al. Mapping in an apple(Malus×domestica)F1 segregating population based on physical clustering of differentially expressed genes[J]. BMC Genomics, 2014, 15(1): 261. DOI:10.1186/1471-2164-15-261 |
| [12] |
Baldi P, Patocchi A, Zini E, et al. Cloning and linkage mapping of resistance gene homologues in apple[J]. Theoretical and Applied Genetics, 2004, 109(1): 231-239. DOI:10.1007/s00122-004-1624-x |
| [13] |
Calenge F, van der Linden C G, Weg E V D, et al. Resistance gene analogues identified through the NBS-profiling method map close to major genes and QTL for disease resistance in apple[J]. Theoretical and Applied Genetics, 2005, 110(4): 660-668. DOI:10.1007/s00122-004-1891-6 |
| [14] |
Parravicini G, Gessler C, Denancé C, et al. Identification of serine/threonine kinase and nucleotide-binding site-leucine-rich repeat(NBS-LRR)genes in the fire blight resistance quantitative trait locus of apple cultivar'Evereste'[J]. Molecular Plant Pathology, 2011, 12(5): 493-505. DOI:10.1111/mpp.2011.12.issue-5 |
| [15] |
Dunemann F, Peil A, Urbanietz A, et al. Mapping of the apple powdery mildew resistance gene Pl1 and its genetic association with an NBS-LRR candidate resistance gene[J]. Plant Breeding, 2007, 126(5): 476-481. DOI:10.1111/pbr.2007.126.issue-5 |
| [16] |
Zhu L, Ni W, Liu S, et al. Transcriptomics analysis of apple leaves in response to Alternaria alternata apple pathotype infection[J]. Front Plant Sci, 2017, 8: 22. |
| [17] |
Audic S, Claverie J M. The significance of digital gene expression profiles[J]. Genome Research, 1997, 7(10): 986-995. DOI:10.1101/gr.7.10.986 |
| [18] |
Jia Y X, Yuan Y, Zhang Y, et al. Extreme expansion of NBS-encoding genes in Rosaceae[J]. BMC Genetics, 2015, 16(1): 48. DOI:10.1186/s12863-015-0208-x |
| [19] |
Grossman A, Mittrücker H W, Nicholl J, et al. Cloning of human lymphocyte-specific interferon regulatory factor(HLSIRF/hIRF4)and mapping of the gene to 6p23-p25[J]. Genomics, 1996, 37(2): 229-233. |
| [20] |
Drouin G, Daoud H, Xia J. Relative rates of synonymous substitutions in the mitochondrial, chloroplast and nuclear genomes of seed plants[J]. Molecular Phylogenetics and Evolution, 2008, 49(3): 827-831. DOI:10.1016/j.ympev.2008.09.009 |
| [21] |
Saito R, Smoot M E, Ono K, et al. A travel guide to Cytoscape plugins[J]. Nature Methods, 2012, 9(11): 1069-1076. DOI:10.1038/nmeth.2212 |
| [22] |
Hu X, Zhong Y, Huang K, et al. Differential expression of 12 NBS-encoding genes in two apple cultivars in response to Alternaria alternata f. sp. mali infection[J]. Canadian Journal of Plant Science, 2017, 98(2): 21-25. |
| [23] |
Arya P, Kumar G, Acharya V, et al. Genome-wide identification and expression analysis of NBS-encoding genes in Malus×domestica and expansion of NBS genes family in Rosaceae[J]. PLoS One, 2014, 9(9): e107987. DOI:10.1371/journal.pone.0107987 |
| [24] |
Li C J, Liu Y, Zheng Y X, et al. Cloning and characterization of an NBS-LRR resistance gene from peanuts(Arachis hypogaea L.)[J]. Physiological and Molecular Plant Pathology, 2013, 84(1): 70-75. |
| [25] |
Li Y, Zhong Y, Huang K, et al. Genomewide analysis of NBS-encoding genes in kiwi fruit(Actinidia chinensis)[J]. Journal of Genetics, 2016, 95(4): 997-1001. DOI:10.1007/s12041-016-0700-8 |
| [26] |
Zhong Y, Zhang X H, Cheng Z M. Lineage-specific duplications of NBS-LRR genes occurring before the divergence of six Fragaria species[J]. BMC Genomics, 2018, 19(1): 128. DOI:10.1186/s12864-018-4521-4 |
| [27] |
Mondragón-Palomino M, Meyers B C, Michelmore R W, et al. Patterns of positive selection in the complete NBS-LRR gene family of Arabidopsis thaliana[J]. Genome Research, 2002, 12(9): 1305-1315. DOI:10.1101/gr.159402 |
| [28] |
Yin R, Zhao M, Wang K, et al. Functional differentiation and spatial-temporal co-expression networks of the NBS-encoding gene family in Jilin ginseng, Panax ginseng C.A. Meyer[J]. PLoS One, 2017, 12(7): e0181596. DOI:10.1371/journal.pone.0181596 |
| [29] |
Kugler K G, Siegwart G, Nussbaumer T, et al. Quantitative trait loci-dependent analysis of a gene co-expression network associated with Fusarium head blight resistance in bread wheat(Triticum aestivum L.)[J]. BMC Genomics, 2013, 14(1): 728. DOI:10.1186/1471-2164-14-728 |
| [30] |
Adhikari B N, Savory E A, Vaillancourt B, et al. Expression profiling of Cucumis sativus in response to infection by Pseudoperonospora cubensis[J]. PLoS One, 2012, 7(4): e34954. DOI:10.1371/journal.pone.0034954 |




