文章信息
- 王磊, 赵霞, 王文静, 张瑞良, 代兴杨, 曾明华, 李琳
- WANG Lei, ZHAO Xia, WANG Wenjing, ZHANG Ruiliang, DAI Xingyang, ZENG Minghua, LI Lin
- 诱导耐药沙门菌CRISPR检测及cas基因表达分析
- Detection of CRISPR and cas expression analysis in induced drug resistance of Salmonella
- 南京农业大学学报, 2018, 41(2): 370-376
- Journal of Nanjing Agricultural University, 2018, 41(2): 370-376.
- http://dx.doi.org/10.7685/jnau.201701021
-
文章历史
- 收稿日期: 2017-01-11
沙门菌(Salmonella)在全球范围内是最重要的食源性病原菌之一, 绝大部分沙门菌对人和动物有致病性, 可导致人和动物的伤寒、副伤寒和胃肠炎等疾病, 给畜牧业带来巨大的损失, 严重威胁人类食品安全。近年来由于抗菌药物的大量和盲目使用, 使沙门菌的多重耐药现象日趋严重, 给沙门菌疾病的防治带来新的挑战[1]。
沙门菌的耐药机制非常复杂, 主要包括基因突变、产生水解酶或钝化酶、药物靶位改变、细胞膜通透性改变、主动外排功能增强和获得外源耐药基因等。近年来研究表明, 成簇间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats sequence, CRISPR)广泛分布于细菌和古细菌的基因组中[2-3], 且CRISPR与细菌耐药性有关[4-5]。CRISPR主要由重复序列(repeat)和间隔序列(spacer)间隔排列构成的R-S结构组成, 其与CRISPR相关基因(CRISPR associated, Cas)组成CRISPR/Cas系统, 为原核生物提供对噬菌体、质粒等外源基因的免疫能力, 从而抵抗噬菌体感染, 限制基因的水平转移[6-7]。耐药基因的水平转移是细菌耐药性产生的主要原因[8], 目前有关CRISPR在原核生物中的功能及应用受到广泛关注, 如基因编辑系统的定点编辑技术[9]、细菌分型[10]、抵御噬菌体[11]和细菌进化分析[12-13]等, 但其与细菌耐药性关系的报道较少, 且不同细菌中CRISPR与耐药性的关系不一致。
cas位于CRISPR位点附近, 是一类较大的多态性家族, 是编码CRISPR相关蛋白的基因, 有研究发现cas基因多达45个家族, 有cas1~cas10等多种类型[14], 其中cas1~cas6基因被认为是核心基因, 这6个cas基因家族广泛存在于不同的CRISPR亚型中。研究发现Cas蛋白具有核酸外切酶、核酸内切酶、聚合酶、解旋酶、多聚核苷酸结合等功能[15-16], 与DNA的重组和修复、间隔序列获得、重复序列维持等有关。目前针对CRISPR序列的研究较少, 对诱导耐药菌株的CRISPR/Cas与耐药性关系的研究尚未见报道。
本试验通过使用不同药物对鼠伤寒沙门菌ATCC13311进行体内和体外耐药性诱导, 对耐药菌株CPISPR序列进行比对, 分析耐药前、后沙门菌CRISPR序列的差异以及cas mRNA表达水平的变化, 为沙门菌CPISPR/Cas与耐药性关系的研究奠定基础。
1 材料与方法 1.1 菌株和线虫鼠伤寒沙门菌ATCC13311(AT)由中国农业大学动物医学院兽医药理学与毒理学实验室惠赠; 秀丽隐杆线虫N2野生型由中国科学院合肥物质科学研究院技术生物与农业工程研究所辐射与环境毒理实验室惠赠。
1.2 药品和试剂DNA回收试剂盒、细菌RNA提取试剂盒, 购自OMEGA Bio-tek公司; PCR相关试剂(Premix Taq、Loading Buffer、2 000 bp DNA Marker)、细菌RNA反转录试剂盒、荧光定量试剂盒, 购自大连宝生物公司; 脑心浸液(BHI)肉汤培养基, 购自杭州微生物试剂有限公司; 琼脂粉, 购自BIOWEST公司; Tris-乙酸缓冲液(50×TAE), 购自上海生工生物工程公司。其余试剂均为国产分析纯。
1.3 沙门菌耐药性的体外诱导使用浓度梯度递增法诱导耐药菌株[17]。将ATCC13311菌接种于MH肉汤中, 37 ℃振荡培养过夜, 分别取5 mL菌液接种于分别含环丙沙星、庆大霉素、氨苄西林(质量浓度分别为0.004、1.00和0.06 mg·L-1)的100 mL MH肉汤中培养。次日, 以相同方法接种。然后, 不断以相同方式接种ATCC13311菌于药物浓度依次翻倍的MH肉汤中, 直至菌株可以在相应抗生素浓度的培养基中生长(环丙沙星4 mg·L-1、庆大霉素16 mg·L-1、氨苄西林32 mg·L-1)。耐药菌株在空白培养基中传5代, 然后进行药物敏感性实验以确定其并非为适应性生长, 最终分别获得环丙沙星体外诱导耐药菌(WC)、庆大霉素体外诱导耐药菌(WG)、氨苄西林体外诱导耐药菌(WA)。
1.4 沙门菌耐药性的体内诱导以秀丽隐杆线虫N2野生型为宿主, 经线虫同步化处理, 用Escherichia.coli OP50培养至L4期后, 将ATCC13311菌定植于线虫体内, 在0.004 mg·L-1环丙沙星S缓冲液(5.85 g NaCl、1 g K2HPO4、6 g KH2PO4、5 g胆固醇、1 L H2O)中培养24 h, 然后使用含1 mmol·L-1叠氮钠的M9缓冲液清洗虫体, 抑制线虫体表细菌, 然后将其转入已灭菌的含有400 mg石英砂的离心管中, 加入1 mL灭菌水, 涡旋振荡2~3 min裂解线虫, 取上清液稀释涂布于沙门菌显色培养基, 在37 ℃下培养观察有无沙门菌落出现。若无则保持该抗生素浓度重复试验直至获得适应菌株; 若长出沙门菌落, 则挑取单菌落, 翻倍抗生素浓度重复上述试验直至抗生素浓度达4 mg·L-1, 获得环丙沙星体内诱导耐药菌株(TN)。耐药菌株在空白培养基中传5代, 然后进行药物敏感性实验以确定其并非为适应性生长。
1.5 药物敏感性试验参照美国临床和实验室标准化协会(Clinical Laboratory Standards Institute, CLSI)指导原则和执行标准, 采用微量肉汤稀释法, 分别测定标准菌和诱导耐药菌对相应抗生素的最小抑菌浓度(minimum inhibitory concentrations, MIC), E.coli ATCC25922作为质控菌株。
1.6 CRISPR的检测 1.6.1 CRISPR引物序列设计根据CRISPR DB数据库公布的沙门菌的CRISPR结构序列以及参考沙门菌CRISPR分子分型的相关文献, 应用Oligo 7.37引物设计软件设计多对引物并经测试最终确定2对引物。引物序列分别为:CRISPR 1-F/R 5′-ATAATGCTGCCGTTGGTAA-3′/5′-TTGATGAGTATGGTGGTTGTGGT-3′; CRISPR 2-F/R 5′-AACGCCATGGCCTCCTCCTG-3′/5′-CAAAATCAGYAAATTAGCTGTTC-3′, 由英潍捷基(上海)公司合成。
1.6.2 CRISPR的PCR扩增分别以WC、WG、WA、TN和AT(ATCC13311)菌液为模板, 对CRISPR进行PCR扩增和检测。PCR扩增体系为25 μL:ddH2O 11 μL, 上、下游引物(20 mmol·L-1)各0.5 μL, 模板DNA 0.5 μL, 2× Premix Taq 12.5 μL。反应条件均为:95 ℃ 10 min; 94 ℃ 45 s, 56 ℃ 45 s, 72 ℃ 45 s, 35个循环; 72 ℃ 10 min。取5 μL PCR产物进行10 g·L-1琼脂糖凝胶电泳。凝胶电泳反应条件:电压100 V, 电流80 mA, 反应时间30 min, 用凝胶成像仪观察并记录结果。
1.6.3 PCR产物的测序及分析参照OMEGA Bio-tek公司的DNA回收试剂盒操作说明, 对WC、WG、WA、TN和AT的PCR扩增产物的DNA进行回收, 并将回收后的DNA送至英潍捷基(上海)贸易有限公司测序。将所测主要序列用CRISPR web server中的CRISPR Finder软件鉴定并分析各菌株的重复序列和间隔序列。
1.7 荧光定量PCR检测沙门菌cas mRNA的表达水平 1.7.1 cas引物设计根据GenBank中公布的沙门菌ATCC13311的核酸序列(NCBI登录号:CP009102.1)分别设计cas1、cas6、casA的RT-qPCR引物, 以16S rRNA为内参基因(表 1)。
| 基因 Genes |
引物对序列(5′→3′) Primer pairs sequences(5′→3′) |
扩增长度/bp Amplification sizes |
退火温度/℃ Annealing temperature |
| cas1 | F:CCCTTCTCCCAGTCTTTCG/R:GCCCGTCGTTCCGTTGA | 131 | 55 |
| cas6 | F:CATCATTCCGCAGCCAAAC/R:TCGGCAGATGATTCAGTTTAG | 127 | 55 |
| casA | F:GCCTCCTGTTTCTTTGCTT/R:GCTGTTTACCCGTCATTACTTC | 153 | 55 |
| 16S | F:TGTAGCGGTGAAATGCGTAG/R:CAAGGGCACAACCTCCAAG | 161 | 55 |
挑取沙门菌WC、WG、WA、TN和AT的单菌落, 于液体培养基37 ℃培养12 h, 再转接于相应的LB培养基培养至对数前期(D600约0.4), 参照E.Z.N.A.TM Bacterial RNA Kit说明书提取细菌的总RNA, 超微量紫外/可见分光光度计检测RNA纯度浓度, 同时用10 g·L-1琼脂糖凝胶电泳检测RNA的完整性, 检测合格后置-80 ℃保存备用。
1.7.3 RNA反转录釆用反转录试剂盒PrimeScriptTM RT消除提取产物中的基因组DNA, 并对RNA进行反转录(RT)。
基因组DNA去除反应:反应体系10.0 μL:5× gDNA Eraser Buffer 2.0 μL, gDNA Eraser 1.0 μL, 总RNA 1.0 μg, ddH2O加至10.0 μL。反应条件:42 ℃ 2 min(或室温5~30 min); 4 ℃保存备用。
RT反应体系(20.0 μL):上一步骤中的反应液10.0 μL, PrimeScript RT Enzyme MixⅠ1.0 μL, RT Primer Mix 1.0 μL, 5×PrimeScript Buffer Ⅱ 4.0 μL, ddH2O为4.0 μL。反应条件为37 ℃ 15 min, 85 ℃ 5 s。
1.7.4 荧光定量PCR(qPCR)采用SYBR荧光定量试剂盒, 对cas1、cas6、casA基因表达进行相对定量的检测, 以16S rRNA为内参基因。将各样品cDNA稀释至40 ng·μL-1。反应体系(20 μL):引物对(10 μmol·L-1)各0.8 μL, Premix Ex Taq Ⅱ(2×)10.0 μL, Rox Reference Dye(50×)0.4 μL, cDNA 2.0 μL, ddH2O 6.8 μL。扩增条件:50 ℃ 2 min, 95 ℃预变性5 min; 94 ℃变性5 s, 55 ℃复性45 s, 循环次数40次; 熔解曲线分析并鉴定引物特异性:95 ℃ 15 s, 55 ℃ 1 min, 95 ℃ 15 s。每个基因3个平行。采用2-ΔΔCT法计算cas mRNA的相对表达量。
2 结果与分析 2.1 药物敏感性试验结果药物敏感性试验结果表明:鼠伤寒沙门菌ATCC13311对环丙沙星、庆大霉素、氨苄西林均敏感, MIC值分别为0.004、1.00和0.25 mg·L-1; WC对环丙沙星的MIC为4 mg·L-1, WG对庆大霉素的MIC为16 mg·L-1, WA对氨苄西林的MIC为32 mg·L-1。
2.2 CRISPR的检测结果分别以WC、WG、WA、TN和AT的菌液为模板, 以CRISPR1和CRISPR2的引物对菌株进行PCR扩增。结果表明, PCR扩增产物均检测出有CRISPR位点, 且在5株菌株中同一CRISPR位点片段大小都相同(图 1)。
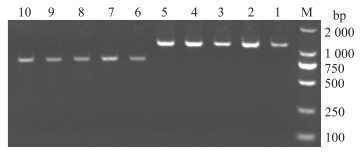
|
图 1 PCR扩增产物电泳图 Figure 1 Electrophoresis of PCR amplification products 1~5分别代表TN、WC、WG、AT、WA菌株的CRISPR1经PCR扩增的产物条带; 6~10分别代表TN、AT、WC、WG、WA菌株的CRISPR2经PCR扩增的产物条带; M为标准品。 1-5 are PCR amplified product bands of CRISPR1, represent the TN, WC, WG, AT, WA strains, respectively; 6-10 are PCR amplified product bands of CRISPR2, represent TN, AT, WC, WG, WA strains, respectively; M.DNA marker. |
用CRISPR Finder软件分析各菌株测序结果, 结果均得到2段重复间隔序列(图 2、3)。

|
图 2 CRISPR1的重复间隔序列 Figure 2 Repeat and spacer sequence of CRISPR1 左为重复序列, 右为间隔序列; 标记碱基为重复序列的退化。下图同此。 The left is repeat sequences, and the right is spaced sequences. Marked bases mean degeneration repeats. The same as follows. |

|
图 3 CRISPR2的重复间隔序列 Figure 3 Repeat and spacer sequence of CRISPR2 |
由图 2、3可知:CRISPR1中含有17条重复序列与16条间隔序列, 重复序列长度为29 bp, 间隔序列长度均为32 bp; CRISPR2由10条重复序列与9条间隔序列组成, 重复序列长度为29 bp, 间隔序列长度均为32 bp。发现CRISPR1与CRISPR2的第1条重复序列中分别有3或4个碱基未按照重复序列相同的原则, 该现象为重复序列的退化。
2.4 CRISPR序列的比对 2.4.1 CRISPR1序列比对对WA、WC、WG与AT的CRISPR1序列进行比对, 发现这4菌株中4~1 135的碱基完全重复, 序列的同源性为99.63%;碱基突变率为0.37%, 而WA与AT序列的1、3和1 136~1 141位的碱基存在差异; WC与AT序列的1和1 136~1 140位的碱基序列存在差异; WG与AT序列的第1个碱基存在差异; TN与AT的基因序列的5~1 138位点碱基相同, 序列的同源性为99.09%, 碱基突变率为0.91%, 其中1~4和1 139~1 144位的碱基存在差异, TN与WC序列的4~1 143位的碱基相同, 序列的同源性为99.63%, 碱基突变率为0.37%, 而1~3和1 144位的碱基存在差异(表 2)。

|
对WA、WC、WG与AT的基因序列比对, 发现这4菌株中15~761位和763~779位的碱基完全重复, 序列的同源性为97.74%, 碱基突变率为2.26%, 其中WA与AT序列的2、5、6、10、11和782~785位的碱基存在差异; WC与AT的5、6、11~14、780和781位的碱基存在差异; WG与AT序列的1、6、9、11、13、762和782~796位的碱基存在差异。对TN与AT的基因序列比对, 发现2菌株中12~779位的碱基完全重复, 序列的同源性为98.99%, 碱基突变率为1.01%, 而1~2、4、10~11和780~782位的碱基存在差异。TN与WC的序列中15~779位的碱基完全重复, 序列的同源性为98.61%, 碱基突变率为1.39%, 而1~2、5、10~14和780~782位的碱基存在差异(表 2)。
2.5 cas基因荧光定量PCR结果由图 4可见:对RT-qPCR所得各基因CT值用2-ΔΔCT法计算cas基因的相对表达量, 结果显示沙门菌在产生耐药性之后cas mRNA相对表达量下调。t检验分析结果显示, WG与AT的cas mRNA表达水平相比差异极显著(P < 0.01), WC与AT的cas mRNA表达水平相比差异显著(P < 0.05)。

|
图 4 cas mRNA相对表达水平 Figure 4 Relative expression level of cas mRNA |
沙门菌作为一种常见的食源性致病菌[18], 是重要的人兽共患病原菌, 随着抗菌药物的滥用, 其耐药谱不断变化, 多重耐药性日趋严重, 因此沙门菌耐药机制的研究引起广大学者的关注。本试验通过检测鼠伤寒沙门菌体内和体外诱导耐药菌的CRISPR, 进行序列比对, 结合分析耐药前、后沙门菌CRISPR相关基因cas mRNA表达水平的变化, 为沙门菌CPISPR与耐药性关系的深入研究奠定基础。
在本试验中, 对WC、WG、WA、TN和AT分别用2种引物序列进行PCR扩增, 获得2个CRISPR位点, 且CRISPR1第1条重复序列GTGTTTATCCCCGCTGGCGCGGGGAACAT中的第1、2和最后1个碱基以及CRISPR2第1条重复序列ACGGTTATCCCCGCTGACGCGGGGAACAC中的第1、2、4和17个碱基出现了重复序列的退化变异, 但是它们的间隔序列数量一致。王琳琳等[19]对志贺菌的研究表明, 不同的菌株间具有相同或相似的重复序列, 可能是因为重复序列在不同微生物间转移所造成的。Kunin等[20]和Grissa等[21]研究发现有的重复序列会有1~3个碱基的差异, 称为重复序列的退化, 在大肠杆菌等菌中重复序列的退化会引起紧连间隔序列的变化。
本试验分别对3株体外诱导耐药沙门菌与AT的CRISPR1和CRISPR2的序列进行分析比对, 结果表明相同的CRISPR序列在耐药菌间同源性较高, 序列差异主要发生在前导区和序列末端。沙门菌耐药株与标准株的CRISPR序列同源性也较高, 重复间隔相对保守。Deveau等[22]研究发现同一CRISPR位点的重复序列相对保守, 在同种细菌的不同菌株间高度相似, 仅在少数菌株中发生变异, 本试验结果与该结论相符。
根据cas qPCR结果, 发现在抗生素压力下沙门菌由敏感株诱导为耐药株后, cas1、cas6、casA的mRNA相对表达量下降且不同耐药菌中有所差异, 推测在不同抗生素压力下沙门菌会通过cas基因的表达变化来应对相应的抗生素侵害。我们在后期的试验中结合UniProt网站, 通过上下游的序列分析和比对找出ATCC13311菌株的CRISPR/Cas系统序列且确定为TypeⅠ-E型。cas1作为系统的核心基因存在于所有的CRISPR/Cas系统中, cas6也存在于绝大多数的Ⅰ型系统中, 而casA是Ⅰ型系统中E亚型特有的基因[14], 由此我们参考沙门菌CRISPR/Cas系统结构选取cas1、cas6和casA作为试验对象。Sampson等[23]对弗朗西斯菌的研究显示, cas9能增强细菌的耐药性, 其通过调控脂蛋白以保护细菌包膜的通透性和完整性, 减少抗生素危害。当cas9基因发生突变时, 弗朗西斯菌的耐药性降低, 对多黏菌素B等抗生素的敏感性增强, 不利于细菌存活。宋春花等[24]通过敏感志贺菌和耐多药大肠杆菌结合转移试验, 将敏感志贺菌株诱导成耐多药株, 通过双向电泳图谱分析发现Cas蛋白为多重耐药菌株新出现的蛋白, 认为Cas蛋白对耐药性起调控作用。本试验通过不同的耐药性诱导方法获得相应的耐药菌, 发现在抗生素压力下, 沙门菌的cas mRNA的表达水平下降, 推测其通过对外膜蛋白的调控导致沙门菌细胞膜通透性下降, 最终使得耐药性增强。
CRISPR/Cas系统最主要的功能是识别处理外源遗传物质, 阻止基因的水平转移[6-7], 但耐药基因的水平传播是细菌耐药性产生的主要形式[8], 由此可以推测CRISPR系统同样可以用于应对外源的耐药基因传播来调控细菌耐药性。Burley等[5]检测了粪肠球菌的CRISPR相关蛋白基因, 研究表明其与细菌多重耐药性有关; Palmer等[25]发现粪肠球菌的CRISPR/Cas与细菌获得性耐药有关。Bikard等[26]研究发现, 在高浓度抗生素环境下肺炎链球菌会通过丢失CRISPR位点来使肺炎链球菌获得相应的耐药基因, 同时特有的间隔序列能有效阻止毒力基因和耐药基因的转移。
本研究通过对3株体外诱导耐药沙门菌、1株体内诱导耐药沙门菌和标准菌株的CRISPR序列比对分析, 发现耐药菌株的CRISPR均有部分碱基的突变, 序列差异主要发生在前导区和序列末端, 间隔序列呈多样性, 均有重复序列的退化现象, 重复间隔序列相对保守, 同时耐药前后沙门菌cas基因的表达水平下降。提示CRISPR序列的变异和cas基因表达水平的下降与沙门菌耐药性有关, 为沙门菌CRISPR/Cas系统与耐药性关系的深入研究提供重要依据。
| [1] |
侯雪娇, 吴科敏, 莫国东, 等. 食源性沙门氏菌耐药表型与耐药基因的研究[J].
食品科学, 2016, 37(19): 166-170.
Hou X J, Wu K M, Mo G D, et al. Evaluation of drug-resistant phenotypes and genes in foodborne Salmonella isolates[J]. Food Science, 2016, 37(19): 166-170. DOI: 10.7506/spkx1002-6630-201619028 (in Chinese with English abstract) |
| [2] | Lillesto P K, Redder P, Barrett A, et al. A putative viral defence mechanism in archaeal cells[J]. Archaea, 2006, 2(1): 59-72. DOI: 10.1155/2006/542818 |
| [3] | Mojica F J, Diez-Villaseonr C, Soria E, et al. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria[J]. Molecular Microbiology, 2000, 36(1): 244-246. DOI: 10.1046/j.1365-2958.2000.01838.x |
| [4] | Touchon M, Charpentier S, Pognard D, et al. Antibiotic resistance plasmids spread among natural isolates of Escherichia coli in spite of CRISPR elements[J]. Microbiology, 2012, 158(12): 2997-3004. DOI: 10.1099/mic.0.060814-0 |
| [5] | Burley K M, Sedgley C M. CRISPR Cas, a prokaryotic adaptive immune system, in endodontic, oral and multidrug-resistant hospital-acquired Enterococcus faecalis[J]. Journal of Endodontics, 2012, 38(11): 1511-1515. DOI: 10.1016/j.joen.2012.07.004 |
| [6] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5926): 167-170. |
| [7] | Wiedenheft B, Sternberg S H, Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482(7385): 331-338. DOI: 10.1038/nature10886 |
| [8] |
孙磊, 裘鹏, 刘晓琳, 等. 细菌耐药性产生对毒力的影响研究进展[J].
畜牧与兽医, 2016, 48(11): 120-123.
Sun L, Qiu P, Liu X L, et al. Research progress onbacterial resistance influencing virulence[J]. Animal Husbandry and Veterinary Medicine, 2016, 48(11): 120-123. (in Chinese with English abstract) |
| [9] |
李东, 左其生, 张亚妮, 等. 基因组编辑技术的研究进展[J].
畜牧与兽医, 2015, 47(7): 124-128.
Li D, Zuo Q S, Zhang Y N, et al. The research progress of genome editing techniques[J]. Animal Husbandry and Veterinary Medicine, 2015, 47(7): 124-128. (in Chinese with English abstract) |
| [10] | Cain A K, Boinett C J. Genmone WATCH A CRISPR view of genome sequences[J]. Nature Reviews Microbiology, 2013, 11(4): 226. DOI: 10.1038/nrmicro2997 |
| [11] | Horvath P, Romero D A, Coûté-Monvoisin A C, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophiles[J]. Journal of Bacteriology, 2008, 190(4): 1401-1412. DOI: 10.1128/JB.01415-07 |
| [12] | Comas I, Homolka S, Niemann S, et al. Genotyping of genetically monomorphic bacteria:DNA sequencing in Mycobacterium tuberculosis highlights the limitations of current methodologies[J]. PLoS ONE, 2009, 4(11): e7815. DOI: 10.1371/journal.pone.0007815 |
| [13] | Sun C L, Tomas B C, Barrangou R, et al. Metagenomic reconstructions of bacterial CRISPR loci constrain population histories[J]. The ISME Journal, 2016, 10(4): 858-870. DOI: 10.1038/ismej.2015.162 |
| [14] | Makarova K S, Haft D H, Barrangou R, et al. Evolution and classification of the CRISPR cas systems[J]. Nat Rev Microbiol, 2011, 9(6): 467-477. DOI: 10.1038/nrmicro2577 |
| [15] | Wright A V, Nunez J K, Doudna J A. Biology and applications of CRISPR systems:harnessing nature's toolbox for genome engineering[J]. Cell, 2016, 164(1): 29-44. |
| [16] | Dixit B, Ghosh K K, Fernandes G, et al. Dual nuclease activity of a Cas2 protein in CRISPR Cas subtype I-B of Leptospira interrogans[J]. FEBS Letters, 2016, 509(7): 1002-1016. |
| [17] |
冯帅, 汪莹, 李琳, 等. 沙门菌体内耐药性诱导模型的建立和体外诱导耐药基因差异分析[J].
中国兽医学报, 2014, 34(12): 1926-1930.
Feng S, Wang Y, Li L, et al. The establishment of the drug-resistance inducing model in vivo of Salmonella enterica typhimurium in the Caenorhabditis elegans and dissimilarity analysis of drug-resistance inducing S.typhimurium in vivo and in vitro[J]. Chinese Journal of Veterinary Science, 2014, 34(12): 1926-1930. (in Chinese with English abstract) |
| [18] |
贾坤, 廖燕红, 徐磊, 等. 酰基-高丝氨酸内酯(AHL)对鼠伤寒沙门氏菌生物菌膜形成的影响[J].
南京农业大学学报, 2017, 40(4): 744-749.
Jia K, Liao Y H, Xu L, et al. Effect of acylated homoserine lactones(AHL)on the biofilm formation of Salmonella typhimurium[J]. Journal of Nanjing Agricultural University, 2017, 40(4): 744-749. DOI: 10.7685/jnau.201611023 (in Chinese with English abstract) |
| [19] |
王琳琳, 王颖芳, 段广才, 等. 志贺菌CRISPR的检测及其与耐药的关系[J].
微生物学报, 2015, 55(4): 476-483.
Wang L L, Wang Y F, Duan G C, et al. Detection of CRISPR and its relationship to drug resistance in Shigella[J]. Atca Microbiologica Sinica, 2015, 55(4): 476-483. (in Chinese with English abstract) |
| [20] | Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biology, 2007, 8(4): 61. DOI: 10.1186/gb-2007-8-4-r61 |
| [21] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8(1): 172. DOI: 10.1186/1471-2105-8-172 |
| [22] | Deveau H, Garneau J E, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Annual Review of Microbiology, 2010, 64: 475-493. DOI: 10.1146/annurev.micro.112408.134123 |
| [23] | Sampson T R, Napier B A, Schroeder M R, et al. A CRISPR cas system enhances envelope integrity mediating antibiotic resistance and inflammasome evasion[J]. Proc Natl Acad Sci USA, 2014, 11l(30): 11163-11168. |
| [24] |
宋春花, 黄学勇, 郗园林, 等. 志贺菌敏感株与基因转移多耐药株蛋白组学分析[J].
中国公共卫生学, 2008, 24(1): 42-45.
Song C H, Huang X Y, Xi Y L, et al. Analysis on proteomics between sensitive strain and gene transferred multi-drug resistant strain of Shigella flexneri[J]. Chin J Public Health, 2008, 24(1): 42-45. (in Chinese with English abstract) |
| [25] | Palmer K L, Gilmore M S. Multidrug-resistant enterococci lack CRISPR-cas[J]. mBio, 2010, 1(4): e00227-10. |
| [26] | Bikard D, Hatoum-Aslan A, Mucida D, et al. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection[J]. Cell Host & Microbe, 2012, 12(2): 177-186. |




