文章信息
- 尤伟名, 朱小龙, 梁永恒
- YOU Weiming, ZHU Xiaolong, LIANG Yongheng
- 酿酒酵母脂滴代谢突变体中细胞自噬缺陷检测条件的比较
- Comparison of the detection conditions for measuring autophagy defects in lipid metabolism mutant in Saccharomyces cerevisiae
- 南京农业大学学报, 2017, 40(3): 425-433
- Journal of Nanjing Agricultural University, 2017, 40(3): 425-433.
- http://dx.doi.org/10.7685/jnau.201611024
-
文章历史
- 收稿日期: 2016-11-24
细胞自噬是真核细胞中的部分物质被自噬体包裹并送到液泡中降解的过程, 这些物质包括受损的蛋白质、糖类以及线粒体、溶酶体、内质网等[1]。近年来, 国内外对细胞自噬的研究越发关注并不断取得进展, 其中日本科学家Yoshinori Ohsumi因在细胞自噬研究领域做出的开创性工作而获得2016年的诺贝尔生理学或医学奖。然而, 细胞自噬研究领域仍有许多未解之谜, 如:除了已报道的线粒体、高尔基体、内质网等细胞器可作为自噬体膜的来源之外[2-5], 是否仍有其他膜性细胞器可作为自噬体膜的来源; 自噬体膜产生延伸后封口时, 哪些因子参与并调控了自噬体膜的封口?这些内容都是本实验室感兴趣并在研究的课题。
由于自噬体膜是含有磷脂的双层膜结构, 因此本研究大胆预测含有丰富磷脂的脂滴可能是自噬体膜的来源之一。我们在进行了简单的检测后发现酿酒酵母中磷脂合成相关单基因突变并不明显影响细胞自噬, 尽管其中CDS1的突变会影响液泡的形态[6]。Shpilka等[7]报道酵母中通过使用与脂滴合成有关的多突变体, 发现脂滴中的甘油三酯(TAG)和甾醇酯对自噬体的生成非常重要。并且饥饿条件下减少脂滴中TAG的合成会影响自噬体组装位点(PAS)对一些自噬蛋白的招募, 最终影响自噬体的形成[8]。鉴于本课题组早期使用脂滴代谢相关单突变体未能检测到自噬缺陷[6], 而后来其他实验室使用脂滴代谢相关多突变体可明显检测到自噬缺陷的情况[7], 我们拓宽了自噬检测的内容和方法, 发现以CDS1为代表的部分脂滴代谢基因在合适的自噬检测条件下, 单基因缺失也是可以检测到自噬缺陷的。本研究结果可为酿酒酵母中一些自噬缺陷不明显的基因缺失导致的自噬缺陷检测提供指导意义, 同时也再次证实了脂滴在细胞自噬中具有重要的作用。
1 材料与方法 1.1 菌株及质粒本研究中所用到的菌株见表 1。
| 菌株Strains | 别名Alias | 基因型Genotype | 菌株来源Source |
| YLY548 | WT(BY4741) | MAT ahis3-1 leu2Δ met15Δ ura3Δ | 文献[9]Reference[9] |
| YLY4718 | BY4741, ura3∷GFP-ATG8-URA3 | 本研究This study | |
| YLY5130 | BY4741, cds1-DAmP∷KanMXura3∷GFP-ATG8-URA3 | ||
| YLY5303 | BY4741, atg1Δ∷Kan ura3∷GFP-ATG8-URA3 | ||
| YLY9432 | BY4741, his3∷GFP-ATG8-HIS3 trp4∷Erg6-mCherry-Trp4 | ||
| YLY9433 | BY4741, vam3Δ∷KanMX his3∷GFP-ATG8-HIS3 trp4∷Erg6-mCherry-Trp4 | ||
| YLY9435 | BY4741, atg1Δ∷Kan his3∷GFP-ATG8-HIS3 trp4∷Erg6-mCherry-Trp4 | ||
| YNY5109 | BY4741, cho1Δ∷KanMX | 酵母菌突变体库[10] Yeast mutant library[10] |
|
| YNY2009 | BY4741, fld1Δ∷KanMX | ||
| YNY5109 | BY4741, cho1Δ∷KanMX | ||
| YNY1225 | BY4741, cho2Δ∷KanMX | ||
| YNY3343 | BY4741, opi3Δ∷KanMX | ||
| YNY985 | BY4741, ino2Δ∷KanMX | ||
| YNY3020 | BY4741, ino4Δ∷KanMX | ||
| YNY4340 | BY4741, rtc2Δ∷KanMX | ||
| YNY1231 | BY4741, mrps35Δ∷KanMX | ||
| YNY2531 | BY4741, ckb1Δ∷KanMX | ||
| YNY525 | BY4741, ckb2Δ∷KanMX | ||
| YEY154 | BY4741, cds1-DAmP∷KanMX | ||
| YNY1703 | BY4741, cax4Δ∷KanMX | ||
| 注:必需基因通过外界介导的方式使mRNA丰度下降获得突变体, 标记为DAmP[10]; 非必需基因通过敲除相应的基因获得突变体, 标记为Δ。 Note: The essential gene was mutated with DAmP(Decreased abundance by mRNA perturbation). The non-essential gene was mutated with deletion(Δ). |
|||
酵母YPD固体培养基(1 L):酵母提取物10 g、蛋白胨20 g、琼脂粉20 g, 加700 mL纯水溶解后定容至950 mL, 121 ℃灭菌20 min, 待培养基冷却至70 ℃左右加入50 mL 400 g·L-1无菌葡萄糖; SD-Trp固体培养基(1 L):酵母氮基质超常粉(YNB super powder)7.1 g, 琼脂粉20 g, 加水溶解后定容至910 mL, 灭菌后待培养基冷却至70 ℃左右加入50 mL 400 g·L-1无菌葡萄糖和40 mL 25×SD-Trp dropout(缺色氨酸氨基酸混合液); SD-Ura固体培养基(1 L):YNB super powder 7.1 g, 琼脂粉20 g, 加水溶解后定容至910 mL, 灭菌后待培养基冷却至70 ℃左右加入50 mL 400 g·L-1无菌葡萄糖和40 mL 25×SD-Ura dropout(缺尿嘧啶氨基酸混合液); 酵母缺氮液体培养基SD-N(1 L):取1.7 g无氨基酸和无过硫酸铵的酵母氮源, 加纯水溶解后定容至950 mL, 灭菌后待培养基冷却至70 ℃左右加入50 mL 400 g·L-1无菌葡萄糖。
1.3 主要试剂与仪器台盼蓝染色细胞存活率检测试剂盒购自上海碧云天生物科技有限公司; G418购自Sigma-Aldrich; Hygromycine B、鲑鱼精DNA(ssDNA)以及实验室所用的氨基酸试剂均购自北京索莱宝科技有限公司(Solarbio); 常用的酶均购自大连宝生物工程有限公司(TaKaRa); 预染蛋白Marker(PageRuler Prestained Protein Ladder)购自Fermentas公司; ECL显色液购自Millpore公司。荧光倒置相差显微镜ECLIPSE Ti-S由日本东京尼康仪器有限公司制造; UltraVIEWVoX活细胞高速共聚焦成像分析系统显微镜由PerkinElmer公司制造, 台式高速冷冻离心机5810R由Eppendorf公司制造。
1.4 构建突变体菌株从-70 ℃冰箱中取出用KanMX或Hyg敲除ATG1、VAM3等基因的菌株, 在YPD板上活化, 26 ℃培养2 d。从板上挑取单菌落到装有2 mL液体YPD的试管中, 26 ℃过夜培养, 最后提取基因组DNA。用PCR扩增出atg1Δ∷KanMX和vam3Δ∷KanMX等目的片段, 最后以BY4741为出发菌株, 通过基因敲除得到突变体菌株。引物由上海生工生物工程技术服务有限公司合成。
1.5 表型检测与方法 1.5.1 自噬表型检测将菌株在YPD平板上进行活化, 30 ℃培养箱培养3 d, 从板上挑取单菌落到2 mL液体YPD中, 26 ℃过夜培养, 菌液的D600值小于2。第2天接种60 μL过夜培养的菌液至装有6 mL液体YPD的三角瓶中, 26 ℃、200 r·min-1摇瓶培养, 使细胞浓度达到D600约0.6即可, 留取1 mL细胞用于荧光观察, 剩余细胞经氮饥饿(SD-N)处理2 h后进行荧光观察。
1.5.2 生长表型检测将酵母单克隆接种到2 mL液体YPD中, 于26 ℃、50 r·min-1过夜培养。取170 μL过夜培养的菌液在96孔板上用无菌水依次进行10倍梯度稀释, 混匀, 将菌液用点样器点在YPD板上, 26 ℃培养并拍照; 将剩余的过夜培养的菌液经SD-N处理15 d, 然后按前面所述的稀释方法将菌液点在YPD板上, 26 ℃培养, 检测菌的生长情况, 并每隔24 h拍照。
1.5.3 台盼蓝染色检测菌体存活率参照1.5.2节中方法培养细胞, 然后收集1 mL饥饿处理过的菌液, 离心去上清液后用100 μL细胞重悬液重悬细胞, 最后在细胞悬浮液中加入100 μL的台盼蓝染液(2×)对细胞染色3 min, 无菌水洗2次, 制片并用荧光显微镜观察细胞的染色情况。存活率=(细胞总数-染上色细胞数)/细胞总数×100%。
1.5.4 蛋白质免疫印迹(Western blot)检测细胞培养与1.5.1节中所述相同, 最后将饥饿处理不同时间的细胞收集起来, 离心去上清液后加入10% TCA, 涡旋混匀, -20 ℃, 30 min, 最后按常规步骤进行Western blot试验来检测细胞中GFP-Atg8的降解以及prApe1的成熟情况。所使用到的抗体有:G6PDH购自Sigma公司; GFP抗体购自Santa Cruz(圣克鲁斯)公司; Ape1抗体由日本东京工业大学Yoshinori Oshumi教授慷慨提供。使用ImageJ软件定量分析GFP-Atg8的降解情况和prApe1的成熟情况[11]。GFP降解率=条带光强度(GFP)/条带光强度(GFP+GFP-Atg8)×100%, mApe1降解率=条带光强度(mApe1)/条带光强度(prApe1+mApe1)×100%。
2 结果与分析 2.1 野生型及自噬相关基因突变体菌株中GFP-Atg8和Erg6-mCherry的定位为了检测脂滴是否能够定位到PAS上, 在野生型菌株(WT)中使用整合到染色体上的GFP-Atg8作为自噬体组装位点(PAS)的标记蛋白和Erg6-mCherry作为脂滴的标记蛋白[12], 以此为出发菌株, 采用同源重组的方法将影响自噬发生的自噬相关基因ATG1进行敲除获得atg1Δ菌株, 将影响自噬体与液泡融合的自噬相关基因VAM3进行敲除获得vam3Δ菌株, 最后通过荧光观察检测Erg6-mCherry与GFP-Atg8的共定位情况。我们首先用普通荧光显微镜观察这些菌株中GFP-Atg8和Erg6-mCherry的定位情况, 发现无论是在营养丰富(YPD)条件下还是在SD-N条件下, GFP-Atg8与Erg6-mCherry在所有菌株中都存在一定比例的重叠(图 1-A)。相同条件下, vam3Δ中重叠比例最高, WT中次之, atg1Δ中最低。与在YPD中相比, 饥饿后的菌株中GFP-Atg8与Erg6-mCherry的重叠比例升高, 尤其是atg1Δ菌株中升高最为明显(图 1-B)。

|
图 1 普通荧光显微镜观察野生型和自噬突变体菌株中GFP-Atg8与Erg6-mCherry的共定位 Figure 1 The co-localization of GFP-Atg8 and Erg6-mCherry in WT and autophagy mutants with an ordinary fluorescence microscopy A:YPD:酵母蛋白胨葡萄糖培养基; SD-N:缺氮酵母培养基。WT:野生型酵母。PhC代表相差图片, 标尺为5 μm。B:统计图A中Erg6-mCherry与GFP-Atg8重叠总数占GFP-Atg8亮点总数的比例。试验进行2次重复, 图中数值为平均值±标准差。 A:YPD:Yeast peptone dextrose medium; SD-N:Yeast synthetic complete dropout medium without amino acids and ammonium sul-fate. WT:Wild type. PhC indicates phase contrast micrograph, and the bar is 5 μm. B:The quantification of percentage of cells with overlay or contact of Erg6-mCherry with GFP-Atg8 based on GFP-Atg8 dots as shown in panel A. The values are the means±SD from two independent experiments. The same as follows. |
在相同培养条件下, 采用激光共聚焦显微镜检测了上述菌株中GFP-Atg8与Erg6-mCherry的共定位情况。由图 2可知:无论是在营养丰富条件下还是在氮饥饿条件下, GFP-Atg8与Erg6-mCherry在所有菌株中都存在一定比例的重叠, 且与在YPD中相比, 饥饿后的菌株中GFP-Atg8与Erg6-mCherry的重叠比例也会升高。相同条件下, 在WT中重叠比例最高, vam3Δ中次之, atg1Δ中最低。但与图 1中的结果相比, 图 2中GFP-Atg8与Erg6-mCherry重叠的结果要偏低, 且以vam3Δ中下降最为明显, 甚至使得该突变体从普通荧光显微镜中高于野生型变为在激光共聚焦显微镜中低于野生型。无论如何, 来自2种拍摄方法的数据均显示脂滴蛋白Erg6可以部分定位到Atg8所标记的PAS或自噬体上, 这说明脂滴可能参与细胞自噬。

|
图 2 激光共聚焦显微镜观察野生型和自噬突变体菌株中GFP-Atg8和Erg6-mCherry的共定位 Figure 2 The co-localization of GFP-Atg8 and Erg6-mCherry in WT and autophagy mutants with a Confocal microscopy A:DIC代表微分干涉对比, 标尺为5 μm。B:统计Erg6-mCherry与GFP-Atg8重叠总数占GFP-Atg8总数的比例。 A:DIC indicates differential interference contrast, the scale bar is 5 μm. B:The amount of overlay of Erg6-mCherry with GFP-Atg8 based on the amount of GFP-Atg8 was quantified. |
饥饿条件下细胞可通过降解脂滴来维持生存[13]。许多典型的atg突变体菌株在饥饿条件下极易死亡, 如:atg1Δ、atg17Δ和atg9Δ突变体菌株长时间的饥饿会使细胞大量死亡[14-15]。一些磷脂合成基因缺失会使得脂滴的形态变大, 从而使得TAG不能被正常利用[16], 那么磷脂合成基因的缺失极有可能会影响细胞自噬。为了验证这一猜想, 我们首先将这些磷脂基因缺失菌株进行15 d的长时间饥饿, 然后检测这些菌株在YPD平板上的生长情况。结果显示:所有饥饿之前的菌株在YPD平板上生长正常且无明显差异, 但将饥饿15 d的菌液收集点在YPD平板上培养1 d以后, 与野生型菌株相比, cho1Δ、mrps35Δ和cds1-DAmP突变体菌株生长相对缓慢, 而atg1Δ和cax4Δ菌株生长非常缓慢, 其他突变体菌株生长正常。这些差异在培养2 d或3 d的平板上均能观察到, 且始终以atg1Δ和cax4Δ菌株生长最差。随着生长时间的延长, 大部分菌与野生型的差异越发缩小(图 3)。这些结果说明磷脂合成基因在酵母细胞耐受氮饥饿方面是具有重要作用的。
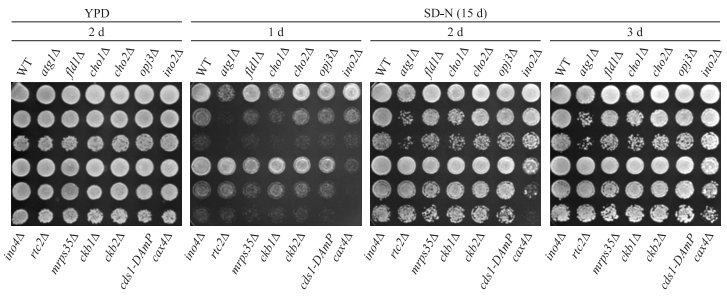
|
图 3 在YPD平板上检测磷脂基因突变体在饥饿15 d后的细胞生长情况 Figure 3 The viability of phospholipid mutants on YPD plates after starvation for 15 days |
在生长检测的基础上, 我们又采用台盼蓝试剂检测图 3中生长缓慢菌株在长时间饥饿条件下的细胞存活率。从图 4可知:在没饥饿之前, 所有菌株的细胞存活率都很高且无明显差异, 但将菌株饥饿15 d以后, 与野生型菌株相比, cho1Δ、mrps35Δ、cds1-DAmP等突变菌株中细胞的存活率明显下降, 而cax4Δ菌株中细胞的存活率无明显下降。这些结果表明:在饥饿条件下, CHO1、MRPS35、CDS1基因对于细胞的生存是非常重要的, 也意味着这些基因的缺失可能会影响细胞自噬。

|
图 4 台盼蓝试剂检测磷脂基因突变体在氮饥饿15 d后细胞的存活情况 Figure 4 The viability of phospholipid mutants with Trypan Blue staining after nitrogen starvation for 15 days A:显微镜观察台盼蓝试剂对饥饿0和15 d的菌株染色情况, 标尺为5 μm。B:统计图A中没被台盼蓝染上色的细胞数占总细胞的比例, 即存活率。 A:The starved cells in SD-N for 0 and 15 days were stained with Trypan Blue and observed by microscopy. The scale bar is 5 μm. B:Quantification for the percentage of cells unstained with Trypan Blue in Figure A. |
CDS1是酵母中的必需基因, 且酵母中大部分CDP-甘油二脂合成酶的活性受其调控[17], 上述2种活性检测结果显示CDS1突变均导致该菌株在氮饥饿条件下的存活下降, 意味着CDS1可能会影响细胞自噬。而我们早前通过免疫印迹检测单一饥饿时间点(2 h)发现CDS1突变并没有影响GFP-Atg8的降解[6]。另外, 笔者所在实验室近期发现饥饿处理之前菌体的生长情况显著影响菌株中细胞自噬的表型, 菌株浓度越高, 自噬表型越弱。为了探讨CDS1突变是否确实影响了细胞自噬的进行, 我们改进了细胞培养条件, 跟踪了不同饥饿处理时间以及增加自噬检测标记的情况下, 重新检测CDS1突变对自噬蛋白变化的影响。我们还检测了CDS1缺失是否会影响细胞自噬中GFP-Atg8的降解成GFP以及prApe1成熟为mApe1。免疫印迹试验结果(图 5)显示:虽然prApe1的成熟在一开始就部分受到抑制, 随着饥饿时间的增加, WT菌株中的GFP-Atg8大部分降解且prApe1完全成熟, atg1Δ菌株中的GFP-Atg8完全不能降解且prApe1完全不能成熟, 但当cds1-DAmP菌株饥饿4 h以上时, 其GFP-Atg8的降解明显低于野生型, 这些结果说明CDS1基因的缺失确实影响了细胞自噬的进行。
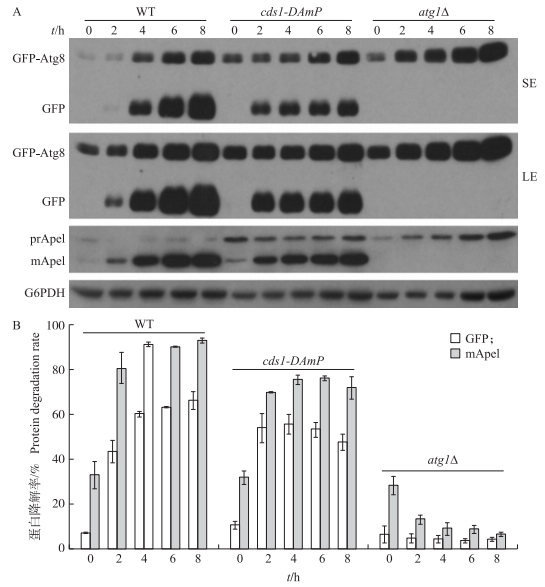
|
图 5 在不同饥饿时间下cds1-DAmP突变体中细胞自噬蛋白降解和成熟的情况 Figure 5 The degradation and maturation of autophagy proteins in cds1-DAmP mutant under different starvation times A:免疫印迹检测cds1-DAmP突变体在不同饥饿时间条件下GFP-Atg8的降解以及prApe1的成熟情况。G6PDH为加样对照。SE为短时间曝光, LE为长时间曝光。B:使用ImageJ软件定量分析条带光强度用于计算GFP-Atg8的降解以及prApe1的成熟情况。 A:The degradation of GFP-Atg8 and the maturation of prApe1 in WT and cds1-DAmP mutant under different starvation times were detected by immuno-bloting. G6PDH served as a loading control. SE:Short exposure. LE:Long exposure. B:ImageJ was used to quantify band intensities of GFP-Atg8 and GFP or prApe1 and mApe1 in panel A to calculate the percentage of GFP-Atg8 degradation and prApe1 maturation. |
本文以脂滴代谢途径中相关基因缺失突变体为研究对象, 运用荧光显微镜、细胞存活率检测、免疫印迹等手段, 优化自噬检测条件, 探究脂滴中的相关基因是否能够参与细胞自噬, 并比较不同自噬检测条件下试验结果的差异。研究结果显示:无论在营养丰富条件下还是在饥饿条件下, 采用普通荧光显微镜或激光共聚焦显微镜均能检测到脂滴标记蛋白Erg6与自噬体或自噬体组装位点标记蛋白GFP-Atg8存在一定比例的重叠或接触, 这表明脂滴可能参与细胞自噬。但用2种显微镜观察所得到的Erg6与GFP-Atg8的重叠比例存在一定的差异, 其中用激光共聚焦显微镜所获得的重叠比例偏低, 尤其是在vam3Δ菌株中, 出现这种结果可能是因为用激光共聚焦显微镜所拍的荧光图片都是通过将单细胞分多层(Z-stack)所拍得到的单层结果, 而用普通荧光显微镜所拍的图片是整个细胞的投影, 所以普通荧光显微镜中看起来重叠的不同蛋白有可能并不在同一层, 因此用普通显微镜所观察到的Erg6与GFP-Atg8重叠比例会偏高。另外, 我们通过比较相同生长条件和检测条件下不同菌株中Erg6与Atg8的重叠比例, 发现自噬过程中atg1Δ菌株中的重叠比例比自噬体大量生成, 而不能与液泡融合的vam3Δ菌株中的重叠比例要低, 说明脂滴本身并不在PAS聚集, 但随着自噬过程的发生, 脂滴与自噬体相关部位发生不同程度的接触或重叠, 并且野生型菌株中重叠比例高于atg1Δ菌株中重叠比例的结果也支持这一推测。由于激光共聚焦显微镜所具有的高清晰度、高灵敏度及所携带的Z-stack拍摄功能, 使其与那些不带Z-stack拍摄功能的普通荧光显微镜相比, 在研究细胞中不同蛋白共定位时具有绝对的优势, 所获得的研究结果更加真实、可靠, 是条件许可时的首选。但是其经济成本较高, 因此建议在前期试验中先用普通荧光显微镜进行观察, 用于摸索试验条件, 在获得初步试验结果后改用激光共聚焦显微镜来获取更加清晰、可靠的试验结果。
上述试验结果暗示脂滴极有可能作为自噬体膜磷脂的来源。另外van Zutphen等[13]报道脂滴可以为自噬细胞的存活提供能量来源, Fei等[16]报道一些磷脂合成基因缺失使得脂滴变大从而不能被细胞利用, 我们预期一些磷脂合成基因缺失突变体在氮饥饿的条件下会因为自噬过程受到影响而使得细胞活性下降。因此采用2种检测方法检测并比较了一些磷脂合成基因缺失突变菌株在长时间氮饥饿后的细胞活性。通过生长表型检测试验发现, cho1Δ、mrps35Δ、cds1-DAmP和cax4Δ突变体菌株在长时间氮饥饿后细胞活性明显下降。此外, 通过台盼蓝染色试验也发现上述前3种突变体菌株在长时间氮饥饿后大部分细胞会被台盼蓝染上色, 但cax4Δ突变体细胞能被染上色的并不多, 与野生型较为接近。出现这种情况有可能是cax4Δ突变体菌株在长时间氮饥饿以后尽管细胞死亡率低, 但细胞的代时变长, 因此在生长表型检测试验中cax4Δ突变体菌株生长缓慢。2种试验方法相比较而言, 台盼蓝染色试验操作简便快速, 成本低, 数据也直观, 是快速检测细胞活性的首选, 但一些未完全死亡的细胞可能会因染色不明显而影响试验结果的准确性, 如本研究中对cax4Δ染色的情况, 从而遗漏掉一些菌株。生长表型检测能更准确地反映出菌株的活性情况, 对试验技能的要求相对要高一些, 试验操作相对台盼蓝染色而言也更加繁琐, 试验成本高。当菌株的量大时, 建议可先采用台盼蓝染色进行初筛, 对那些不能染上色的菌株, 进一步采用生长表型检测进行确认; 而当菌株的量不大时, 可同时采用台盼蓝染色和生长表型检测来查看活性, 并最终以生长表型检测试验所获得的试验结果为准。
我们之前通过免疫印迹法检测cds1-DAmP菌株经饥饿2 h后GFP-Atg8的降解, 发现该菌株并不存在自噬缺陷, 但上述的试验结果均显示其参与了自噬过程。通过延长氮饥饿时间, 我们发现cds1-DAmP菌株随着饥饿时间的延长, GFP-Atg8的降解缺陷清晰可见, 而Ape1的成熟缺陷在各个时间段均可观察到。这些结果说明:cds1-DAmP菌株中不同自噬蛋白对环境的感受程度是不一样的, 短时间氮饥饿后细胞中的营养成分仍能够支持细胞进行代谢, 此时仅表现为部分细胞自噬过程受损, 即prApe1的成熟受到少量的影响。但当长时间饥饿的细胞将体内的能量全部消耗且TAG不能够被降解时, 细胞自噬过程的进程就会受到更严重的影响, 如GFP-Atg8的降解受阻。这些结果表明:当采用常规诱导时间不能检测到缺陷表型时, 并不表明这些突变体一定不影响自噬, 可以通过改变饥饿诱导时间和检测多种自噬标记物来进行进一步的确认。但关于CDS1是如何具体调控细胞自噬的机制仍不清楚, 未来可以对更多的与脂滴代谢有关的突变体在自噬中的作用及机制进行深入研究。
综上所述, 本研究再次证实了脂滴代谢基因参与细胞自噬, 并发现不同的自噬检测条件以及方法对试验结果会产生较大影响, 有时甚至出现不同的结论, 因此建议在以后类似的试验中采用多种方法和手段进行检测, 以便得出更为准确的结论。
| [1] | Feng Y, He D, Yao Z, et al. The machinery of macroautophagy[J]. Cell Research, 2014, 24(1): 24–41. DOI: 10.1038/cr.2013.168 |
| [2] | Hayashi-Nishino M, Fujita N, Noda T, et al. A subdomain of the endoplasmic reticulum forms a cradle for autophagosome formation[J]. Nature Cell Biology, 2009, 11(12): 1433–1437. DOI: 10.1038/ncb1991 |
| [3] | Hamasaki M, Furuta N, Matsuda A, et al. Autophagosomes form at ER-mitochondria contact sites[J]. Nature, 2013, 495(7441): 389–393. DOI: 10.1038/nature11910 |
| [4] | Yen W L, Shintani T, Nair U, et al. The conserved oligomeric Golgi complex is involved in double-membrane vesicle formation during autophagy[J]. Journal of Cell Biology, 2010, 188(1): 101–114. DOI: 10.1083/jcb.200904075 |
| [5] | Shibutani S T, Yoshimori T. A current perspective of autophagosome biogenesis[J]. Cell Research, 2014, 24(1): 58–68. DOI: 10.1038/cr.2013.159 |
| [6] |
赵秀, 朱小龙, 何亚文, 等. 酿酒酵母中磷脂合成相关基因突变对细胞自噬和液泡形态的影响[J].
南京农业大学学报, 2015, 38(1): 70–77.
Zhao X, Zhu X L, He Y W, et al. CDS1 is required for proper vacuole morphology but not for autophagy in Saccharomyces cerevisiae[J]. Journal of Nanjing Agricultural University, 2015, 38(1): 70–77. DOI: 10.7685/j.issn.1000-2030.2015.01.011 (in Chinese) |
| [7] | Shpilka T, Welter E, Borovsky N, et al. Lipid droplets and their component triglycerides and steryl esters regulate autophagosome biogenesis[J]. EMBO Journal, 2015, 34(16): 2117–2131. DOI: 10.15252/embj.201490315 |
| [8] | Li D, Song J Z, Li H, et al. Storage lipid synthesis is necessary for autophagy induced by nitrogen starvation[J]. FEBS Letters, 2015, 589(2): 269–276. DOI: 10.1016/j.febslet.2014.11.050 |
| [9] | Segev N, Botstein D. The ras-like yeast YPT1 gene is itself essential for growth, sporulation, and starvation response[J]. Molecular and Cellular Biology, 1987, 7(7): 2367–2377. DOI: 10.1128/MCB.7.7.2367 |
| [10] | Yan Z, Costanzo M, Heisler L E, et al. Yeast Barcoders:a chemogenomic application of a universal donor-strain collection carrying bar-code identifiers[J]. Nature Methods, 2008, 5(8): 719–725. DOI: 10.1038/nmeth.1231 |
| [11] | Zou S, Chen Y, Liu Y, et al. Trs130 participates in autophagy through GTPases Ypt31/32 in Saccharomyces cerevisiae[J]. Traffic, 2013, 14(2): 233–246. DOI: 10.1111/tra.12024 |
| [12] | Szymanski K M, Binns D, Bartz R, et al. The lipodystrophy protein seipin is found at endoplasmic reticulum lipid droplet junctions and is important for droplet morphology[J]. Proc Natl Acad Sci USA, 2007, 104(52): 20890–20895. DOI: 10.1073/pnas.0704154104 |
| [13] | van Zutphen T, Todde V, de Boer R, et al. Lipid droplet autophagy in the yeast Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2014, 25(2): 290–301. DOI: 10.1091/mbc.E13-08-0448 |
| [14] | Noda T, Kim J, Huang W P, et al. Apg9p/Cvt7p is an integral membrane protein required for transport vesicle formation in the Cvt and autophagy pathways[J]. Journal of Cell Biology, 2000, 148(3): 465–480. DOI: 10.1083/jcb.148.3.465 |
| [15] | Kabeya Y, Kamada Y, Baba M, et al. Atg17 functions in cooperation with Atg1 and Atg13 in yeast autophagy[J]. Molecular Biology of the Cell, 2005, 16(5): 2544–2553. DOI: 10.1091/mbc.E04-08-0669 |
| [16] | Fei W, Shui G, Zhang Y, et al. A role for phosphatidic acid in the formation of"supersized"lipid droplets[J]. PLoS Genetics, 2011, 7(7): e1002201. DOI: 10.1371/journal.pgen.1002201 |
| [17] | Shen H, Heacock P N, Clancey C J, et al. The CDS1 gene encoding CDP-diacylglycerol synthase in Saccharomyces cerevisiae is essential for cell growth[J]. Journal of Biological Chemistry, 1996, 271(2): 789–795. DOI: 10.1074/jbc.271.2.789 |




