文章信息
- 王彦杰, 陈叶清, 薛泽云, 周华, 金奇江, 徐迎春
- WANG Yanjie, CHEN Yeqing, XUE Zeyun, ZHOU Hua, JIN Qijiang, XU Yingchun
- 荷花花瓣着色过程实时荧光定量PCR内参基因的筛选及验证
- Selection and validation of reference genes for RT-qPCR normalization in lotus (Nelumbo nucifera) during petal coloration
- 南京农业大学学报, 2017, 40(3): 408-415
- Journal of Nanjing Agricultural University, 2017, 40(3): 408-415.
- http://dx.doi.org/10.7685/jnau.201609033
-
文章历史
- 收稿日期: 2016-09-24
2. 江西省观赏植物遗传改良重点实验室/江西省科学院生物资源研究所, 江西 南昌 330096
2. Key Laboratory of Horticultural Plant Genetic and Improvement of Jiangxi/Institute of Biology and Resources, Jiangxi Academy of Sciences, Nanchang 330096, China
荷花 (Nelumbo nucifera) 属于莲科 (Nelumbonaceae) 莲属的多年生挺水花卉, 其外观清新优雅、花大叶丽, 观赏价值极高, 是广泛用于公园、庭院等水景布置中的园林景观植物[1]。花色是植物重要的表型性状之一, 在很大程度上决定了其观赏及园林应用价值。目前已经登记在册的荷花品种有800多个, 但仅含有红、粉、白、黄、复色等5种花色, 色彩单一。因此, 培育更多新奇的花色品种是荷花育种的重要目标之一[2]。随着分子生物技术的不断进步与发展, 基因工程逐渐成为花色改良的重要育种途径, 也是荷花花色改良创新的主要手段。至今国内外关于荷花花色形成的分子机制研究很少, 已有的报道大多停留在花色素成分分析及花青素合成途径关键基因的克隆上[3-4], 缺乏更全面深入的分子研究。探索荷花中花色素合成相关基因的表达, 有助于明晰花色形成的调控机制, 对于利用基因工程手段改良荷花花色具有重要意义。
实时荧光定量PCR (quantitative real-time PCR, RT-qPCR) 技术具有定量准确、灵敏度高、反应迅速等特点, 已被广泛应用于生物及医学研究等领域, 成为分子生物学中研究基因表达的主要方法[5-6]。也正是这些优点, 使得它的结果往往受到RNA质量、反转录效率和初始模板量等多种因素的影响, 因此, 需要引入内参基因对其进行校正[7-9]。实际RT-qPCR分析中常常选择稳定表达的看家基因, 如肌动蛋白基因 (ACT)、18S核糖体RNA (18S rRNA)、甘油醛-3-磷酸脱氢酶基因 (GAPDH)、转录延伸因子基因 (EF1) 等作为内参基因。然而, 近年来大量研究表明, 任一看家基因的稳定表达都只是一定范围内的相对稳定, 总是恒定表达的理想内参基因几乎是不存在的[5, 8]。因此, 根据不同试验条件筛选合适的内参基因是精确分析RT-qPCR基因表达的重要前提。
迄今, 国内外已对菊花[10]、甘菊[11]、牡丹[12-13]等多种观赏植物在不同试验条件下进行了内参基因的筛选, 但对荷花的相关研究尚未见报道, 制约了荷花重要代谢进程的分子机制探究。本试验选取了8个常用看家基因 (ACT、EF1α、GAPDH、FPGS、TUA、LEU、CUL和TRY), 利用RT-qPCR检测了这些候选内参基因在荷花不同花色品种及不同花发育期花瓣中的表达水平, 并结合基因稳定性分析统计学软件geNorm[8]、NormFinder[9]和BestKeeper[14]对各候选内参基因的表达稳定性进行了评价, 以期筛选出相应试验背景下稳定表达的荷花内参基因, 为后续探索荷花花色形成关键基因的表达提供校正依据。
1 材料与方法 1.1 材料供试的4个不同花色的荷花品种为‘中国红·北京’(红色)、‘秣陵秋色’(黄色)、‘杏花粉’(粉色) 和‘珠峰翠影’(白色), 均取材于江苏省南京市艺莲苑花卉有限公司生产基地。采集各品种盛花期 (图 1-A) 的花瓣样品, 以及红色品种蕾期 (S1)、初花期 (S2)、盛花期 (S3)、末花期 (S4) 等不同花发育期 (图 1-B) 的花瓣样品, 液氮速冻后, 于-80 ℃冰箱保存备用。

|
图 1 不同花色荷花品种盛花期 (A) 及‘中国红·北京’不同花发育期 (B) 的形态 Figure 1 Lotus cultivars with different flower colors at full opening stage (A) and'Zhongguohong·Beijing' at different developmental stages (B) S1:蕾期Bud stage; S2:初花期Initial opening stage; S3:盛花期Full opening stage; S4:末花期Wilting stage. The same as follows. |
荷花花瓣总RNA的提取参照Zhang等[15]的CTAB-LiCl法, 并用RNase-free DNaseⅠ消化以去除DNA污染。利用琼脂糖凝胶电泳及Agilent 2100生物分析仪检测总RNA的质量。以各样品中1 μg总RNA为模板, 使用PrimeScript® RT reagent Kit (TaKaRa, 大连) 反转录成cDNA第一链, 于-20 ℃保存备用。
1.3 内参基因的选择和RT-qPCR引物设计在前人研究基础上[16-20], 选择荷花的ACT、EF1α、GAPDH、FPGS、TUA、LEU、CUL、TRY等8个看家基因作为候选内参基因。根据RT-qPCR引物设计原则, 采用Primer Premier 5.0和Beacon designer 7等软件设计RT-qPCR引物 (表 1), 并在NCBI Primer-BLAST上对引物的特异性进行检测。引物由上海捷瑞生物工程有限公司合成。
| 基因 Gene |
基因描述 Gene description |
GenBank登录号 GenBank accession No. |
引物序列 (5′→3′) Primer sequences |
扩增长度/bp Amplicon length |
| ACT | Actin 3 | XM_010252745 | F:GCGTTCTGCCGTCTTCTAAA R:CCCTCTTGGATTGTGCCTC |
228 |
| EF1α | Elongation factor 1-alpha | XM_010255256 | F:GTGGACAAGAAGGACGCA R:GGCATAGTAAACAACAGACC |
104 |
| GAPDH | Glyceraldehyde-3-phosphate dehydrogenase | XM_010267325 | F:GGGATTCACTTGACTCTCTGG R:AAACGGAAGGCTTGGACT |
98 |
| FPGS | Folylpoly glutamate synthase | XM_010247955 | F:TGTGCTTCCTCTGGTGTTCA R:GCCTCTGGGTTATCTATCTTGG |
212 |
| TUA | Tubulin alpha-3 chain | XM_010259330 | F:ACCGCCTCGTCTCTCTTTGG R:CGACCTGAATCCCCGCTTGT |
179 |
| LEU | Leunig | XM_010251616 | F:AAATGCGACTGGGTTGGTAG R:CTCGTGTTAGATGGAAGGGA |
246 |
| CUL | Cullin | XM_010261385 | F:TTCTGTATGGAGGTGGGA R:CGAAGTAGTAGCAAGGG |
279 |
| TRY | Tryptophan synthase alpha chain | XM_010275911 | F:TCCCTCAATCAGAGTCGTCA R:AGCCACAACCATAGAAGCCA |
96 |
| CHS | Chalcone synthase | KX176839 | F:ATTGACGGACACCTGAGAG R:AGCACACGAGCACTTGAC |
269 |
内参基因的普通PCR扩增反应在T100TM热循环仪 (Bio-Rad, USA) 中进行, 反应体系为:10×Easy Taq Buffer 2.5 μL, dNTPs (2.5 mmol·L-1)2 μL, 上、下游引物 (10 μmol·L-1) 各1 μL, cDNA模板2 μL, Easy Taq DNA Polymerase (5 U·μL-1)0.3 μL, 加ddH2O补足至25 μL。普通PCR扩增程序为:94 ℃ 5 min, 94 ℃ 30 s, 57 ℃ 30 s, 72 ℃ 30 s, 35个循环; 72 ℃ 10 min。最后用琼脂糖凝胶电泳检测扩增产物。
1.5 内参基因RT-qPCR分析内参基因RT-qPCR反应在Eppendorf Real-time PCR仪中完成, 操作参照TaKaRa公司的SYBR Premix Ex TaqTM Ⅱ的试剂盒说明书进行。反应体系如下:SYBR Premix Ex TaqTM 10 μL, 上、下游引物 (10 μmol·L-1) 各0.4 μL, cDNA模板2 μL, 加ddH2O补足至20 μL。每个样品重复3次, 并设置不加模板的阴性对照。RT-qPCR扩增程序为:95 ℃ 30 s, 95 ℃ 5 s, 55 ℃ 30 s, 72 ℃ 30 s, 40个循环, 反应结束后进行熔解曲线分析。
1.6 数据分析分别采用geNorm[8]、NormFinder[9]和BestKeeper[14]软件对8个候选内参基因在荷花不同花色品种以及不同花发育时期花瓣中的表达稳定性进行统计学分析, 从而筛选出相应试验条件下最合适的内参基因。
geNorm和NormFinder软件在导入数据时, 先将每个样品的原始CT值根据公式 (Q=2CTmin-CTsample) 转换成相对表达量Q(CTmin为依次从各候选内参基因中选出的各样品中的最小CT值; CTsample为该候选内参基因的各样品CT值); 随后, 将转换后的Q值输入geNorm或NormFinder软件, 计算得到各内参基因的平均表达稳定值 (M或S), 该值的大小反映了基因稳定性的高低。另外, geNorm软件能通过分析内参基因的配对差异值Vn/n+1, 确定试验所需内参基因的最佳数目。配对差异值以0.15为默认取舍值, 当Vn/n+1小于0.15时, 说明n个内参基因已能准确校正目的基因的表达量, 无需引入第n+1个内参。BestKeeper软件直接分析各候选内参基因原始CT值之间的标准差 (SD) 和变异系数 (CV) 即可筛选出最适内参基因。SD和CV值越小, 基因表达越稳定; 若SD值大于1, 说明该基因稳定性较差。
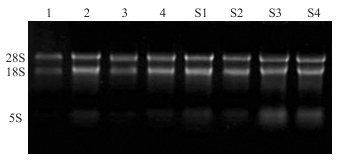
2 结果与分析 2.1 总RNA质量检测及引物特异性鉴定从荷花不同花色品种、不同花发育期花瓣中提取的总RNA的A260/A280为2.0~2.2, A260/A230为2.0~2.4;总RNA的琼脂糖凝胶电泳检测显示28S、18S条带清晰, 无降解现象 (图 2), 说明荷花各样品的总RNA提取完整性好、纯度高, 符合后续试验要求。
|
图 2 荷花各样品总RNA琼脂糖凝胶电泳图 Figure 2 Agarose gel electrophoresis analysis of total RNA extracted from different samples 1-4:分别为红、黄、粉、白色品种盛花期花瓣Petals of red, yellow, pink, and white flower cultivar at full opening stage, respectively. |
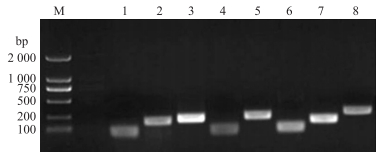
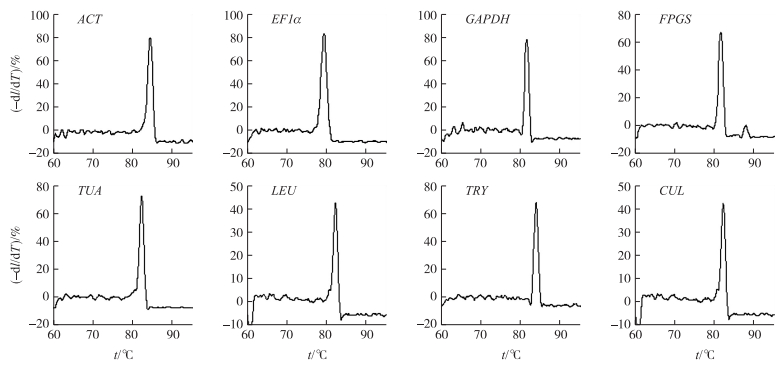
以红色荷花品种盛花期花瓣cDNA为模板, 通过普通PCR分别扩增出与8个候选内参基因预期大小一致的片段, 且产物条带单一, 没有非特异性扩增及引物二聚体 (图 3)。进一步以荷花不同花色品种及不同花发育期花瓣的cDNA为模板, 进行RT-qPCR分析, 结果 (图 4) 发现各候选内参基因的熔解曲线有且只有明显的单一信号峰, 且重复样品间扩增曲线重复性好, 说明8对引物均能特异性地扩增相应片段, RT-qPCR反应专一性高, 结果准确可靠。
|
图 3 8个候选内参基因普通PCR扩增产物的凝胶电泳检测 Figure 3 Agarose gel electrophoresis analysis 8 candidate reference genes for PCR amplification M:DNA marker; 1-8:GAPDH, FPGS, ACT, TRY, LEU, EF1α, TUA, CUL |
|
图 4 8个候选内参基因的熔解曲线 Figure 4 Melting curves of 8 candidate reference genes |
为了筛选合适的内参基因, 采用geNorm软件计算各候选内参基因在不同试验条件下的M值, M值越小表明基因表达稳定性越高[8]。geNorm分析结果 (表 2) 显示:8个候选内参基因在不同样品中的表达稳定性存在差异。在荷花不同花色品种花瓣样品中, ACT与EF1α的M值最小 (M=0.058), 表明二者表达最为稳定, 其次是FPGS, 而GAPDH的M值大于1.5, 表达变化最剧烈, 因此该基因应该被排除在候选内参基因之外; 而在不同花发育期花瓣样品中, ACT与EF1α的M值仅为0.132, 表达最稳定, 其次为LEU, 而TRY和GAPDH的表达稳定性相对较差。
| 基因 Gene |
不同花色品种Different color cultivars | 不同花发育期Different developmental stages | |||
| 排名Rank | 平均表达稳定值 (M) Average expression stability | 排名Rank | 平均表达稳定值 (M) Average expression stability | ||
| ACT | 1 | 0.058 | 1 | 0.132 | |
| EF1a | 1 | 0.058 | 1 | 0.132 | |
| FPGS | 2 | 0.211 | 4 | 0.525 | |
| LEU | 3 | 0.383 | 2 | 0.286 | |
| CUL | 4 | 0.423 | 3 | 0.399 | |
| TUA | 5 | 0.504 | 5 | 0.626 | |
| TRY | 6 | 0.842 | 7 | 0.898 | |
| GAPDH | 7 | 1.547 | 6 | 0.702 | |
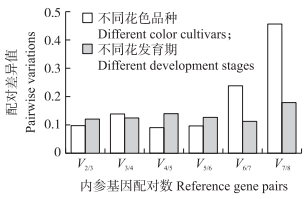
利用geNorm软件对内参基因的配对差异值Vn/n+1进行分析, 结果 (图 5) 显示, 在所检测的2个试验中V2/3均小于程序默认取舍值0.15, 说明在荷花不同花色品种及不同花发育时期花瓣样品中, 都没有必要引入第3个基因进行校正, 选择2个表达最为稳定的内参基因组合 (ACT和EF1α) 即可准确地校正目的基因的表达。
|
图 5 利用geNorm软件分析不同试验中8个候选内参基因的配对差异值 Figure 5 Pairwise variations of 8 candidate reference genes in different experiments calculated by geNorm software Vn/n+1:配对差异值Pairwise variations |
利用NormFinder软件对内参基因的稳定值 (S) 进行评价, S值越小表示基因表达越稳定[9]。分析结果 (表 3) 显示, 在不同花色品种花瓣样品中, 各候选内参基因中EF1α的S值最小 (S=0.020), 表达稳定性最高; 其次为FPGS和ACT; 而GAPDH的S值最大 (S=2.518), 表达最不稳定。在不同花发育期花瓣样品中, ACT、LEU和EF1α的表达稳定性均较高; 而TRY的S值最大 (S=0.977), 表达稳定性最差。
| 基因 Gene |
不同花色品种Different color cultivars | 不同花发育期Different developmental stages | |||
| 排名Rank | 稳定值 (S) Stability value | 排名Rank | 稳定值 (S) Stability value | ||
| EF1a | 1 | 0.020 | 3 | 0.188 | |
| FPGS | 2 | 0.096 | 4 | 0.308 | |
| ACT | 3 | 0.110 | 1 | 0.027 | |
| CUL | 4 | 0.127 | 5 | 0.429 | |
| LEU | 4 | 0.127 | 2 | 0.108 | |
| TUA | 5 | 0.380 | 7 | 0.582 | |
| TRY | 6 | 1.396 | 8 | 0.977 | |
| GAPDH | 7 | 2.518 | 6 | 0.564 | |
BestKeeper软件主要是通过比较内参基因CT值的SD和CV, 进而筛选出相应试验条件下最适合的内参基因[14]。由表 4可知:在不同花色品种花瓣样品中, 各候选内参基因中ACT的SD和CV最小, 说明该基因表达最稳定, 其次是EF1a, 而GAPDH的SD值大于1, 稳定性最差; 在不同花发育期, EF1a的SD和CV都最小, 表达最稳定; 其次是FPGS, 而GAPDH、ACT、TRY、LEU、CUL和TUA的SD值都大于1, 说明这6个基因在不同花发育期间稳定性均较差。
| 基因 Gene |
不同花色品种Different color cultivars | 不同花发育期Different developmental stages | |||
| SD | CV | SD | CV | ||
| ACT | 0.39 | 1.73 | 2.27 | 9.34 | |
| EF1a | 0.40 | 1.93 | 0.32 | 1.53 | |
| FPGS | 0.54 | 1.95 | 0.54 | 1.95 | |
| CUL | 0.68 | 2.86 | 1.06 | 4.35 | |
| TUA | 0.69 | 2.56 | 1.01 | 3.76 | |
| LEU | 0.77 | 2.82 | 1.39 | 4.91 | |
| TRY | 0.83 | 2.61 | 1.40 | 4.34 | |
| GAPDH | 3.37 | 10.93 | 3.56 | 11.59 | |
为了进一步验证各候选内参基因对RT-qPCR结果的影响, 我们分别以8个看家基因及筛选出的表达相对最稳定的EF1a+ACT组合为内参, 采用RT-qPCR技术分析了红色荷花品种不同花发育期花瓣中查耳酮合成酶基因 (CHS) 的相对表达量。由图 6所示:当以EF1a+ACT组合为内参时, 目的基因CHS在不同花发育期中的表达模式与分别以EF1a、ACT为内参时的变化规律一致, 呈现先波动增加后减少的趋势, 在盛花期时其相对表达量达到最大; 当以LEU和FPGS为内参时, CHS的表达模式与前2个内参基因的略有不同, 但仍可以基本反映基因的表达趋势; 当以其他候选看家基因 (尤其是GAPDH和TRY) 为内参时, 其表达趋势与前几个内参明显不同, 说明GAPDH和TRY这2个基因最不适合作为荷花花瓣着色过程中基因表达研究的内参基因。
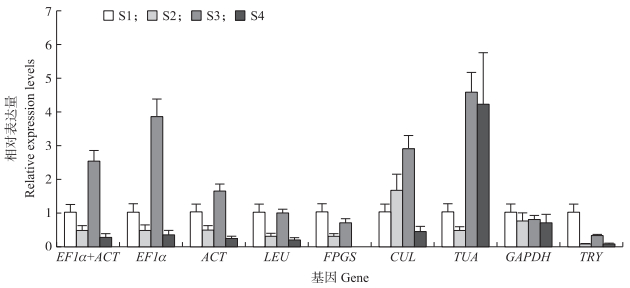
|
图 6 以不同候选基因作为内参分析不同花发育期CHS的表达 Figure 6 Expression analysis of CHS in different flower developmental stages using various candidate genes as references |
利用RT-qPCR技术检测基因表达水平是了解基因功能及其调控机制的重要前提。为了获得准确的RT-qPCR结果, 往往需要引入内参基因作为校正标准[8-9]。实际应用中常选用看家基因作为RT-qPCR分析中的内参基因, 由于这些基因广泛存在于真核细胞中, 是维持细胞结构或代谢所必需的, 常被认为在所有细胞及生理状态下表达都较稳定。然而, 越来越多的研究证实这些看家基因也受到不同试验条件的调节, 如不加以筛选盲目以一种看家基因作为任何试验条件下的内参, 容易得到精确度低甚至错误的结果[5, 8]。因此, 针对特定的试验材料和试验条件选择合适的内参基因极其重要。
本研究结果表明:geNorm和NormFinder的评价结果基本一致, 但BestKeeper的分析结果与前两者略有不同。这种差异在大豆[17]、紫花苜蓿[21]、芹菜[22]等内参基因筛选中也被报道, 推测可能是由于软件的统计学原理不同而造成的[17, 23]。
虽然3个软件的评价结果存在差异, 但大部分候选内参基因的稳定性较为一致。综合软件的分析结果, 发现GAPDH在本研究各试验条件下表达变化都相当剧烈。相似的, 以往研究发现在红三叶草不同器官[18]以及玉米不同品种和不同谷粒发育期间[24], GAPDH表达也极不稳定。此外, TRY也不是本研究各试验的合适内参, 这与Chandna等[20]对芥菜不同发育期中内参基因的筛选结果类似。本研究认为EF1α和ACT在荷花不同花色品种间及不同花发育期中表达都相对最为稳定。有趣的是, 这2个基因也是另一种重要水生观赏植物——睡莲不同组织间表达最稳定的内参基因[19]。前人研究还发现, EF1α在冷处理下马铃薯不同品种间[25], 牡丹不同花发育期中[12], 荔枝遭受弱光、环剥刻伤等胁迫时[16]都表现出很高的表达稳定性; 而ACT在紫花苜蓿茎、叶的生长期间[21], 大豆根侵染大豆疫霉过程中[17]表达也很稳定, 这些说明EF1α、ACT能在许多试验背景下作为合适的内参基因。当然, 这2个基因在某些特定条件下也表现出较差的稳定性[10-11, 13]。
本试验结果表明, 当以表达相对最稳定的EF1α、ACT以及EF1α+ACT组合分别作为内参时, 目的基因CHS表达趋势一致, 呈现先增加后减少的变化趋势, 这与牡丹、芍药花瓣CHS的表达模式[26-27]相似, 且与本课题组前期观测到的荷花花瓣总花青素含量变化基本一致[28]; 而以其他候选基因作为内参时并没有表现出相似的规律。可见, 在进行基因表达研究时, 根据不同的植物种类、处理条件等选择合适的内参基因是十分必要的。
综上所述, 当分析荷花不同花色品种间及不同花发育期中花瓣的基因表达时, 可以选择EF1α或ACT任一基因作为校正RT-qPCR数据的合适内参。然而, 不少研究证实选择2个或2个以上的内参基因, 可以减弱单一内参变化的影响, 增加结果的可靠性和精确度[8]。据此, 本研究根据对内参基因的配对差异分析, 认为同时选择相应试验条件下表达最稳定的EF1α和ACT作为内参即可。本研究的结果为研究荷花花色形成关键基因的RT-qPCR表达分析提供了可靠的校正依据。
| [1] |
王其超, 张行言.
中国荷花品种图志[M]. 北京: 中国林业出版社, 2005.
Wang Q C, Zhang X Y. Lotus Flower Cultivars in China[M]. Beijing: China Forestry Publishing House, 2005. (in Chinese) |
| [2] |
张行言.
中国荷花新品种图志Ⅰ[M]. 北京: 中国林业出版社, 2011.
Zhang X Y. New Lotus Flower Cultivars in ChinaⅠ[M]. Beijing: China Forestry Publishing House, 2011. (in Chinese) |
| [3] | Deng J, Chen S, Yin X J, et al. Systematic qualitative and quantitative assessment of anthocyanins, flavones and flavonols in the petals of 108 lotus (Nelumbo nucifera) cultivars[J]. Food Chemis TRY, 2013, 139: 307–312. DOI: 10.1016/j.foodchem.2013.02.010 |
| [4] | Dong C, Yu A Q, Wang M L, et al. Identification and characterization of chalcone synthase cDNA (NnCHS) from Nelumbo nucifera[J]. Cell Mol Biol, 2015, 61(8): 112–117. |
| [5] | Bustin S A, Benes V, Garson J A, et al. The MIQE guidelines:minimum information for publication of quantitative real-time PCR experiments[J]. Clinical Chemistry, 2009, 55(4): 611–622. DOI: 10.1373/clinchem.2008.112797 |
| [6] | Postollec F, Falentin H, Pavan S, et al. Recent advances in quantitative PCR (qPCR) applications in food microbiology[J]. Food Microbiology, 2011, 28(5): 848–861. DOI: 10.1016/j.fm.2011.02.008 |
| [7] | Bustin S A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays[J]. Journal of Molecular Endocrinology, 2000, 25: 169–193. DOI: 10.1677/jme.0.0250169 |
| [8] | Vandesompele J, de Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): research0034. |
| [9] | Andersen C L, Jensen J L, Orntoft T F. Normalization of real-time quantitative reverse transcription PCR data:a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64: 5245–5250. DOI: 10.1158/0008-5472.CAN-04-0496 |
| [10] | Fu J X, Wang Y, Huang H, et al. Reference gene selection for RT-qPCR analysis of Chrysanthemum lavandulifolium during its flowering stages[J]. Mol Breeding, 2013, 31: 205–215. DOI: 10.1007/s11032-012-9784-x |
| [11] | Qi S, Yang L W, Wen X H, et al. Reference gene gelection for RT-qPCR analysis of flower development in Chrysanthemum morifolium and Chrysanthemum lavandulifolium[J]. Frontiers in Plant Science, 2016, 7: 1–12. |
| [12] | Li J, Han J G, Hu Y H, et al. Selection of reference genes for quantitative real-time PCR during flower development in tree peony (Paeonia suffruticosa Andr.)[J]. Frontiers in Plant Science, 2016, 7: 1–9. |
| [13] |
王彦杰, 董丽, 张超, 等. 牡丹实时定量PCR分析中内参基因的选择[J].
农业生物技术学报, 2012, 20(5): 521–528.
Wang Y J, Dong L, Zhang C, et al. Reference gene selection for real-time quantitative PCR normalization in tree peony (Paeonia suffruticosa Andr.)[J]. Journal of Agricultural Biotechnology, 2012, 20(5): 521–528. (in Chinese) |
| [14] | Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes, and sample integrity:Bestkeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26: 509–515. DOI: 10.1023/B:BILE.0000019559.84305.47 |
| [15] | Zhang Y J, Hao X Y, Liang Z S, et al. Efficient isolation of high-quality RNA from lotus Nelumbo nucifera ssp.nucifera tissues[J]. Genetics and Molecular Research, 2013, 12: 223–229. DOI: 10.4238/2013.January.24.14 |
| [16] | Zhong H Y, Chen J W, Li C Q, et al. Selection of reliable reference genes for expression studies by reverse transcription quantitative real-time PCR in litchi under different experimental conditions[J]. Plant Cell Rep, 2011, 30: 641–653. DOI: 10.1007/s00299-010-0992-8 |
| [17] |
曾文韬, 柴春月, 窦道龙. 适用于大豆实时荧光定量PCR分析的内参基因的筛选和验证[J].
南京农业大学学报, 2015, 38(5): 787–795.
Zeng W T, Chai C Y, Dou D L. Selection and validation of reference genes for quantitative RT-PCR analysis in soybean[J]. Journal of Nanjing Agricultural University, 2015, 38(5): 787–795. DOI: 10.7685/j.issn.1000-2030.2015.05.013 (in Chinese) |
| [18] | Khanlou K M, Bockstaele E V. A critique of widely used normalization software tools and an alternative method to identify reliable reference genes in red clover (Trifolium pratense L.)[J]. Planta, 2012, 236(1381): 1393. |
| [19] | Luo H L, Chen S M, Wan H J, et al. Candidate reference genes for gene expression studies in water lily[J]. Analytical Biochemis TRY, 2010, 404: 100–102. DOI: 10.1016/j.ab.2010.05.002 |
| [20] | Chandna R, Augustine R, Bisht N C. Evaluation of candidate reference genes for gene expression normalization in Brassica juncea using real time quantitative RT-PCR[J]. PLoS ONE, 2012, 7(5): e36918. DOI: 10.1371/journal.pone.0036918 |
| [21] |
张微, 崔为体, 段星亮, 等. UV-B胁迫下紫花苜蓿qRT-PCR内参基因的筛选[J].
南京农业大学学报, 2015, 38(2): 248–254.
Zhang W, Cui W T, Duan X L, et al. Reference gene selection for qRT-PCR normalization in alfalfa under UV-B irradiation[J]. Journal of Nanjing Agricultural University, 2015, 38(2): 248–254. DOI: 10.7685/j.issn.1000-2030.2015.02.011 (in Chinese) |
| [22] | Li M Y, Wang F, Jiang Q, et al. Validation and comparison of reference genes for QPCR normalization of celery (Apium graveolens) at different development stages[J]. Original Research, 2016, 7: 1–12. |
| [23] | Hu R B, Qi G, Kong Y Z, et al. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa[J]. BMC Plant Biology, 2010, 10: 145. DOI: 10.1186/1471-2229-10-145 |
| [24] | Galli V, Messias R S, Silva SDA, et al. Selection of reliable reference genes for quantitative real-time polymerase chain reaction studies in maize grains[J]. Plant Cell Rep, 2013, 32: 1869–1877. DOI: 10.1007/s00299-013-1499-x |
| [25] | Lopez-Pardo R, de Galarreta J I R, Ritter E. Selection of housekeeping genes for qRT-PCR analysis in potato tubers under cold stress[J]. Mol Breeding, 2013, 31: 39–45. DOI: 10.1007/s11032-012-9766-z |
| [26] | Zhao D Q, Tao J, Han C X, et al. Flower color diversity revealed by differential expression of flavonoid biosynthetic genes and flavonoid accumulation in herbaceous peony (Paeonia lactiflora Pall.)[J]. Mol Biol Rep, 2012, 39: 11263–11275. DOI: 10.1007/s11033-012-2036-7 |
| [27] | Zhang C, Wang W N, Wang Y J, et al. Anthocyanin biosynthesis and accumulation in developing flowers of tree peony (Paeonia suffruticosa)'Luoyang Hong'[J]. Postharvest Biology and Technology, 2014, 97: 11–22. DOI: 10.1016/j.postharvbio.2014.05.019 |
| [28] | Wang Y J, Chen Y Q, Yuan M, et al. Flower color diversity revealed by differential expression of flavonoid biosynthetic genes in sacred lotus[J]. J Amer Soc Hort Sci, 2016, 141(6): 1–10. |




