文章信息
- 林莹, 韩玉辉, 熊思嘉, 李泽斌, 张欣, 顾婷婷
- LIN Ying, HAN Yuhui, XIONG Sijia, LI Zebin, ZHANG Xin, GU Tingting
- 森林草莓组蛋白去乙酰化酶基因的鉴定和分析
- Identification and analysis of histone deacetylases in Fragaria vesca
- 南京农业大学学报, 2017, 40(2): 225-233
- Journal of Nanjing Agricultural University, 2017, 40(2): 225-233.
- http://dx.doi.org/10.7685/jnau.201607018
-
文章历史
- 收稿日期: 2016-07-15
在真核生物中, 基因表达受到染色质结构的调控。组蛋白修饰可以通过影响DNA与组蛋白的结合, 或者募集其他转录因子和染色质蛋白而调控基因的活跃/沉默状态, 其中乙酰化修饰与基因的活跃表达有着密切的联系。在真核生物中, 组蛋白的乙酰化和去乙酰化是一个动态可逆的过程, 而且直接与基因的转录激活与沉默相关[1]。核心组蛋白的乙酰化能诱导一个“开放”的染色质结构而有利于基因表达, 相反的, 组蛋白去乙酰化能造成一个“关闭”的染色质结构而导致基因沉默[2]。
组蛋白乙酰化修饰是一个由组蛋白乙酰化酶和去乙酰化酶共同调控的动态过程, 其中组蛋白去乙酰化酶 (histone deacetylases-HDACs) 可以催化组蛋白和非组蛋白的去乙酰化[3]。根据序列相似性, 在植物中, HDACs可以被划分为3个家族:RPD3/HDA1家族, SIR2家族和植物特有的HD2家族[4]。RPD3/HDA1家族蛋白有核心结构域Hist_deacety, 其催化功能需要Zn2+的辅助[3]; SIR2家族蛋白则需要核心结构域SIR2与NAD辅助因子发挥其去乙酰化功能。模式植物拟南芥中共有18个HDAC基因, 其中12个基因编码蛋白属于RPD3/HDA1家族 (其中2个仅有部分去乙酰化功能), 2个属于SIR2家族, 4个属于HD2家族[4]。
HDACs在植物的生长发育过程中有着重要作用。RPD3/HDA1家族通过调控重要基因的表达而参与很多重要的生理生化途径。譬如, 在拟南芥中, AtHDA56参与调控开花时间[5], AtHDA在维持DNA甲基化和基因沉默中发挥了一定作用[6-7], AtHDA19和AtHDA6参与调控开花和逆境胁迫响应[8-10], AtHDA18与根的形成密切相关[11-12]。SIR2家族基因参与能量代谢和代谢物转运[13], 其中, AtSRT2可能在线粒体能量代谢中有重要的调节作用。此外, SIR2家族可能在拟南芥对病原菌的抗性具有重要作用[14]。HD2家族在拟南芥花、茎、叶、胚等均具有较高表达水平[1], 在干旱、盐等逆境胁迫中也具有重要调控作用[14]。HDACs在玉米、水稻中的功能和作用也有少量报道, 玉米的RPD3/HDA1家族基因ZmHDA101通过参与特定DNA序列的组蛋白修饰来调控基因表达和玉米的发育过程[15]; 水稻中HD2家族基因OsHDT701主导H4的去乙酰化, 并通过调节H4的乙酰化水平而参与调控水稻自身免疫系统[16]。HDACs在其他植物中的报道也说明了HDACs在植物中结构和功能的保守性和多样性。
草莓是一种重要的非呼吸跃变型果实作物, 而表观遗传修饰作为一种重要的基因调控机制, 在草莓这种肉质果实中的研究仍相对欠缺。目前已有关于组蛋白甲基化修饰基因以及DNA甲基化相关基因在草莓中的研究[17-18], 但组蛋白乙酰化修饰在草莓中的研究仍很少。二倍体森林草莓的测序为从基因组水平上挖掘森林草莓HDAC基因提供了可靠数据和重要参考[19]。同时, 草莓也是一种重要的经济作物, 且我国是世界上草莓种植面积最大、产量最高的国家, 高温和低温等逆境胁迫作为常见的不可控环境因素, 对草莓生长发育以及产量有着重要的影响。而乙酰化修饰作为一种重要的表观调控机制参与了植物逆境胁迫的响应过程, 因此, 森林草莓中乙酰化修饰酶的鉴定和研究具有重要的意义。本研究利用生物信息学方法, 鉴定出森林草莓中的HDAC基因, 并对这些基因进行了进化分析和表达分析, 为进一步研究组蛋白去乙酰化在草莓生长发育中的作用以及分子育种提供了可靠参考。
1 材料与方法 1.1 数据来源森林草莓 (Fragaria vesca) 和中国莲 (Nelumbo nucifera) 的数据来源于NCBI, 桃 (Prunus persica)、黄瓜 (Cucumis sativus)、拟南芥 (Arabidopsis thaliana)、葡萄 (Vitis vinifera) 和番茄 (Solanum lycopersicum) 数据来源于Phytozome (version 10.3;http://phytozome.jgi.doe.gov/pz/portal.html)。
1.2 HDAC基因的鉴定与保守序列分析从Pfam网站分别下载RPD3/HDA1和SIR2家族关键结构域序列 (Hist_deacety domain, PF00850;SIR2 domain, PF02146), 利用隐马尔科夫模型软件HMMER进行比对搜索所选物种中的HDAC基因。对于HD2家族HDAC基因, 通过MEME网站分析得到拟南芥中所有HD2蛋白的保守序列, 并构建HMM文件, 然后利用HMMER软件搜索所选物种中HD2基因。将所有候选HD2基因提交MEME网站进行motif分析, 保留与拟南芥HD2蛋白motif相似的序列, 得到所有的HD2基因。将得到的所有候选基因提交Pfam[20]、CDD[21]网站, 保留其中包含HDAC基因关键结构域或保守序列的基因, 并得到HDAC基因中包含的其他保守结构域, 其中设置e不大于10-4。数据处理中, 保留同一基因不同可变剪切中最长的氨基酸序列。
1.3 多序列比对和系统进化树构建对RPD3/HDA1基因家族运用MAFFT (V7.158b) 软件对全蛋白序列进行比对, 得到的比对数据运用Raxml (Version 8.1.16) 软件构建系统发生进化树。参数设置为:MAFFT比对设置为默认参数, Raxml设置为γ分布和1 000次Bootstrap重复; SIR2家族和HD2家族蛋白序列, 运用MEGA 5.0进行序列比对, 并用邻近法Neighbor-joining构建进化树, Bootstrap设置为1 000。
1.4 基因染色体定位与基因复制类型分析从森林草莓基因组注释信息提取HDAC基因相关染色体位置信息, 运用MapInspect软件构建基因染色体定位图; 运用McscanX软件[22]对HDAC基因进行复制类型分析, McscanX参数设置为默认参数。
1.5 森林草莓中HDAC基因的表达分析根据森林草莓中HDAC基因的基因ID从SGR (http://bioinformatics.towson.edu/strawb-erry/Default.aspx) 网站下载相应基因的表达数据。数据处理过程为:计算生物学重复平均值, 并取其值log2, 运用Mev9(Version 4.9.0) 软件将数据可视化。
1.6 材料处理、RNA提取和实时荧光定量PCR将所有生长于MS培养基上的30 d大小的二倍体森林草莓苗 (‘Rugen F7-4’) 避光开盖24 h后, 分别进行冷胁迫处理和热胁迫处理。其中, 低温胁迫处理:在避光、4 ℃低温条件下分别处理0、1和8 h。高温胁迫处理方法:在避光、38 ℃高温条件下分别处理0、1和3 h, 并在热处理3 h结束后分别进行1 h (共4 h) 和5 h (共8 h) 恢复处理。采用CTAB法提取对照组和处理组草莓苗RNA, 利用TaKaRa公司的PrimeScript® RT reagent Kit进行反转录, 获得用于实时荧光定量PCR的模板cDNA。选用森林草莓的FvGAPDH2作为内参基因[23], 试验中所用特异性引物序列见表 1, 数据处理采用2-ΔΔCT方法[24]。每个数据2个生物学重复、3个技术重复。显著性检验由SPSS (IBM SPSS Statistics 19) 软件完成。
| 基因名Gene name | 上游引物 (5′→3′) Forward primer | 下游引物 (5′→3′) Reverse primer |
| FvGAPDH2 | CCCAAGTAAGGATGCCCCCATGTTCG | TTGGCAAGGGGAGCAAGACAGTTGGTAG |
| FvHDA1 | GGAGCAGGAATTGCCTTAATTG | TGGTGTCCTGGAGGTCTTAT |
| FvHDA2 | GTCCCGTATTCGATGACCTATTC | CGATATCAGCGTCTCCTTGATT |
| FvHDA3 | GATGCTGGACTGGATGAGATTAC | CATCTGATCCGCAGGCTTATT |
| FvHDA4 | AAGTTTGCTCGGGAGAGAAATA | GAGAGGTTGGTTATCGAGTCTG |
| FvHDA5 | AGGCCCAATCTCTCCTTACA | GCATCTTGCCTCCCTCTTTATC |
| FvHDA6 | GATGGAGTTGAGGAGGCATTT | CTGCTCCGACATCCTTGATATG |
| FvHDA7 | GCATCCAACATGCTCCTAGT | CATCAGGGTTCTGCTCATCTT |
| FvHDA8 | GGAACGGCACCCAAGAAATA | CAGCTCCAGTACCAGGATAGAA |
| FvHDA9 | GAGAAGGGTTGGTACCGTAATG | GGATTTGAAGCCCTCGATGT |
| FvHDT1 | GGGAAGACTCCTGCTACTGATA | CATCTGAACCGAATGCCTTACT |
| FvSRT1 | GGACAGAGCTGCTAAGGTAATC | TGACAAGTCGAAAGGCAGAAA |
| FvSRT2 | ATGGAAGTCTGCCCTTCTTG | CACCTTCTGGAAGTCTCCTTTAG |
通过全基因组的搜索, 鉴定出所选7个物种中所有的HDAC基因, 并根据拟南芥中基因命名方式, 对不同家族基因以相应物种拉丁名和基因名结合的方式命名, 并对各基因名从1开始顺序编号 (表 2)。森林草莓中, 共鉴定出HDAC基因12个, 其中, RPD3家族9个、SIR2家族2个、HD2家族1个 (图 1)。其他6个双子叶物种中鉴定出13~18个。所选取的物种中HDAC基因的数目在经历几次全基因组复制事件后并没有发生巨大变化, 而是维持在一个相对稳定的数目 (图 1)。
| 基因家族 Gene family |
分类 Class |
拟南芥Arabidopsis thaliana | 森林草莓Fragaria vesca | |||||
| 基因名 Gene name |
基因号 GenBank accession No. |
染色体 Chromosome |
基因名 Gene name |
基因号 GenBank accession No. |
染色体 Chromosome |
|||
| RPD3/HDA1 | Ⅰ | AtHDA6(RPD3B) | AT5G63110 | 5 | FvHDA6 | gene09844 | 5 | |
| AtHDA7 | AT5G35600 | 5 | FvHDA2 | gene12349 | 1 | |||
| AtHDA9 | AT3G44680 | 3 | FvHDA7 | gene29263 | 5 | |||
| AtHDA19(RPD3A) | AT4G38130 | 4 | FvHDA3 | gene17127 | 2 | |||
| Ⅱ | AtHDA5 | AT5G61060 | 5 | FvHDA8 | gene26215 | 5 | ||
| AtHDA15 | AT3G18520 | 3 | FvHDA9 | gene25808 | 5 | |||
| AtHDA18 | AT5G61070 | 5 | ||||||
| Ⅲ | AtHDA2 | AT5G26040 | 5 | FvHDA4 | gene02678 | 2 | ||
| Ⅳ | AtHDA14 | AT4G33470 | 4 | FvHDA1 | gene16203 | 1 | ||
| V | AtHDA8 | AT1G08460 | 1 | FvHDA5 | gene27053 | 2 | ||
| SIR2 | Ⅰ | AtSRT1 | AT5G55760 | 5 | FvSRT2 | gene15261 | 1 | |
| Ⅱ | AtSRT2 | AT5G09230.7 | 5 | FvSRT1 | gene16117 | 2 | ||
| HD2 | AtHDT1 | AT3G44750 | 3 | FvHDT1 | gene14356 | 3 | ||
| AtHDT2 | AT5G22650 | 5 | ||||||
| AtHDT3 | AT5G03740 | 5 | ||||||

|
图 1 物种进化树和HDAC基因数目 Figure 1 Phylogenetic tree of 7 species and the number of HDAC genes |
为进一步探究HDAC基因的进化历程, 对每个HDAC基因家族进行了系统进化分析。结果发现RPD3家族中基因可进一步划分为5个分类 (图 2), 除了已有文献报道的3类, 拟南芥中未分类的基因AtHDA8和AtHDA14分别在不同分支内 (图 2)。这2个基因被划分为2个不同的分类, 与之前拟南芥中的报道略有差异[1, 4]。对RPD3家族中蛋白保守结构域分析发现, 该家族蛋白仅包含去乙酰化酶核心结构域 (Hist_deacetyl domain, PF00850)。大部分Hist_deacetyl结构域长度为200~400个氨基酸。也有少数蛋白的Hist_deacetyl结构域长度小于200个氨基酸, 这些蛋白可能丧失了组蛋白去乙酰化的功能。

|
图 2 森林草莓与其他物种中RPD3家族氨基酸序列系统进化树 Figure 2 Phylogenetic tree of the amino acid sequences of RPD3 family in Fragaria vesca and other species At:拟南芥Arabidopsis thaliana; Fv:森林草莓Fragaria vesca; Nn:中国莲Nelumbo nucifera; Pp:桃Prunus persica. The same as follows. |
SIR2家族氨基酸序列进化树显示, SIR2家族中基因可以明显划分为两类, 这与之前报道一致[4]。尽管在SIR2家族进化树中部分Bootstrap值较小, 但对该家族蛋白序列保守结构域分析的结果支持了SIR2家族进化树的2种分类 (图 3)。通过进化树分析, 将位于同一进化树分支上的森林草莓HDAC基因与拟南芥中基因相对应 (表 2), 通过拟南芥中HDAC基因的已知功能可以推测森林草莓中直系同源HDAC基因的可能功能。

|
图 3 森林草莓与其他物种中SIR2家族氨基酸序列系统进化树分析 Figure 3 Phylogenetic tree of the amino acid sequences of SIR2 family in Fragaria vesca and other species |
在HD2家族中, 目前仅发现其N端序列比较保守。Motif分析发现, 所选取的4个物种中所有的HD2基因, 其N端均包含相同的保守motif序列, 分别为motif 1、motif 3、motif 4、motif 11和motif 14(图 4)。同时morif 2、motif 5、motif 7、motif 8、motif 10和motif 12在大多数HD2家族基因中相对保守, 表明这些motif对于HD2家族基因的功能可能有重要作用。
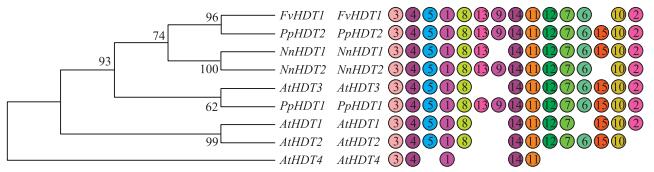
|
图 4 森林草莓和其他物种HD2家族motif组成 Figure 4 Motif construction of HD2 family in Fragaria vesca and other species 相同颜色的圆圈代表不同物种间相同的motif, 圆圈中的数字代表不同的motif。The same colors of circles refer to identical motifs among different species, and the numbers in circles represent different motifs. |
利用森林草莓基因组注释信息, 将HDAC基因定位到森林草莓相应的染色体上。结果 (图 5) 表明, HDAC基因随机分布在1号、2号、3号和5号染色体上, 4号、6号和7号染色体上没有HDAC基因, 3号染色体上仅仅发现1个植物中特有的HDAC基因。为进一步探究森林草莓中组蛋白去乙酰化酶基因的进化历程, 运用McscanX软件对森林草莓中所有HDAC基因的复制进行了分析。结果 (表 3) 表明, 在RPD3这一最大的HDAC基因家族中, 所有的森林草莓基因均由离散复制产生; HD2家族中的单个HDAC基因也均由离散复制产生。森林草莓中各个HDAC家族中均没有通过WGD/大片段复制方式而扩张 (图 1), 这与森林草莓在近期没有发生过WGD事件相一致[25]。对其他研究中物种HDAC基因的复制类型分析发现, 在这6个双子叶植物共86个HDAC基因中, 56个与离散复制相关, 16个与WGD相关, 其余14个与近端复制、串联复制相关或为单基因 (表 3), 这表明离散复制和WGD/片段复制对于HDAC基因在植物中的扩张有重要作用。
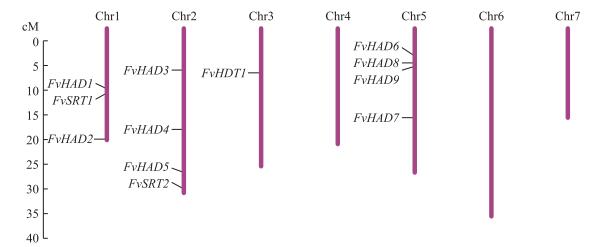
|
图 5 森林草莓HDAC基因染色体定位 Figure 5 Chromosomal location of FvHDAC genes in Fragaria vesca |
| 基因家族 Gene faminly |
物种 Species |
单基因 Singleton |
离散复制 Dispersed |
近端复制 Proximal |
串联复制 Tandem |
全基因组/片段复制 WGD/segmental |
| RPD3/HDA1 | 桃Prunus persica | 0 | 7 | 0 | 2 | 0 |
| 森林草莓Fragaria vesca | 0 | 9 | 0 | 0 | 0 | |
| 黄瓜Cucumis sativus | 0 | 7 | 0 | 0 | 2 | |
| 拟南芥Arabidopsis thaliana | 1 | 7 | 2 | 2 | 0 | |
| 葡萄Vitis vinifera | 0 | 10 | 0 | 0 | 0 | |
| 番茄Solanum lycopersicum | 0 | 7 | 0 | 0 | 2 | |
| 中国莲Nelumbo nucifera | 1 | 8 | 0 | 0 | 2 | |
| SIR2 | 桃Prunus persica | 2 | 0 | 0 | 0 | 0 |
| 森林草莓Fragaria vesca | 2 | 0 | 0 | 0 | 0 | |
| 黄瓜Cucumis sativus | 0 | 1 | 2 | 0 | 0 | |
| 拟南芥Arabidopsis thaliana | 2 | 0 | 0 | 0 | 0 | |
| 葡萄Vitis vinifera | 0 | 2 | 0 | 0 | 0 | |
| 番茄Solanum lycopersicum | 0 | 2 | 0 | 0 | 0 | |
| 中国莲Nelumbo nucifera | 0 | 2 | 0 | 0 | 0 | |
| HD2 | 桃Prunus persica | 0 | 0 | 0 | 0 | 2 |
| 森林草莓Fragaria vesca | 0 | 1 | 0 | 0 | 0 | |
| 黄瓜Cucumis sativus | 0 | 1 | 0 | 2 | 0 | |
| 拟南芥Arabidopsis thaliana | 0 | 2 | 0 | 0 | 1 | |
| 葡萄Vitis vinifera | 0 | 0 | 0 | 0 | 2 | |
| 番茄Solanum lycopersicum | 0 | 0 | 0 | 0 | 3 | |
| 中国莲Nelumbo nucifera | 0 | 0 | 0 | 0 | 2 |
从SGD下载森林草莓花发育不同阶段、果实发育早期以及不同组织的转录组数据 (http://bioinformatics.towson.edu/strawberry/)[26-28], 对数据进行进一步处理后, 通过Mev软件将数据可视化, 得到森林草莓中HDAC基因的表达图 (图 6)。
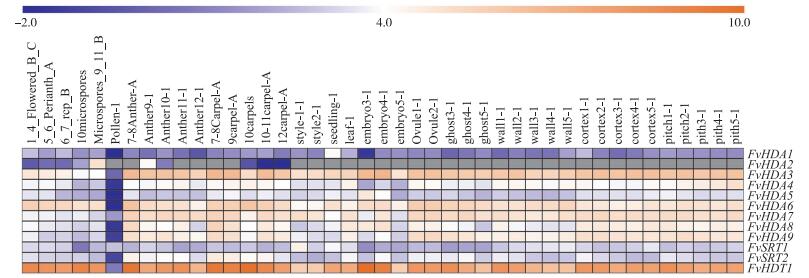
|
图 6 森林草莓HDAC基因表达图 Figure 6 Expression patterns of FvHDAC genes in Fragaria vesca |
在森林草莓HDAC基因中, 3个基因家族的表达模式各有特点。RPD3家族中, 大部分基因在花药、心皮、皮质和髓中表达水平较高, 表明该家族基因可能参与调控了森林草莓果实的发育。同时发现, 与拟南芥AtHDA6和AtHDA19对应的森林草莓FvHAD3和FvHAD6基因在花发育过程中表达水平显著高于其他基因, 表明这2个基因可能与森林草莓花发育密切相关。已知拟南芥中AtHDA6和AtHDA19参与调控拟南芥开花时间, 推测FvHAD3和FvHAD6的功能可能与拟南芥中AtHDA6和AtHDA19基因具有相似性[1]。在SIR2家族中, FvSRT1在花粉中表达水平显著高于其他HDAC基因, 表明该基因可能参与调控森林草莓花粉发育。而FvSRT2在花药和心皮中的表达水平高于在其他组织中的表达水平, 说明FvSRT2可能在森林草莓花药和心皮发育中有特定的作用。植物特有的一类HDAC基因FvHDT1, 除了花粉外, 在整个森林草莓花发育和果实发育过程中, 表达水平均显著高于其他HDAC基因。以上分析表明, 森林草莓HDAC基因在不同发育阶段呈现组织特异性表达模式, 各个HDAC基因可能发生了特定的功能分化。
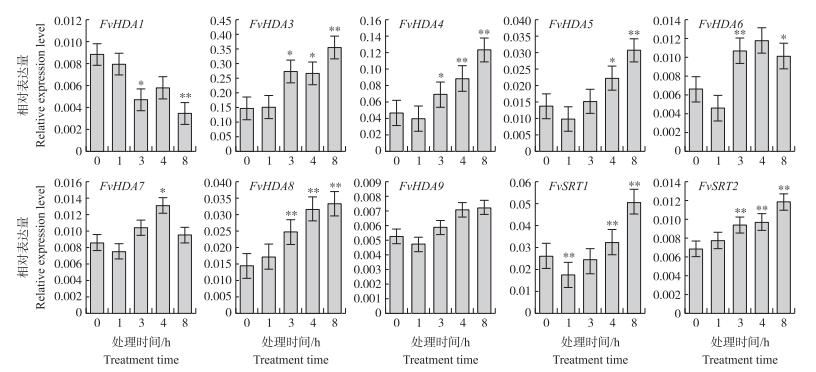
2.5 低温胁迫和高温胁迫中森林草莓HDAC基因的表达分析为了探究森林草莓HDAC基因在逆境胁迫中的表达变化, 对森林草莓幼苗分别进行4 ℃低温胁迫和38 ℃高温胁迫处理, 并利用实时荧光定量PCR检测相关基因的表达水平。由图 7可见:在低温胁迫8 h后, FvHDA1表达水平显著升高, 而其他基因没有发生显著变化。表明FvHDA1在森林草莓低温胁迫响应过程中可能发挥了一定调控作用。由图 8可见:在高温胁迫中, FvHDA1、FvHDA3、FvHDA4、FvHDA6、FvHDA8、FvSRT2在高温胁迫3 h后表达显著升高; FvHDA1、FvHDA3、FvHD4、FvHD5、FvHDA6、FvHDA8、FvSRT1和FvSRT2在高温胁迫3 h并恢复5 h后表达显著升高, 说明这些基因可能参与森林草莓对高温胁迫的响应。FvHDA1在低温和高温胁迫中其表达水平都有显著变化, 表明该基因可能在森林草莓响应逆境胁迫过程中有重要作用。
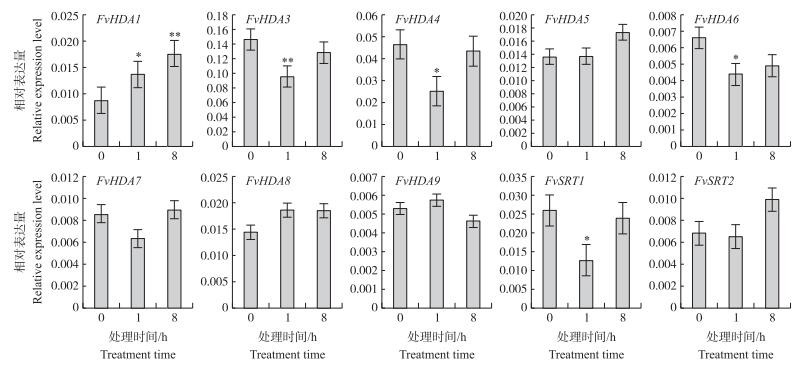
|
图 7 低温胁迫条件下森林草莓HDAC基因表达分析 Figure 7 Expression patterns of FvHDAC genes in response to cold stress *P < 0.05, **P < 0.01. The same as follows. |
|
图 8 高温胁迫条件下森林草莓HDAC基因表达分析 Figure 8 Expression patterns of FvHDAC genes in response to heat stress |
组蛋白乙酰化修饰在拟南芥生长发育过程中具有重要作用[1, 28], 而乙酰化修饰相关基因在草莓中的研究还非常有限。在本研究中, 我们运用生物信息学方法, 对森林草莓中HDAC基因进行了鉴定, 并从进化、功能预测和基因表达方面进行了分析。在森林草莓中我们共鉴定出12个HDAC基因, 其中RPD3/HDA1家族9个, SIR2家族2个, HD2家族1个。结构域和motif分析表明, RPD3/HDA1家族和SIR2家族都只含有去乙酰化核心结构域, 而HD2家族在N端有与去乙酰化功能相关的保守motif。染色体定位和基因复制类型分析发现, 森林草莓不同HDAC家族中基因复制类型有较大差异, 离散复制对HDAC基因在森林草莓的进化中具有重要作用。对HDAC基因在森林草莓不同组织和不同发育阶段及逆境胁迫响应中的表达模式研究表明, 这些基因可能在不同组织/发育阶段逆境响应过程中有不同的作用方式, 但这些HDAC基因的具体调控功能还有待于进一步的试验验证。
我们研究的7个物种在进化历程中经历了不同次数的全基因组复制事件, 但是HDAC基因的数目并不与WGD发生的次数呈绝对的正相关关系。可见, 在双子叶进化过程中, HDAC基因的数目是相对稳定的。同时, 从进化树的结构看, 在每一类HDACs中, 每个分支内部的结构并不严格与物种系统进化树完全相同。尤其是拟南芥中的HDAC基因常因序列差异较大而分布在外围, 这可能与拟南芥中近期发生了两次WGD事件相关。WGD发生后, 基因拷贝数目增多, 选择压力下降, 序列多样性被保留的可能性也越大。因此, 拟南芥与草莓直系同源基因的差异就有可能大于中国莲与草莓直系同源基因的差异。由此可见, WGD不但对HDAC基因在双子叶植物中的扩张有贡献, 而且可能对HDACs的序列多样性和功能分化有重要的作用。
对比模式植物拟南芥和森林草莓的花和果实早期发育转录组发现, 无论在序列上还是在功能上森林草莓HDAC基因与拟南芥都具有相似性。譬如, 森林草莓FvHDT1在整个花发育后期和果实发育早期阶段都有较高的表达水平, 而且在花药、心皮和胚中表达量更高, 这与拟南芥中4个HDT基因表达模式相吻合[1], 表明FvHDT1可能参与草莓果实发育过程。同时, 拟南芥和森林草莓中某些HDAC基因又有其表达模式上的差异。譬如, 拟南芥HD2家族有4个基因, 其在拟南芥不同生长发育阶段具有不同的表达水平[1]。而森林草莓中仅对应一个FvHDT1基因, 其在森林草莓整个生长发育中均具有较高表达量。由HD家族在不同物种中的进化树可以推测, 拟南芥中HD家族发生了特异性扩张并可能产生了功能分化, 而草莓中HD家族基因变化相对较小。
另一方面, 拟南芥和草莓对低温和高温胁迫的响应既有相似之处也有不同之处。拟南芥HDAC基因对低温胁迫的响应不大, 而在高温胁迫条件下, AtHDA8、AtHDA14、AtHDA19、AtHDT3、AtHDT4和AtSRT2基因表达水平均发生了变化 (http://jsp.weigelworld.org/expviz/exp-viz.jsp)。而在草莓中, 和拟南芥相似, 低温胁迫下HDAC基因变化幅度比较小。而在高温胁迫下, FvHDA1、FvHDA3、FvHDA4、FvHDA5、FvHDA6、FvHDA8、FvSRT1和FvSRT2基因表达发生了显著变化。拟南芥和草莓各个HDAC直系同源基因在各发育阶段以及低温和高温胁迫响应中有所异同, 表明在物种分化过程中HDAC基因发生了功能分化以适应各个物种的发育及环境的需要。
我们从物种进化、染色体定位、基因进化、保守序列与保守结构域以及基因表达方面, 对森林草莓HDAC基因进行了较为详细的分析, 为HDAC基因在森林草莓中的深入研究奠定了基础。尤其是HDAC基因在森林草莓花发育和果实发育早期以及逆境胁迫中的转录水平分析, 为组蛋白去乙酰化在森林草莓果实发育和逆境胁迫响应中的研究提供了可靠的数据支持。
| [1] | Hollender C, Liu Z. Histone deacetylase genes in Arabidopsis development[J]. J Integr Plant Biol, 2008, 50(7): 875–885. DOI: 10.1111/jipb.2008.50.issue-7 |
| [2] | Liu X, Yang S, Zhao M, et al. Transcriptional repression by histone deacetylases in plants[J]. Mol Plant, 2014, 7(5): 764–772. DOI: 10.1093/mp/ssu033 |
| [3] | Yang X J, Seto E. HATs and HDACs:from structure, function and regulation to novel strategies for therapy and prevention[J]. Oncogene, 2007, 26(37): 5310–5318. DOI: 10.1038/sj.onc.1210599 |
| [4] | Pandey R, Muller A, Napoli C A, et al. Analysis of histone acetyltransferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes[J]. Nucleic Acids Res, 2002, 30(23): 5036–5055. DOI: 10.1093/nar/gkf660 |
| [5] | Luo M, Tai R, Yu C, et al. Regulation of flowering time by the histone deacetylase HDA5 in Arabidopsis[J]. The Plant Journal, 2015, 82(6): 925–936. DOI: 10.1111/tpj.12868 |
| [6] | Aufsatz W, Mette M F, van der Winden J, et al. HDA6, a putative histone deacetylase needed to enhance DNA methylation induced by double-stranded RNA[J]. EMBO J, 2002, 21(24): 6832–6841. DOI: 10.1093/emboj/cdf663 |
| [7] | Probst A V, Fagard M, Proux F, et al. Arabidopsis histone deacetylase HDA6 is required for maintenance of transcriptional gene silencing and determines nuclear organization of rDNA repeats[J]. Plant Cell, 2004, 16(4): 1021–1034. DOI: 10.1105/tpc.018754 |
| [8] | Tian L, Chen Z J. Blocking histone deacetylation in Arabidopsis induces pleiotropic effects on plant gene regulation and development[J]. Proc Natl Acad Sci USA, 2001, 98(1): 200–205. DOI: 10.1073/pnas.98.1.200 |
| [9] | Long J A, Ohno C, Smith Z R, et al. TOPLESS regulates apical embryonic fate in Arabidopsis[J]. Science, 2006, 312(5779): 1520–1523. DOI: 10.1126/science.1123841 |
| [10] | Zhou Y, Tan B, Luo M, et al. HISTONE DEACETYLASE19 interacts with HSL1 and participates in the repression of seed maturation genes in Arabidopsis seedlings[J]. Plant Cell, 2013, 25(1): 134–148. DOI: 10.1105/tpc.112.096313 |
| [11] | Xu C R, Liu C, Wang Y L, et al. Histone acetylation affects expression of cellular patterning genes in the Arabidopsis root epidermis[J]. Proc Natl Acad Sci USA, 2005, 102(40): 14469–14474. DOI: 10.1073/pnas.0503143102 |
| [12] | Liu C, Li L C, Chen W Q, et al. HDA18 affects cell fate in Arabidopsis root epidermis via histone acetylation at four kinase genes[J]. Plant Cell, 2013, 25(1): 257–269. DOI: 10.1105/tpc.112.107045 |
| [13] | Konig A C, Hartl M, Pham P A, et al. The Arabidopsis class Ⅱ sirtuin is a lysine deacetylase and interacts with mitochondrial energy metabolism[J]. Plant Physiol, 2014, 164(3): 1401–1414. DOI: 10.1104/pp.113.232496 |
| [14] | Ma X, Lü S, Zhang C, et al. Histone deacetylases and their functions in plants[J]. Plant Cell Rep, 2013, 32(4): 465–478. DOI: 10.1007/s00299-013-1393-6 |
| [15] | Rossi V, Locatelli S, Varotto S, et al. Maize histone deacetylase hda101 is involved in plant development, gene transcription, and sequence-specific modulation of histone modification of genes and repeats[J]. Plant Cell, 2007, 19(4): 1145–1162. DOI: 10.1105/tpc.106.042549 |
| [16] | Ding B, Bellizzi M R, Ning Y, et al. HDT701, a histone H4 deacetylase, negatively regulates plant innate immunity by modulating histone H4 acetylation of defense-related genes in rice[J]. Plant Cell, 2012, 24(9): 3783–3794. DOI: 10.1105/tpc.112.101972 |
| [17] | Gu T, Han Y, Huang R, et al. Identification and characterization of histone lysine methylation modifiers in Fragaria vesca[J]. Scientific Reports, 2016, 6: 23581. DOI: 10.1038/srep23581 |
| [18] | Gu T, Ren S, Wang Y, et al. Characterization of DNA methyltransferase and demethylase genes in Fragaria vesca[J]. Molecular Genetics and Genomics, 2016, 291(3): 1333–1345. DOI: 10.1007/s00438-016-1187-y |
| [19] | Shulaev V, Sargent D J, Crowhurst R N, et al. The genome of woodland strawberry (Fragaria vesca)[J]. Nat Genet, 2011, 43(2): 109–116. DOI: 10.1038/ng.740 |
| [20] | Finn R D, Mistry J, Tate J, et al. The Pfam protein families database[J]. Nucleic Acids Research, 2010, 38: D211–D222. DOI: 10.1093/nar/gkp985 |
| [21] | Marchler-Bauer A, Derbyshire M K, Gonzales N R, et al. CDD:NCBI's conserved domain database[J]. Nucleic Acids Research, 2015, 43: D222–D226. DOI: 10.1093/nar/gku1221 |
| [22] | Wang Y, Tang H, Debarry J D, et al. MCScanX:a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Res, 2012, 40(7): e49. DOI: 10.1093/nar/gkr1293 |
| [23] | Amil-Ruiz F, Garrido-Gala J, Blanco-Portales R, et al. Identification and validation of reference genes for transcript normalization in strawberry (Fragaria×ananassa) defense responses[J]. PLoS ONE, 2013, 8(8): e70603. DOI: 10.1371/journal.pone.0070603 |
| [24] | Heid C A, Stevens J, Livak K J, et al. Real time quantitative PCR[J]. Genome Res, 1996, 6(10): 986–994. DOI: 10.1101/gr.6.10.986 |
| [25] | Shulaev V, Sargent D J, Crowhurst R N, et al. The genome of woodland strawberry (Fragaria vesca)[J]. Nat Genet, 2011, 43(2): 109–116. DOI: 10.1038/ng.740 |
| [26] | Kang C, Darwish O, Geretz A, et al. Genome-scale transcriptomic insights into early-stage fruit development in woodland strawberry Fragaria vesca[J]. The Plant Cell, 2013, 25(6): 1960–1978. DOI: 10.1105/tpc.113.111732 |
| [27] | Darwish O, Slovin J P, Kang C, et al. SGR:an online genomic resource for the woodland strawberry[J]. BMC Plant Biol, 2013, 13: 223. DOI: 10.1186/1471-2229-13-223 |
| [28] | Hollender C A, Geretz A C, Slovin J P, et al. Flower and early fruit development in a diploid strawberry, Fragaria vesca[J]. Planta, 2012, 235(6): 1123–1139. DOI: 10.1007/s00425-011-1562-1 |




