文章信息
- 於丙军, 刘振, 卫培培
- YU Bingjun, LIU Zhen, WEI Peipei
- 基于盐逆境下植物CLC同源基因的发掘和功能解析的研究进展
- Research progresses on whole genome discovery and function analysis of CLC homologous genes in plants based on salt stress
- 南京农业大学学报, 2017, 40(2): 187-194
- Journal of Nanjing Agricultural University, 2017, 40(2): 187-194.
- http://dx.doi.org/10.7685/jnau.201701020
-
文章历史
- 收稿日期: 2017-01-11
迄今对植物耐盐生理及分子机制的研究大都集中在Na+毒害和适应方面, 但植物盐害的阴离子毒害主要由Cl-造成, 但有关Cl-毒害问题受到研究者的关注较少。就高等植物而言, 位于植物各种细胞器膜 (如质膜、液泡膜、内质网膜、线粒体膜、叶绿体膜等) 上, 以介导Cl-、NO3-为主的阴离子通道 (anion channels) 或转运蛋白 (transporters), 在参与植株营养运输, 细胞动作电位和膨压调节, 胞内pH调节, 气孔运动, 信号识别与转导, 生物或非生物 (盐分、重金属、低温等) 逆境下阴离子 (包括Cl-、NO3-) 吸收、转运、稳态重建和抗性表现等方面发挥着非常重要的功能[1-3]。其中Cl-通道蛋白 (chloride channels, CLCs) 同源基因家族受到人们的关注程度最高[4]。研究表明, 盐逆境下通过主动向细胞外排Cl-, 进行细胞 (如液泡) 或不同器官 (如根和地上部) 内Cl-区域化分配, 实现细胞或植株内Cl-稳态是植物抵抗盐分胁迫和提高耐氯 (盐) 性的重要生理策略之一[5-9]。本文从盐逆境下植物Cl-毒害效应的角度, 对目前有关植物CLC同源基因的发掘及其耐氯 (盐) 功能的解析方面的研究进展进行归纳、综述。
1 盐逆境下Cl-吸收、转运和耐氯性是植物耐盐性研究不可忽视的重要方面离子毒害、渗透胁迫、营养失衡和活性氧伤害是造成植物或作物盐害的主要原因, 其中离子毒害是造成盐害的原初和根本因素。NaCl是构成植物或作物盐害的最主要盐类, Na+和Cl-也就构成了盐分逆境下离子毒害的最主要盐分离子。通过排出胞外和液泡内区域化, 以达到降低地上部Na+和Cl-浓度是农作物盐适应的主要有效策略[7, 10]。一般植物对Na+较Cl-胁迫更敏感, 如棉花、水稻、小麦、大麦等作物的盐胁迫伤害主要是由Na+引起的[11-12]。迄今人们对植物耐盐生理及其分子生物学机制的研究大都集中在阳离子——Na+毒害和适应方面, 如控制Na+进入细胞内的非选择性阳离子通道蛋白 (nonselective cation channels, NSCCs) 和高亲和K+转运蛋白 (high-affinity K+ transporters, HKTs), 质膜和液泡膜上分别控制Na+外排转运和液泡中区域化累积的Na+/H+逆向转运蛋白 (Na+/H+antiporter, 分别如AtSOS1和AtNHX1)[13-14]。
氯 (Cl) 是高等植物生长和发育所必需的重要微量元素之一, 在植物体内主要以离子 (Cl-) 形式存在。作为微量营养元素, Cl-具有参与光合作用、调节气孔运动、维持细胞渗透压和电荷平衡以及抑制植物病害等作用。然而, Cl-是植物组织和细胞中除NO3-外的主要矿质阴离子之一, 在盐逆境下植物盐害的阴离子毒害主要由Cl-造成。有关Cl-毒害的问题在过去很少受到关注和研究, 过量的Cl-对N、P、K等重要营养元素吸收、叶片细胞水势、光合放氧等诸多生理过程产生不良效应, 导致气孔关闭和叶绿体活性氧积累, 进而影响植物生长、产量和品质[3, 15]。实际上, 1980年Greenway等[16]在划分植物盐害或适应类型时, 把植物分成四类:极端盐生植物 (IA), 其生长受过量Cl-(一般小于400 mmol·L-1 NaCl) 促进, 如碱蓬, 滨藜 (高达700 mmol·L-1 NaCl), 盐角草 (高达1 020 mmol·L-1 NaCl) 等; 一般盐生植物 (IB), 其生长仅在外界介质Cl-浓度达200 mmol·L-1时略受影响, 如戟叶滨藜, 大米草等; 一般耐盐植物 (Ⅱ), 在外界介质Cl-浓度达100 mmol·L-1时生长才受到影响的植物, 包括部分盐生植物 (如紫羊茅、碱茅等) 和淡土植物 (如棉花、大麦、大豆等); 敏感淡土植物 (Ⅲ), 如柑桔等木本植物、禾谷类和果蔬类作物, 对Cl-敏感植物和耐Cl-植物组织中临界Cl-浓度分别为4~7 mg·g-1和15~50 mg·g-1, 可以看出他们划分的主要依据就是不同植物对Cl-过量时的忍受能力。Teakle等[6]指出Cl-毒害是植物盐害研究领域“被忽视的敌人 (the forgotten enemy)”, 生长介质中增加NO3-水平可降低盐胁迫下植株叶片内Cl-含量, 减轻植物盐害程度, NO3-/Cl-关系甚至可与制约植物耐钠 (盐) 性的K+/Na+选择性相提并论。
研究表明, 烟草、葡萄、马铃薯、柑橘、栽培大豆等的胁迫作用主要是由Cl-引起, 被称为“忌氯植物”[6-8, 17]。以葡萄、柑橘为代表的这类“忌氯类”果树在采用砧木-接穗育种时, 首先是所选砧木 (rootstock) 的类型对苗木的耐氯 (盐) 能力起关键性作用[18-20], 其次是由根木质部薄壁细胞调控Cl-向导管的转运, 进而限制盐胁迫下植株根部吸收的Cl-向地上部 (主要是叶片) 的运输, 并通过根部细胞Cl-的主动外排及液泡内的Cl-区域化, 使根中Cl-积累量尽量降低[21]。就大豆而言, 它起源于中国, 是我国主要农作物之一, 也是世界上种植最广泛的谷类豆科植物, 是人类生产和生活中植物源蛋白和油脂的主要来源[22]。栽培大豆 (Glycine max) 属中度盐敏感植物[23-24], 由于其遗传基础已越来越窄, 局限在栽培大豆品种范围内的常规耐盐育种工作难以取得较大的突破。野生大豆 (Glycine soja) 是栽培大豆的近缘种, 它具有高蛋白、高油分、耐盐、耐旱、耐阴、抗蚜虫、抗大豆孢囊线虫、抗灰斑病等多种优良性状。目前, 我国收集到的野生大豆种群达6 000余份, 占世界总数的90%以上, 被认为是大豆优质、高产和多抗育种的重要遗传种质资源[25-26]。我们前期多年的研究工作率先明确:在NaCl胁迫下, Na+和Cl-都是构成大豆盐害的毒性离子, 栽培大豆和野生大豆对Na+和Cl-的敏感性存在差异; 就栽培大豆而言, 由于其植株根部对盐胁迫下Na+吸收和转运具有较强的控制作用, 所以往往是Cl-更容易积累到对植株具有毒害作用的程度, 因此栽培大豆的Cl-胁迫作用大于Na+, 其受害程度与其叶片和茎中的Cl-含量呈显著正相关, 而作为其近缘种的盐生野大豆及其与栽培大豆的杂交后代对Cl-表现颇强的抗性[7, 23, 27-29]。因此, 研究盐分逆境下不同种类大豆或其他种类植物体内Cl-的吸收、运输及其调控机制, 也就成为植物耐盐性研究不可忽视的重要方面。
2 从CLC同源基因角度研究植物耐氯 (盐) 性的必要性和科学意义CLCs是一类广泛分布于原核和真核生物细胞膜 (主要是内膜系统) 上介导以Cl-为代表的阴离子运输的重要通道 (channels) 或转运蛋白 (transporters) 家族[30]。第1个CLC家族蛋白 (CLC-0) 是利用爪蟾 (Xenopus) 卵母细胞基因表达技术在石纹电鳐 (Torpedo marmorata) 中分离得到的, 之后在大肠杆菌 (Escherichia coli)、酿酒酵母 (Saccharomyces cerevisiae)、哺乳动物和植物中相继发现其新成员。细菌CLC蛋白 (CLC-ec1) 主要起致电性Cl-/H+ antiporter作用, 而对于动物而言, 位于细胞质膜上的CLCs主要起Cl-通道作用, 位于细胞内膜上的CLCs主要发挥2Cl-/1H+ antiporter功能[31]。酿酒酵母GEF1基因编码1个CLC蛋白 (即ScCLC或GEF1), 驱动Cl-和H+的跨膜交换, 该基因的突变体 (Δgef1) 因向其细胞内囊泡 (液泡或高尔基体) 中运输Cl-受阻, 从而使其对许多胞外阳离子 (如Fe3+和Mn2+)、潮霉素B、氯化四甲铵等表现超敏性, 呼吸和生长受阻[32]。
高等植物中现在研究最多也最清楚的拟南芥7种AtCLCs中, AtCLCa-d和AtCLCg的阴离子选择属性与高度保守的氨基酸序列基元GxGIPE中x有关, x若为P (Pro), 则首选阴离子为NO3-, x若为S (Ser) 则首选阴离子为Cl- [4, 33-34]。定位于液泡膜上的AtCLCa, 主要作为2NO3-/1H+ antiporter驱动NO3-(超过50%) 在液泡中的积累, 其NO3-/H+逆向转运活性与该蛋白保守氨基酸基元GP160GIPE中第160位的P (Pro) 密切有关, 当P160突变为S时, AtCLCa丧失对NO3-的优先权, 即优先转运Cl-或以同样的速率转运NO3-和Cl- [30, 35]。与AtCLCa同源性 (约80%) 最接近的AtCLCb也是如此, 此氨基酸位点 (P159) 略有不同, 两者在功能上可能有重复, 因为AtCLCb基因的突变不会影响拟南芥植株体内NO3-和Cl-含量水平[36]。AtCLCc (定位于液泡膜)、AtCLCd (定位于高尔基体膜)、AtCLCg (定位于液泡膜) 的相应位置氨基酸则为S (丝氨酸) 取代, 主要表现Cl-转运活性, 其中AtCLCc可在叶片气孔保卫细胞中高效表达, 参与气孔运动调节, 也有利于植株耐盐性的增强[1]。Chi等[3]研究表明, 定位于液泡膜上的AtCLCg蛋白, 主要分布于叶片叶肉、排水器和韧皮部细胞, 该基因突变体atclcg在NaCl胁迫下盐敏感性增强, 地上部积累Cl-显著多于野生型, 但在渗透胁迫 (甘露醇) 下, atclcg突变体植株生长与野生型没有差异。当AtCLCg与蛋白一致性很高 (62%)、与NaCl胁迫下液泡内Cl-稳态调节和耐盐性也密切相关的AtCLCc双突变时, 拟南芥植株 (atclcc-1atclcg-2) 的盐敏感性和地上部Cl-含量未表现叠加效应, 表明两者在调控植株耐Cl-(盐) 性方面同等重要, 且没有表现基因功能冗余现象。AtCLCe定位于叶绿体类囊体膜上, 主要负责叶肉细胞叶绿体基质中NO2-(由NO3-还原而来) 或Cl-向叶绿体类囊体腔中运输, 与胞质中NO3-同化有关, 和定位于液泡膜上的AtCLCa存在功能 (NO3-吸收、转运和同化) 协同效应[37]。AtCLCf定位于高尔基体上, 主要负责Cl-转运, AtCLCe和AtCLCf不具有拟南芥其他5种CLC蛋白中的保守氨基酸序列基元GxGIPE, 分别代之以ESAGK和EILDQ[4]。已发现的拟南芥7种CLC蛋白 (AtCLCa-g) 中, AtCLCa、AtCLCd和AtCLCf等转入酵母Δgef1突变体中表达, 可实现酵母CLC蛋白的功能或表型互补[30, 32]。AtCLCd还被证明参与了拟南芥先天病菌免疫的负调控[38]。
科学家们于2000年、2002年和2010年相继完成拟南芥、水稻和大豆基因组测序工作之后, 除NCBI (http://www.ncbi.nlm.nih.gov/) 外, 相关的水稻或大豆基因组数据库或网站 (如http://www.ricedata.cn/gene/, http://www.phytozome.net/soybean, http://soybase.org/, http://soykb.org/) 相继出现或完善。因此, 从盐胁迫下Cl-毒害效应和CLC同源基因的角度, 比较系统地发掘和筛选决定大豆、水稻等作物耐氯 (盐) 性差异的关键CLC同源基因, 解析和验证它们参与盐逆境下氯 (盐) 胁迫适应过程的生理和分子机制, 是对植物耐盐机制研究领域的创新拓展和重要补充。)
3 植物CLC同源基因和编码蛋白的生物信息学分析目前已在拟南芥、烟草、水稻、马铃薯、玉米、大豆中陆续发现CLC蛋白[2, 33-34]。关于各种植物CLC基因或蛋白家族的命名或编号, 除在基因或蛋白名称前冠以该种植物拉丁学名的属和种的首字母组合外, 在各类网站数据库中习惯上多以英文字母CLCa-g排列, 但在许多的研究文献中, 学者们好象又偏好以数字, 如CLC1-n表示。以研究最多也最清楚的拟南芥为例, 其7种CLCs在各类数据库和研究文献中都是以英文字母AtCLCa-g排列的。当然, 由于不同研究者及年代更新, 各类数据库和研究文献中某种植物某个CLC基因或蛋白的命名或编号也会存在一定的差异, 甚至显得有些凌乱更缺乏统一 (如水稻OsCLCs编号), 这就要求我们在关注和研究该种植物的某个CLC基因或蛋白家族成员时, 需要全面收集并注意辨别以尽可能获得最权威、最新的数据资料。我们以GmCLC-1或GmCLC1基因编码蛋白的氨基酸序列为基准, 从Pfam数据库 (http://pfam.xfam.org/) 中检索出多种CLC蛋白家族的保守结构域序列, 并结合Uniprot数据库 (http://pfam.xfam.org/) 中检索出多种CLC蛋白家族的保守结构域序列, 并结合Uniprot数据库 (http://www.uniprot.org/) 从NCBI数据库 (https://www.ncbi.nlm.nih.gov) 中检索出CLC同源蛋白氨基酸序列, 进行整理和分析后共检索出34种植物的217个蛋白或基因序列 (主要采用英文字母*CLCa-g编号, 同一英文字母下出现2种以上类似序列时, 再附加数字, 以示区别, 如*CLCb-1和CLCb-2, 其中木本植物10种, 草本植物23种, 以及藤本植物1种 (表 1)。
| 植物类型 Plant species |
植物名称 Plant names |
| 木本植物 Woody plant |
毛果杨 (Populus trichocarpa, 以Pt表示, 下同)、橙 (Citrus sinensis, Cs)、核桃 (Juglans regia, Jr)、胡杨 (Populus euphratica, Pe)、巨桉 (Eucalyptus grandis, Eg)、可可 (Theobroma cacao, Tc)、麻疯树 (Jatropha curcas, Jc)、梅 (Prunus mume, Pe)、树棉 (Gossypium arboreum, Ga)、棕榈 (Elaeis guineensis, Eg) |
| 草本植物 Herbaceous plant |
芭蕉 (Musa acuminata subsp, Ma)、白菜 (Brassica rapa, Br)、蓖麻 (Ricinus communis, Rc)、大豆 (Glycine max, Gm)、短柄草 (Brachypodium distachyon, Bd)、番茄 (Solanum lycopersicum, Sl)、甘蓝 (Brassica oleracea, Bo)、黄瓜 (Cucumis sativus, Cus)、辣椒 (Capsicum annuum, Ca)、莲 (Nelumbo nucifera, Nn)、萝卜 (Raphanus sativus, Rs)、马铃薯 (Solanum tuberosum, St)、蒺藜苜蓿 (Medicago truncatula, Mt)、拟南芥 (Arabidopsis thaliana, At)、日本牵牛 (Ipomoea nil, In)、甜菜 (Beta vulgaris, Bv)、亚麻荠 (Camelina sativa, Cas)、烟草 (Nicotiana tabacum, Nt)、油菜 (Brassica napus, Bn)、醉蝶花 (Tarenaya hassleriana, Th)、谷子 (Setaria italica, Si)、水稻 (Oryza sativa L.spp.japonica, Os)、玉米 (Zea mays, Zm) |
| 藤本植物Liana plant | 葡萄 (Vitis vinifera, Vv) |
将这217个基因或蛋白序列进行生物信息学分析, 其中包括使用Bioinformatics数据库 (http://www.bioinformatics.nl/) 中的GEECE工具分析各基因的GC含量, 使用PEPSTATS工具统计各蛋白质的相对分子质量、理论等电点等理化特性, 使用Npsa-prabi数据库中的SOPMA工具 (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_server.html) 进行各蛋白质的二级结构预测等。检索结果表明:上述34种植物中, 毛果杨 (Populus trichocarpa) 可检索到的PtCLC同源基因成员的数量最少, 仅为3种, 蒺藜苜蓿 (Medicago truncatula) 中为5种, 谷子 (Setaria italica) 和玉米 (Zea mays) 及大豆 (Glycine max) 中为8种, 其余多数种类植物中为6或7种, CDS序列碱基长度为1 758~2 853 bp, 平均为2 322 bp; GC含量除少数大于60%, 多数在45%左右。各CLC蛋白氨基酸残基数和相对分子质量分别为773.1和84.18×103, 理论等电点平均值为7.5, 可见它们多数为碱性蛋白质; 二级结构预测结果显示, 均包含α-螺旋、β-折叠片、β-转角、无规则卷曲4种结构元件, 所占比例均值分别为40.45%、22.01%、9.10%和29.39%(资料未列出)。将这些蛋白氨基酸序列采用Mega 6.0软件进行比对, 由构建的N-J进化树 (图 1) 可知:整个进化树呈现2个大的分支, 即分支1和分支2, 其中CLC-e和CLC-f主要分布在分支1中, 其他均分布在分支2中; 分支2又衍生出2个亚分支, 即分支3和分支4, 分支3中主要分布着CLC-a、CLC-b、CLC-c和CLC-g, 而分支4主要分布CLC-d。
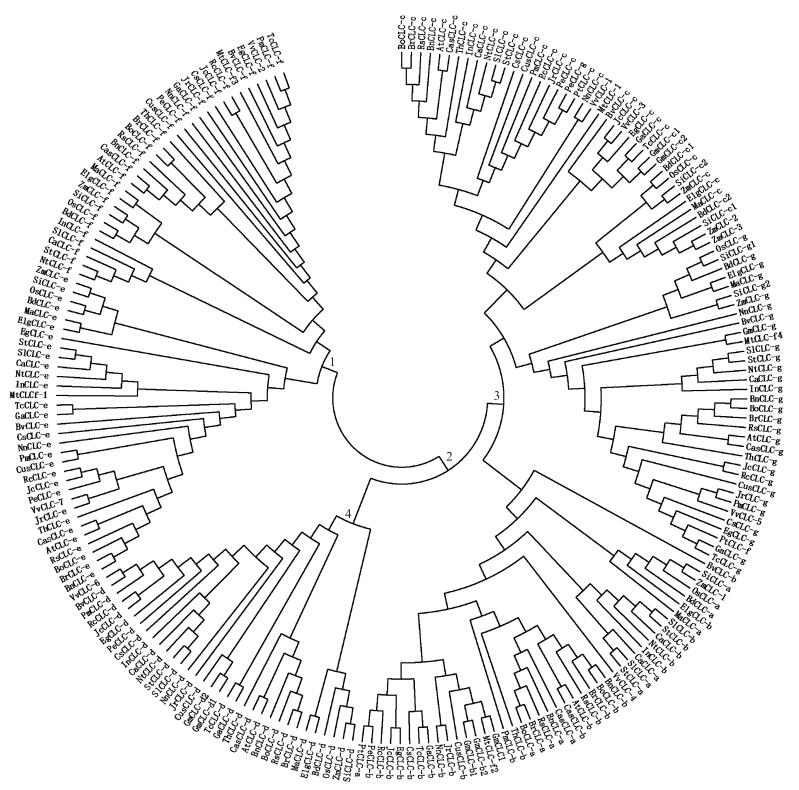
|
图 1 已报道的植物CLC蛋白的进化树 Figure 1 Phylogenetic tree of the reported plant CLC proteins |
在大豆中, 除GmCLC1基因 (GenBank登录号为AY972079, Phytozome数据库中对应序列号为Glyma05g14760) 外, 我们在大豆基因组数据库 (www.phytozome.com) 中又检索到其他7个CLC同源基因序列, 序列号分别为Glyma16g06190、Glyma19g25680、Glyma09g28620、Glyma16g33351、Glyma01g44950、Glyma11g00690、Glyma13g23080, 被依次命名为GmCLC-b1、GmCLC-b2、GmCLC-c1、GmCLC-c2、GmCLC-d1、GmCLC-d2和GmCLC-g, 并分别以栽培大豆代表品种N23674和野生大豆品系BB52为供试材料, 已全部完成基因克隆、测序验证和比较, 部分成员在上述2个材料间存在少数核苷酸和氨基酸序列差异位点 (资料未列出)。李伟等[34]利用有关生物信息学软件, 对大豆CLC同源基因及其编码蛋白家族进行了系统的预测分析和比较, 其中大豆CLC同源基因CDS序列的聚类分析显示, Glyma01g44950(GmCLC-d1) 和Glyma11g00690(GmCLC-d2) 二者一致性高达98.05%, 列为第一类; Glyma05g14760(GmCLC1)、Glyma09g28620(GmCLC-c1)、Glyma13g23080(GmCLC-g)、Glyma16g33351(GmCLC-c2)、Glyma16g06190(GmCLC-b1)、Glyma19g25680(GmCLC-b2) 则为第二类, CDS序列整体一致性超过50%, 其中GmCLC-b1与GmCLC-b2, GmCLC-c1与GmCLC-c2分别显示97.47%、96.24%的一致性。所编码大豆CLC蛋白均为跨膜蛋白 (含9~12个跨膜区), 都与拟南芥CLC蛋白亚家族Ⅰ关系较近, 都具有 (Ⅰ) GS/PGIPE、(Ⅱ) GKE/AGPLVH和 (Ⅲ) PVGGVLFALEE/G 3个氨基酸残基保守区, 除GmCLC-g可能是一种阴离子通道外, 其他均可能发挥H+/阴离子共转运体功能。
4 植物CLC同源基因参与了盐逆境下耐盐功能的发挥在盐胁迫下, 植物为了应对Cl-毒害, 会采用诸如CLC蛋白家族之类的转运蛋白来减轻细胞质中的Cl-积累。为此我们需要借助已完成的拟南芥、水稻和大豆等植物的基因组数据库信息资料, 去关注和研究植物CLC同源基因参与植株体内Cl-或NO3-吸收、转运及耐氯 (盐) 性的生理和分子调控功能。基因功能的研究方法一般包括:通过表型寻找功能异常突变的传统遗传学分析, 用物理或化学诱变产生突变表型来克隆控制该性状基因的正向遗传学 (forward genetics) 方法, 以及研究某个基因敲除 (knockout或knockdown)、反义RNA (anti-sense)、RNA干扰 (RNA interference, RNAi)、基因沉默 (gene silencing) 或过表达 (overexpression) 后 (可统称为转基因技术, transgenic technology) 的生物性状改变从而反推基因功能的反向遗传学 (reverse genetics) 手段。其中病毒诱导的基因沉默 (virus-induced gene silencing, VIGS) 是指利用携带目标基因片段的病毒侵染植物后, 通过特异性降解寄主内源mRNA序列, 诱导植物内源基因沉默的现象[39], 近年来已被用作研究植物基因功能的反向遗传学新技术, 与转基因、基因敲除、反义抑制等基因功能研究方法相比, VIGS技术具有周期短、低成本、高通量和不需要遗传转化等优点。我们可以利用该技术沉默与目标基因同源性较高的非靶基因, 解析大豆、水稻等作物CLC同源基因成员是否存在功能冗余现象。
在水稻 (Oryza sativa L.ssp.japonica, ‘Nipponbare’) 中也鉴定出2个CLC基因成员 (OsCLC-1和OsCLC-2), 它们均为单拷贝, 分别定位于4号和2号染色体上。OsCLC-1在植株多数组织中都有表达, 且受盐处理促进, 而OsCLC-2只在根、节及节间、叶鞘等组织中表达, 两者都可恢复酵母CLC基因突变体 (Δgef1) 在各种离子胁迫下的生长表型, 所编码蛋白都定位在液泡膜上, 插入逆转录转座子Tos17后的水稻突变体的生长发育明显受阻[40]。盐胁迫下敏感水稻品种IR29植株体内, 尤其是叶片中Cl-积累明显高于耐盐品种Pokkali, 这可能与OsCLC1基因在2个水稻植株根中的转录分别被抑制或诱导增强的差异有关[41]。大豆GmCLC1基因编码蛋白定位于细胞的液泡膜上, 可把细胞质中的Cl-向液泡内运输和累积, 在盐胁迫条件下植株体内Cl-稳态保持或重建方面发挥重要作用, 从而有利于减轻大豆盐害[42]。进一步采用GmCLC1基因转烟草BY-2细胞 (台盼蓝染色法) 和爪蟾卵母细胞 (膜片钳仪电生理法)、大豆根 (mRNA转录水平差异) 等技术方法, 研究发现GmCLC1基因的表达及其编码蛋白的Cl-转运活性表现出对胞质环境中pH值的依赖性, 表明该蛋白很可能是一种Cl-/H+逆向转运体[43]。本课题组采用转基因拟南芥、发根农杆菌转化大豆和突变体酵母互补等技术手段, 探究了GmCLC1基因在盐胁迫响应过程中的功能[9], 结果表明:转GmCLC1拟南芥幼苗在盐胁迫下比其野生型表现出明显的发芽和生长优势, 转基因拟南芥植株地上部Cl-的动态积累量一直明显低于野生型, 而两者根部Cl-积累量无明显差异; 通过发根农杆菌介导将GmCLC1基因在大豆子叶发根或组合植株中过表达后盐害明显减轻, 其中组合植株地上部 (含茎、叶) 中Cl-积累也显著低于空载对照; GmCLC1基因还可以使酵母CLC基因突变体 (Δgef1) 在NaCl或MnCl2胁迫下的生长抑制得到不同程度的恢复, 表现出良好的互补效应。过表达橙CsCLCc基因或玉米ZmCLC-d基因可以分别明显增强转基因拟南芥突变体atclcc或atclcd的耐盐性[2, 44]。还有研究表明, 在烟草花粉萌发和花粉管伸长过程中, 通过其Cl-通道而进行的跨细胞膜Cl-转运, 对花粉的极性生长和渗透调节发挥了关键性作用[37]。目前可查到的关于植物CLC同源基因家族成员及其编码蛋白参与耐氯 (盐) 功能解析方面的文献还为数不多。不过, 上述的研究结果也说明, CLC蛋白作为一类重要的阴离子通道或转运蛋白, 在参与盐逆境下植株体内Cl-稳态和耐氯 (盐) 性调控方面可以发挥重要作用。
5 结语与展望随着研究的不断深入, 人们发现植物CLC蛋白家族的许多成员实际上也是NO3-通道或运输蛋白, 这与其早前Cl-通道 (chloride channels) 的本义已不太符合。也有些离子转运蛋白, 虽然名称上与Cl-没有关系, 却具有Cl-运输功能, 比如ALMT (aluminum activated malate transporters) 蛋白家族, 本义是指受Al3+激活、转运苹果酸或柠檬酸的一类蛋白质, 与提高植物的耐铝性有关, 如定位于小麦(Triticum aestivum)根尖细胞质膜上的ALMT1[31]。但ALMT这一缩略语是早期的用法, 现在看来这类蛋白并不是都受Al3+激活, 也不仅仅局限于转运苹果酸或柠檬酸[37], 如AtALMT9就是1个定位于液泡膜上, 受胞质苹果酸激活的转运Cl-的通道蛋白, 参与植物气孔开关的调控过程[45]。Li等[46]在盐敏感植物拟南芥中发现的NPF2.4(nitrate transporter 1/peptide transporter family) 基因, 编码1个定位于根中柱细胞质膜上介导Cl-向木质部装载的蛋白, 其表达受盐、ABA处理所抑制, 当被敲除 (knockdown) 或过表达 (overexpression) 时, 盐胁迫下拟南芥植株地上部Cl-积累量分别明显下降或上升, 表明该基因编码蛋白介导了盐胁迫下Cl-由拟南芥植株根部向地上部的长距离转运; 把该基因导入爪蟾卵母细胞表达后的电生理试验结果显示, 该基因编码蛋白对Cl-的选择性明显强于NO3-。尽管如此, 这没有削弱人们对以CLC蛋白家族为代表的植物阴离子通道或运输蛋白研究的浓厚兴趣, 相信今后通过功能基因组学、细胞器标记基因编码蛋白亚细胞共定位、表达载体构建、酵母突变体互补、植物转基因 (根癌农杆菌诱导、发根农杆菌侵染)、VIGS和膜片钳电生理技术等多种分子生物学和生理生化分析技术与方法, 一定会为植物CLC同源基因的发掘, 尤其是耐氯基因及其功能的解析, 进一步丰富并深入揭示植物耐盐性的生理和分子机制, 寻求提高大豆、水稻等作物耐氯 (盐) 性可行的分子育种手段, 提供重要的科学理论依据和实践指导途径。
| [1] | Jossier M, Kroniewicz L, Dalmas F, et al. The Arabidopsis vacuolar anion transporter, AtCLCc, is involved in the regulation of stomatal movements and contributes to salt tolerance[J]. The Plant Journal, 2010, 64: 563–576. DOI: 10.1111/tpj.2010.64.issue-4 |
| [2] | Wei Q, Liu Y, Zhou G, et al. Overexpression of CsCLCc, a chloride channel gene from Poncirus trifoliata, enhances salt tolerance in Arabidopsis[J]. Plant Molecular Biology Reporter, 2013, 31: 1548–1557. DOI: 10.1007/s11105-013-0592-1 |
| [3] | Chi T N, Agorio A, Jossier M, et al. Characterization of the chloride channel-like, AtCLCg, involved in chloride tolerance in Arabidopsis thaliana[J]. Plant Cell Physiol, 2016, 57(4): 764–775. DOI: 10.1093/pcp/pcv169 |
| [4] | Zifarelli G, Pusch M. CLC transport proteins in plants[J]. FEBS Letters, 2010, 584: 2122–2127. DOI: 10.1016/j.febslet.2009.12.042 |
| [5] | Munns R, Tester M. Mechanisms of salinity tolerance[J]. Annual Review of Plant Biology, 2008, 59: 651–681. DOI: 10.1146/annurev.arplant.59.032607.092911 |
| [6] | Teakle N L, Tyerman S D. Mechanisms of Cl- transport contributing to salt tolerance[J]. Plant Cell and Environment, 2010, 33: 566–589. DOI: 10.1111/pce.2010.33.issue-4 |
| [7] | Zhang X K, Zhou Q H, Cao J H, et al. Differential Cl-/salt tolerance and NaCl-induced alternations of tissue and cellular ion fluxes in Glycine max, Glycine soja and their hybrid seedlings[J]. Journal of Agronomy and Crop Science, 2011, 197: 329–339. DOI: 10.1111/jac.2011.197.issue-5 |
| [8] | Henderson S W, Baumann U, Blackmore D H, et al. Shoot chloride exclusion and salt tolerance in grapevine is associated with differential ion transporter expression in roots[J]. BMC Plant Biology, 2014, 14: 273. DOI: 10.1186/s12870-014-0273-8 |
| [9] | Wei P P, Wang L C, Liu A, et al. GmCLC1 confers enhanced salt tolerance through regulating chloride accumulation in soybean[J]. Frontiers in Plant Science, 2016. DOI: 10.3389/fpls.2016.01082 |
| [10] | Hasegawa P M. Sodium (Na+) homeostasis and salt tolerance of plants[J]. Environmental and Experimental Botany, 2013, 92: 19–31. DOI: 10.1016/j.envexpbot.2013.03.001 |
| [11] | Qu Y N, Zhou Q, Yu B J. Effects of Zn2+, niflumic acid on photosynthesis in Glycine soja and Glycine max seedlings under NaCl stress[J]. Environmental and Experimental Botany, 2009, 65: 304–309. DOI: 10.1016/j.envexpbot.2008.11.005 |
| [12] | Henderson S W, Wege S, Qiu J, et al. Grapevine and arabidopsis cation-chloride cotransporters localize to the Golgi and trans-Golgi network and indirectly influence long-distance ion transport and plant salt tolerance[J]. Plant Physiology, 2015, 169: 2215–2229. |
| [13] | Adams E, Shin R. Transport, signaling, and homeostasis of potassium and sodium in plants[J]. Journal of Integrative Plant Biology, 2014, 56: 231–249. DOI: 10.1111/jipb.v56.3 |
| [14] | Deinlein U, Stephan A B, Horie T, et al. Plant salt-tolerance mechanisms[J]. Trends in Plant Science, 2014, 19(6): 371–379. DOI: 10.1016/j.tplants.2014.02.001 |
| [15] |
於丙军, 刘友良. 植物中的氯、氯通道和耐氯性[J].
植物学通报, 2004, 21(4): 402–410.
Yu B J, Liu Y L. Chlorine, chloride channel and chlorine tolerance in plants[J]. Chinese Bulletin of Botany, 2004, 21(4): 402–410. (in Chinese) |
| [16] | Greenway H, Munns R. Mechanisms of salt tolerance in non-halophytes[J]. Annual Review of Plant Physiology, 1980, 31: 149–190. DOI: 10.1146/annurev.pp.31.060180.001053 |
| [17] | Karaivazoglou N A, Papakosta D K, Divanidis S. Effect of chloride in irrigation water and form of nitrogen fertilizer on Virginia (flue-cured) tobacco[J]. Field Crops Research, 2005, 92: 61–74. DOI: 10.1016/j.fcr.2004.09.006 |
| [18] | Brumós J, Colmenero-Flores J M, Conesa A, et al. Membrane transporters and carbon metabolism implicated in chloride homeostasis differentiate salt stress responses in tolerant and sensitive Citrus rootstocks[J]. Functional and Integrative Genomics, 2009, 9: 293–309. DOI: 10.1007/s10142-008-0107-6 |
| [19] | Tregeagle J M, Tisdall J M, Tester M, et al. Cl- uptake, transport and accumulation in grapevine rootstocks of differing capacity for Cl--exclusion[J]. Functional Plant Biology, 2010, 37: 665–673. DOI: 10.1071/FP09300 |
| [20] | Alvarez-Gerding X, Cortés-Bullemore R, Medina C, et al. Improved salinity tolerance in Carrizo citrange rootstock through overexpression of glyoxalase system genes[J]. BioMed Research International, 2015. DOI: 10.1155/2015/827951 |
| [21] | Abbaspour N, Kaiser B, Tyerman S. Chloride transport and compartmentation within main and lateral roots of two grapevine rootstocks differing in salt tolerance[J]. Trees, 2013, 7: 1317–1325. |
| [22] | Lam H M, Xu X, Liu X, et al. Resequencing of 31 wild and cultivated soybean genomes identifies patterns of genetic diversity and selection[J]. Nature Genetics, 2010, 42(12): 1053–1059. DOI: 10.1038/ng.715 |
| [23] | Luo Q Y, Yu B J, Liu Y L. Differential sensitivity to chloride and sodium ions in seedlings of Glycine max and Glycine soja under NaCl stress[J]. Journal of Plant Physiology, 2005, 162: 1003–1012. DOI: 10.1016/j.jplph.2004.11.008 |
| [24] | Guan R, Qu Y, Guo Y, et al. Salinity tolerance in soybean is modulated by natural variation in GmSALT3[J]. The Plant Journal, 2014, 80: 937–950. DOI: 10.1111/tpj.12695 |
| [25] |
伍应保, 於丙军. 栽培和野生大豆亲本及其杂交后代幼苗Cl-通道基因表达及其与耐氯性的关系[J].
南京农业大学学报, 2009, 32(4): 67–71.
Wu Y B, Yu B J. Gene expression of Cl- channel and its relation to chloride tolerance of Glycine max, Glycine soja and their hybrid seedlings[J]. Journal of Nanjing Agricultural University, 2009, 32(4): 67–71. DOI: 10.7685/j.issn.1000-2030.2009.04.013 (in Chinese) |
| [26] | Qi X, Li M W, Xie M, et al. Identification of a novel salt tolerance gene in wild soybean by hole-genome sequencing[J]. Nature Communications, 2014. DOI: 10.1038/ncomms5340 |
| [27] | Luo Q Y, Yu B J, Liu Y L. Stress of Cl- is stronger than that of Na+ on Glycine max seedling under NaCl stress[J]. Agricultural Science in China, 2002, 1(12): 1404–1409. |
| [28] |
陈宣钦, 於丙军, 刘友良. 栽培和野生大豆及其杂交后代幼苗的耐氯性与多胺积累的关系[J].
植物生理与分子生物学学报, 2007, 33(1): 46–52.
Chen X Q, Yu B J, Liu Y L. Relationship between chloride tolerance and polyamine accumulation in Glycine max, Glycine soja, and their hybrid seedlings[J]. Journal of Plant Physiology and Molecular Biology, 2007, 33(1): 46–52. (in Chinese) |
| [29] | Chen X Q, Yu B J. Ionic effects of Na+and Cl- on photosynthesis in Glycine max seedlings under isoosmotic salt stress[J]. Journal of Plant Physiology and Molecular Biology, 2007, 33(4): 294–300. |
| [30] | Barbier-Brygoo H, de Angeli A, Filleur S, et al. Anion channels/transporters in plants:from molecular bases to regulatory networks[J]. Annual Review of Plant Biology, 2011, 62: 25–51. DOI: 10.1146/annurev-arplant-042110-103741 |
| [31] | Martinoia E, Meyer S, de Angeli A, et al. Vacuolar transporters in their physiological context[J]. Annual Review in Plant Biology, 2012, 63: 183–213. DOI: 10.1146/annurev-arplant-042811-105608 |
| [32] | Lü Q, Tang R, Liu H, et al. Cloning and molecular analyses of the Arabidopsis thaliana chloride channel gene family[J]. Plant Science, 2009, 176: 650–651. DOI: 10.1016/j.plantsci.2009.02.006 |
| [33] | Zhou C, Wang H, Zhu J, et al. Molecular cloning, subcellular localization and functional analysis of ThCLC-a from Thellungiella halophila[J]. Plant Molecular Biological Reporter, 2013, 31: 783–790. DOI: 10.1007/s11105-012-0545-0 |
| [34] |
李伟, 王龙超, 曹金花, 等. 大豆基因组CLC同源基因家族的生物信息学分析[J].
南京农业大学学报, 2014, 37(3): 35–43.
Li W, Wang L C, Cao J H, et al. Bioinformatics analysis of CLC homologous genes family in soybean genome[J]. Journal of Nanjing Agricultural University, 2014, 37(3): 35–43. DOI: 10.7685/j.issn.1000-2030.2014.03.005 (in Chinese) |
| [35] | Wege S, Jossier M, Filleur S, et al. The proline 160 in the selectivity filter of the Arabidopsis NO3-/H+ exchanger AtCLCa is essential for nitrate accumulation in planta[J]. The Plant Journal, 2010, 63: 861–869. DOI: 10.1111/tpj.2010.63.issue-5 |
| [36] | von der Fecht-Bartenbach J, Bogner M, Dynowski M, et al. CLC-b-mediated NO3-/H+ exchange across the tonoplast of Arabidopsis vacuoles[J]. Plant Cell Physiology, 2010, 51(6): 960–968. DOI: 10.1093/pcp/pcq062 |
| [37] | Tavares B, Domingos P, Dias P N, et al. The essential role of anionic transport in plant cells:the pollen tube as a case study[J]. Journal of Experimental Botany, 2011, 62(7): 2273–2298. DOI: 10.1093/jxb/err036 |
| [38] | Guo W, Zuo Z, Cheng X, et al. The chloride channel family gene CLCd negatively regulates pathogen-associated molecular pattern (PAMP)-triggered immunity in Arabidopsis[J]. Journal of Experimental Botany, 2014, 65: 1205–1215. DOI: 10.1093/jxb/ert484 |
| [39] | Manmathan H, Shaner D, Snelling J, et al. Virus-induced gene silencing of Arabidopsis thaliana gene homologues in wheat identifies genes conferring improved drought tolerance[J]. Journal of Experimental Botany, 2013, 64: 1381–1392. DOI: 10.1093/jxb/ert003 |
| [40] | Nakamura A, Fukuda A, Sakai S, et al. Molecular cloning, functional expression and subcellular localization of two putative vacuolar voltage-gated chloride channels in rice (Oryza sativa L.)[J]. Plant Cell Physiology, 2006, 47: 32–42. |
| [41] | Diédhiou C J, Golldack D. Salt-dependent regulation of chloride channel transcripts in rice[J]. Plant Science, 2006, 170: 793–800. DOI: 10.1016/j.plantsci.2005.11.014 |
| [42] | Li W Y, Wong F L, Tsai S N, et al. Tonoplast-located GmCLC1 and GmNHX1 from soybean enhance NaCl tolerance in transgenic bright yellow (by)-2 Cells[J]. Plant Cell and Environment, 2006, 29: 1122–1137. DOI: 10.1111/pce.2006.29.issue-6 |
| [43] | Wong T H, Li M W, Yao X Q, et al. The GmCLC1 protein from soybean functions as a chloride ion transporter[J]. Journal of Plant Physiology, 2013, 170: 101–104. DOI: 10.1016/j.jplph.2012.08.003 |
| [44] | Wang S, Su S Z, Wu Y, et al. Overexpression of maize chloride channel gene ZmCLC-d in Arabidopsis thaliana improved its stress resistance[J]. Biologia Plantarum, 2015, 59(1): 55–64. DOI: 10.1007/s10535-014-0468-8 |
| [45] | de Angeli A, Zhang J, Meyer S, et al. AtALMT9 is a malate-activated vacuolar chloride channel required for stomatal opening in Arabidopsis[J]. Nature Communications, 2013, 4: 1804. DOI: 10.1038/ncomms2815 |
| [46] | Li B, Byrt C, Qiu J, et al. Identification of a stelar-localized transport protein that facilitates root-to-shoot transfer of chloride in Arabidopsis[J]. Plant Physiology, 2016, 170: 1014–1029. DOI: 10.1104/pp.15.01163 |




