文章信息
- 王莉, 黄亚楠, 郭满园, 苏彦华
- WANG Li, HUANG Yanan, GUO Manyuan, SU Yanhua
- OsKAT3 RNAi转基因水稻植株的构建
- Construction of RNAi transgenic rice plants of OsKAT3
- 南京农业大学学报, 2016, 39(6): 979-985
- Journal of Nanjing Agricultural University, 2016, 39(6): 979-985.
- http://dx.doi.org/10.7685/jnau.201603027
-
文章历史
- 收稿日期: 2016-03-16
2. 中国科学院南京土壤研究所土壤与农业可持续发展国家重点实验室, 江苏 南京 210008
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
钾是植物生长发育所必需的三大矿质营养元素之一[1]。钾参与了植物生长发育过程中的诸多生理生化反应, 如调节细胞彭压、维持细胞电荷平衡、控制膜电位、调节酶活性及参与蛋白质的合成等[2]。植物对钾离子的吸收转运过程主要是通过钾通道和钾转运体来完成的[3]。
Epstein等[4]指出植物体内可能存在两种不同类型的钾吸收机制:机制Ⅰ主要在低钾浓度下起作用, 对钾具有较高的亲和力, 称为高亲和吸收系统(high-affinity systems); 机制Ⅱ主要在高钾浓度下起作用, 对钾具有较低的亲和力, 称为低亲和吸收系统(low-affinity systems)。现在普遍认为高亲和性钾吸收系统主要是细胞质膜上的钾转运体(transporter), 低亲和性钾吸收系统主要是细胞质膜上的钾通道(channel), 并且二者之间存在一定的功能性重叠。植物正是利用这两种不同的钾吸收系统来适应外界及体内变化的钾浓度环境。在不同类型钾通道中, Shaker类钾通道被认为是介导钾离子吸收、转运和细胞内动态平衡最重要的一类通道[5]。AKT1是Shaker钾通道GroupⅠ的典型成员, 它对K+具有双亲和性, 是拟南芥根中负责钾吸收和转运的主要通道[6-10]。GroupⅢ中的弱整流性通道AKT2/3在拟南芥韧皮部表达时, 其通过介导K+在其中的装载和卸载实现了K+在植株体内的长距离运输[11-12]。GroupⅣ的AtKC1单独存在时不产生电流, 但可以和AKT1形成异源复合K+通道, 并调控AKT1通道的活性[13-14]。GroupⅤ中的外向K+通道SKOR能将根中的K+转运至木质部, 再以质流的方式向地上部运输[15], 而保卫细胞中的外向钾离子通道GORK参与调控气孔的关闭[16]。GroupⅡ的KAT类钾通道是目前研究最为透彻的气孔调控型钾吸收通道。KAT型K+通道是一种内向型钾离子通道, 主要在叶中表达, 特别是在叶中的保卫细胞中表达, 通过介导K+的内流, 引起保卫细胞渗透压增加, 吸水使其保卫细胞体积增大, 气孔开放[17-20], 与外向K+通道GORK共同高效调控气孔的开闭来平衡植物蒸腾对水分的丧失和植物光合作用对CO2的需求[21-22]。在模式植物拟南芥中已经证明:当将2个KAT型K+通道(KAT 1和KAT2)敲掉1个时, 保卫细胞内向钾电流明显减小, 但不影响其气孔的开放程度及植株的生长[23]; 当同时使KAT1和KAT2散失功能时, 几乎检测不到保卫细胞内流型钾电流, 突变体植株在气孔的开闭与野生型相比都明显延迟, 显著降低了植物生物量的积累[24]。
目前, 水稻(Oryza sativa)基因组测序已经完成, 但其K+通道研究较少, 主要集中在对OsATK 1生理功能及调控机制的研究。OsATK1负责水稻根中钾离子的吸收, 当缺失该通道时, 会严重影响水稻对K+的吸收, 进而出现缺钾症状并影响其生长, 该通道还受CBL1和CIPK23磷酸化过程的调控[25-26]。到目前为止, 对水稻气孔型钾吸收通道的研究较少, 仅Hwang等[27]对水稻的3个KAT类的钾通道进行了基本组织定位, 并利用异源表达系统对其基本功能特性进行了研究, 结果显示OsKAT 1、OsKAT2和OsKAT3都是内向型K+通道, 但OsKAT 1主要在根中表达, 而OsKAT2和OsKAT3主要在叶片中的保卫细胞中表达, 且OsKAT 3的表达丰度明显高于OsKAT2。因气孔型钾吸收通道在水稻体内的生理功能作用还没有研究报道, 此外水稻是单子叶禾本科植物, 且生长在水生环境中, 所以我们提出了这样的科学问题:与拟南芥相比, 它是否会存在特殊的调控机制?
RNAi干扰现象(RNA interference, RNAi)是正常生物体内抑制特定基因表达的一种现象, 是生物体内一种通过双链RNA (double stranded RNA, dsRNA)分子使特异性序列的mRNA发生降解而导致基因表达沉默的过程, 这种现象发生在转录后水平, 即为序列特异性转录后基因沉默(post-transcriptional gene silencing, PTGS)[28-29]。陈忠正等[30]通过RNAi转基因技术, 沉默水稻sbe 3基因, 导致胚乳中sbe3基因的表达水平明显受到抑制。Yan等[31]的研究表明沉默OsNAR 2.1的同时, 也抑制了其他高亲和和低亲和硝酸根转运体OsNRT2.1、OsNRT2.2和OsNRT2.3a的表达。Ai等[32]通过RNAi技术沉默OsPT 2或者OsPT6都大幅度降低了磷从根向地上部的长距离运输。因此, 本试验的目的是构建OsKAT3基因的RNAi载体, 用于转化水稻, 为进一步研究OsKAT 3基因奠定基础。
1 材料与方法 1.1 材料水稻品种‘日本晴’(Orazy sativa‘Nipponbare’)、大肠杆菌(Escherichia coli Top10)、根癌农杆菌(Agrobacterium tumefaciens LBA4404)、RNAi载体pTCK303(14621bp)由本实验室提供。DNA限制性内切酶SacⅠ、SpeⅠ、BamHⅠ-HF、KpnⅠ和T4 DNA Ligase购自NEB公司, 高保真聚合酶PrimeSTAR、pMD19-T、DL2000 DNA Marker、250 bp DNA Marker、琼脂糖、DNA回收试剂盒和质粒小提试剂盒等购自宝生物工程(大连)有限公司。引物合成由上海英骏生物技术有限公司合成, 测序由北京六合华大基因完成。
1.2 OsKAT 3特异片段的克隆选取OsKAT 3的3′端的一段特异区域用于RNAi载体的构建, 命名为C, 正向片段命名为C1, 反向片段命名为C2, 利用Primer Premier 5.0软件设计带有RNAi载体pTCK303上相应酶切位点的引物, 引物序列见表 1。
| 引物名称 Name |
序列(5′→3′) Sequence |
| C1-F | GACTAGTAGCTGAAGCTACGAGAAAG |
| C1-R | CGAGCTCTGTAGTACTCATCTGGAATTC |
| C2-F | GGGGTACCAGCTGAAGCTACGAGAAAG |
| C2-R | CGGGATCCTGTAGTACTCATCTGGAATTC |
| M13-47 | CGCCAGGGTTTTCCCAGTCACGAC |
| pTCK303-F | ATGATAATCATCGCAAGACC |
| pTCK303-R | TCGATGCTCACCCTGTTGTTTGG |
| OsKAT3-DL-F | CCAGACCGAAGATTCTTGAG |
| OsKAT3-DL-R | CTGTCACCTCCAAATACTGG |
| 注:下划线代表此区域为酶切位点。Underline of the sequence represents this area is the place of enzyme cutting sites. | |
以本实验室已克隆的OsKAT 3为模板, 以C1-F/C1-R和C2-F/C2-R为引物分别扩增片段C1和C2。PCR反应体系:1 μL OsKAT 3模板, 1.6 μL dNTP, 4 μL 5×Buffer, 上、下游引物分别为0.5 μL, 0.3 μL Prime STAR® HS DNA Polymerase, 11.9 μL ddH2O。PCR扩增程序:94 ℃ 3 min, 94 ℃ 30 s; 58 ℃ 30 s, 72 ℃ 30 s, 28个循环; 72 ℃延伸5 min。PCR产物切胶回收并用10 g · L-1的琼脂糖凝胶检测, 然后用rTaq酶在72 ℃对纯化的PCR产物加A尾。继而与pMD19-T载体在4 ℃下连接过夜, 最后转化至大肠杆菌Top10感受态细胞, 用pMD19-T载体上的通用引物M13-47和反向引物C1-R/C2-R鉴定有插入的阳性单克隆(pMD19-T-C1和pMD19-T-C2)后送华大基因测序。
1.3 RNAi载体的构建将测序正确的pMD19-T-C1和pMD19-T-C2, 分别通过SacⅠ/SpeⅠ和BamHⅠ/KpnⅠ限制性内切酶将同一特异性片段以正向和反向连接到RNAi表达载体pTCK303中。1)首先将正向片段C1连接到pTCK303载体上, 用SacⅠ/SpeⅠ双酶切pMD19-T-C1和载体pTCK303, 切胶回收纯化C1片段和pTCK303载体, 然后将纯化的片段和载体在4 ℃下连接过夜, 继而将连接产物转化大肠杆菌Top10感受态细胞, 菌落PCR鉴定挑选有插入的阳性克隆, 并提质粒通过SacⅠ/SpeⅠ进行酶切验证。2)片段C反向连接到RNAi载体上, 用BamHⅠ/KpnⅠ双酶切pMD19-T-C2和含有正向片段的载体pTCK303-C1。切胶回收C2和pTCK303-C1, 然后将pTCK303-C1与C2在4 ℃下连接过夜, 转化Top10感受态细胞。菌落PCR鉴定挑选有插入的阳性克隆, 提质粒后分别用SacⅠ/SpeⅠ和BamHⅠ/KpnⅠ限制性内切酶对重组质粒进行酶切验证, 最后将酶切验证正确的质粒转化根癌农杆菌LBA4404, 用于水稻遗传转化。
1.4 农杆菌介导的水稻转化参考Hiei等[33]和林拥军[34]的方法, 采用农杆菌介导的方法转化水稻。农杆菌转化的受体是‘日本晴’水稻。首先用电击法将pTCK303-C质粒转化至农杆菌LBA4404, 然后用该菌液侵染‘日本晴’种子诱导得到的愈伤组织, 继而进行愈伤组织的抗性筛选及抗性愈伤组织的诱导分化和生根, 最后进行转基因水稻苗的炼苗和移栽。
1.5 T0代OsKAT 3 RNAi转基因水稻阳性植株的鉴定用TPS (100 mmol · L-1 Tris-HCl、10mmol · L-1 EDTA、1 mol · L-1 KCl, pH8.0)法[35]提取水稻叶片的DNA, 然后用pTCK303载体上的通用引物进行PCR扩增, 通用引物是在Primer Premier 5.0软件辅助下, 以pTCK303质粒多克隆位点两侧的载体序列为基础设计(表 1)的, 用于检测正、反片段是否都已转入植物体内。
1.6 定量PCR对T0代OsKAT 3 RNAi转基因水稻OsKAT 3基因表达丰度的鉴定用Trizol提取不同株系的OsKAT 3 RNAi转基因水稻和野生型水稻叶片的RNA, 然后反转录为cDNA, 以此cDNA为模板进行定量PCR, 在3′端设计特异引物(表 1)进行定量PCR扩增。
2 结果与分析 2.1 水稻KAT类通道与拟南芥中KAT类通道的同源性比对水稻中的3个KAT类钾通道和拟南芥中的2个KAT类钾通道同源性分析结果(图 1)表明:水稻中的3个钾通道在结构上也是高度保守, 都有6个跨膜结构域和1个通道孔区域, 而OsKAT 1是一个天然的C端缺失的钾通道, OsKAT 2的C端也较OsKAT 3短, 所以水稻中的3个KAT类的钾通道存在差异最大的结构域为C端。

|
图 1 OsKAT 3与拟南芥和水稻KAT类通道的同源性比对 Figure 1 Amino acid sequence alignments between OsKAT 3 and KAT channels among the Arabidopsis and rice 图中的实线框代表钾通道的6个跨膜区域(S1~S6)和1个通道孔区域, 虚线框代表环核苷酸结合域(cNBD)。 The regions in the boxes drawn with solid lines are transmembrane domains (S1-S6) and pore regions, and the boxes drawn with dotted lines arecyclic nucleotide biding domain (cNBD). |
由于水稻中的3个KAT类的钾通道同源性较高, 因此设计RNAi引物时应避开其保守区域对应的mRNA区域。图 1的氨基酸序列比对显示水稻这3个钾通道存在差异最大的区域为C端, 所以我们在OsKAT 3的cDNA序列的3′端选择一段特异区域(C)作为沉默OsKAT 3的特异RNAi片段, 以本实验室已克隆的OsKAT 3为模板, 按照表 1所示的2对引物(C1-F/C1-R和C2-F/C2-R) PCR扩增得到片段C1和C2(图 2)。将C1与C2连到pMD19-T载体上, 挑选单克隆进行菌落PCR鉴定, 结果显示所挑选的均为阳性克隆(图 3), 然后挑选3~4个单克隆送北京六合华大基因测序, 选取测序正确的单克隆富集培养后并提取质粒, 用于后续RNAi载体的构建。
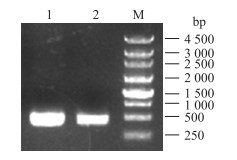
|
图 2 C1和C2的PCR产物电泳检测 Figure 2 Electrophoresis validation of PCR amplification products of C1 and C2 1.C1片段PCR products of C1;2.C2片段PCR products of C2;M.250 bp DNA Marker |
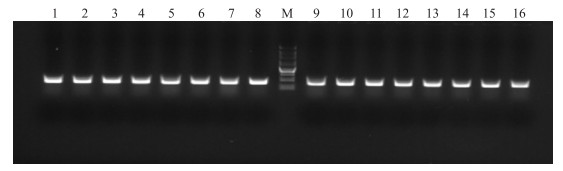
|
图 3 重组质粒pMD19-T-C1和pMD19-T-C2的菌落PCR验证 Figure 3 Bacterial colony PCR confirmation of recombinant plasmids of pMD19-T-C1 and pMD19-T-C2 1~8为C1阳性克隆。1-8 represent the positive clones of pMD19-T-C1;9~16为C2阳性克隆。9-16 represent the positive clones of pMD19-T-C2;M. 250 bp DNA Marker |
将从pMD19-T载体上切下来的正向片段C1和反向片段C2用于RNAi载体的构建, 将正、反片段连到RNAi载体pTCK303上, 然后对构建好的pTCK303-C重组质粒进行酶切验证, 结果如图 4所示, 1泳道代表SacⅠ/SpeⅠ酶切结果, 表明正向片段已连到载体上, 2泳道表示BamHⅠ/KpnⅠ双酶切鉴定的结果, 显示反向片段已连接到载体上, 酶切结果表明RNAi载体构建成功。
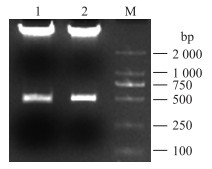
|
图 4 pTCK303-C酶切鉴定 Figure 4 The double restriction enzyme of pTCK303-C 1和2分别为pTCK303-CSacⅠ/SpeⅠ和BamHⅠ/KpnⅠ酶切电泳图。1 and 2 represent enzyme electrophoresis diagram of pTCK303-C digested with SacⅠ/SpeⅠ and BamHⅠ/KpnⅠ, respectively; M:DL2000 DNA marker |
通过TPS法提取OsKAT 3 RNAi转基因水稻和野生型水稻叶片的基因组DNA, 用pTCK303载体上的通用引物(pTCK303-F和pTCK303-R)进行转基因阳性植株鉴定, 以野生型(WT)作为对照, 预期转基因阳性水稻扩增片段中包括正、反片段共计888 bp, 载体上的序列片段(包括中间内含子)扩增大小为653 bp, 所以转基因水稻采用pTCK303载体上的通用引物扩增出来的片段大小为1 541 bp。图 5所示的鉴定结果显示27株转基因水稻中只有2株为假阳性。
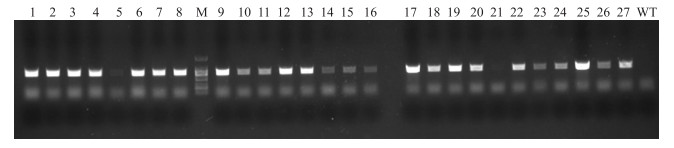
|
图 5 T0代转基因水稻的阳性检测 Figure 5 PCR detection of the positive clones in T0 generation RNAi transgenic rice plants 1~27为不同的T0代RNAi转基因植株。1-27 represent different T0 generation RNAi transgenic rice plants; WT:野生型水稻Wild type rice; M:DL2000 DNA marker |
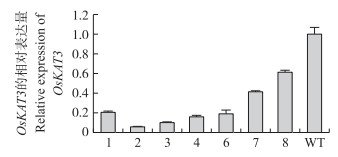
利用定量PCR对T0代部分RNAi转基因水稻植株的OsKAT 3基因表达量进行分析, 结果(图 6)显示, 与野生型‘日本晴’相比, 检测的7株T0代转基因水稻植株表达量都有不同程度的下降, 其中前5个株系的表达量下降达80%, 株系2和3下降达90%左右, 而株系7和8下降比例相对较少, 所以选择株系2和3作为后续试验研究的材料。
|
图 6 RT-PCR检测T0代RNAi转基因植株中OsKAT 3基因表达量 Figure 6 RT-PCR detection of the OsKAT 3 gene expression in the T0 generation transgenic rice plants 1~8为不同转基因株系。1-8 represent different T0 generation RNAi transgenic rice plants; WT:野生型Wild type |
通过将水稻中的KAT类钾通道与拟南芥的KAT类通道进行氨基酸序列比对, 我们发现无论在氨基酸序列和关键结构元件上都与拟南芥中的通道高度同源。尽管玉米中的ZmK2.1与拟南芥KAT1高度同源, 跨膜区的相似性达88%, 且通道孔区域只有1个氨基酸的差异, 但玉米中的ZmK2.1却形成了不同于KAT1的钾感应机制和酸调控机制[36]。因此, 长期生长在淹水、高温环境中的水稻, 其钾离子通道在长期适应生长环境的过程中是否会形成有别于陆生且弱光照植物拟南芥的独特功能特性有待研究。水稻中有3个KAT类钾通道, 其中OsKAT 1是一个天然的C端缺失的钾通道, 且与以往报道的KAT1钾通道主要在叶片中表达不一样, 该通道主要在根中表达, 而OsKAT 2和OsKAT3在叶片保卫细胞中表达, 负责调控气孔的开。本研究中, 我们克隆了叶片中的OsKAT 2和OsKAT3, 发现OsKAT2与其他KAT类通道相比少了一段S3-S4 linker, 且在蛙卵表达系统中没有检测到电流, 而OsKAT3能检测到电流, OsKAT2是否没有功能或是否需要与OsKAT3形成异源四聚体才表现出介导K+吸收的功能还需进一步的验证。本研究主要研究水稻中KAT类通道中表达丰度最高的OsKAT 3的功能特性, 而本试验的目的主要是构建OsKAT 3的RNAi转基因水稻。
RNAi技术的序列特异性、高效性和多效性, 为大规模高效研究复杂的基因功能提供了技术平台。我们知道植物界基因家族现象很普遍, RNAi既可用于不同成员进行分别沉默, 也可用于对整个家族进行有效沉默。因此当我们将KAT家族中的一个基因OsKAT 3进行沉默时, 选择了该基因C端的特异性片段。近期研究表明, 选择发夹结构臂序列的最佳片段应该为200~600 bp。片段太短不利于构建操作, 并且太短的dsRNA不能被DICER酶很好的识别; 太长片段形成的dsRNA在结构上不太稳定[37]。本研究所构建的RNAi载体2个反向重复序列臂之间有内含子, 且序列长度为444 bp (在最佳片段范围), 可以预测此载体进行转化获得的转基因植株, 将会对靶基因产生较好的基因沉默效果。
在RNAi表达载体构建时, 通常需要在质粒中同时插入序列相同、方向相反的2个目的片段, 在表达载体的PCR检测中, 任何一条与目的基因序列一致的鉴定引物都可以同时与RNAi载体中反向重复的2个插入片段相结合, 导致非特异性扩增概率急剧增大。因而使RNAi表达载体的PCR鉴定难度增大, 单纯的目的基因序列引物扩增已经无法满足鉴定需要。所以我们采用的是载体上的通用引物进行PCR鉴定[38]。可以根据PCR扩增片段的大小确定正、反片段是否已成功整合到植物体中。定量PCR结果显示:OsKAT 3 RNAi转基因植株出现了OsKAT 3表达丰度不同程度的降低, 获得的OsKAT 3 RNAi T0代种子为后续研究该基因功能奠定了一定的基础。
我们用OsKAT 3 RNAi株系和OsKAT 3的过表达株系对OsKAT 3在水稻中的生理功能进行了后续研究, 将获得的OsKAT 3 RNAi T0代种子继续进行纯系筛选, 并用OsKAT 3 RNAi纯系和过表达纯系进行水培和大田试验。OsKAT 3负责调控气孔的开放, 气孔开度的大小进一步影响植株胞间CO2浓度、光合速率和蒸腾速率。蒸腾速率大小的变化又会进一步影响植株对营养元素的吸收和运输, 影响植株的长势; 光合速率的变化影响植株干物质的积累和向籽粒中碳水化合物的转移; 蒸腾速率和光合速率的变化最终反映在水稻产量的变化。因此, 我们在分蘖盛期和抽穗期测定比较OsKAT 3 RNAi转基因水稻和野生型水稻的气孔导度、光合速率、蒸腾速率和胞间CO2浓度, 并对分蘖数、有效分蘖数、茎粗和株高进行统计, 于收获期对产量、穗质量、穗粒数、饱满穗粒百分数和千粒质量等指标进行统计。通过对OsKAT 3 RNAi株系、过表达株系与野生型水稻的这些指标的比较分析来解释OsKAT 3对气孔调控的作用和对水稻产量形成的影响。
| [1] | Leigh R A, Jones R G W. A hypothesis relating critical potassium concentrations for growth to the distribution and functions of this ion in the plant cell[J]. New Phytologist, 1984,97(1): 1–13. DOI: 10.1111/nph.1984.97.issue-1 |
| [2] | Walker D J, Leigh R A, Miller A J. Potassium homeostasis in vacuolate plant cells[J]. Proc Natl Acad Sci USA, 1996,93(19): 10510–10514. DOI: 10.1073/pnas.93.19.10510 |
| [3] | Véry A A, Nieves-Cordones M, Daly M, et al. Molecular biology of K+ transport across the plant cell membrane:what do we learn from comparison between plant species[J]. Plant Physiol, 2014,171(9): 748–769. DOI: 10.1016/j.jplph.2014.01.011 |
| [4] | Epstein E, Rains D W, Elzam O E. Resolution of dual mechanisms of potassium absorption by barley roots[J]. Proc Natl Acad Sci USA, 1963,49(5): 684–692. DOI: 10.1073/pnas.49.5.684 |
| [5] | Pilot G, Gaymard F, Mouline K, et al. Regulated expression of Arabidopsis shaker K+ channel genes involved in K+ uptake and distribution in the plant[J]. Plant Mol Biol, 2003,51(5): 773–787. DOI: 10.1023/A:1022597102282 |
| [6] | Sentenac H, Bonneaud N, Minet M, et al. Cloning and expression in yeast of a plant potassium ion transport system[J]. Science, 1992,256(5057): 663–665. DOI: 10.1126/science.1585180 |
| [7] | Lagarde D, Basset M, Lepetit M, et al. Tissue-specific expression of Arabidopsis AKT1 gene is consistent with a role in K+nutrition[J]. Plant J, 1996,9(2): 195–203. DOI: 10.1046/j.1365-313X.1996.09020195.x |
| [8] | Hirsch R E, Lewis B D, Spalding E P, et al. A role for the AKT1 potassium channel in plant nutrition[J]. Science, 1998,280(5365): 918–921. DOI: 10.1126/science.280.5365.918 |
| [9] | Spalding E P, Hirsch R E, Lewis D R, et al. Potassium uptake supporting plant growth in the absence of AKT1 channel activity inhibition by ammonium and stimulation by sodium[J]. The Journal of General Physiology, 1999,113(6): 909–918. DOI: 10.1085/jgp.113.6.909 |
| [10] | Xu J, Li H D, Chen L Q, et al. A protein kinase, interacting with two calcineurin B-like proteins, regulates K+ transporter AKT1 in Arabidopsis[J]. Cell, 2006,125(7): 1347–1360. DOI: 10.1016/j.cell.2006.06.011 |
| [11] | Marten I, Hoth S, Deeken R, et al. AKT3, a phloem-localized K+ channel, is blocked by protons[J]. Proc Natl Acad Sci USA, 1999,96(13): 7581–7586. DOI: 10.1073/pnas.96.13.7581 |
| [12] | Lacombe B, Pilot G, Michard E, et al. A shaker-like K (+) channel with weak rectification is expressed in both source and sink phloem tissues of Arabidopsis[J]. Plant Cell, 2000,12(6): 837–851. |
| [13] | Duby G, Hosy E, Fizames C, et al. AtKC1, a conditionally targeted shaker-type subunit, regulates the activity of plant K+channels[J]. Plant J, 2008,53(1): 115–123. DOI: 10.1111/tpj.2008.53.issue-1 |
| [14] | Geiger D, Becker D, Vosloh D, et al. Heteromeric AtKC1·AKT1 channels in Arabidopsis roots facilitate growth under K+-limiting conditions[J]. J Biol Chem, 2009,284(32): 21288–21295. DOI: 10.1074/jbc.M109.017574 |
| [15] | Gaymard F, Pilot G, Lacombe B, et al. Identification and disruption of a plant shaker-like outward channel involved in K+ release into the xylem sap[J]. Cell, 1998,94(5): 647–655. DOI: 10.1016/S0092-8674(00)81606-2 |
| [16] | Ache P, Becker D, Ivashikina N, et al. GORK, a delayed outward rectifier expressed in guard cells of Arabidopsis thaliana, is a K (+)-selective, K (+)-sensing ion channel[J]. FEBS Lett, 2000,486(2): 93–98. DOI: 10.1016/S0014-5793(00)02248-1 |
| [17] | Dietrich P, Sanders D, Hedrich R. The role of ion channels in light-dependent stomatalopening[J]. J Exp Bot, 2001,52(363): 1959–1967. DOI: 10.1093/jexbot/52.363.1959 |
| [18] | Dreyer I, Blatt M R. What makes a gate?The ins and outs of Kv-like K+ channels in plants[J]. Trends Plant Sci, 2009,14(7): 383–390. DOI: 10.1016/j.tplants.2009.04.001 |
| [19] | Lebaudy A, Véry A A, Sentenac H. K+channel activity in plants:genes, regulations and functions[J]. FEBS Lett, 2007,581(12): 2357–2366. DOI: 10.1016/j.febslet.2007.03.058 |
| [20] | Müller-R ber B, Ehrhardt T, Plesch G. Molecular features of stomatal guard cells[J]. J Exp Bot, 1998,49: 293–304. DOI: 10.1093/jxb/49.Special_Issue.293 |
| [21] | Roelfsema M R G, Hedrich R. In the light of stomatal opening:new insights into'the Watergate'[J]. New Phytol, 2005,167(3): 665–691. DOI: 10.1111/nph.2005.167.issue-3 |
| [22] | Schroeder J I, Allen G J, Hugouvieux V, et al. Guard cell signal transduction[J]. Annu Rev Plant Physiol Plant Mol Biol, 2001,52(1): 627–658. DOI: 10.1146/annurev.arplant.52.1.627 |
| [23] | Szyroki A, Ivashikina N, Dietrich P, et al. KAT1 is not essential for stomatalopening[J]. Proc Natl Acad Sci USA, 2001,98(5): 2917–2921. DOI: 10.1073/pnas.051616698 |
| [24] | Lebaudy A, Vavasseur A, Hosy E, et al. Plant adaptation to fluctuating environment and biomass production are strongly dependent on guard cell potassium channels[J]. Proc Natl Acad Sci USA, 2008,105(13): 5271–5276. DOI: 10.1073/pnas.0709732105 |
| [25] | Fuchs I, St lzle S, Ivashikina N, et al. Rice K+ uptake channel OsAKT1 is sensitive to salt stress[J]. Planta, 2005,221(2): 212–221. DOI: 10.1007/s00425-004-1437-9 |
| [26] | Li J, Long Y, Qi G N, et al. The Os-AKT1 channel is critical for K+ uptake in rice roots and is modulated by the rice CBL1-CIPK23 complex[J]. Plant Cell, 2014,26(8): 3387–3402. DOI: 10.1105/tpc.114.123455 |
| [27] | Hwang H, Yoon J, Kim H Y, et al. Unique features of two potassium channels, OsKAT2 and OsKAT3, expressed in rice guard cells[J]. PLoS ONE, 2013,8(8): e72541. DOI: 10.1371/journal.pone.0072541 |
| [28] | Fire A, Xu S Q, Montgomery M K, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans[J]. Nature, 1998,391(6669): 806–811. DOI: 10.1038/35888 |
| [29] | Timmons L, Fire A. Specific interference by ingested dsRNA[J]. Nature, 1998,395(6705): 854. DOI: 10.1038/27579 |
| [30] | 陈忠正, 郭健, 李斌, 等. RNAi沉默淀粉分支酶sbe3基因对水稻直链淀粉的影响[J]. 食品科学, 2007, 28(7): 291–295. Chen Z Z, Guo J, Li B, et al. Effects of RNAi silencing starch branch enzyme 3(sbe3) gene on rice amylose content[J]. Food Science, 2007,28(7): 291–295. (in Chinese) |
| [31] | Yan M, Fan X, Feng H, et al. Rice OsNAR2.1 interacts with OsNRT2.1, OsNRT2.2 and OsNRT2. 3a nitrate transporters to provide uptake over high and low concentration ranges[J]. Plant Cell Environ, 2011,34(8): 1360–1372. DOI: 10.1111/pce.2011.34.issue-8 |
| [32] | Ai P, Sun S, Zhao J, et al. Two rice phosphate transporters, OsPht1;2 and OsPht1;6, have different functions and kinetic properties in uptake and translocation[J]. Plant J, 2009,57(5): 798–809. DOI: 10.1111/tpj.2009.57.issue-5 |
| [33] | Hiei Y, Ohta S, Komari T, et al. Efficient transformation of rice (Oryza sativa L.) mediated by Agrobacterium and sequenceanalysis of the boundaries of the T-DNA[J]. Plant J, 1994,6(2): 271–282. DOI: 10.1046/j.1365-313X.1994.6020271.x |
| [34] | 林拥军.农杆菌介导的水稻转基因研究[D].武汉:华中农业大学, 2001. Lin Y J. The study of transgenic rice mediated by agrobacterium[D]. Wuhan:Huazhong Agricultural University, 2001(in Chinese with English abstract). |
| [35] | 穆春华, 张发军, 李文才, 等. 玉米叶片基因组快速提取方法研究[J]. 玉米科学, 2010, 18(3): 170–172. Mu C H, Zhang F J, Li W C, et al. A method of genomic DNA extraction of maize[J]. Journal of Maize Sciences, 2010,18(3): 170–172. (in Chinese) |
| [36] | Su Y H, North H, Grignon C, et al. Regulation by external K+in a maize inward shaker channel targets transport activity in the high concentration range[J]. Plant Cell, 2005,17(5): 1532–1548. DOI: 10.1105/tpc.104.030551 |
| [37] | 冯光秀, 陈惠. 水稻OsNCED3基因的RNAi载体构建[J]. 生物学杂志, 2012, 29(1): 47–50. Feng G X, Chen H. Construction of RNAi vector of OsNCED3 gene[J]. Journal of Biology, 2012,29(1): 47–50. (in Chinese) |
| [38] | 王华新, 曹家树, 向珣, 等. pBI121表达载体及其转化植株的快速鉴定[J]. 浙江大学学报(农业与生命科学版), 2008, 34(2): 137–142. Wang H X, Cao J S, Xiang X, et al. Rapid method to identify expression vectors and tansgenic plants based on pBI121 plasmid[J]. Journal of Zhejiang University (Agric Life Sci Edit), 2008,34(2): 137–142. (in Chinese) |




