文章信息
- 曲晓旭, 刘洪霞, 高玲, 陆兆新, 吕凤霞, 张充, 赵海珍, 别小妹
- QU Xiaoxu, LIU Hongxia, GAO Ling, LU Zhaoxin, LÜ Fengxia, ZHANG Chong, ZHAO Haizhen, BIE Xiaomei
- 芽孢杆菌产抗菌脂肽调控基因快速检测
- Rapid identification of the encoding genes of antimicrobial lipopeptides producted by Bacillus
- 南京农业大学学报, 2016, 39(5): 858-864
- Journal of Nanjing Agricultural University, 2016, 39(5): 858-864.
- http://dx.doi.org/10.7685/jnau.201601042
-
文章历史
- 收稿日期: 2016-01-21
抗菌脂肽类物质一般是由革兰氏阳性芽孢杆菌产生的一类代谢物, 随着现代生物技术的迅猛发展以及抗菌脂肽类物质在环保、石油开采、制药、食品加工等方面的潜在应用价值不断被发现[1-2], 人们对于在工业规模上通过生物技术方法生产抗菌脂肽产生了强烈兴趣。
目前已发现的抗菌活性物质主要有:通过非核糖体途径合成的低分子质量的抗菌脂肽, 如Surfactin、Fengycin、Iturin和Bacillomycin、Mycosubtilin、Plipstatin等[3]; 还有通过核糖体途径合成的细菌素类物质, 如TasA、Subtilin、Ericin、Mersacidin、Sublancin和Subtilosin A等[3]。抗菌脂肽的产生菌主要集中在芽孢杆菌属的不同菌株之间[4], 除枯草芽孢杆菌(Bacillus.subtilis)外, 淀粉液化芽孢杆菌(B.amyloliquefaciens)、短小芽孢杆菌(B.pumilus)、地衣芽孢杆菌(B.lichemformis)、蜡样芽孢杆菌(B.cereus)、苏云金芽孢杆菌(B.thuringiensis)、多黏类芽孢杆菌(Paenibacillus paenibacillus)和环状芽孢杆菌(B.circulan)等也能产生抗菌脂肽类物质。
目前关于脂肽类化合物的研究主要集中于它的物理、化学性质和生物学特性, 但是对于脂肽类化合物合成表达的分子调控机制的研究报道较少[5]。对抗菌脂肽产生菌早期鉴定方法[3, 6]主要通过菌种发酵、有机溶液萃取、抑菌试验、高效液相色谱法和核磁共振结合的方法进行的, 过程繁琐且成本较高, 不适于快速筛选。因此, 通过PCR技术进行编码调控基因的检测为实现抗菌脂肽产生菌快速识别、鉴定提供了一种有效方法[7-8]。
本研究选用多株枯草芽孢杆菌、淀粉液化芽孢杆菌及多黏类芽孢杆菌的典型菌株作为研究对象, 针对几种典型脂肽类抗菌物质合成酶基因进行PCR扩增, 并通过基因测序的方法确保PCR扩增结果的正确性, 旨在基于PCR技术建立了一种抗菌脂肽产生菌快速识别、筛选方法。
1 材料与方法 1.1 材料 1.1.1 菌种本试验所用菌株为Bacillus subtilis PB2、PB198、fmbR、fmbR-2、fmb4、fmb3、fmb2、fmbj, Bacillus amyloliquefaciens ES-2和Paenibacillus polymyxa JSa-9、JSa-9-81, 所有菌株均由南京农业大学酶工程实验室保藏; Escherichia coli DH5α菌株及克隆载体pMD19-T购自大连宝生物(TaKaRa)有限公司。
1.1.2 培养基及溶液配制LB培养基:蛋白胨10.0 g·L-1, 酵母提取物5.0 g·L-1, NaCl 10.0 g·L-1, pH调至7.0~7.2, 121 ℃ 20 min灭菌备用。固体LB培养基加入15~20 g·L-1琼脂。氨苄青霉素(Amp)溶液:将1.0 g Amp溶解于10 mL蒸馏水中, 配成100 g·L-1的Amp母液, 用无菌滤膜过滤除菌, -20 ℃保存使用。
1.1.3 主要生化及分子生物学试剂柱式细菌基因组提取试剂盒、柱式胶回收试剂盒、柱式质粒提取试剂盒均购自上海生工生物工程有限公司。各种分子生物学酶试剂、T4连接酶、2×Taq Master Mix DNA聚合酶等均购自南京诺唯赞(Vazyme)生物科技有限公司。蛋白胨、NaCl、琼脂糖、无水乙醇、EB染液等生化试剂及普通化学试剂均为进口或国产分析纯。
1.1.4 实验仪器JS-380C全自动数码凝胶成像分析仪购于上海培清科技有限公司; PTC-100TM和PTC-200TM PCR仪购于MJ Research公司; Eppendorf 5418离心机、Eppendorf移液枪购于德国Eppendorf公司; SW-CJ-IBU型净化工作台购于江苏安泰空气技术有限公司; HYG-A全温摇瓶柜购于太仓实验设备厂; BS-200S电子天平购于Sartorius公司。
1.2 检测基因的PCR扩增及测序选择6种常见的抗菌脂肽Surfactin、Fengycin、Iturin、Subtilosin A、Bacillomycin D和Polymyxin的编码和调控基因作为检测对象, 进行PCR扩增。选用菌株B. subtilis 168(GenBank登录号:AL009126.3)、B.amyloliquefaciens FZB42(CP000560.1)和P.polymyxa M1(HE577054.1)的相关待测调控基因信息, 设计出几种基因的克隆引物, PCR引物委托上海生工生物工程有限公司合成。其序列见表 1。
| 抗菌脂肽 Antibacterial lipopeptide |
靶基因 Target gene |
引物名称 Primers name |
引物序列(5′→3′) Primers sequences(5′→3′) |
扩增长度/bp Size |
| Surfactin | sfp | sfp-F sfp-R |
ATGAAGTTTACGGAATTTA TTATAAAAGCTCTTCGTACG |
675 |
| Surfactin | srfAA | srfAA-F srfAA-R |
GCCCGT GAGCCGAAT GGATAAG CCGTTTCAGGGACACAAGCTCCG |
1 600 |
| Surfactin | srfAD | srfAD-F srfAD-R |
CCGTTCGCAGGAGGCTATTCC CGCCCATCCTGCTGAAAAAGCG |
1 300 |
| Fengycin | fenA | fenA-F fenA-R |
GCTGTCCGTTCTGCTTTTTC GTCGGTGCATGAAATGTACG |
1 000 |
| Fengycin | fenB | fenB-F fenB-R |
CTATAGTTTGTTGACGGCTC CAGCACTGGTTCTTGTCGCA |
1 600 |
| Fengycin | fenD | fenD-F fenD-R |
TTTGGCAGCAGGAGAAGTTT GACAGTGCTGCCTGATGAAA |
1 600 |
| Iturin | ituD | ituD-F ituD-R |
ATGAACAATCTTGCCTTTTTA TTATTTTAAAATCCGCAATT |
1 203 |
| Iturin | ituA | ituA-F ituA-R |
ATGTATACCAGTCAATTCC GATCCGAAGCTGACAATAG |
1 047 |
| Iturin | ituB | ituB-F ituB-R |
CAACGGTATCGAAGCA CGTCTCGGGTATCATTT |
449 |
| Subtilosin A | albF | albF-F albF-R |
GCTGGATGAACGAACCG GATGCGATGTCCCAATAA |
822 |
| Subtilosin A | albA | albA-F albA-R |
ATGCGAATGGATGGG GCAATAGCTGCTGAACG |
1 086 |
| Bacillomycin D | bmyA | bmyA-F bmyA-R |
AAAGCGGCTCAAGAAGCGAAACCC CGATTCAGCTCATCGACCAGGTAGGC |
1 200 |
| Bacillomycin D | bmyB | bmyB-F bmyB-R |
AATAGAAGAACTGCTGGCGT GCCTTCCCGACACGACACT |
983 |
| Bacillomycin D | bmyC | bmyC-F bmyC-R |
GAAGGACACGGCAGAGAGTC CACTGATGACTGTTCATGCT |
875 |
| Polymyxin | pmxB | pmxB-F pmxB-R |
CAACACCGTCGCATCCTT CCGCCCGCTTTGAGAAT |
527 |
| Polymyxin | pmxC | pmxC-F pmxC-R |
TTCCAAACGCCAAACCG TCGTGCCGCTAAACAAAT |
1 348 |
| Polymyxin | pmxE | pmxE-F pmxE-R |
GCTGGAAGACGGCAACG GACCACCCAAGTCAAAGAAA |
464 |
提取检测菌株总DNA作为模板, PCR反应体系(50 μL):2×Taq Master Mix DNA聚合酶(1 U·μL-1)1 μL, 模板DNA 2 μL, 上、下游引物各2 μL, ddH2O 17 μL。反应条件:94 ℃ 3 min; 94 ℃ 15 s, 55 ℃ 30 s, 72 ℃ 30 s, 共30个循环; 72 ℃ 10 min。PCR产物经琼脂糖凝胶电泳进行鉴定后回收目的片段, 将目的片段连接到克隆载体pMD19-T上, 转化E.coli DH5α, 随机挑选阳性转化子若干提取质粒, 委托南京金斯瑞生物有限科技公司完成测序。
2 结果与分析 2.1 菌株Surfactin合成酶基因的PCR扩增检测Surfactin由25 kb的srfA操纵子编码合成, 该操纵子编码Surfactin合成酶的4个亚基srfAA、srfAB、srfAC和srfAD[9]。同时sfp基因在Surfactin合成过程中同样扮演着重要角色[10]。在srfA操纵子中, srfAA基因负责编码Surfactin合成酶的第1个肽单体酶。而srfAD基因与脂肽链的内酯化相关。在本研究中, 我们选择sfp基因(675 bp)、srfAA基因(1 600 bp)和srfAD基因(1 300 bp)作为研究对象, 由于srfAA基因和srfAD基因的完整序列过长, 仅选择其保守序列进行PCR扩增反应。由图 1可见:所有检测菌株均扩增出与目的片段相同大小的明亮条带。结合测序结果, 我们可以确定所有检测菌株均具有Surfactin合成酶编码基因。而从目前报道已知, B.subtilis fmbj[11]、B.amyloliquefaciens ES-2[12]和B.subtilis fmbR[13]菌株均具有合成Surfactin的能力, 其他菌株产抗菌脂肽Surfactin能力尚未报道。
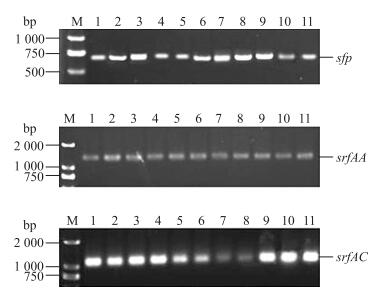
|
图 1 Surfactin合成酶基因的PCR产物 Figure 1 PCR products of genes required for Surfactin biosynthesis M:DL2000 marker; 1:B.subtilis PB2;2:B.subtilis fmbj; 3:B.subtilis PB198;4:B.subtilis fmbR-2;5:B.amyloliquefaciens ES-2;6:B.subtilis fmbR; 7:P.polymyxa JSa-9;8:P.polymyxa JSa-9-81;9:B.subtilis fmb4;10:B.subtilis fmb2;11:B.subtilis fmb3 |
编码Fengycin合成的调控基因由fen操纵子组成, 包括fenC、fenD、fenE、fenA和fenB 5个基因, 分别控制合成编码Fengycin合成酶的5个单体酶:FenC、FenD、FenE、FenA和FenB。Fengycin合成酶一共包括10个模块, 每个模块单独活化一种氨基酸, 这些氨基酸依次连接成完整的脂肽链。Fengycin合成过程由FenC开始, 经FenD、FenE、FenA的传递, 最终由FenB的TE结构域终止合成, 释放完整肽链。因此, 本研究选择fenA基因(1 000 bp)、fenB基因(1 600 bp)和fenD基因(1 600 bp)作为Fengycin合成酶的代表基因进行PCR扩增。由图 2可见:所有检测菌株均扩增得到目的片段大小的明亮条带, 但经过测序验证, 菌株P.polymyxa JSa-9的PCR扩增结果为假阳性, 其他菌株测序正确。除B.subtilis fmbj和B.amyloliquefaciens ES-2菌株外, 其他菌株尚未发现具有合成Fengycin的能力。
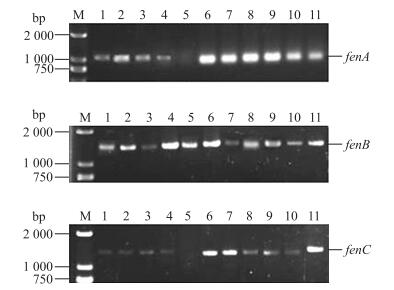
|
图 2 Fengycin合成酶基因的PCR产物 Figure 2 PCR products of genes required for Fengycin biosynthesis M:DL2000 marker; 1:B.subtilis PB2;2:B.subtilis fmbj; 3:B.subtilis PB198;4:B.subtilis fmbR-2;5:P.polymyxa JSa-9;6:B.amyloliquefaciens ES-2;7:B.subtilis fmbR; 8:P.polymyxa JSa-9-81;9:B.subtilis fmb4;10:B.subtilis fmb2;11:B.subtilis fmb3 |
Iturin A合成酶操纵子由ituD、ituA、ituB和ituC 4个开放性读码框构成, 它们在Iturin合成过程中各司其职:ituD基因可能与丙二酰辅酶A转酰基酶有关, 该酶负责编码脂肪酸的合成[9]。当细菌缺乏ituD基因时, Iturin合成产量明显不足。ituA基因能够编码合成一段蛋白, 这段蛋白与脂肪酸合成酶、氨基酸转移酶以及肽合成酶都具有很高的同源性, 因此推测ituA基因可能与β-氨基脂肪酸的形成有关。而ituB基因则负责编码合成具有4个氨基酸模块的肽合成酶。本研究我们选择ituA基因(1 047 bp)、ituB基因(449 bp)和ituD基因(1 203 bp)的保守序列作为靶基因进行PCR扩增。由图 3可见:只有B.subtilis fmbj和B.amyloliquefaciens ES-2菌株扩增得到目的条带大小的片段, B.subtilis fmbR菌株仅扩增得到ituA和ituD基因大小的片段。大多数淀粉液化芽孢杆菌都具有合成抗菌肽Iturin的能力[14], 比如B.amyloliquefaciens ES-2就是一株已报道的Iturin产生菌[12]。但是结合测序结果证明B.subtilis fmbj与B.subtilis fmbR菌株的PCR扩增结果属于假阳性。因为ituDABC操纵子与bmyDABC操纵子具有高度同源性[15], 测序结果表明, PCR扩增得到的目的条带与bmyDABC操纵子的相似性更高。
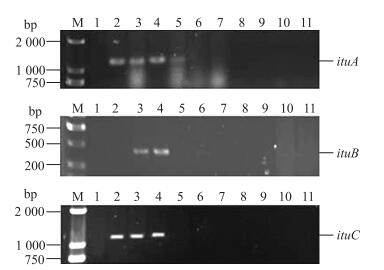
|
图 3 Iturin合成酶基因的PCR产物 Figure 3 PCR products of genes required for Iturin biosynthesis M:DL2000 marker; 1:B.subtilis PB2;2:B.subtilis fmbR; 3:B.subtilis fmbj; 4:B.amyloliquefaciens ES-2;5:B.subtilis PB198;6:B.subtilis fmbR-2;7:P.polymyxa JSa-9;8:P.polymyxa JSa-9-81;9:B.subtilis fmb4;10:B.subtilis fmb2;11:B.subtilis fmb3 |
Bacillomycin D是Iturin家族的一类抗菌脂肽, 它与Iturin结构相似, 但是其氨基酸组成不同。Bacillomycin D是由非核糖体肽合成酶的多载体巯基化模板机制控制合成的, 是一个由β-氨基脂肪酸组成的七肽环状物质[16]。Bacillomycin D由bmyDABC操纵子控制合成, 该操纵子包括bmyD、bmyA、bmyB和bmyC 4个基因。bmyA、bmyB和bmyC基因包含可以编码脂肽合成的氨基酸活化模块, 它们组成了Bacillomycin D合成酶的单体结构。bmyA基因负责Bacillomycin D合成的起始反应, 经由bmyB基因传递至bmyC基因, bmyC基因包含TE结构域, 可能参与Bacillomycin D的终止合成与环化过程[17]。我们选择bmyA基因(1 200 bp)、bmyB基因(983 bp)和bmyC(875 bp)与ituDABC操纵子不同的保守序列进行PCR扩增, 结果(图 4)表明, 在菌株B.subtilis fmbj、B.amyloliquefaciens ES-2、B.subtilis fmbR、P.polymyxa JSa-9-81和B.subtilis fmbR-2中扩增得到了目的条带且测序结果正确。B.subtilis fmbj菌株的基因检测结果从分子角度验证了其具有合成Bacillomycin D的能力[16, 18]。其他菌株均属于首次扩增得到Bacillomycin D合成酶基因, 推测其具有产生Bacillomycin D的能力。
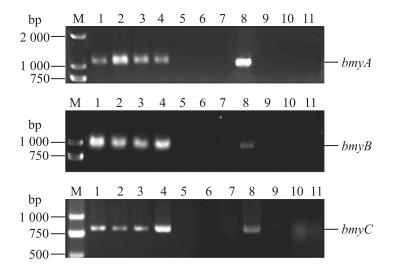
|
图 4 Bacillomycin D合成酶基因的PCR产物 Figure 4 PCR products of genes required for Bacillomycin D biosynthesis M:DL2000 marker; 1:B.subtilis fmbj; 2:B.amyloliquefaciens ES-2;3:B.subtilis fmbR; 4:P.polymyxa JSa-9-81;5:B.subtilis PB2;6:B.subtilis PB198;7:P.polymyxa JSa-9;8:B.subtilis fmbR-2;9:B.subtilis fmb4;10:B.subtilis fmb2;11:B.subtilis fmb3 |
sbo-alb基因簇负责Subtilosin A的编码合成, 该基因簇包括sbo、albA、albB、albC、albD、albE、albF和albG 8个基因[19]。因为Subtilosin A合成酶的操纵子序列较短, 本研究仅选用albA基因(1 086 bp)和albF基因(822 bp)作为研究对象。选择这2个基因的原因如下:albA基因是alb基因簇的第1个基因, 该基因负责Subtilosin A前体的翻译后修饰, 它的合成产物对金属离子和酶辅因子的结合有一定作用。而albF基因则负责Subtilosin A合成的后期加工过程。由图 5可见:B.subtilis fmbR、PB2、fmbj、PB198、fmb4、fmb2和fmb3菌株中发现了Subtilosin A合成酶基因albA和albF的存在, 这些菌株均具有合成Subtilosin A的能力。
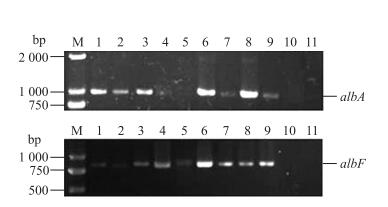
|
图 5 Subtilosin A合成酶基因的PCR产物 Figure 5 PCR products of genes required for Subtilosin A biosynthesis M:DL2000 marker; 1:B.subtilis PB2;2:B.subtilis fmbj; 3:B.subtilis PB198;4:B.subtilis fmbR-2;5:B.amyloliquefaciens ES-2;6:B.subtilis fmbR; 7:B.subtilis fmb4;8:B.subtilis fmb2;9:B.subtilis fmb3;10:P.polymyxa JSa-9;11:P.polymyxa JSa-9-81 |
Choi等[20]报道了Polymyxin合成酶的完整基因簇序列及其功能, Polymyxin的基本结构是一个具有氨基末端脂肪酸酰化三肽侧链的环状七肽物质。Polymyxin合成酶的操纵子包括5个开放性读码框:pmxA、pmxB、pmxC、pmxD和pmxE。其中pmxC基因负责编码运输蛋白, 与Polymyxin的外化有关。而pmxB和pmxE基因则负责编码Polymyxin合成酶。pmxB基因末端的TE结构域负责终止和释放完整肽链。本研究选择pmxB基因(527 bp)、pmxC基因(1 348 bp)和pmxE基因(464 bp)作为目的基因进行PCR扩增。由图 6可知:只有已知的Polymyxin产生菌P.polymyxa JSa-9和P.polymyxa JSa-9-81菌株扩增得到目的基因[21-22], 其他菌株尚未发现具有Polymyxin合成酶基因片段。
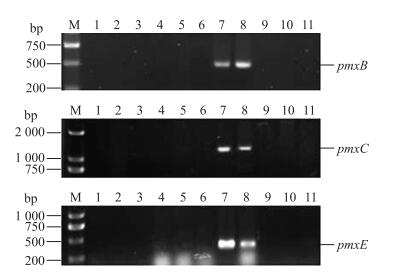
|
图 6 Polymyxin合成酶基因的PCR产物 Figure 6 PCR products of genes required for Polymyxin biosynthesis M:DL2000 marker; 1:B.subtilis PB2;2:B.subtilis fmbj; 3:B.subtilis PB198;4:B.subtilis fmbR-2;5:B.amyloliquefaciens ES-2;6:B.subtilis fmbR; 7:P.polymyxa JSa-9;8:P.polymyxa JSa-9-81;9:B.subtilis fmb4;10:B.subtilis fmb2;11:B.subtilis fmb3 |
非核糖体肽、肽类抗生素以及细菌素都具有很强的抗真菌、抗细菌及抗病毒效果, 在抗菌领域发挥着重要作用。目前, 这种抗菌活性物质的研究集中在芽孢杆菌领域[4], 同时大量研究表明, 不同种属的芽孢杆菌可以合成不同或相同的抗菌物质。而同一种属细菌的不同菌株合成的抗菌物质也不尽相同, 如枯草芽孢杆菌的某些菌株只能合成释放Surfactin[23], 而有些菌株却能同时产生Surfactin、Fengycin和Iturin[24]。而且, 即使同一菌株产生的同一种抗菌活性物质也并非单一的、纯的化合物, 而是由几种结构类似的同系物组成的复杂化合物[25]。本研究中, 我们应用了更加简便快捷的方法, 通过PCR扩增技术对抗菌物质的编码调控基因进行检测, 从而初步判断该菌株是否具有产生抗菌物质的能力。
我们对11株检测菌株进行测定, 结果证实它们在不同程度上对多种真菌、细菌具有一定的抗菌效果, 但其具体的抗菌机制尚不清楚。
基于对抗菌物质编码基因的PCR扩增技术与序列测定, 本次检测结果从分子水平上验证了前期相关研究结果的可靠性。例如, B.subtilis fmbj菌株可以合成Surfactin、Fengycin、Bacillomycin D等抗菌脂肽[11, 16, 18], 同时针对它的全基因组测序结果也同样佐证了这一研究结果的正确性。而B.amyloliquefaciens ES-2菌株能够合成Surfactin、Fengycin和Iturin[12]; P.polymyxa JSa-9和P.polymyxa JSa-9-81两株菌株能够产生Polymyxin[21-22]; B.subtilis fmbR菌株可以合成Surfactin和Subtilosin A[13], 前期的研究成果与本次研究结果相互印证, 证实了基因检测方法在抗菌脂肽产生菌鉴定方面具有较强的可行性。同时, 通过克隆检测方法, 还在某些菌种中发现一些尚未报道的脂肽合成酶相关基因, 例如检测发现, B.subtilis fmbR具有Fengycin和Bacillomycin D的编码基因, B.amyloliquefaciens ES-2具有Bacillomycin D的合成调控基因等等, 推测它们可能具有合成此类抗菌物质的潜在能力。本研究证实了枯草芽孢杆菌、淀粉液化芽孢杆菌以及多黏类芽孢杆菌均具有许多抗菌脂肽生物合成所需要的编码基因, 推测它们均可以合成多种抗菌活性物质。同样证实了不同种属的菌株具有不同或相同的抗菌物质编码基因, 而同一种细菌的不同菌株也具有几种不同抗菌物质的基因。因此, 本次基因检测技术同样可以运用于寻找不同种属之间的同源性基因。
目前, 芽孢杆菌的抗菌脂肽用于生物防治领域已经引起了广泛关注, 然而抗菌脂肽类物质却因为其较高的生产成本, 在食品领域和化工领域的应用缺乏竞争力。抗菌脂肽未来的研究发展需要兼顾生产力与经济需求, 而基因检测这一快速、简单、可靠的抗菌脂肽产生菌鉴定方法的建立, 帮助我们更快地实现脂肽物质在农业、医药等众多领域的大范围生产使用。
| [1] | 翟少伟, 李剑, 史庆超. 抗菌脂肽Surfactin的抗菌活性及应用[J]. 动物营养学报, 2015, 27(5): 1333–1340. Zhai S Q, Li J, Shi Q C. Antimicrobial lipopeptide surfactin:antimicrobial activity and applications[J]. Chinese Journal of Animal Nutrition, 2015,27(5): 1333–1340. (in Chinese) |
| [2] | Naing K W, Yong S L, Xuan H N, et al. Isolation and characterization of an antimicrobial lipopeptide produced by Paenibacillus ehimensis MA2012[J]. Journal of Basic Microbiology, 2015,55(7): 857–868. DOI: 10.1002/jobm.v55.7 |
| [3] | Leclère V, Béchet M, Adam A, et al. Mycosubtilin overproduction by Bacillus subtilis BBG100 enhances the organism's antagonistic and biocontrol activities[J]. Applied and Environmental Microbiology, 2005,71(8): 4577–4584. DOI: 10.1128/AEM.71.8.4577-4584.2005 |
| [4] | 陈萍, 冯芬, 杨恬然, 等. 脂肽类抗生素及其作用机制[J]. 微生物学杂志, 2015, 35(5): 89–93. Chen P, Feng F, Yang T R, et al. Lipopeptide antibiotics and its action mechanism[J]. Journal of Microbiology, 2015,35(5): 89–93. (in Chinese) |
| [5] | 陈哲, 黄静, 赵佳, 等. 解淀粉芽孢杆菌抑菌机制的研究进展[J]. 生物技术通报, 2015, 31(6): 37–41. Chen Z, Huang J, Zhao J, et al. Research advances on antibacterial mechanism of Bacillus amyloliquefaciens[J]. Biotechnology Bulletin, 2015,31(6): 37–41. (in Chinese) |
| [6] | 姬婧媛, 杨洁, 高小宁, 等. 植物内生枯草芽孢杆菌E1R-j脂肽类化合物的分离鉴定及抑菌作用[J]. 农药学学报, 2015(2): 172–178. Ji J Y, Yang J, Gao X N, et al. Isolation and identification of lipopeptides produced by endophytic bacteria Bacillus subtilis E1R-j and its anti-fungal mechanism studies[J]. Chinese Journal of Pesticide Science, 2015(2): 172–178. (in Chinese) |
| [7] | 李全胜, 谢宗铭, 张国丽, 等. 棉花黄萎病拮抗芽孢杆菌S12的筛选鉴定及拮抗机制的分析[J]. 南京农业大学学报, 2015, 38(3): 402–408. Li Q S, Xie Z M, Zhang G L, et al. Screening and identification of antagonistic spore bacterium S12 against cotton Verticillium wilt and preliminary study on its antagonistic mechanisms[J]. Journal of Nanjing Agricultural University, 2015,38(3): 402–408. DOI: 10.7685/j.issn.1000-2030.2015.03.008 (in Chinese) |
| [8] | 韦巧婕, 郑新艳, 邓开英, 等. 黄瓜枯萎病拮抗菌的筛选鉴定及其生物防效[J]. 南京农业大学学报, 2013, 36(1): 40–46. Wei Q J, Zheng X Y, Deng K Y, et al. Screening and identification of antagonistic Bacillus vallismortis B against cucumber Fusarium wilt and its biological effect[J]. Journal of Nanjing Agricultural University, 2013,36(1): 40–46. DOI: 10.7685/j.issn.1000-2030.2013.01.008 (in Chinese) |
| [9] | 侯红漫, 靳艳, 金美芳, 等. 环脂肽类生物表面活性剂结构、功能及生物合成[J]. 微生物学通报, 2006, 33(5): 122–128. Hou H M, Jin Y, Jin M F, et al. Structure function and biosynthesis of cyclic lipopeptidic biosurfactant[J]. Microbiology China, 2006,33(5): 122–128. (in Chinese) |
| [10] | 刘丽霞, 高玲, 别小妹, 等. Bacillus subtilis 168菌株产surfactin改造[J]. 南京农业大学学报, 2014, 37(6): 97–102. Liu L X, Gao L, Bie X M, et al. Reconstruction of Bacillus subtilis 168 to produce surfactin[J]. Journal of Nanjing Agricultural University, 2014,37(6): 97–102. DOI: 10.7685/j.issn.1000-2030.2014.06.014 (in Chinese) |
| [11] | 别小妹, 吕凤霞, 陆兆新, 等. Bacillus subtilis fmbJ脂肽类抗菌物质的分离和鉴定[J]. 生物工程学报, 2006, 22(4): 328–333. Bie X M, Lü F X, Lu Z X, et al. Isolation and identification of lipopeptides produced by Bacillus subtilis fmbJ[J]. Chinese Journal of Biotechno-logy, 2006,22(4): 328–333. (in Chinese) |
| [12] | Sun L J, Lu Z X, Bie X M, et al. Isolation and characterization of a co-producer of fengycins and surfactins, endophytic Bacillus amyloliquefaciens ES-2, from Scutellaria baicalensis Georgi[J]. World Journal of Microbiology and Biotechnology, 2006,22(12): 1259–1266. DOI: 10.1007/s11274-006-9170-0 |
| [13] | 别小妹, 陆兆新, 吕凤霞, 等. Bacillus subtilis fmbR抗菌物质的分离和鉴定[J]. 中国农业科学, 2006, 39(11): 2327–2334. Bie X M, Lu Z X, Lü F X, et al. Isolation and identification of the antimicrobiaI substance produced by Bacillus subtilis fmbR[J]. Scientia Agricultura Sinica, 2006,39(11): 2327–2334. (in Chinese) |
| [14] | Hiradate S, Yoshida S, Sugie H, et al. Mulberry anthracnose antagonists(iturins)produced by Bacillus amyloliquefaciens RC-2[J]. Phytochemistry, 2002,61(6): 693–698. DOI: 10.1016/S0031-9422(02)00365-5 |
| [15] | Moyne A L, Cleveland T E, Tuzun S. Molecular characterization and analysis of the operon encoding the antifungal lipopeptide bacillomycin D[J]. FEMS Microbiology Letters, 2004,234(1): 43–49. DOI: 10.1111/fml.2004.234.issue-1 |
| [16] | Gong Q W, Zhang C, Lu F X, et al. Identification of bacillomycin D from Bacillus subtilis fmbJ and its inhibition effects against Aspergillus flavus[J]. Food Control, 2014,36(1): 8–14. DOI: 10.1016/j.foodcont.2013.07.034 |
| [17] | Chen X H, Koumoutsi A, Scholz R, et al. Genome analysis of Bacillus amyloliquefaciens FZB42 reveals its potential for biocontrol of plant pathogens[J]. Journal of Biotechnology, 2009,140(1): 27–37. |
| [18] | Qian S Q, Lu H D, Meng P P, et al. Effect of inulin on efficient production and regulatory biosynthesis of bacillomycin D in Bacillus subtilis fmbJ[J]. Bioresource Technology, 2015,179: 260–267. DOI: 10.1016/j.biortech.2014.11.086 |
| [19] | Stein T, Düsterhus S, Stroh A, et al. Subtilosin production by two Bacillus subtilis subspecies and variance of the sbo-alb cluster[J]. Applied and Environmental Microbiology, 2004,70(4): 2349–2353. DOI: 10.1128/AEM.70.4.2349-2353.2004 |
| [20] | Choi S K, Park S Y, Kim R, et al. Identification of a polymyxin synthetase gene cluster of Paenibacillus polymyxa and heterologous expression of the gene in Bacillus subtilis[J]. Journal of Bacteriology, 2009,191(10): 3350–3358. DOI: 10.1128/JB.01728-08 |
| [21] | Deng Y, Lu Z X, Lu F X, et al. Identification of LI-F type antibiotics and di-n-butyl phthalate produced by Paenibacillus polymyxa[J]. Journal of Microbiological Methods, 2011,85(3): 175–182. DOI: 10.1016/j.mimet.2011.02.013 |
| [22] | Deng Y, Lu Z X, Bi H, et al. Isolation and characterization of peptide antibiotics LI-F04 and polymyxin B 6 produced by Paenibacillus polymyxa strain JSa-9[J]. Peptides, 2011,32(9): 1917–1923. DOI: 10.1016/j.peptides.2011.08.004 |




