文章信息
- 刘红蕊, 吴诚诚, 单衍可, 谢青云, 邢刚, 雷静, 施志玉, 孙海凤, 刘斐
- LIU Hongrui, WU Chengcheng, SHAN Yanke, XIE Qingyun, XING Gang, LEI Jing, SHI Zhiyu, SUN Haifeng, LIU Fei
- RNA解螺旋酶A表达系统的探究及其动力学研究
- Investigation of expression system and dynamics properties of RNA helicase A
- 南京农业大学学报, 2016, 39(5): 831-837
- Journal of Nanjing Agricultural University, 2016, 39(5): 831-837.
- http://dx.doi.org/10.7685/jnau.201511048
-
文章历史
- 收稿日期: 2015-11-28
RNA解螺旋酶是一类利用NTP供能使双链RNA解旋或使RNA-蛋白质复合体结构发生重组的酶类, 对确保RNA分子的正确折叠以及其特定的二、三级结构的保持和修饰必不可少[1]。研究发现, RNA解螺旋酶存在于所有有细胞生物以及许多病毒中[2-4]。
目前研究结果表明, RNA解螺旋酶不仅可以由病毒核酸自身编码合成, 也可以在宿主的细胞内经过编码合成。这些RNA解螺旋酶作为病毒RNA合成过程中起关键作用的酶, 除参与了病毒的复制之外, 还可刺激宿主细胞诱导宿主产生抗病毒效应[2, 5-6], 但其作用机制尚不清楚。
解螺旋酶根据保守区氨基酸序列可分为6个超家族(SF1-SF6)[7], RNA解螺旋酶A(RNA helicase A, RHA), 又称DHX9、NDHⅡ[8], 属于SF2。RHA是由哺乳动物细胞编码翻译的一类酶, 在哺乳动物胚胎的形成和发育过程中发挥着重要的作用。目前研究已经证实RHA参与不同的细胞功能, 包括转录、翻译、RNA干扰途径以及先天性免疫反应[9-11]。RHA还可以促进HIV-1等一些病毒的复制[12-13], 改变局部pH值可能对于病毒的复制产生抑制作用。RHA与NPH-Ⅱ属于SF2家族中的DExH亚家族, 均有3′-tailed依赖的解螺旋极性[14-15]。NPH-Ⅱ解旋活性对温度不敏感, 而同样属于SF2家族的DED1, 位于DEAD-box亚家族, 其解旋活性受温度影响较大[16]。因此, 推测不同家族的RNA解螺旋酶对温度可能有不同的敏感度。
本文在对RHA全长CDs区进行克隆的基础上, 分别构建了原核与真核表达系统, 通过对两种表达系统的比较, 最终对真核表达系统表达纯化的RHA蛋白的解RNA双螺旋的活性进行了研究。通过对在不同温度、pH值条件下RHA解双螺旋活性变化的探究, 为进一步深入研究RHA作用机制奠定了基础。
1 材料与方法 1.1 试验材料 1.1.1 质粒、菌种、细胞含全长RHA序列的质粒(Thermo, 美国), pET-28a(笔者所在实验室保存), pColdⅠ和Rosetta2感受态细胞(南京农业大学茅翔教授馈赠), pFastBac Dual(南京农业大学姜平教授馈赠), BL21感受态细胞(TaKaRa, 大连), BL21star(DE3)(Invitrogen, 美国), DH10 Bac(博迈德生物有限公司, 北京), sf9细胞(江苏省农业科学院国家兽用生物技术工程技术研究中心馈赠)。
1.1.2 工具酶与试剂DNALigation Kit、BamHⅠ、EcoRⅠ、NdeⅠ、Prime STAR® HS DNA Polymerase、RNAase Recombinant Inhibitor(RRI)(TaKaRa, 日本), 质粒提取试剂盒(Omega, 美国), Cellfectin Ⅱ Reagent(Invitrogen, 美国), DHX9抗体(ABGENT, 美国), HRP标记羊抗兔IgG(鼎国生物技术有限公司, 北京)。
1.2 试验方法 1.2.1 原核表达载体的构建根据全长RHA基因序列设计引物F1/R1(表 1)。以含全长RHA基因的质粒为模板, 进行PCR。PCR程序为:98 ℃ 10 s; 55 ℃ 15 s, 72 ℃ 4 min, 35个循环; 最后72 ℃延伸10 min。扩增产物经10 g·L-1琼脂糖凝胶电泳分析, 利用分子克隆技术将目的基因连接至pET-28a、pCold-Ⅰ载体上, 得到重组质粒RHA-pET-28a。用内切酶NdeⅠ、EcoRⅠ对重组质粒RHA-pET-28a、RHA-pCold-Ⅰ进行双酶切和测序鉴定。
| 序列名称 Sequence name |
引物(对)序列(5′→3′) Primer(pairs)sequence |
酶切位点 Restriction site |
| F1/R1 | GGGAATTCCATATGATGGGTGACGTAAAAATTTTCTG/CGGGAATTCCTTAATAGCCGCCACCTCCTCTTCCCTG | NdeⅠ/EcoRⅠ |
| F2/R2 | CGGGATCCGCCATGGGCCATCATCATCATCATCATATGGGTGACGTTAAAAATTTTCTG/CGGAATTCTTAATAGCCGCCACCTCCTCTTCCCTG | BamHⅠ/EcoRⅠ |
| F3/R3 | CCCAGTCACGACGTTGTAAAACG/AGCGGATAACAATTTCACACAGG | |
| Top strand | 6FAM-GCGUCGUAUCGAUCUGGUC | |
| Bottom strand | GACCAGAUCGAUACGACGCUAGGACAUGCCACAGACGUACUAACAGCAUCAAUG |
将上述鉴定正确的重组质粒RHA-pET-28a转化至BL21 star(DE3)中, 重组RHA-pCold-Ⅰ转化至BL21、Rosetta2中。加入IPTG(终浓度为0.5 mmol·L-1), BL21 star(DE3)于28 ℃振荡培养, BL21、Rosetta2于15 ℃振荡培养。
收集表达菌体, 用500 μL lysis buffer重悬后进行超声破碎。12 000 r·min-1离心10 min, 取上清液, 进行80 g·L-1 SDS-PAGE凝胶电泳, 将经SDS-PAG凝胶电泳后的蛋白转印至PVDF膜上, 用10%脱脂乳封闭后依次孵育DHX9抗体和HRP标记的羊抗兔二抗, 经化学发光显色后观察结果。
1.2.3 重组杆状病毒表达系统的构建根据全长RHA基因序列设计引物F2/R2(表 1)。按照1.2.1节方法构建重组杆状质粒RHA-pFastBac Dual。经酶切和测序鉴定正确的RHA-pFastBac Dual转化到DH10Bac感受态细胞中, 经过3次蓝白斑筛选后挑选出白色菌落。
合成杆状质粒通用引物F3/R3(表 1)。取F2/R2引物和F3/R3引物分别以野生型杆状病毒穿梭质粒和重组杆状质粒为模板, 进行PCR反应。将鉴定为阳性的重组杆状质粒利用CellfectinⅡ Reagent转染至sf9昆虫细胞, 感染72 h后收集细胞, 裂解细胞取上清液, 进行SDS-PAG电泳, 然后按照1.2.2节方法进行Western blot分析。
1.2.4 RHA蛋白活性检测设计一对RNA序列(Top strand/Bottom strand, 表 1), 作为RHA的解螺旋底物, 其中Top strand全部序列可以和Bottom strand部分序列退火为双链, 形成具有3′-tailed结构的双链RNA。6FAM为荧光分子。
构建体外解螺旋反应体系(10 μL):30 mmol·L-1 Tris(pH7.5)、0.1 mg·mL-1 BSA、2 mmol·L-1 DTT、2 U·μL-1重组RNA水解酶抑制剂(recombinant RNA inhibitor, RRI)、25 mmol·L-1 NaCl、3 mmol·L-1 ATP、3 mmol·L-1 MgCl2、4 nmol·L-1 dsRNA、270 nmol·L-1 RHA。将上述混合液后置于37 ℃反应40 min, 与等体积的2×stop buffer(50 mmol·L-1 EDTA、1% SDS、0.01%溴酚蓝、0.01%二甲苯青、10%甘油)混合后置于冰上, 经120 g·L-1 PAGE电泳2 h后, 用Typhoon多功能激光扫描仪成像分析。
1.2.5 trap RNA浓度的滴定为避免解开的双链RNA(Top strand/Bottom strand)自动退火为双链, 本试验设计一段trap RNA序列。该序列与Top strand序列相同, 但无荧光分子(6FAM)标记, 可与荧光标记的Top strand序列竞争结合Bottom strand序列。本试验分别采用2、4、8 nmol·L-1 trap RNA与4 nmol·L-1 ds RNA(共10 μL)进行反应。95 ℃作用3 min, 用120 g·L-1 PAGE进行电泳分析, 采用Image J对结果中RNA进行定量。
1.2.6 温度对RHA活性的影响构建体外解螺旋反应体系(90 μL):30 mmol·L-1 Tris(pH7.5)、0.1 mg·mL-1 BSA、2 mmol·L-1 DTT、2 U·μL-1 RRI、25 mmol·L-1 NaCl、3 mmol·L-1 ATP、3 mmol·L-1 MgCl2、4 nmol·L-1 dsRNA、50 nmol·L-1 RHA、2 nmol·L-1 trap RNA。其中, 除ATP、trap RNA外其余成分混合后分别置于19 ℃和28 ℃反应5 min; ATP和trap RNA混合后分别置于19 ℃和28 ℃作用5 min。然后两部分混匀后分别置于19 ℃和28 ℃, 且分别于混合后0 s、15 s、45 s、1.5 min、5 min、10 min、30 min、60 min迅速取反应液10 μL与10 μL 2×stop buffer混合, 冰上放置。经120 g·L-1 PAGE电泳并经Typhoon成像后, 利用Image J进行RNA定量, 使用KaleidaGraph进行一级反应方程(A=A0-A0·e-k·t, A为反应幅度, A0给定初始值为1, k为速率常数, t为反应时间)拟合, 可得到速率常数和反应幅度。
1.2.7 pH对RHA活性的影响按照1.2.6节构建pH值分别为6.5、7.5、8.0、8.5的体外解螺旋反应体系。在19 ℃条件下, 按照1.2.6节方法处理样品并进行相应的分析。
2 结果与分析 2.1 RHA的原核表达结果鉴定HeLa细胞可以表达RHA蛋白, 提取HeLa细胞表达的RHA蛋白作为对照。含有重组质粒的菌体经IPTG诱导后, 表达产物进行SDS-PAGE和Western blot鉴定。利用SDS-PAGE分析RHA的表达, 不能确定其表达量是偏低还是无表达(图 1-A, 图 2-A)。利用Western blot技术, 进一步验证RHA在大肠杆菌中的表达情况。BL21 star(DE3)在28 ℃下经诱导的表达产物中未检测到RHA条带(图 1-B), BL21和Rosetta2在15 ℃下经诱导表达后能检测到目的蛋白RHA(图 2-B), 说明RHA可以在BL21和Rosetta2中在15 ℃诱导表达。大量培养BL21和Rosetta2后进行蛋白纯化, 但只有很少量的蛋白纯化出来, 所以我们还需要表达量高的系统来表达RHA蛋白。
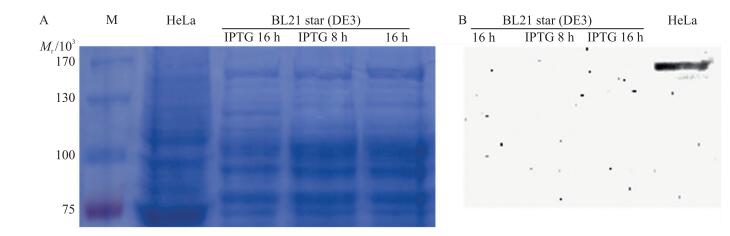
|
图 1 RHA蛋白在BL21 star(DE3)中表达SDS-PAGE电泳图(A)和Western blot分析(B) Figure 1 The analysis of expression of RHA proteinin BL21 star(DE3)by SDS-PAGE(A)and Western blot(B) |
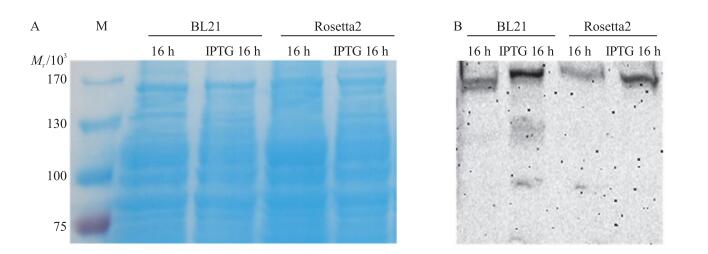
|
图 2 15 ℃时RHA蛋白在BL21和Rosetta2中表达SDS-PAGE电泳图(A)和Western blot分析(B) Figure 2 The analysis of expression of RHA protein in BL21 and Rosetta2 at 15 ℃ by SDS-PAGE(A)and Western blot(B) |
挑取重复3次筛选出的白色菌落, 提取重组杆粒, 进行PCR分析。将PCR鉴定为阳性的重组杆粒转染至sf9细胞中进行表达并对表达的蛋白进行分析和纯化。结果显示:以野生型杆粒为模板, F3/R3引物扩增, 在300 bp附近有单一条带; 以重组杆粒为模板, F3/R3引物扩增, 在7 000 bp处有单一条带; 以重组杆粒为模板, F2/R2引物扩增, 在4 000 bp附近有单一条带(图 3)。以上结果表明含有RHA的重组杆状质粒构建成功。转染后的sf9细胞经破碎后, 利用Western blot可检测到RHA条带(图 4)。经过Ni-NTA-agarose和Capto DEAE Sepharose纯化并经考马斯亮蓝染色后, 检测到单一相对分子质量为140×103左右的条带(图 4)。
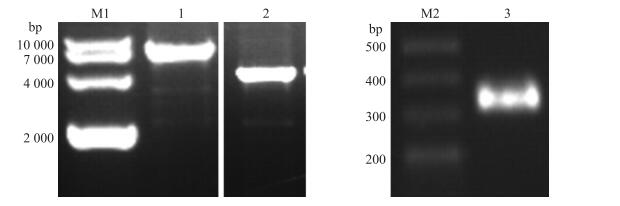
|
图 3 重组杆粒的PCR分析结果 Figure 3 Analyzing recombinant bacmid DNA by PCR M1:DNA标准品DL10000;M2:DNA标准品DL500;1:重组杆粒为模板, F3/R3为引物; 2:重组杆粒为模板, F2/R2为引物; 3:空杆粒为模板, F3/R3为引物 M1:DL10000 DNA marker; M2:DL500 DNA marker; 1:Recombinant bacmid DNA and F3/R3 primer; 2:Recombinant bacmid DNA and F2/R2 primer; 3:Bacmid alone and F3/R3 primer |
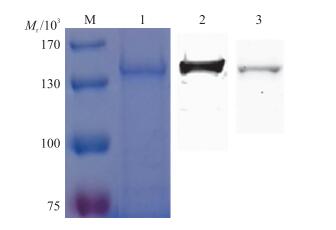
|
图 4 RHA在sf9细胞中表达的分析 Figure 4 Analyzing RHA expression in sf9 M:蛋白质标准; 1:纯化后RHA的SDS-PAGE分析; 2:RHA的Western blot分析; 3:HeLa细胞中RHA(对照) M:Protein maker; 1:SDS-PAGE analysis for purified RHA; 2:Analyze RHA by Western blot; 3:RHA expression in HeLa as control |
对利用真核sf9细胞表达的RHA蛋白进行活性检测, 如图 5所示:有RHA和ATP共同存在的体系在反应40 min后有单链条带产生; 只有RHA或只有ATP的体系在反应40 min后不能产生单链条带; 双链RNA经过95 ℃处理后会变性生成单链条带, 具有对照作用。以上结果表明, 纯化得到的RHA具有ATP依赖的解螺旋活性。
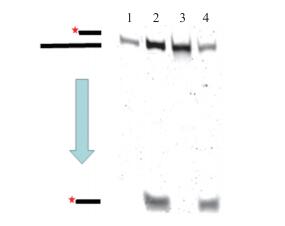
|
图 5 RHA解螺旋活性鉴定 Figure 5 Unwinding activity assay of RHA 1:有RHA和ATP; 2:有RHA无ATP; 3:无RHA有ATP; 4:95 ℃处理对照(黑色线段表示RNA链; 五角星代表 6FAM) 1:Added RHA and ATP; 2:Added RHA, non-ATP; 3:Added ATP, non-RHA; 4:95 ℃ control(RNA, black lines; 6FAM, pentangle) |
将构建的体外解旋体系分别置于19 ℃和28 ℃进行反应。扫描成像后, 对RNA条带进行定量, 然后利用一级反应方程进行拟合(图 6-A)。结果显示, 在50和100 nmol·L-1 2个高浓度下, 速率常数随温度的升高明显增加(图 6-B); 在10、50和100 nmol·L-1 3个浓度下, 反应幅度随温度的升高均变大(图 6-C)。上述结果表明, 将RHA分别置于19~28 ℃时, 在较高温度(28 ℃)条件下RHA解螺旋活性较高。
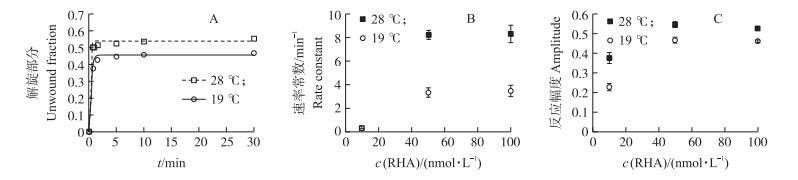
|
图 6 温度对RHA活性的影响 Figure 6 The effect of temperature on RHA activity A:拟合结果; B:速率常数趋势图; C:反应幅度趋势图 A:The result of fitting; B:The trend of rate constant with different temperature; C:The trend of amplitude with different temperature |
建立2个除反应pH外其他成分完全相同的反应体系, 置于19 ℃反应。扫描成像后对RNA进行定量, 利用一级反应方程进行拟合(图 7-A)。结果显示, pH值在6.5~8.0的范围内, 随着pH值的升高, 速率常数变大, RHA活性增加; 但如果pH值继续升高, 速率常数变小, RHA活性降低(图 7-B); 此外, 4个pH值下的反应幅度相差不是很大(图 7-C), 表明在pH值为6.5~8.0的范围内, 足够的反应时间可以达到几乎相同的反应幅度。表明pH值的变化并未使RHA变性失活, 可能在RHA所带电荷量、溶解性方面发挥作用而降低RHA的活性。
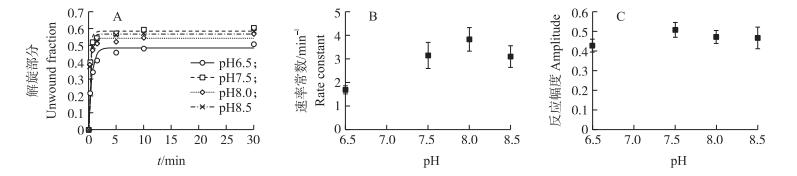
|
图 7 pH值对RHA活性的影响 Figure 7 RHA activity affected by pH value A:拟合结果; B:速率常数趋势图; C:反应幅度趋势图。 A:The result of fitting; B:The trend of rate constant with different pH value; C:The trend of amplitude with different pH value. |
pColdⅠ利用了大肠杆菌的低温表达基因(冷休克基因), 当培养温度切换到低温时, 大肠杆菌暂时停止生长, 大部分内源性蛋白质表达减少, 但是一类称为冷休克蛋白的蛋白质却能被特异性诱导表达[17]。但不同物种有不同的密码子偏爱性, 如果外源蛋白中含大量大肠杆菌的稀有密码子, 特别当这些稀有密码子呈连续分布的时候, 就会导致蛋白表达量极低, 或者翻译提前终止[18]。大肠杆菌稀有密码子为CUA、AGG、AUA、AGA、CCC和GGA, RHA基因上对应的序列数分别为8、9、11、25、15、47。其中, AGA、GGA含量偏高, 尤其是GGA的含量最高, 且在RHA全长序列里多处是连续存在的。
蛋白质序列亲水性越高, 可溶性表达的可能性越大。用DNA star(Protean)对RHA蛋白序列进行分析, 480~563处的氨基酸形成疏水性区域, 640~1 050处的氨基酸随机形成间隔疏水性区域, 且分别在492、661、813、996处氨基酸形成高度疏水区。
综合密码子的稀有性及疏水性, 可知RHA在大肠杆菌中是很难大量可溶性表达的, 证明重组载体RHA-pColdⅠ在Rosetta2和BL21中可微量表达。
3.2 RHA机制研究分析本试验使用6FAM荧光分子标记双链RNA中的一条链, 使用激光扫描仪采集信号。当荧光单链与另外一条链退火形成双链时, 由于分子质量的改变, 导致在相同时间内迁移的距离不同, 从而可区分出单双链的位置。由于荧光分子较同位素灵敏度低, 需使用较高浓度的RNA以采集到足量信号, 但较高浓度的双链RNA在解旋后自动退火的概率变大, 对解旋数据采集造成影响。本试验加入trap RNA(序列与荧光标记的RNA相同, 无荧光分子标记)与荧光RNA竞争[19], 使解旋产生的单链荧光RNA的量更接近实际值。通过对trap RNA浓度进行滴定以确定正确的trap RNA浓度。
温度升高, 分子碰撞率增大, 活化能降低, 速率增大[20]。与RHA具有相同解旋极性的NPH-Ⅱ解螺旋酶, 其活性受温度影响较小。无解旋极性的Ded1解螺旋酶, 其活性受温度影响较大。针对RHA解螺旋酶, 对19和28 ℃两个温度下的数据进行分析, 温度升高, 速率常数变大, 且在50和100 nmol·L-1 2个高浓度下效果更明显。本试验系统地证明了具有3-tailed解旋极性的RHA活性受温度影响较大。选用19 ℃为后续试验的反应温度, 改变反应pH值, 测定速率常数、反应幅度。
蛋白质多肽链上含有许多极性基团, 它们具有高度的亲水性, 在溶液中与水分子相互作用形成一层水化膜。蛋白质在低于等电点(pI)的缓冲液中带正电, 在高于pI的缓冲液中带负电。当pH值过高时, 溶液中的氢氧根偏多, 与溶液中水分子作用增强, 破坏蛋白质的水化膜, 降低其稳定性, 进而影响其活性。但如果pH值过高会使蛋白变性失活。RHA的pI为6.4, 试验结果表明pH值在6.5~8.0范围内, 随着pH值的升高, RHA活性增加。但随着pH值继续升高, RHA活性出现降低趋势, 推测pH值过高会导致蛋白质稳定性降低从而降低活性, 引起RHA活性降低的原因还有待进一步探究。
| [1] | Byrd A K, Raney K D. Superfamily 2 helicases[J]. Front Biosci, 2013,17(2070-2088). |
| [2] | Frick D N. The Hepatitis C virus NS3 protein:a model RNA helicase and potential drug target[J]. [J]Current Issues in Molecular Biology, 2007,9: 1–20. |
| [3] | Jankowsky E. RNA helicases at work:binding and rearranging[J]. Trends in Biochemical Sciences, 2011,36(1): 19–29. DOI: 10.1016/j.tibs.2010.07.008 |
| [4] | Ranji A, Boris-Lawrie K. RNA helicases:emerging roles in viral replication and the host innate response[J]. RNA Biol, 2010,7(6): 775–787. DOI: 10.4161/rna.7.6.14249 |
| [5] | Courtney W, Michael G. Recognition of viruses by cytoplasmic sensors[J]. Current Opinion in Immunology, 2010,22(1): 41–47. DOI: 10.1016/j.coi.2009.12.003 |
| [6] | Kwong A D, B Govinda R, Kuan-Teh J. Viral and cellular RNA helicases as antiviral targets[J]. Dressnature Reviews Drug Discovery, 2005,4(10): 845–853. DOI: 10.1038/nrd1853 |
| [7] | Fairman-Williams M E, Guenther U P, Jankowsky E. SF1 and SF2 helicases:family matters[J]. Current Opinion in Structural Biology, 2010,20(3): 313–324. DOI: 10.1016/j.sbi.2010.03.011 |
| [8] | Koh H R, Xing L, Kleiman L, et al. Repetitive RNA unwinding by RNA helicase A facilitates RNA annealing[J]. Nucleic Acids Res, 2014,42(13): 8556–8564. DOI: 10.1093/nar/gku523 |
| [9] | Lee C G, Soares V D, newberger C, et al. RNA helicase A is essential for normal gastrulation[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998,95(23): 13709–13713. DOI: 10.1073/pnas.95.23.13709 |
| [10] | Robb G B, Rana T M. RNA Helicase A interacts with RISC in human cells and functions in RISC loading[J]. Molecular Cell, 2007,26(4): 523–537. DOI: 10.1016/j.molcel.2007.04.016 |
| [11] | Hartman T R, Qian S, Bolinger C, et al. RNA helicase A is necessary for translation of selected messenger RNAs[J]. Nature Structural & Molecular Biology, 2006,13(6): 509–516. |
| [12] | Jeang K T, Yedavalli V. Role of RNA helicases in HIV-1 replication[J]. Nucleic Acids Res, 2006,34(15): 4198–4205. DOI: 10.1093/nar/gkl398 |
| [13] | Li X, Meijuan N, Lawrence K. Role of the OB-fold of RNA helicase A in the synthesis of HIV-1 RNA[J]. Biochimica et Biophysica Acta(BBA), 2014,1839(11): 1069–1078. DOI: 10.1016/j.bbagrm.2014.08.008 |
| [14] | Li X, Xia Z, Niu M, et al. Helicase associated 2 domain is essential for helicase activity of RNA helicase A[J]. Biochimica et Biophysica Acta, 2014,1844(10): 1757–1764. DOI: 10.1016/j.bbapap.2014.07.001 |
| [15] | Jankowsky E, Gross C H, Shuman S, et al. The DExH protein NPH-Ⅱ is a processive and directional motor for unwinding RNA[J]. Nature, 2000,403(6768): 447–451. DOI: 10.1038/35000239 |
| [16] | Yang Q, Jankowsky E, Yang Q, et al. The DEAD-box protein Ded1 unwinds RNA duplexes by a mode distinct from translocating helicases[J]. Nature Structural and Molecular Biology, 2006,13(11): 981–986. DOI: 10.1038/nsmb1165 |
| [17] | 周鹰, 孙金霞, 王运刚, 等. 粉尘螨变应原第16组分基因克隆及表达[J]. 中国病原生物学杂志, 2012, 7(7): 519–522. Zhou Y, Sun J X, Wang Y G, et al. Cloning and expression of the group 16 allergen of Dermatophagoides farinae[J]. Journal of Pathogen Biology, 2012,7(7): 519–522. (in Chinese) |
| [18] | 蒋岚, 杨永华, 龚毅, 等. 稀有密码子对proUK基因在大肠杆菌中高表达的影响[J]. 生物工程学报, 1999, 15(1): 64–67. Jiang L, Yang Y H, Gong Y, et al. The rare coden effects the overexpression of human proUK gene in Escherichia coli[J]. Chinese Journal of Biotechnology, 1999,15(1): 64–67. (in Chinese) |
| [19] | Putnam A, Jankowsky E. Analysis of duplex unwinding by RNA helicases using stopped-flow fluorescence spectroscopy[J]. Method Enzymol, 2012,511: 1–27. DOI: 10.1016/B978-0-12-396546-2.00001-2 |
| [20] | 王利侠. 碰撞理论和过渡态理论[J]. 中学化学, 2014(9): 20–21. Wang L X. Collision theory and transition state theory[J]. Middle School Chemistry, 2014(9): 20–21. (in Chinese) |




