文章信息
- 董丽丽, 龚凌燕, 陈磊, 涂佳丽, 张水明
- DONG Lili, GONG Lingyan, CHEN Lei, TU Jiali, ZHANG Shuiming
- 石榴肉桂酰辅酶A还原酶基因的克隆与表达分析
- Cloning and expression analysis of PgCCR from pomegranate
- 南京农业大学学报, 2016, 39(5): 747-753
- Journal of Nanjing Agricultural University, 2016, 39(5): 747-753.
- http://dx.doi.org/10.7685/jnau.201602011
-
文章历史
- 收稿日期: 2016-02-15
石榴(Punica granatum L.)为石榴科石榴属多年生落叶果树, 在阿富汗、中国、伊朗、印度、日本、地中海国家、俄罗斯、美国等地广泛栽培[1]。果实富含多种维生素、糖类、有机酸、矿物质和抗氧化剂以及多酚类物质[2], 因此具有较高的营养价值和保健功能。目前市场上的石榴多以硬籽品种为主, 可食用部分仅占果实总质量的45%~52%[3]。而软籽品种核软可食, 可食率高, 是珍贵的石榴种质资源。研究发现, 籽粒中木质素含量与籽粒硬度具有一定相关性[4]。因此, 降低石榴籽粒中的木质素含量, 能够有效降低籽粒的硬度, 提高石榴的可食率。
木质素在植物体内的生物合成主要包括莽草酸途径、苯丙氨酸途径及木质素特异合成途径。肉桂酰辅酶A还原酶(cinnamoyl-CoA reductase, CCR)是木质素特异途径的第一个关键酶, 能够催化羟基肉桂酸CoA酯的还原反应, 生成相应的肉桂醛(主要将阿魏酰辅酶A催化成松柏醛)[5]。目前认为CCR可能对木质素生物合成途径的碳流具有潜在的调控作用[6], 因此在木质素的合成途径中起着重要作用。随着部分突变体的鉴定以及基因克隆、转基因、RNAi等分子生物学技术的发展, CCR基因的功能研究逐步深入, 并演变成木质素合成途径研究的一个热点。CCR基因首先在桉树[7]中被鉴定, 后来陆续在杨树[8]、烟草[9]、玉米[10]、拟南芥[11]、黑麦草[12]、番茄[13]、丹参[14]、象草[15]等物种中被鉴定。在CCR下调的烟草株系中, 木质素含量下降50%[9, 16]。敲除拟南芥CCR 1 能够导致木质素含量的降低[17-19]。转基因杨树(Populus tremula×Populus alba)中CCR基因的下调导致木质素含量下降50%[20]。玉米CCR 1 基因的突变同样导致木质素含量的下降和结构的改变[21]。以上研究说明改变CCR基因的表达水平能够引起木质素含量和组成的变化。因此, 调节CCR基因以调控CCR蛋白活性被认为是降低木质素合成以达到改良植物品质的最重要途径之一[22]。
本研究从石榴中克隆了CCR基因的同源基因PgCCR, 并采用实时荧光定量PCR对该基因在不同石榴品种、组织和籽粒发育阶段的表达水平进行检测, 旨在为进一步研究PgCCR的分子生物学功能奠定基础, 也为软籽石榴的新品种培育提供了候选基因。
1 材料与方法 1.1 试验材料石榴品种‘红玉石籽’‘会理软籽’和‘突尼斯软籽’均采自安徽农业大学农业园石榴种质资源圃。‘红玉石籽’由‘玉石籽’变异得到, 核硬度为3.31 kg·cm-2, 属软籽, 可食率达55.7%;其果实扁圆且具4~5棱, 果皮表面光滑, 具深红色, 果皮厚度约0.32 cm; 籽粒顶端淡红、色泽淡黄[23]。采集长势相似且成熟度一致的‘红玉石籽’‘会理软籽’和‘突尼斯软籽’的果实带回实验室, 剥离出籽粒, 去除假种皮、种仁, 仅留下种皮部分, 浸于液氮中后贮存于-80 ℃冰箱中备用。在多株‘红玉石籽’上采集花、嫩茎、叶片, 浸于液氮中带回实验室, 贮存于-80 ℃冰箱中备用。分别采集开花后20、40、60、80、100和120 d的‘红玉石籽’果实带回实验室, 同理去除假种皮、种仁, 仅留下种皮部分贮存于-80 ℃冰箱中备用。
1.2 基因全长序列的克隆以去除假种皮、种仁后的‘红玉石籽’的种皮部分为材料, 参照王壮伟等[24]的方法提取RNA, 以去除DNA后的RNA为模板, 使用MMLV(TaKaRa)反转录酶反转录合成cDNA。将从石榴籽粒EST库中获得的部分序列作为中间片段, 并以此设计引物, 使用SMARTerTM RACE cDNA Amplification Kit(Clontech)分别获得5′和3′端序列。将3部分序列进行拼接, 最终获得基因的全长序列。使用PrimeSTAR HS(TaKaRa)高保真聚合酶扩增目的基因, 加A反应后连接到pGEM-Teasy载体, 由上海生工生物工程公司进行测序。所用引物见表 1。PCR扩增反应程序为:95 ℃ 5 min; 95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 2 min, 34个循环; 72 ℃ 5 min, 12 ℃保存。
| 引物Primers | 序列(5′→3′)Sequence | 用途Usage |
| UPM Long | CTAATACGACTCACTATAGGGCAA- | RACE通用引物 |
| GCAGTGGTATCAACGCAGAGT | RACE universal primer | |
| NUP | AAGCAGTGGTATCAACGCAGAGT | RACE巢式引物RACE nested primer |
| CCR-PF | GTGAACCCAGTGCTGGTGCT | PgCCR中间片段扩增 |
| CCR-PR | ATGGGGTACTCGGGGAAGAA | Amplification for internal fragment of PgCCR |
| CCR-5′GSP1 | GAAGGACGCTCTCGGCGCAGAGGT | PgCCR 5′端扩增 |
| CCR-5′GSP2 | GAGGGGGTCTCGAAGACAAGGAT | Amplification for 5′ race of PgCCR |
| CCR-3′GSP1 | TGCTTCAGCCCACGATCAATGCC | PgCCR 3′端扩增 |
| CCR-3′GSP2 | CCAACTCCGTGCAGGCGTATGTA | Amplification for 3′ race of PgCCR |
| CCR-QC-F | CTGCCCGATCGTGAGCTCCCA | PgCCR全长扩增 |
| CCR-QC-R | GCGGAAGTTTCCTTTTACTAA | Amplification for full length of PgCCR |
| CCR-RTPF | CGCCAAGTTCTTTCCCGAGTATCC | PgCCR荧光定量PCR |
| CCR-RTPR | ATGCCAAGGTCTCTGAGCTTCTGC | qRT-PCR primers |
| ACTIN-PF | AGTCCTCTTCCAGCCATCTC | PgACTIN扩增 |
| ACTIN-PR | CACTGAGCACAATGTTTCCA | Amplification for PgACTIN |
利用DNAMAN 2.6软件将基因编码的蛋白序列与NCBI数据库中其他物种的同源序列进行比对; 使用ClustalX 1.8和MEGA 5.05软件, 构建最大似然系统发育树[25], 并设置Bootstrap重复分析500次。
1.4 基因表达分析根据获得的基因序列, 设计引物CCR-RTPF和CCR-RTPR。提取不同样品的RNA, 分别取2 μg经DNaseⅠ(TaKaRa)消化的RNA, 利用MMLV反转录酶(TaKaRa)进行反转录获得cDNA模板。荧光定量PCR使用ABI7500 Fast实时荧光定量PCR系统以及SYBR® Premix Ex TaqTM Ⅱ(TaKaRa)荧光染料。反应程序如下:95 ℃ 90 s, 95 ℃ 15 s, 60 ℃ 30 s, 40个循环。以石榴PgACTIN作为内参基因, 数据分析使7500软件(Version 2.0.4), 相对表达量的计算使用2-ΔΔCT方法[26]。
2 结果与分析 2.1 石榴PgCCR基因cDNA全长的克隆与序列分析根据石榴籽粒EST库中的PgCCR基因片段, 利用通用引物UPM Long和特异引物CCR-5′GSP1进行第1轮扩增, 并以此PCR产物为模板, 使用NUP与CCR-5′GSP2作为引物进行第2轮扩增, 获得CCR基因的5′端序列, 测序得到1个长为883 bp的片段(图 1)。利用UPM-Long和CCR-3′GSP1引物进行CCR基因3′端的第1轮扩增, 以此PCR产物为模板, 使用NUP和CCR-3′GSP2引物进行第2轮扩增, 获得CCR基因的3′端序列, 测序得到1个515 bp的带有多聚A尾巴的片段。将获得的3部分序列进行拼接, 再次设计引物CCR-QC-F/CCR-QC-R, 利用高保真酶进行全长扩增, 最终得到1 269 bp的PgCCR基因序列(登录号:KM881713), 其中包含5′非编码区118 bp, 3′非编码区137 bp和开放阅读框1 017 bp。该基因编码一个含有338个氨基酸的蛋白质。利用ExPaSy ProtParam在线工具预测相对分子质量为59.7×103, 理论等电点为5.65。
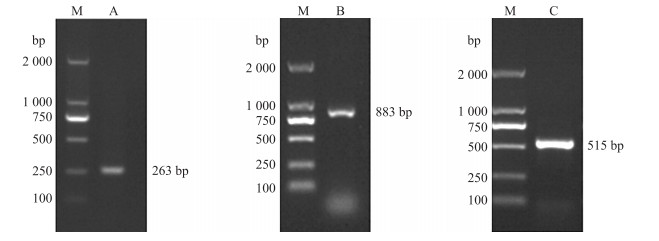
|
图 1 PgCCR基因的扩增 Figure 1 The detection of PgCCR gene amplification M.DL2000 marker; A.中间片段产物Internal fragments; B.5′Race产物5′Race fragments; C.3′Race产物3′Race fragments |
将PgCCR基因编码的氨基酸序列与其他物种的氨基酸序列进行比对, 发现PgCCR与葡萄的VvCCR蛋白相似性最高(图 2), 达到86.69%。与红麻HcCCR、橡胶HbCCR和古尼桉EgCCR相似性分别为82.94%、83.73%和82.25%。

|
图 2 PgCCR基因推导的氨基酸与其他物种的比对 Figure 2 Alignment of the predicted amino acid sequences of PgCCR with other species 1) Eg:古尼桉Eucalyptus gunnii; Vv:葡萄Vitis vinifera; Hc:红麻Hibiscus cannabinus; Hb:橡胶树Hevea brasiliensis. The same as follows.2) 保守功能域KNWYCYGK由黑框标出。黑色、红色及蓝色分别代表氨基酸完全保守、部分保守及相似。The conserved functional domain KNWYCYGK was marked by black frame. Amino acids highlighted in black, red and blue represent residues completely conserved, partially conserved and similar to consensus, respectively. |
PgCCR氨基酸序列中60~67氨基酸位点存在一段保守序列(KNWYCYGK), 该序列在所有已知的CCR基因中都存在, 被认为是CCR的催化位点[7]。
2.3 PgCCR同源蛋白系统发育分析为了确定PgCCR与其他物种CCR的亲缘关系, 利用MEGA 5.05软件将PgCCR与已克隆的番茄、油菜、亚麻、拟南芥、白桦、马铃薯、矮牵牛、毛白杨、欧洲菘蓝、小麦、黑麦草、北高丛越橘、羊乳、玉米、橡胶、红麻、沙梨、葡萄、陆地棉、古尼桉等物种的CCR进行多重序列比对和系统进化分析(图 3)。结果显示:PgCCR与其他物种的CCR起源相同, 确实是CCR家族的同源基因。但在后来进化的不同时期, 与其他物种分离开来。PgCCR和葡萄VvCCR的亲缘关系最近, 聚为一支, 而与玉米ZmCCR、黑麦草LpCCR、小麦TaCCR亲缘关系最远(图 3)。

|
图 3 不同物种CCR的系统进化分析 Figure 3 Phylogenetic analysis among different CCR Ph:矮牵牛Petunia hybrida; Sl:番茄Solanum lycopersicum; St:马铃薯Solanum tuberosum; Cl:羊乳Codonopsis lanceolata; Vc:北高丛越橘Vaccinium corymbosum; Pp:沙梨Pyrus pyrifolia; Pg:石榴Punica granatum; La:亚麻Linum album; Gh:陆地棉Gossypium hirsutum; Pt:毛白杨Populus tomentosa; Bp:白桦Betula platyphylla; It:欧洲菘蓝Isatis tinctoria; At:拟南芥Arabidopsis thaliana; Bn:油菜Brassica napus; Lp:黑麦草Lolium perenne; Ta:小麦Triticum aestivum; Zm:玉米Zea mays |
为探讨不同石榴品种中PgCCR基因的表达水平, 利用荧光定量PCR对‘红玉石籽’‘会理软籽’和‘突尼斯软籽’3个品种籽粒中PgCCR基因的相对表达量进行检测。结果表明:PgCCR基因在不同品种的石榴籽粒中均有表达, 其中在‘红玉石籽’中表达量最高, ‘会理软籽’表达量最低(图 4)。
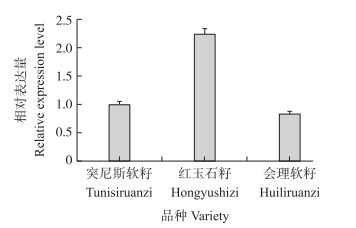
|
图 4 不同品种石榴籽粒中PgCCR基因的相对表达量 Figure 4 The relative expression level of PgCCR in seeds of different pomegranate varieties |
以品种‘红玉石籽’为材料, 对石榴不同组织中PgCCR基因的表达水平进行检测。由图 5可见:PgCCR基因在叶、花、茎3个组织中均有表达。其中, 茎和花中的表达量较高, 而叶片中的表达量较低, 花和茎中的表达量分别是叶片的4.43和4.90倍。
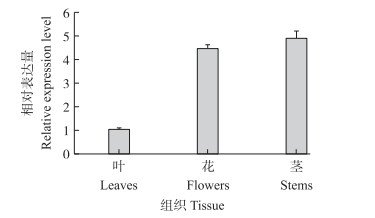
|
图 5 ‘红玉石籽’不同组织PgCCR基因的相对表达量 Figure 5 The relative expression level of PgCCR in different tissues of'Hongyushizi' |
为揭示PgCCR基因在籽粒不同发育时期的表达特性, 分别对开花后20、40、60、80、100和120 d的‘红玉石籽’籽粒进行取样, 去除假种皮和种仁后, 对剩下的种皮部分进行检测。结果(图 6)表明:PgCCR在籽粒的不同时期均有表达, 整体呈现先升高后下降的趋势, 20~80 d相对表达量逐渐上升, 并在80 d时相对表达量达到最高, 80~120 d表达量逐渐降低。
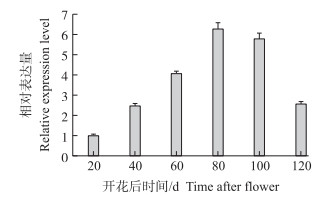
|
图 6 ‘红玉石籽’籽粒不同时期PgCCR基因的相对表达量 Figure 6 The relative expression level of PgCCR in 'Hongyushizi'seeds of different periods |
目前为止, CCR已从多种植物中被鉴定, 但其研究热点集中于植物的抗病、抗逆及作物品质改良[6], 而对于种皮, 尤其是籽粒中的研究较为少见。本研究从石榴籽粒中克隆了CCR的同源基因, 该基因推导的氨基酸序列含有CCR蛋白共有的保守域:KNWYCYGK, 因此命名为PgCCR。构建的进化树分为单、双子叶植物两个类群, PgCCR与双子叶植物中的葡萄VvCCR的亲缘关系最近, 而与单子叶植物玉米ZmCCR、黑麦草LpCCR、小麦TaCCR亲缘关系较远。
本试验中, ‘红玉石籽’中PgCCR表达量最高, ‘会理软籽’与‘突尼斯软籽’中表达量较低。之前的研究结果显示:‘红玉石籽’种皮总木质素含量达到9.25%, ‘会理软籽’和‘突尼斯软籽’的木质素含量较低, 分别为7.57%与6.36%[4]。因此, PgCCR基因的相对表达量与对应的木质素含量有一定相关性。说明PgCCR基因在石榴籽粒木质素合成中可能具有重要作用。
对其他植物的研究发现, CCR基因主要在木质部及韧皮部中表达, 这就决定CCR基因在茎中尤其是成熟茎中的表达量最高[7, 11, 27]。但不同植物CCR基因的表达特性并不相同, 例如:甘蔗和黑麦草中, CCR基因在茎和根中表达量较高, 而在叶和花中几乎不表达[12, 28]; 马铃薯StCCR 1基因主要集中在根中表达, 在叶和茎中表达量较少[29]; 美洲山杨中, 叶片中无法检测到CCR基因的表达, 而在成熟的茎杆中表达量极高[27]。以上研究表明CCR基因尽管在多个物种中保守存在, 但其表达特性并不完全相同。本研究发现, PgCCR在3个组织中均有表达, 而在茎中表达量最高, 花中表达量次之, 叶中表达量最低。这与其他物种CCR的表达具有相似性, 而花中表达量较高可能说明石榴中CCR基因参与除木质素合成外的其他发育过程, 这为进一步研究PgCCR的功能奠定了基础。
本研究对不同时期籽粒中的PgCCR基因的表达量进行检测, 发现PgCCR基因的表达量呈现先升高后下降的趋势。在开花后20~80 d, PgCCR基因的表达量逐渐升高, 说明这段时期籽粒中的木质素迅速合成并积累。而当木质素含量达到一定范围时, 木质素合成减弱, PgCCR基因的表达量也随之下降, 因此呈现出80~120 d逐步下降的趋势。说明石榴籽粒中木质素的合成与PgCCR的表达量密切相关。综上所述, PgCCR基因是石榴籽粒中木质素合成的重要调控位点, 其生物学功能值得深入解析。
| [1] | Alighourchi H, Barzegar M, Abbasi S. Anthocyanins characterization of 15 Iranian pomegranate(Punica granatum L.)varieties and their variation after cold storage and pasteurization[J]. European Food Research and Technology, 2008,227(3): 881–887. DOI: 10.1007/s00217-007-0799-1 |
| [2] | Jalikop S H, Venugopalan R, Kumar R. Association of fruit traits and aril browning in pomegranate(Punica granatum L.)[J]. Euphytica, 2010,174(1): 137–141. DOI: 10.1007/s10681-010-0158-3 |
| [3] | Sarkhosh A, Zamani Z, Fatahi R, et al. Evaluation of genetic diversity among Iranian soft-seed pomegranate accessions by fruit characteristics and RAPD markers[J]. Scientia Horticulturae, 2009,121(3): 313–319. DOI: 10.1016/j.scienta.2009.02.024 |
| [4] | 张水明, 龚凌燕, 曹丹琴, 等. 石榴种皮总木质素含量及PgCOMT基因的克隆与表达[J]. 热带亚热带植物学报, 2015, 23(1): 65–73. Zhang S M, Gong L Y, Cao D Q, et al. Total lignin content in pomegranate seed coat and cloning and expression analysis of PgCOMT gene[J]. Journal of Tropical and Subtropical Botany, 2015,23(1): 65–73. (in Chinese) |
| [5] | van Acker R, Leplé J C, Aerts D, et al. Improved saccharification and ethanol yield from field-grown transgenic poplar deficient in cinnamoyl-CoA reductase[J]. Proc Natl Acad Sci USA, 2014,111(2): 845–850. DOI: 10.1073/pnas.1321673111 |
| [6] | 李波, 梁颖, 柴友荣. 植物肉桂酰辅酶A还原酶(CCR)基因的研究进展[J]. 分子植物育种, 2006, 4(3S): 55–65. Li B, Liang Y, Chai Y R. Achievements in research on plant cinnamoyl-CoA reductase(CCR)genes[J]. Molecular Plant Breeding, 2006,4(3S): 55–65. (in Chinese) |
| [7] | Lacombe E, Hawkins S, van Doorsselaere J, et al. Cinnamoyl CoA reductase, the first committed enzyme of the lignin branch biosynthetic pathway:cloning, expression and phylogenetic relationships[J]. Plant Journal, 1997,11(3): 429–441. DOI: 10.1046/j.1365-313X.1997.11030429.x |
| [8] | Leplé J C, Grima-Pettenati J, van Montagu M, et al. A cDNA encoding cinnamoyl-CoA reductase from Populus trichocarpa[J]. Plant Physiology, 1998,117(3): 1126. |
| [9] | Piquemal J, Lapierre C, Myton K, et al. Down-regulation of cinnamoyl-CoA reductase induces significant changes of lignin profiles in transgenic tobacco plants[J]. Plant Journal, 1998,13(1): 71–83. |
| [10] | Pichon M, Courbou I, Beckert M, et al. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase(CCR) and differential expression of the corresponding genes[J]. Plant Mol Biol, 1998,38(4): 671–676. DOI: 10.1023/A:1006060101866 |
| [11] | Lauvergeat V, Lacomme C, Lacombe E, et al. Two cinnamoyl-CoA reduetase(CCR)genes from Arabidopsis thaliana are differentially expressed during development and inresponse to infection with pathogenic bacteria[J]. Phytochemistry, 2001,57(7): 1187–1195. DOI: 10.1016/S0031-9422(01)00053-X |
| [12] | Mcinnes R, Lidgett A, Lynch D, et al. Isolation and characterization of a cinnamoyl-CoA reductase gene from perennial ryegrass(Lolium perenne)[J]. Journal of Plant Physiology, 2002,159(4): 415–422. DOI: 10.1078/0176-1617-00719 |
| [13] | van der Rest B, Danoun S, Boudet A M, et al. Down-regulation of cinnamoyl-CoA reductase in tomato(Solanum lycopersicum L.)induces dramatic changes in soluble phenolic pools[J]. Journal of Experimental Botany, 2006,57(6): 1399–1411. DOI: 10.1093/jxb/erj120 |
| [14] | 陈尘, 王政军, 曹鑫林, 等. 丹参肉桂酰辅酶还原酶基因克隆与生物信息学分析[J]. 西北植物学报, 2011, 31(10): 1963–1968. Chen C, Wang Z J, Cao X L, et al. Bioinformatics and expression analysis of cinnamoyl-CoA reductase gene from Salvia miltiorrhiza Bunge[J]. Acta Botanica Boreali-Occidentalia Sinica, 2011,31(10): 1963–1968. (in Chinese) |
| [15] | Zhu Q H, Zhang X Q, Huo S, et al. Cloning and bioinformatic analysis of cinnamoyl-CoA reductase gene(CCR)from Pennisetum purpureum[J]. Agricultural Science and Technology, 2012,13(2): 284–291. |
| [16] | Ralph J, Hatfield R D, Piquemal J, et al. NMR characterization of altered lignins extracted from tobacco plants down-regulated for lignification enzymes cinnamylalcohol dehydrogenase and cinnamoyl-CoA reductase[J]. Proc Natl Acad Sci USA, 1998,95(22): 12803–12808. DOI: 10.1073/pnas.95.22.12803 |
| [17] | Laskar D D, Jourdes M, Patten A M, et al. The Arabidopsis cinnamoyl CoA reductase irx4 mutant has a delayed but coherent(normal)program of lignification[J]. Plant Journal, 2006,48(5): 674–686. DOI: 10.1111/tpj.2006.48.issue-5 |
| [18] | Derikvand M, Berrio Sierra M, Ruel J, et al. Redirection of the phenylpropanoid pathway to feruloyl malate in Arabidopsis mutants deficient for cinnamoyl-CoA reductase 1[J]. Planta, 2008,227(5): 943–956. DOI: 10.1007/s00425-007-0669-x |
| [19] | Ruel K, Berrio-Sierra J, Derikvand M M, et al. Impact of CCR1 silencing on the assembly of lignified secondary walls in Arabidopsis thaliana[J]. New Phytologist, 2009,184(1): 99–113. DOI: 10.1111/nph.2009.184.issue-1 |
| [20] | Leplé J C, Dauwe R, Morreel K, et al. Downregulation of cinnamoyl-coenzyme A reductase in poplar:multiple-level phenotyping reveals effects on cell wall polymer metabolism and structure[J]. Plant Cell, 2007,19(11): 3669–3691. DOI: 10.1105/tpc.107.054148 |
| [21] | Tamasloukht B, Wong Quai Lam M S, Martinez Y, et al. Characterization of a cinnamoyl-CoA reductase 1(CCR1)mutant in maize:effects on lignification, fibre development, and global gene expression[J]. Journal of Experimental Botany, 2011,62(11): 3837–3848. DOI: 10.1093/jxb/err077 |
| [22] | Anterola A M, Lewis N G. Trends in lignin modification:a comprehensive analysis of the effects of genetic manipulations mutations on lignification and vascular integrity[J]. Phytochemistry, 2002,61(3): 221–294. DOI: 10.1016/S0031-9422(02)00211-X |
| [23] | 朱立武, 张水明, 巩雪梅, 等. 软籽石榴新品种'红玉石籽'[J]. 园艺学报, 2005, 32(5): 965. Zhu L W, Zhang S M, Gong X M, et al. A new soft-seeded pomegranate variety 'Hongyushizi'[J]. Acta Horticulturae Sinica, 2005,32(5): 965. (in Chinese) |
| [24] | 王壮伟, 渠慎春, 章镇, 等. 苹果属RNA高效快速提取新方法[J]. 果树学报, 2004, 21(4): 385–387. Wang Z W, Qu S C, Zhang Z, et al. A fast method for total RNA extraction from the tissue culture material of Malus sp.[J]. Journal of Fruit Science, 2004,21(4): 385–387. (in Chinese) |
| [25] | Saitou N, Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987,4(4): 406–425. |
| [26] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real time quantitative PCR and the 2-ΔΔCT methods[J]. Methods, 2001,25(4): 402–408. DOI: 10.1006/meth.2001.1262 |
| [27] | Li L G, Cheng X F, Lu S, et al. Clarification of einnamoyl co-enzymea reduetase catalysis in monolignol biosynthesis of aspen[J]. Plant Cell Physiology, 2005,46(7): 1073–1082. DOI: 10.1093/pcp/pci120 |
| [28] | Larsen K. Cloning and characterization of a ryegrass(Lolium perenne)gene encoding einnamoyl-CoA reduetase(CCR)[J]. Plant Science, 2004,166(3): 569–581. DOI: 10.1016/j.plantsci.2003.09.026 |
| [29] | Larsen K. Molecular cloning and characterization of cDNAs encoding cinnamoyl CoA reduetase(CCR)from barley(Hordeum vulgate) and potato(Solarium tuberosum)[J]. Journal of Plant Physiology, 2004,161(1): 105–112. DOI: 10.1078/0176-1617-01074 |




