文章信息
- 叶文武, 张萌, 曹明娜, 翟春花, 李爱宁, 王源超. 2016.
- YE Wenwu, ZHANG Meng, CAO Mingna, ZHAI Chunhua, LI Aining, WANG Yuanchao. 2016.
- 大豆疫霉PsMPK1沉默突变体的基因表达谱分析
- Gene expression profiling of a PsMPK1-silenced mutant of Phytophthora sojae
- 南京农业大学学报, 39(3): 386-393
- Journal of Nanjing Agricultural University, 39(3): 386-393.
- http://dx.doi.org/10.7685/jnau.201509026
-
文章历史
- 收稿日期:2015-09-19
促有丝分裂原蛋白激酶(mitogen-activated protein kinase,MAPK)是真核生物细胞信号转导途径中的关键蛋白激酶,对植物病原菌的生长发育与侵染过程具有重要作用[1]。我们经过前期研究发现,MAPK基因PsMPK 1 参与调控大豆疫霉的细胞壁完整性、菌丝生长、游动孢子形成,以及对大豆的致病性等生物学性状[2]。
细胞壁在维持细胞形态、物质吸收、传递信号,以及抵御环境胁迫等方面具有重要的作用。细胞壁不仅作为细胞的保护屏障,还作为骨架将细胞壁蛋白(cell wall proteins,CWPs)支撑于细胞表面。对于植物病原菌(如真菌和卵菌等)来说,细胞壁蛋白具有细胞壁生物合成和帮助病菌黏附到寄主组织表面的作用,还能够帮助病原菌抵御植物的防卫反应[3, 4]。
游动孢子的释放和游动是疫霉菌传播扩散的主要方式,也是它们区别于其他生物的重要特征之一。游动孢子无细胞壁,可以在水中游动并通过趋化性寻找寄主,之后形成具有细胞壁的休止孢黏附于寄主组织表面。休止孢萌发形成芽管穿透寄主表皮,并在植物细胞内进一步扩展[5]。休止孢的形成、黏附以及萌发形成芽管都需要细胞壁相关蛋白的参与。
为了探究PsMPK1如何调控大豆疫霉的细胞壁完整性以及致病性等生物学性状,我们通过数字基因表达谱(digital gene expression profiling,DGE)方法,对PsMPK 1 沉默后的大豆疫霉差异表达基因进行分析,以期揭示PsMPK1的调控网络。由于大豆疫霉PsMPK 1基因沉默导致游动孢子的形成受到影响且病原菌的致病性丧失,因此无法获得游动孢子阶段和侵染寄主过程的样品。鉴于PsMPK1沉默后影响细胞壁完整性和菌丝生长等,且PsMPK 1 在游动孢子囊阶段的表达量略高于菌丝阶段[2],本研究选择游动孢子囊阶段的大豆疫霉样品进行DGE分析。
1 材料与方法 1.1 样品的准备供试的大豆疫霉菌株为野生型菌株P6497与PsMPK 1 基因沉默突变体T21[2]。将保存在10 ℃冰箱的大豆疫霉菌株转接至10% V8固体培养基上,25 ℃黑暗培养72 h。切取边缘2 mm×2 mm菌丝块,移至10% V8液体培养液中,黑暗培养48 h。用灭菌超纯水反复冲洗菌丝3~5次后,将菌丝浸没于灭菌超纯水中,4 h 后在显微镜下观察孢子囊的产生情况。用镊子将产生孢子囊的菌丝夹出,在滤纸上将菌丝挤干液体后在液氮中研磨,使用Total RNA Purification System(Invitrogen,Carlsbad,CA,USA)试剂盒提取RNA。
1.2 基因表达量的计算将提取的RNA送交深圳华大基因科技服务有限公司进行3′-标签数字基因表达谱分析(3′-tag digital gene expression),建库、测序与分析方法详见文献[6]。利用Bowtie(v. 2.1.0)软件(http://bowtie-bio.sourceforge.net)将高质量的转录本标签与大豆疫霉的基因组与基因序列(下载自JGI数据库http://genome.jgi.doe.gov;版本:v. 1.1)[7]进行比对,比对过程中允许有1个碱基的错配。分析结果中基因的相对表达量指的是每100万条转录本标签中匹配到目标基因的总数量(transcripts per million clean tags,TPM),即:TPM=(匹配到基因的标签数/匹配到参考序列的总标签数)×1 000 000。
1.3 实时定量PCR验证采用与DGE分析相同的方法准备RNA样品,使用SYBR Premix ExTaq(TaKaRa Inc.,Dalian,China)试剂盒进行定量PCR分析。引物详见表 1。反应体系包括:cDNA 50 ng,上、下游引物各0.2 μmol,SYBR Green PCR Master Mix 10 μL,并加灭菌超纯水至20 μL。分析仪器为ABI PRISM 7300 fast real-time PCR system(Applied Biosystems,Foster City,CA,USA)。使用7300 system sequence detection software(v.1.4)软件进行数据分析。
| 基因编号Gene ID | 数字基因表达谱数据 DGE data | 实时定量PCR结果 qRT-PCR data | 两组数据中变化特征是否一致Consistentchangpattern | 定量PCR的正向引物序列Sequence of forward primerfor qRT-PCR(5′→3′) | 定量PCR的反向引物序列Sequence of reverse primerfor qRT-PCR(5′→3′) | |||||
| 野生型中基因的表达量TPM ofWT-SP | 突变体中基因的表达量TPM ofMPK1-SP | 基因表达量变化倍数(log2)Ratio ofMPK1-SP/WT-SP(log2) | 基因表达量变化特征Changpattern | 平均值±标准差 x± SD | log2转换后的平均值log2convertedmean value | 基因表达量变化特征Changpattern | ||||
| Ps138161 | 17.39 | 0.55 | -4.98 | Down | 0.52±0.25 | -0.94 | Down | Yes | ATGGCTTTCGTTCGCTCTTC | GGTCGGTGTCATCGCTGGT |
| Ps157780 | 583.56 | 19.53 | -4.90 | Down | 0.56±0.03 | -0.84 | Down | Yes | CTGCAGTCACAGGCATTCG | GGTACTTCTCGTGGTTGTCC |
| Ps139315 | 95.52 | 3.58 | -4.74 | Down | 0.40±0.02 | -1.31 | Down | Yes | CAGTTATGAAGGTTGTTTAGGC | GGATTAACGCGTTCGCTCATAC |
| Ps134056 | 73.31 | 3.85 | -4.25 | Down | 0.80±0.13 | -0.33 | Down | Yes | GACGCAGGAACGAAGTCGC | CACCAGGCTTGATCCGTTG |
| Ps109539 | 618.08 | 52.82 | -3.55 | Down | 0.21±0.02 | -2.28 | Down | Yes | GAACAGTGACAAGGTCGTC | CTACAATTCACCTACAGCAG |
| Ps136243 | 123.62 | 12.38 | -3.32 | Down | 0.37±0.03 | -1.45 | Down | Yes | GAAGTGTCGAGACGTGGCAG | GACACGTCGTGGCTTCAGC |
| Ps109281 | 180.07 | 29.99 | -2.59 | Down | 0.39±0.05 | -1.36 | Down | Yes | CTAAGCAGACCACGTACACG | GTGGGTCAGGTACTGGGTC |
| Ps158856 | 39.33 | 8.80 | -2.16 | Down | 0.45±0.04 | -1.14 | Down | Yes | TACGACGAGAACGTGGCCAAC | TACAGCTTGTCCTCGTCCGTG |
| Ps128024 | 35.05 | 7.98 | -2.13 | Down | 0.86±0.04 | -0.22 | Down | Yes | GTTCCTCGTGACGACCAATC | GTAGACGAGAGAGGCTACG |
| Ps139525 | 55.65 | 12.93 | -2.11 | Down | 0.69±0.04 | -0.53 | Down | Yes | CTCAAGAAGCTGGACAGATG | CCAGAGGGAAGCTTGAGTC |
| Ps143590 | 288.44 | 67.12 | -2.10 | Down | 0.58±0.04 | -0.79 | Down | Yes | CTACGGCAACGACTGCATG | GGTTCCGGTGCTTCCAGTC |
| Ps132587 | 14.18 | 3.85 | -1.88 | Down | 0.95±0.08 | -0.07 | Down | Yes | TGAGCAGTCAATCAGGAACC | CTCTGAGACGAGCTGGTATC |
| Ps134912 | 1 257.30 | 345.24 | -1.86 | Down | 0.51±0.05 | -0.97 | Down | Yes | CAGGAGACGTGGAACGTTG | TCTCCTCCTCGTCCTTCTG |
| Ps139852 | 45.49 | 165.33 | 1.86 | Up | 3.45±0.25 | 1.79 | Up | Yes | CAAGCTGTACACGCGCATGC | AGCACCTGCCAGTACAGCAC |
| Ps109296 | 27.29 | 104.54 | 1.94 | Up | 2.22±0.29 | 1.15 | Up | Yes | GACGCTGACATCATGAGGAAG | GTAGTACATGTACCAGTACGC |
| Ps109378 | 4.28 | 18.43 | 2.11 | Up | 1.26±0.11 | 0.34 | Up | Yes | CTGGTCGCACAAGTGCAACGT | CGAACTGCAAGAATGTGTAC |
| Ps108266 | 19.00 | 83.35 | 2.13 | Up | 1.84±0.17 | 0.88 | Up | Yes | CGAAGAAGTACTTCTCCAAC | ATCAATCCAGCTTGCAATGG |
| Ps137459 | 2.94 | 13.20 | 2.17 | Up | 1.24±0.21 | 0.31 | Up | Yes | ATATGAGCACGGAATGGTAC | CAAGATCACCTTCTTCTTGC |
| Ps155247 | 2.14 | 9.63 | 2.17 | Up | 5.81±0.50 | 2.54 | Up | Yes | GATTGTTCACTGCTGAGCAG | GAAGCTCTTCGTTCGGCTG |
| Ps132981 | 0.80 | 13.48 | 4.07 | Up | 3.86±0.41 | 1.95 | Up | Yes | GACGACCTGAAGGCCATTC | CCAGTCACACCAACACATTC |
| Ps110713 | 103.82 | 14.85 | -2.81 | Down | 1.59±0.14 | 0.67 | Up | No | GTCGCTGTAAGCAGCCTCAG | GGTTATACGTCTGATACCAGC |
| Ps127835 | 58.60 | 21.73 | -1.43 | Down | 1.40±0.28 | 0.48 | Up | No | CTCGACTACTGCATGAACAG | CGCAGTACGTGACGATGGC |
| Ps116268 | 234.39 | 87.75 | -1.42 | Down | 2.32±0.18 | 1.21 | Up | No | CTCAACAACAACTCGTAC | GCGTAGCGGTCCACAAGC |
| Ps141900 | 574.73 | 218.70 | -1.39 | Down | 3.25±0.38 | 1.70 | Up | No | CAAGTTCGTGATGGGTCTGTTC | GTGCAGTTCACGAACATGTAGG |
| 注:TPM:基因表达量Gene expression level;WT-SP:野生型Wild type;MPK1-SP:突变体Mutant. The same as follows. | ||||||||||
使用Microsoft Excel 2007中的PEARSON函数计算样品间基因表达水平的皮尔森相关性(Pearson correlation)。使用MultiExperiment Viewer(MeV,v. 4.6)软件进行主成分分析(principal component analysis,PCA)。差异表达基因需要满足:变化倍数在2倍以上,且经卡方检验和Bonferroni校正后P≤0.01。基因的gene ontology(GO)信息下载自大豆疫霉基因组数据库(http://genome.jgi.doe.gov),利用WEGO进行分析和作图(http://wego.genomics.org.cn)。使用SeqHunter[8]中的BLASTp程序(E-value阈值为1×10-10)搜索序列相似的蛋白。使用NCBI的BLASTp工具搜索序列相似的蛋白以注释基因及所属的基因家族(http://blast.ncbi.nlm.nih.gov,使用默认参数)。
2 结果与分析 2.1 基本数据分析对大豆疫霉PsMPK 1 沉默突变体(样品名称:MPK1-SP)在游动孢子囊阶段的RNA样品进行数字基因表达谱分析,得到约364万条高质量的转录本标签序列(Clean Tags)(表 2)。这些转录本标签中,90%能够精确比对到大豆疫霉基因组上。10 022个基因具有表达数据,占大豆疫霉19 027个预测基因的53%(表 2)。
| 统计项目 Statistical item | 野生型 WT-SP* | 突变体 MPK1-SP |
| 原始序列标签总数Total number of raw tags | 3 889 220 | 3 739 010 |
| 高质量序列标签总数Total number of clean tags | 3 737 336 | 3 635 145 |
| 比对到参考基因组的高质量序列标签总数Total number of clean tags mapping to genome | 3 394 930 | 3 257 045 |
| 比对到参考基因组的高质量序列标签比例/% Clean tags mapping to genome rate | 90.84 | 89.60 |
| 未比对到参考基因组的高质量序列标签总数Total number of unmapped clean tags | 342 406 | 378 100 |
| 未比对到参考基因组的高质量序列标签比例/% Unmapped clean tags rate | 9.16 | 10.40 |
| 比对到参考基因的高质量序列标签总数Total number of clean tags mapping to genes | 2 450 639 | 2 384 913 |
| 比对到参考基因的高质量序列标签比例/% Clean tags mapping to genes rate | 65.57 | 65.61 |
| 比对到唯一参考基因的高质量序列标签总数Total number of clean tags mapping to unique gene | 2 210 175 | 2 176 838 |
| 比对到唯一参考基因的高质量序列标签比例/% Clean tags mapping to unique gene rate | 59.14 | 59.88 |
| 被高质量序列标签比对到的基因总数Total number of clean-tag-mapped genes | 12 146 | 12 326 |
| 被高质量序列标签比对到的基因比例/% Clean-tag-mapped genes rate | 62.23 | 64.78 |
| 被明确的高质量序列标签比对到的基因总数Total number of unambiguous-clean-tag-mapped genes | 9 960 | 10 022 |
| 被明确的高质量序列标签比对到的基因比例/% Unambiguous-clean-tag-mapped genes rate | 52.35 | 52.67 |
| 注:*数据来自文献[6]。*Data were obtained from the reference[6]. | ||
基于已发表的野生型大豆疫霉无性发育与侵染大豆过程中共10个阶段或时间点(菌丝MY、产孢子囊菌丝SP、游动孢子ZO、休止孢CY、萌发的休止孢GC,以及菌丝侵染大豆叶片后1.5、3、6、12和24 h,即IF1.5h~IF24h)的基因表达谱数据[6],比较MPK1-SP与上述10个样品中基因表达水平的整体相关性。皮尔森相关性分析结果表明:MPK1-SP中的基因表达水平整体上与野生型的游动孢子囊阶段样品(SP)最接近(皮尔森相关系数R为0.89;R值越接近1表明越趋近正相关),其次是菌丝阶段(MY,R=0.85),而与游动孢子阶段(ZO,R=0.57)、休止孢阶段(CY,R=0.63)和休止孢萌发阶段(GC,R=0.69)相关性最低(图 1-A)。利用主成分分析方法得到相同的样品间关系(图 1-B)。

|
图 1 样品间基因表达水平的整体相关性和不同表达水平的基因数量分布
Fig. 1 Correlation of the whole-library genes expression patterns and the distribution of gene expression levels
A. 皮尔森相关性分析;B. 主成分分析中不同样品在主成分1和2的相对距离;C. 不同表达水平区间的基因数量分布,x轴上数值(n)代表“n<基因表达水平 ≤ n+1”。MY:菌丝;SP:产孢子囊菌丝;ZO:游动孢子;CY:休止孢;GC:萌发的休止孢;IF1.5h~IF24h:菌丝侵染大豆叶片后1.5、3、6、12和24 h。 A. Pearson correlation analysis;B. Principal component analysis plot of principal components 1 and 2;C. The distribution of gene transcription levels,based on the number of genes falling in each log2 gene expression category. On x-axis,digit(n)refers to n<expression level ≤n+1. MY:Mycelia;SP:Zoosporangia;ZO:Zoospores;CY:Cysts;GC:Germinating cysts;IF1.5h-IF24h:1.5,3,6,12 and 24 h post-mycelia-inoculation of soybean leaves. |
对SP和MPK1-SP的分析数据进行比较,发现2个样品中分别得到约374万和364万条Clean Tags,其中分别有91%和90%均比对到大豆疫霉基因组上,并分别有9 960和10 022个基因获得表达数据(表 2)。对具有不同表达水平的基因进行数量统计,发现SP和MPK1-SP 2个样品中检测到的基因表达水平大部分都在1和5之间;随着表达水平数值的继续升高,基因数量呈递减趋势;大于0且小于1区间略少于大于5且小于6区间的基因数量(图 1-C)。该结果表明,对SP和MPK1-SP的DGE分析得到的基本数据基本一致。
2.2 差异表达基因对SP和MPK1-SP之间进行差异表达基因的鉴定,发现相对于SP,1 451个基因在MPK1-SP中显著下调超过2倍,占所有检测到基因的14%;显著上调的基因相对较少,为981个,占9%;77%的被检测基因(8 332个)则没有显著的转录变化(图 2-A)。大部分(80%)的差异表达基因呈现2至8倍的变化倍数,随着倍数增加,基因的数量逐渐减少(图 2-A)。
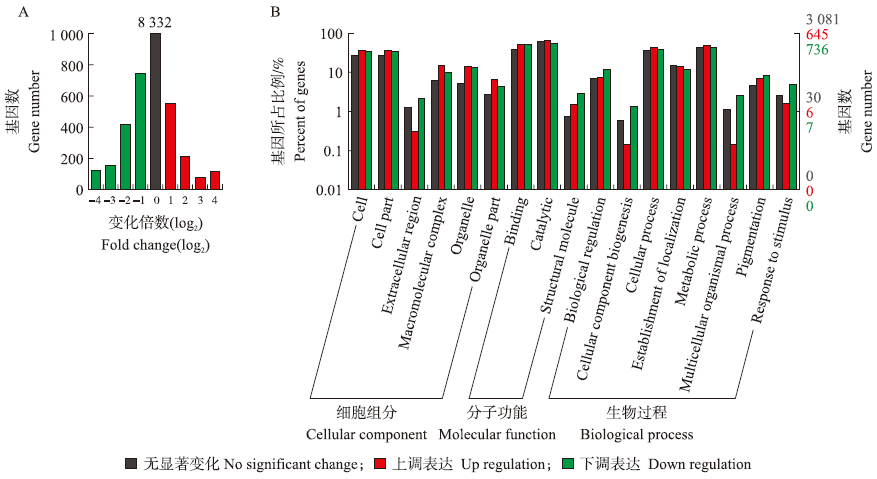
|
图 2 差异表达基因(A)及基因功能的GO分析(B)
Fig. 2 Differentially expressed genes(A)and GO(gene ontology)analysis for gene functions(B)
A.SP和MPK1-SP之间差异表达基因的分布,x轴上代表MPK1-SP/SP倍数的log2值,数值(n)代表“n<变化倍数 ≤n+1”;B.基因功能的GO分析,图中只呈现基因比例具有显著变化的GO条目。 A.Differentially expressed genes between SP and MPK1-SP,x-axis refers to log2 values for MPK1-SP/SP and digit(n)refers to n< fold change ≤n+1. B.GO analysis for gene functions,the figure only displays the GO terms with significant difference in percent of genes. |
PsMPK 1 在SP中的相对表达水平为1.36(TPM值),而在PsMPK 1 突变体的样品MPK1-SP中没有发现该基因的转录本,表明MPK1-SP中PsMPK 1 确实已经被沉默。
为了验证基因差异表达的可靠性,随机挑选了24个基因进行实时定量PCR的验证。结果发现,20个基因呈现相同的基因表达变化特征,占所有验证基因数的83%(表 1)。对20个变化特征一致的基因进行变化倍数的皮尔森相关性分析,发现R值为0.84,表明DGE和实时定量PCR分析得到的基因表达数据具有较高的一致性。
2.3 基因功能的GO分析GO分析是根据相似序列的比对将基因或其编码产物归类到与分子功能(molecular function)、生物过程(biological process)和细胞组分(cellular component)等3个方面对应的GO条目。比较无显著变化、显著上调和显著下调3组基因对应的GO条目,发现3组基因之间对应到17个GO条目的基因比例具有显著差异(图 2-B)。其中4个条目对应到较多的显著下调基因、较少的显著上调基因,包括:Extracellular region(GO:0005576,胞外区)、Cellular component biogenesis(GO:0044085,细胞成分的生物合成)、Multicellular organismal process(GO:0032501,多细胞生物体的生物过程)、Response to stimulus(GO:0050896,对刺激或胁迫的应答);2个条目对应到较少的显著下调基因、较多的显著上调基因,包括:Catalytic activity(GO:0003824,催化活性)、Obsolete molecular function(GO:0008369,已废除的条目)。其他11个条目对应到较多的差异表达基因,例如:Metabolic process(GO:0008152,代谢过程)、Binding(GO:0005488,结合活性)、Structural molecule(GO:0005198,结构分子活性)、Biological regulation(GO:0065007,生物体调节过程)、Pigmentation(GO:0043473,色素积累)、Cell and organelle(GO:0005623、GO:0044422、GO:0044464,细胞与细胞器组成)等(图 2-B)。
2.4 细胞壁相关基因在GO分析中,发现与“细胞成分的生物合成”和“细胞与细胞器组成”等相关的基因更多地出现在差异表达基因中(图 2-B)。鉴于PsMPK 1 的沉默导致大豆疫霉的细胞壁完整性受损,进一步对细胞壁相关基因进行分析。橡树疫霉中已报道了17个细胞壁相关蛋白,分别属于激发素(elicitin)、葡聚糖酶(glucanase)、谷氨酰胺转胺酶(transglutaminase)、黏液素(mucin)、kazal、膜联蛋白(annexin)和其他等7个家族或类别[4]。基于该结果,通过序列相似性比对方法在表达的10 764个大豆疫霉基因中找到了126个可能的细胞壁相关基因。分析发现:37%的基因(46个)在SP与MPK1-SP之间差异表达(图 3),该比例显著高于所有10 764个基因中差异表达基因的比例(23%,超几何分布P<0.001)。此外,46个差异表达的细胞壁相关基因中,40个(87%)表现为显著下调(图 3),明显大于所有10 764个被检测的大豆疫霉基因中显著下调的基因比例(14%)。上述结果表明:PsMPK 1 的沉默导致许多细胞壁相关基因的表达被抑制。
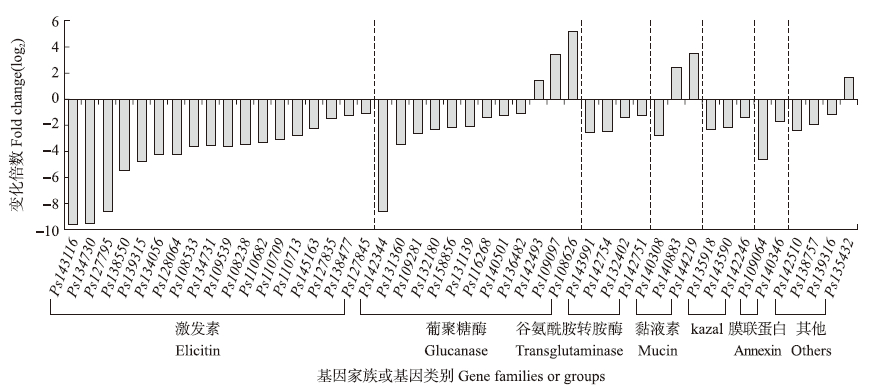
|
图 3 细胞壁相关基因的表达变化倍数
Fig. 3 Fold change of expression level for the cell wall associated genes
变化倍数指的是MPK1-SP/SP倍数的log2值,该值大于0或小于0分别表示基因上调或者下调。 Fold change refers to log2 values for MPK1-SP/SP,and values > 0 and values < 0 mean up regulation and down regulation,respectively. |
上述结果中发现分别有14%和9%的被检测基因在MPK1-SP中显著下调和上调表达,占所分析基因总数的23%。与编码转录因子、蛋白激酶和分泌蛋白的基因进行比较,发现14%和9%的转录因子编码基因分别显著下调和上调表达,与所有被检测基因的结果相近;17%和9%的蛋白激酶编码基因分别显著下调和上调表达,即有略多的该类基因在PsMPK 1 沉默后转录水平下降;22%和6%的分泌蛋白编码基因分别显著下调和上调表达,差异表达基因的比例明显比所有被检测基因中的比例高(28%>23%),且更多的基因呈现下调表达,但相对较少的基因上调表达(图 4)。由于病原菌的外泌蛋白中包含许多致病相关蛋白或效应因子(effectors),推测外泌蛋白编码基因表达水平的下降可能是导致PsMPK 1 沉默后大豆疫霉致病性丧失的重要原因之一。
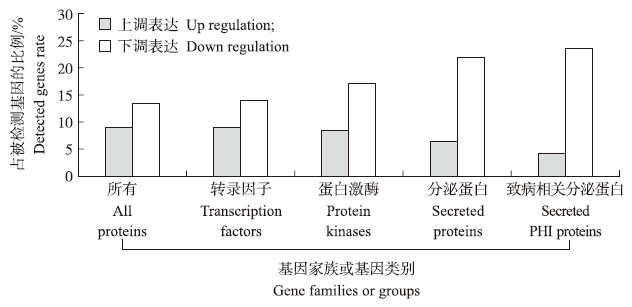
|
图 4 不同基因家族中差异表达基因的比例 Fig. 4 Proportion of differentially expressed genes for certain gene families |
病原菌与寄主互作数据库(Pathogen-Host Interactions database,PHI database)收集了真菌、卵菌和细菌中已试验证明的致病相关基因和效应分子编码基因[9]。通过比对,发现被检测到的分泌蛋白编码基因中有118个与PHI基因编码相似的蛋白序列。其中28%的基因显著下调表达,4%为下调表达。与所有分泌蛋白的编码基因相比,更多基因呈现下调表达,更少基因上调表达(图 4)。该结果进一步说明,PsMPK 1 沉默后导致致病相关基因或效应分子基因转录水平的降低,这可能是大豆疫霉丧失致病性的原因。
在与PHI基因比对的基础上,进一步对PsMPK 1 沉默突变体中显著下调的分泌蛋白编码基因进行功能注释和分析。除了鉴定到27个外泌的细胞壁相关基因之外(包括elicitin、glucanase、transglutaminase、kazal等),还发现37个可能与致病过程相关的基因。如表 3所示:这37个基因主要编码RxLR、NLP和CRN等家族的效应分子,CBEL蛋白,富含半胱氨酸的蛋白,氧化还原相关蛋白,以及水解酶等。效应分子包括RxLR家族的PsAvh50、PsAvh113、PsAvh158、PsAvh172、PsAvh177、PsAvh268、PsAvh389、PsAvr3a1和PsAvr1d,CRN家族的PsCRN63和PsCRN179,以及NLP家族的PsojNIP等。氧化还原相关蛋白包括过氧化物酶(Ps140211、Ps140214和Ps140215)、谷胱甘肽过氧化物酶(Ps133117)、多铜氧化酶(Ps137983和Ps137984)、过氧化氢酶(Ps143739)和酪氨酸酶(Ps131976、Ps135423和Ps133889)等。水解酶包括纤维素酶(Ps132587)、葡聚糖酶(Ps128950)、金属依赖性水解酶(Ps135377)等。
| 基因家族(基因名)Gene family(Gene name) | 基因编号Gene ID | 野生型中基因的表达量TPM of WT-SP | 突变体中基因的表达量TPM of MPK1-SP | 基因表达量变化倍数(log2)Ratio of MPK1-SP/WT-SP(log2) |
| RxLR(PsAvh50) | Ps134001 | 23.28 | 0 | -11.18 |
| RxLR(PsAvh113) | Ps135182 | 5.08 | 0 | -8.99 |
| RxLR(PsAvh158) | Ps140196 | 9.10 | 0 | -9.83 |
| RxLR(PsAvh172) | Ps144029 | 13.11 | 1.93 | -2.76 |
| RxLR(PsAvh177) | Ps135098 | 38.26 | 10.45 | -1.87 |
| RxLR(PsAvh268) | Ps136243 | 123.62 | 12.38 | -3.32 |
| RxLR(PsAvh389) | Ps109021 | 56.72 | 1.65 | -5.10 |
| RxLR(PsAvr3a1) | Ps139995 | 8.83 | 1.93 | -2.19 |
| RxLR(PsAvr1d) | Ps142795 | 51.64 | 8.53 | -2.60 |
| CRN(PsCRN63) | Ps108741 | 61.54 | 23.93 | -1.36 |
| CRN(PsCRN179) | Ps125578 | 202.82 | 28.61 | -2.83 |
| NLP(PsojNIP) | Ps109198 | 8.03 | 1.65 | -2.28 |
| CBEL protein | Ps134874 | 47.63 | 5.50 | -3.11 |
| CBEL protein | Ps134006 | 248.03 | 39.61 | -2.65 |
| CBEL protein | Ps108819 | 410.45 | 149.10 | -1.46 |
| CBEL protein | Ps140695 | 250.18 | 100.68 | -1.31 |
| Small cysteine rich protein | Ps156264 | 263.82 | 89.96 | -1.55 |
| Cystatin-like cysteine protease inhibitor | Ps138161 | 17.39 | 0.55 | -4.98 |
| Glucanase inhibitor protein | Ps139134 | 8.29 | 0 | -9.70 |
| Glucanase inhibitor protein | Ps139135 | 6.96 | 0 | -9.44 |
| Glucanase inhibitor protein | Ps139985 | 15.52 | 6.60 | -1.23 |
| Peroxidase protein | Ps140214 | 4.28 | 0 | -8.74 |
| Peroxidase protein | Ps140215 | 10.44 | 0.55 | -4.25 |
| Peroxidase protein | Ps140211 | 14.45 | 1.38 | -3.39 |
| Glutathione peroxidase | Ps133117 | 25.15 | 11.83 | -1.09 |
| Multicopper oxidase | Ps137984 | 6.69 | 0 | -9.39 |
| Multicopper oxidase | Ps137983 | 11.51 | 0.55 | -4.39 |
| Catalase-peroxidase | Ps143739 | 67.43 | 17.06 | -1.98 |
| Tyrosinase | Ps131976 | 17.39 | 0.55 | -4.98 |
| Tyrosinase | Ps135423 | 21.67 | 2.75 | -2.98 |
| Tyrosinase | Ps133889 | 55.92 | 26.41 | -1.08 |
| Cellulase | Ps132587 | 14.18 | 3.85 | -1.88 |
| Alpha-glucosidase | Ps130345 | 8.29 | 1.38 | -2.59 |
| 1,3-beta-glucanosyltransferase | Ps128950 | 22.48 | 2.20 | -3.35 |
| Metal-dependent hydrolase | Ps135377 | 9.90 | 0 | -9.95 |
| Calreticulin | Ps109612 | 1 665.34 | 506.44 | -1.72 |
| Sterol esterase | Ps128947 | 7.22 | 1.10 | -2.71 |
本文首次通过转录组学方法揭示了大豆疫霉中特定的功能基因PsMPK 1 沉默后基因组的转录变化。DGE分析在MPK1-SP中没有检测到转录本,再次表明PsMPK 1 已经沉默。样品间基因表达水平的整体相关性分析和实时定量PCR验证的结果表明,数据具有较高的可靠性。
分析发现突变体菌株中许多细胞壁相关基因以及分泌蛋白(特别是致病相关蛋白)编码基因的表达显著降低,解释了PsMPK 1 沉默后大豆疫霉细胞壁完整性受损和致病性丧失等生物学性状[2]产生的原因。细胞壁在维持细胞内外传递信号以及抵御环境胁迫等方面具有重要的作用[10],除了发现大量细胞壁相关基因下调表达之外,在GO分析中也发现较多的对刺激或胁迫应答相关基因显著下调。因此,细胞壁完整性受损可能导致病原菌无法有效感知与植物的相互作用并抵御植物的免疫反应,这可能也是许多致病相关基因无法正常表达的原因。
MAPK与蛋白激酶之间通过磷酸化和去磷酸化实现信号的激活与关闭,如经典的MAPK级联途径中,信号通过上游MAPKKK和MAPKK等蛋白的逐级磷酸化传递给MAPK,被激活的MAPK通过调控下游的转录因子等进而影响相关基因的转录[11]。本研究分析表明:PsMPK 1 的沉默造成大豆疫霉基因组约1/5的基因发生明显的转录变化,且相对较多的基因显著下调表达;差异表达基因在蛋白激酶和转录因子等信号因子和调控因子编码基因中的比例基本一致,然而细胞壁相关基因和分泌蛋白编码基因中更高比例的基因发生转录变化且更多的为下调表达。由于信号因子和调控因子可能影响细胞中生物过程的各个方面,且往往存在反馈调节机制,推测这些基因的转录变化维持在一个相对较小的、细胞正常生命活动可接受的程度;而特定信号因子或调控因子的转录变化或变化效应往往造成其调控的一系列基因的显著转录变化,如大量的细胞壁相关基因和分泌蛋白编码基因等在PsMPK 1 沉默后转录显著下调。研究与PsMPK1信号途径相关的候选基因的功能与相互作用关系,将进一步揭示大豆疫霉中MAPK的调控网络。
| [1] | Turra D,Segorbe D,Pietro A D. Protein kinases in plant-pathogenic fungi:conserved regulators of infection[J]. Annual Review of Phytopathology,2014,52:267-288. |
| [2] | Li A N,Zhang M,Wang Y L,et al. PsMPK1,an SLT2-type mitogen-activated protein kinase,is required for hyphal growth,zoosporogenesis,cell wall integrity,and pathogenicity in Phytophthora sojae[J]. Fungal Genetics and Biology,2014,65:14-24. |
| [3] | de Groot P W J,Ram A F,Klis F M. Features and functions of covalently linked proteins in fungal cell walls[J]. Fungal Genetics and Biology,2005,42(8):657-675. |
| [4] | Meijer H J G,van de Vondervoort P J,Yin Q Y,et al. Identification of cell wall-associated proteins from Phytophthora ramorum[J]. Molecular Plant-Microbe Interactions,2006,19(12):1348-1358. |
| [5] | Tyler B M. Phytophthora sojae:root rot pathogen of soybean and model oomycete[J]. Molecular Plant Pathology,2007,8(1):1-8. |
| [6] | Ye W W,Wang X L,Tao K,et al. Digital gene expression profiling of the Phytophthora sojae transcriptome[J]. Molecular Plant-Microbe Interactions,2011,24(12):1530-1539. |
| [7] | Tyler B M,Tripathy S,Zhang X M,et al. Phytophthora genome sequences uncover evolutionary origins and mechanisms of pathogenesis[J]. Science,2006,313(5791):1261-1266. |
| [8] | 叶文武,王源超,窦道龙. SeqHunter:序列搜索与分析的生物信息学软件包[J]. 生物信息学,2010,8(4):364-367,377. Ye W W,Wang Y C,Dou D L. SeqHunter:a bioinformatics toolbox for local Blast and sequence analysis[J]. China Journal of Bioinformatics,2010,8(4):364-367,377(in Chinese with English abstract). |
| [9] | Winnenburg R,Baldwin T K,Urban M,et al. PHI-base:a new database for pathogen host interactions[J]. Nucleic Acids Research,2006,34(Database issue):D459-D464. |
| [10] | Valiante V,Macheleidt J,Foge M,et al. The Aspergillus fumigatus cell wall integrity signaling pathway:drug target,compensatory pathways,and virulence[J]. Frontiers in Microbiology,2015,6:325. |
| [11] | Krishna M,Narang H. The complexity of mitogen-activated protein kinases(MAPKs)made simple[J]. Cellular and Molecular Life Sciences,2008,65(22):3525-3544. |




