文章信息
- 黄欣梅, 李银, 赵冬敏, 刘宇卓, 杨婧, 韩凯凯.
- HUANG Xinmei, LI Yin, ZHAO Dongmin, LIU Yuzhuo, YANG Jing, HAN Kaikai.
- 11株H9N2亚型禽流感病毒神经氨酸酶基因遗传分析
- Genetic analysis of neuraminidase genes of 11 H9N2 subtype avian influenza viruses
- 南京农业大学学报, 2016, 39(01): 150-155
- Journal of Nanjing Agricultural University, 2016, 39(01): 150-155.
- http://dx.doi.org/10.7685/jnau.201503052
-
文章历史
- 收稿日期:2015-03-26
禽流感(avian influenza,AI)是由正黏病毒科、流感病毒属的A型流感病毒引起的以禽类呼吸系统和全身败血为特征的禽类传染病。H9N2亚型禽流感病毒(avian influenza virus,AIV)属于低致病性禽流感病毒,家禽感染后发病率和死亡率较低,但是因为分布广泛,能造成宿主的免疫抑制及引起人类感染,其危害性不容忽视。1966年Homme等[1]从火鸡体内分离到第一株H9N2亚型AIV,之后该亚型病毒在世界范围内广泛流行。我国是1994年首次报道鸡感染H9N2亚型AIV,目前其已成为我国主要的禽流感流行毒株,给家禽养殖业造成了巨大的经济损失。1999年以来,国内外已报道多起人感染H9N2亚型AIV病例[2]。2013年爆发的人感染H7N9亚型AIV与H9N2亚型AIV关系密切,已证实其内部基因是通过基因片段重组从H9N2亚型AIV获得的[3]。H9N2亚型AIV对人类健康是一个潜在的威胁,因此加强对H9N2亚型AIV的监测还具有重要的公共卫生意义。
神经氨酸酶(neuraminidase,NA)是流感病毒粒子表面的一种重要的糖蛋白,可水解细胞表面受体特异性糖蛋白末端的N-乙酰基神经氨酸,帮助成熟的病毒粒子脱离宿主细胞并感染新的细胞。NA还能协助病毒穿透宿主呼吸道表面的黏膜层,侵入呼吸道上皮细胞内,并将诱捕受体从上皮细胞表面的糖萼上移除,从而促进病毒在体内的传播[4]。有报道[5]称NA能激活转化生长因子β(transforming growth factor-β,TGF-β),进而抑制炎症和免疫损伤的发展,有利于病毒的持续感染和传播。NA还具有较强的免疫原性,能有效诱导机体产生特异性的免疫力,用NA免疫小鼠可减轻疾病症状,减少肺脏内病毒效价以及排毒量[6]。NA不仅是制备疫苗的重要抗原成分,还是流感治疗药物的作用靶点,抗流感药物通过结合NA的酶活性位点抑制NA的活性,对于治疗流感病毒感染有显著效果。但由于NA具有较高的变异性,易发生抗原漂移以及产生耐神经氨酸酶抑制剂的毒株。本文分析了华东地区2008—2013年分离的H9N2亚型AIV的NA基因序列,从分子水平了解本地区H9N2亚型AIV NA基因的变异特点和耐药性情况,为H9N2亚型禽流感的防控提供理论依据。
1 材料与方法 1.1 病毒11株H9N2亚型AIV毒株由农业部兽用生物制品工程技术重点实验室于2008—2013年从江苏、安徽、浙江等省有温和流感症状或无明显病症的家禽中分离,并通过血清学鉴定。将病毒接种10日龄SPF鸡胚,72 h后收集尿囊液,测定血凝效价后置于-70 ℃保存备用。
1.2 主要试剂RNase 抑制剂、AMV反转录酶、Ex Taq酶、pMD18-T载体克隆试剂盒、限制性内切酶等购自大连宝生物有限公司;体液病毒DNA/RNA抽提试剂盒、胶回收试剂盒、小量提取质粒试剂盒等购自Axygen公司。
1.3 引物设计与合成根据GenBank已发表的H9N2亚型AIV NA基因序列,设计1对引物用以扩增NA基因全长序列:上/下游引物NAF/NAR:5′-AGCAAAAGCAGGAGT-3′/5′-AGTAGAAACAAGGAGTTTTT-3′。反转录引物使用流感病毒通用引物Uni12:5′-AGCAAAAGCAGG-3′。扩增片段长度约为1.4 kb。引物由南京金斯瑞公司合成。
1.4 NA基因的RT-PCR扩增与测定用体液病毒DNA/RNA抽提试剂盒提取病毒RNA,按AMV反转录酶说明书进行反转录。以合成的cDNA为模板进行PCR 扩增,PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。反应结束后取PCR产物在10 g·L-1琼脂糖凝胶中进行电泳。回收目的片段,与pMD18-T克隆载体连接,转化大肠杆菌DH5α感受态细菌。经酶切鉴定后,挑选阳性克隆质粒送南京金斯瑞公司进行基因序列测定。
1.5 NA基因的序列分析用Lasergene 7.1 DNAStar软件分析NA基因的同源性,并对唾液酸结合位点、 蛋白酶活性位点、潜在糖基化位点、抗原决定簇的氨基酸差异进行分析。应用MEGA 5.05软件对本研究获得的11株NA基因序列与GenBank中已发表的H9N2亚型禽流感经典毒株和疫苗毒株进行遗传进化关系分析,构建系统进化树。采用基于默认参数的邻接法(Neighbor-joining)计算遗传距离,自举检验值(Bootstrap test)设置为重复1 000次。参考毒株的名称和GenBank登录号如下:TU/WS/1/66(CY130056)、CK/GD/SS/94(DQ874395)、CK/BJ/1/94(AF156398)、CK/SD/6/96(AF508591)、CK/HK/G9/97(AF156391)、DK/HK/Y280/97(AF156394)、DK/HK/Y439/97(AF156395)、QU/HK/G1/97(AF156396)、CK/SH/F/98(AY253754)、DK/NJ/01/99(DQ681205)、QU/ST/11195/05(EF155120)、DK/HN/S4111/11(CY146662)、CK/RZ/267/13(KF259593)、CK/WZ/606/13(KF259563)。
2 结果与分析 2.1 NA基因的扩增及序列测定分别提取11株H9N2亚型AIV的RNA,用RT-PCR方法扩增NA基因片段,大小约为1.4 kb,与预期相符。测序结果经NCBI在线比对确认为H9N2亚型AIV的NA基因。分离毒株名称、宿主、分离时间和分离地点见表1。
| 病毒名称Virus name | 宿主Host | 分离时间Isolation time | 分离地点Isolation site | 病毒名称Virus name | 宿主Host | 分离时间Isolation time | 分离地点Isolation site | |
| CK/AH/FT/08 | Chicken | 2008 | Anhui Province | CK/ZJ/JX1603/13 | Chicken | 2013 | Zhejiang Province | |
| CK/AH/MG08/08 | Chicken | 2008 | Anhui Province | CK/JS/WX0201/13 | Chicken | 2013 | Jiangsu Province | |
| CK/AH/HF1004/10 | Chicken | 2010 | Anhui Province | CK/JS/WX0204/13 | Chicken | 2013 | Jiangsu Province | |
| CK/JS/JT1005/10 | Chicken | 2010 | Jiangsu Province | DK/JS/WX5001/13 | Duck | 2013 | Jiangsu Province | |
| CK/JS/SZ0207/13 | Chicken | 2013 | Jiangsu Province | DK/JS/KS0802/13 | Duck | 2013 | Jiangsu Province | |
| CK/ZJ/JX1605/13 | Chicken | 2013 | Zhejiang Province |
通过DNAStar 软件比对分析,发现本试验分离的11株毒株NA基因核苷酸序列同源性为86.2%~99.5%,推导的氨基酸同源性为86.9%~99.1%。2株鸭源毒株DK/JS/KS0802/13和DK/JS/WX5001/13与H9N2亚型AIV欧亚分支中G1-like亚系的代表株QU/HK/G1/97的同源性较高,核苷酸序列同源性为91.6%~92.6%,氨基酸同源性为90.9%~92.2%;其余9株鸡源毒株与Y280-like亚系的代表株DK/HK/Y280/97的同源性较高,核苷酸序列同源性为92.2%~94.5%,氨基酸同源性为92.5%~95.9%。
2.3 NA基因的系统进化分析将11株分离毒株的NA基因核苷酸序列与参考毒株进行比较,并绘制系统进化树。如图1所示:11株分离毒株均属于欧亚分支,其中2株鸭源分离株位于以QU/HK/G1/97为代表株的G1-like亚分支内,其余9株鸡源分离株位于以DK/HK/Y280/97为代表株的Y280-like亚分支内,但与疫苗株亲缘关系较远,位于不同的进化分支。
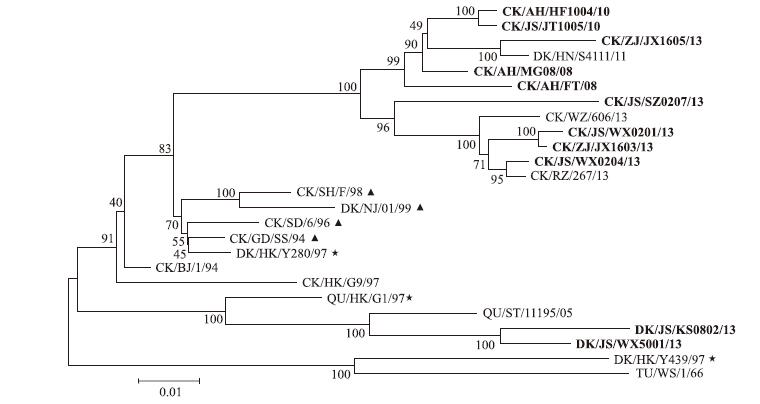 |
图 1 H9N2亚型AIV NA基因系统进化树分析 Fig. 1 Phylogenetic tree of H9N2 subtype AIV based on the NA gene
本研究分离的11株毒株用粗体标记,疫苗株用▲标记,欧亚分支内各亚系的代表毒株用★标记。 The virus strains characterized in this study were marked in bold,vaccine strains were marked with triangles,and the isolates marked with stars stand for representative strains of sublineage group. |
本研究中11株H9N2亚型AIV的NA基因中,2株鸭源毒株DK/JS/KS0802/13和DK/JS/WX5001/13的开放阅读框(ORF)含1 404个核苷酸,编码467个氨基酸,其余9株鸡源毒株的ORF含1 401个核苷酸,编码466个氨基酸。流感病毒NA蛋白的颈部区域可以有不同长度氨基酸的缺失。分析分离毒株的NA基因序列后,发现2株鸭源毒株的氨基酸序列在颈部第39、40位有Q、N 2个氨基酸的缺失,G1-like亚系的毒株QU/HK/G1/97和QU/ST/11195/05在该位点也有相同的氨基酸缺失。9株鸡源毒株在NA蛋白颈部第63、64、65位有T、E、I 3个氨基酸的缺失,2株鸭源毒株在该位点没有发生氨基酸缺失,但第65位氨基酸由I突变为V(表2)。
| 毒株Virus | 颈部缺失Deletion in the stalk | N-糖基化位点Potential N-glycosylation sites | |||||||||||||
| 39-40 | 63-65 | 61 | 69 | 70 | 86 | 143 | 146 | 155 | 200 | 234 | 264 | 306 | 368 | 402 | |
| CK/AH/FT/08 | QN | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | NIS | - | - | - |
| CK/AH/MG08/08 | QN | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | NIS | - | - | - |
| CK/AH/HF1004/10 | QN | - | - | NST | - | - | - | NGT | - | NAT | NGT | NIS | - | - | - |
| CK/JS/JT1005/10 | QN | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | NIS | - | - | - |
| CK/JS/SZ0207/13 | KD | - | - | NNT | NTT | NWS | - | NGT | - | NAT | NGT | NIS | - | NGS | - |
| CK/ZJ/JX1605/13 | QT | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | NIS | - | - | - |
| CK/ZJ/JX1603/13 | QN | - | - | NST | - | NWS | NHS | NGT | - | NAT | NGT | NIS | - | NNS | NWS |
| CK/JS/WX0201/13 | QN | - | - | NST | - | - | NHS | NGT | - | NAT | NGT | NIS | - | NNS | NWS |
| CK/JS/WX0204/13 | QN | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | NIS | - | NDS | NWS |
| DK/JS/WX5001/13 | - | TEV | NTT | NNT | - | NWS | - | NGT | - | NAT | NGT | - | - | - | NWS |
| DK/JS/KS0802/13 | - | TEV | NTT | NNT | - | NWS | - | NGT | - | NAT | NGT | - | - | - | NWS |
| QU/HK/G1/97 | - | TEI | NIT | NNT | NTT | NWS | - | NGT | - | NAT | NGT | - | - | - | - |
| DK/HK/Y280/97 | QN | - | - | NST | - | NWS | - | NGT | - | NAT | NGT | - | - | - | NWS |
| DK/HK/Y439/97 | QN | TEI | NIT | NST | - | NWS | - | NGT | NRT | NAT | NGT | - | NMT | - | - |
| 注:“-”表示颈部缺失或失去潜在的N-糖基化位点。 | |||||||||||||||
| Note:“-”means stalk deletions or no potential N-glycosylation sites. | |||||||||||||||
糖基化位点被证实对于维持NA蛋白分子的结构和正常生物功能具有重要意义[7]。如表2所示:对本研究获得的分离毒株及欧亚分支内3个亚系代表毒株的NA蛋白潜在的N-糖基化位点(拥有N-X-T/S基序,X为除P以外的氨基酸)进行分析,发现在69、146、200、234位氨基酸的糖基化位点比较保守;所有分离毒株均失去了155、306位点的糖基化位点;9株鸡源毒株由于在63位氨基酸发生了缺失,因而失去了61位点处的潜在糖基化位点;CK/JS/SZ0207/13毒株的第70位氨基酸由S突变为N,因此在该处增加了一个糖基化位点;CK/AH/HF1004/10和CK/JS/WX0201/13失去了86位点处的糖基化位点;CK/ZJ/JX1603/13和CK/JS/WX0201/13毒株的第143位氨基酸突变为N,出现了一个新的糖基化位点;9株鸡源毒株在264位点增加了一个糖基化位点;4株毒株在368位点出现新的糖基化位点;6株毒株由于402位氨基酸发生突变而失去了该处的糖基化位点。
唾液酸结合位点,即红细胞结合位点(hemadsorbing site,HB)位于NA蛋白的表面,远离神经氨酸酶酶活性区域,对病毒的复制具有重要作用。本试验分析了NA蛋白HB位点的氨基酸变异情况,如表3所示,11株分离毒株在431~433氨基酸位点区域比较保守,均为PQE。而在366~373位点区域的367、368和369氨基酸位点,以及399~404位点区域的401、402氨基酸位点表现出较大的变异。
| 毒株Virus | 红细胞结合位点 Hemadsorbing(HB)sites | 毒株Virus | 红细胞结合位点 Hemadsorbing(HB)sites | |||||
| 366-373 | 399-404 | 431-433 | 366-373 | 399-404 | 431-433 | |||
| CK/AH/FT/08 | IKSDSRSG | DSDSWS | PQE | CK/JS/WX0201/13 | -RNN---- | --EN-- | --- | |
| CK/AH/MG08/08 | -------- | E----- | --- | CK/JS/WX0204/13 | -RN----- | --EN-- | --- | |
| CK/AH/HF1004/10 | ----L--- | ------ | --- | DK/JS/WX5001/13 | -NK----- | --NN-- | --- | |
| CK/JS/JT1005/10 | -------- | ------ | --- | DK/JS/KS0802/13 | -NK----- | --NN-- | --- | |
| CK/JS/SZ0207/13 | --NG---- | ---D-- | --- | QU/HK/G1/97 | --K----- | --DIR- | --- | |
| CK/ZJ/JX1605/13 | -------- | ------ | --- | DK/HK/Y280/97 | --E----- | --DN-- | --- | |
| CK/ZJ/JX1603/13 | -RNN---- | --EN-- | --- | CK/HK/Y439/97 | -SK----- | -NNN-- | -K- | |
当流感病毒NA蛋白酶活性位点中的氨基酸发生突变时,病毒将获得抗神经氨酸酶抑制剂如扎那米韦和奥赛米韦的抗药性。对本研究中分离毒株NA蛋白的酶活性位点进行分析,发现这些位点都非常保守,没有发生突变,表明这些毒株还没有对神经氨酸酶抑制剂产生抗药性(表3)。
NA蛋白含有7个抗原决定簇,均位于其头部区域,分别为位于153、197~199、328~336、339~347、367~370、400~403和431~434位氨基酸[8]。经过氨基酸序列比对分析,发现11株分离毒株NA蛋白抗原决定簇中的153、199、328、331、346、136、368、369、401、402和434位氨基酸变异较为活跃。
3 讨论H9N2亚型AIV遗传进化关系分为北美种系和欧亚种系两大分支,欧亚分支进一步分为Y280-like、G1-like以及Y439-like 3个亚系,其 代 表 株 分 别 为DK/HK/Y280/97、QU/HK/G1/97和DK/HK/Y439/97[9]。本研究中的11株分离毒株均属于欧亚分支,其中9株鸡源病毒属于Y280-like亚系,但与疫苗株之间存在一定的遗传差距,说明近年来华东地区鸡群中H9N2亚型AIV正在不断变异演化,与疫苗株之间的遗传距离不断增加,应考虑筛选与现阶段流行毒株相匹配的疫苗候选毒株。值得注意的是,2株鸭源病毒与分离自鹌鹑的QU/HK/G1/97亲缘关系较近,属于G1-like亚系。而QU/HK/G1/97毒株的内部基因与同期在香港感染人的H5N1亚型AIV高度同源,使得G1-like亚系病毒备受关注,被认为可能对哺乳动物具有潜在致病性。之前G1-like亚系主要在鹌鹑中流行[10],此次从鸭体内分离到该亚系毒株,说明G1-like亚系毒株已在华东地区水禽中流行,很有可能会传入鸡群中,鉴于该亚系病毒的公共卫生意义,应加强对该类病毒的监测。
在不同亚型和同亚型流感病毒之间,NA蛋白颈部的氨基酸序列差异较大,并且长度也不尽相同。NA 蛋白颈部长度的变化会影响神经氨酸酶的活性,引起病毒复制能力的变化,进而影响病毒的毒力[11]。Y280-like 分支的毒株在颈部63~65位点缺失3个氨基酸,G1-like分支在颈部38、39位点缺失2个氨基酸。Liu等[12]报道NA蛋白颈部63~65位点缺失3个氨基酸是大陆鸡群中H9N2亚型AIV的分子标志,本研究中分离的9株鸡源毒株符合这一特点。2株鸭源毒株在38、39位点缺失 2个氨基酸,符合G1-like分支的特点。NA蛋白颈部氨基酸的缺失是否会影响病毒毒力尚待进一步研究。
N-糖基化位点对病毒毒力、宿主免疫反应以及神经氨酸酶的作用都至关重要。较早的研究表明,中国大陆地区H9N2亚型AIV的NA蛋白中第86、146、200、234和402位潜在糖基化位点高度保守[13],但本试验中有2株毒株缺失了第86位糖基化位点,6株毒株失去了第402位糖基化位点。与Y439-like亚系的代表株相比,所有分离毒株均失去了155、306位点的糖基化位点;9株鸡源毒株属于Y280-like亚系,与2株G1-like亚系的鸭源毒株相比存在较大差异,分别在61位缺失了1个糖基化位点,在264位增加了1个糖基化位点;此外,CK/JS/SZ0207/13毒株在第70位增加了1个糖基化位点,2株毒株的第143位点出现了1个新的糖基化位点,4株毒株在368位点出现新的糖基化位点。与早期报道相比,本研究中分离毒株NA蛋白潜在的糖基化位点出现了较多的变异,这是否会影响病毒的生物学特性还有待进一步的研究。
NA蛋白通过唾液酸结合位点,即红细胞结合位点(HB)与另一表面糖蛋白血凝素(HA)结合,当HA结合到感染细胞的受体上后,NA蛋白就裂解细胞受体上的唾液酸残基,使病毒完全进入到细胞中。如果HB位点发生变异,可能就会影响病毒的繁殖特性。Kobasa等[14]报道早期北美和韩国水禽中H9N2亚型AIV NA蛋白的HB位点在366~373区域和399~404区域非常保守,氨基酸序列分别是ISKDSRSG和DNNNWS。而本研究中分离毒株的HB位点氨基酸序列差异较大,主要表现在第367位氨基酸残基由S突变为K/R/N,第368位由K突变为S/N,第369位由D突变为G/N,第400位由N突变为S,第401位由N突变为D/E,第402位由N突变为S/D。
分离毒株的NA蛋白抗原决定簇中有部分氨基酸序列变异较为活跃。由于本地区普遍使用H9N2亚型AIV灭活疫苗,推测抗原决定簇的变异可能与抗体选择有关。HB位点中变异较大的位点恰好处于抗原决定簇区域,其发生突变可能也与疫苗的使用有关。
当流感病毒NA蛋白酶活性位点中的氨基酸发生特定突变时,病毒将获得抗神经氨酸酶抑制剂的抗药性[15]。本研究中分离毒株NA蛋白的酶活性位点都非常保守,没有发生突变,表明这些毒株还没有产生对神经氨酸酶抑制剂的抗药性。
本试验对华东地区2008—2013年分离的11株H9N2亚型禽流感毒株的NA基因进行了序列分析。由于采用灭活疫苗对H9N2亚型禽流感进行防控,在长期的免疫选择压力下,病毒更易发生基因突变和重组,遗传演化愈加复杂。本试验结果表明,本地区家禽中的H9N2亚型禽流感毒株NA蛋白的糖基化位点、HB位点以及抗原决定簇都发生较大变异,遗传关系与疫苗株愈来愈远,在今后的防控工作中应考虑当前流行毒株与疫苗株之间的遗传差异。此次从鸭群中分离到2株G1-like亚系的毒株,鉴于该亚系毒株具有重要的公共卫生学意义,应加强水禽中G1-like亚系AIV的监测,密切关注其是否传入鸡群并发生重组。加强对本地区H9N2亚型AIV的持续监测,对掌握该类病毒的变异情况,制定防控措施和筛选疫苗毒株具有重要意义。
| [1] | Homme P J,Easterday B C. Avian influenza virus infections:Ⅰ. Characteristics of influenza A-turkey-Wisconsin-1966 virus[J]. Avian Dis,1970,14:66-74. |
| [2] | 郭元吉,谢健屏,王敏,等. 从我国人群中再次分离到H9N2亚型流感病毒[J]. 中华实验和临床病毒学杂志,2000,14(3):209-212. Guo Y J,Xie J P,Wang M,et al. A strain of influenza A H9N2 virus repeatedly isolated from human population in China[J]. Chinese J Exp Clin Virol,2000,14(3):209-212(in Chinese with English abstract). |
| [3] | Lam T T,Wang J,Shen Y,et al. The genesis and source of the H7N9 influenza viruses causing human infections in China[J]. Nature,2013,502(7470):241-244. |
| [4] | Cohen M,Zhang X Q,Senaati H P,et al. Influenza A penetrates host mucus by cleaving sialic acids with neuraminidase[J]. Virol J,2013,10:321. |
| [5] | Carlson C M,Turpin E A,Moser L A,et al. Transforming growth factor-β:activation by neuraminidase and role in highly pathogenic H5N1 influenza pathogenesis[J]. PLoS Pathog,2010,6:e1001136. |
| [6] | Schulman J L,Khakpour M,Kilbourne E D. Protective effects of specific immunity to viral neuraminidase on influenza virus infection in mice[J]. J Virol,1968,2:776-778. |
| [7] | Skehel J J,Stevens D J,Daniels R S,et al. A carbohydrate side chain on hemagglutinims of Hong Kong influenza viruses inhibits recognition by a monoclonal antibody[J]. Proc Natl Acad Sci USA,1984,81(6):1779-1783. |
| [8] | 李向忠,方芳,陈则. 流感病毒神经氨酸酶不同区域的作用[J]. 生命科学研究,2005,9(2):55-61. Li X Z,Fang F,Chen Z. Roles of different domains of influenza virus neuraminidase[J]. Life Science Research,2005,9(2):55-61(in Chinese with English abstract). |
| [9] | Guo Y J,Krauss S,Senne D A. et al. Characterization of the pathogenicity of members of the newly established H9N2 influenza virus lineages in Asia[J]. Virology,2000,267(2):279-288. |
| [10] | Xu K M,Li K S,Smith G J,et al. Evolution and molecular epidemiology of H9N2 influenza A viruses from quail in southern China,2000 to 2005[J]. J Virol,2007,81:2635-2645. |
| [11] | Castrucci M R,Kawaoka Y. Biologic importance of neuraminidase stalk length in influenza A virus[J]. J Virol,1993,67(2):759-764. |
| [12] | Liu J H,Okazaki K,Wu Q M,et al. Phylogenetic analysis of neuraminidase gene of H9N2 influenza viruses prevalent in chickens in China during 1995-2002[J]. Virus Genes,2003,27:197-202. |
| [13] | 郭霄峰,廖明,辛朝安. H9N2亚型禽流感病毒纤突蛋白基因的分析[J]. 中国人兽共患病杂志,2002,18(4):17-19. Guo X F,Liao M,Xin C A. Analysis of glycoprotein gene of H9N2 AIV[J]. Chinese Journal of Zoonoses,2002,18(4):17-19(in Chinese with English abstract). |
| [14] | Kobasa D,Rodger M E,Well K,et al. Neuraminidase hemadsorption activity,conserved in avian influenza A viruses,does not influence viral replication in ducks[J]. J Virol,1997,71:6706-6713. |
| [15] | Hurt A C,Holien J K,Parker M,et al. Zanamivir-resistant influenza viruses with a novel neuraminidase mutation[J]. J Virol,2009,83:10366-10373. |




