文章信息
- 朱业培, 王玮, 吕青骎, 范巧君, 徐幸莲, 周光宏. 2015.
- ZHU Yepei, WANG Wei, LÜ Qingqin, FAN Qiaojun, XU Xinglian, ZHOU Guanghong. 2015.
- 基于基因芯片技术检测6种动物源性成分
- Detection of six kinds of animal-derived ingredients using gene chips
- 南京农业大学学报, 38(6): 1003-1008
- Journal of Nanjing Agricultural University, 38(6): 1003-1008.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.06.020
-
文章历史
- 收稿日期: 2015-06-30
肉类食品的安全追溯及种属鉴别已逐渐受到各国政府部门、食品生产企业和消费者的关注[1]。为保证肉类产品质量安全,保障消费者的合法权益,避免市场不公平竞争现象的发生,亟待建立一种能快速准确鉴别、检测动物源性成分的方法。当前,国内外对于肉类品种的鉴定方法主要有蛋白质鉴定(ELISA[2]、色谱法[3]等)和核酸鉴定(PCR方法[4]等)两大类。蛋白质技术已成功应用于生鲜肉类物种鉴别,但当肉类经过切碎、混合、蒸煮、熏烤等深加工后,肉中蛋白质的结构和稳定性受到破坏,物种特异性蛋白或抗原决定簇也遭到破坏[5]。因此,蛋白质技术鉴定肉类物种的可靠性和稳定性较差,无法满足现代肉类掺杂掺假检测的要求。相比之下,由于DNA的相对稳定性,使核酸技术在深加工产品的肉类物种检测方面具有很大的优势,只要知道动物物种的特异性基因或蛋白的基因序列就能实现检测[6]。此外,核酸方法的检测灵敏度通常要高于蛋白质方法,现已成为肉类种属鉴别最常用的方法[7, 8]。但当前无论是蛋白质检测还是核酸检测,通常只能同时鉴别一种或少量几种动物种类,而在实际检测中,由于肉类食品种类繁多、成分复杂,更需要对多种动物源性成分进行同时检测鉴别。基因芯片技术作为一种新型的检测平台,因其具有高准确性、高灵敏度、高通量等优势已逐渐成为肉类种属鉴别研究的热点[9, 10]。为此,本试验以物种特异性DNA为检测目标,选取牛、羊、猪、马、鹿、兔6种动物样品,建立基于基因芯片技术同时检测6种动物源性成分的方法,弥补常规检测方法的缺陷,旨在为我国肉及肉制品快速检测和种属鉴别提供有效的技术支持。
1 材料与方法
1.1 样品
1.1.1 试验材料
各种市售食品,如牛肉丸、羊肉卷、牛肉干、马肉肠、酱鹿肉和猪肉松以及牛肉、羊肉、猪肉、马肉、鹿肉、兔肉、鸡肉等样品,均购于南京市农贸市场或超市。
1.1.2 引物和探针
试验所采用引物和探针来自文献报道和本课题组自主研发,质控探针包括阳性对照探针(NH2-(T)10-CCAAATTGATCCCACCC)和Cy5标记互补链(Cy5-(T)10-GGGTGGGATCAATTTGG)。引物和探针采用Oligo 6.0软件设计,由上海英骏生物技术有限公司合成和修饰,具体序列见表 1。
| 物种Species | 靶基因Target gene | 序列Sequence | 扩增长度/bp Size |
| 鹿Deer | cytb | F:5′-ATCATCGCAGCACTCGCTATAGTACACT-3′ R:5′-ATCTCCAAGCAGGTCTGGTGCGAATAATA-3′ P:5′-NH2-(T)10-GGGTATCTTACTTCTAATACTCTTCCTAAT-3′ | 195 |
| 兔Rabbit | cytb | F:5′-GTCTTAATTCACCTCCTCTTTC-3′ R:5′-GGAGAAGAATGGCTACAAGGAAA-3′ P:5′-NH2-(T)10-AGGAATTCCTTCAAACTCACATAAAAT-3′ | 139 |
| 牛Beef | mtDNA | F:5′-GCCATATACTCTCCTTGGTGACA-3′ R:5′-GTAGGCTTGGGAATAGTACGA-3′ P:5′-NH2-(T)10-CACAACTTTTATCACAATCCAGAACTGACACCAAC-3′ | 271[11] |
| 马Horse | cytb | F:5′-ACAACGAAGCATAATATTCCGGCCTCT-3′ R:5′-CAGTTGGCCGATAATTACGTATGGGT-3′ P:5′-NH2-(T)10-GACTCTTAGTGGCAGACTTACTGACACTAACAT-3′ | 127 |
| 羊Sheep | cytb | F:5′-TATTACACCATTAAAGACATCCTAGGT-3′ R:5′-GGTCTCCGAGTAAGTCAGGC-3′ P:5′-NH2-(T)10-TACTAATCCTCATCCTCATGCTA-3′ | 91 |
| 猪Pork | cytb | F:5′-TTGCAAATCCTAACAGGCCTG-3′ R:5′-GTAACTGATGAGAAAGCTGT-3′ P:5′-NH2-(T)10-TAGCAATACATTACACATCAGACACA-3′ | 74 |
| 注:F.正向引物Forward primer;R.反向引物Reverse primer;P.探针Probe | |||
Taq DNA聚合酶、dNTPs、溴化乙锭和50 bp DNA Ladder Marker购自宝生物工程(大连)有限公司;DMSO、SDS、NaBH4、琼脂糖(电泳纯)和三氯甲烷购自上海生工公司;Cy5-dNTPs购自美国Amersham biosciences公司;无水乙醇、异丙醇购自北京六合通公司;醛基化DNA芯片基片及盖片、4孔围栏购自北京 博奥生物有限公司;CTAB提取缓冲液(20 g · L-1 CTAB,1.4 mol · L-1 NaCl,0.1 mol · L-1 Tris-HCl,0.02 mol · L-1 Na2EDTA,pH 8.0)、CTAB沉淀液(5 g · L-1 CTAB,40 mmol · L-1 NaCl)、蛋白酶K溶液(20 mg · mL-1)、TE缓冲液(10 mmol · L-1 Tris-HCl,1 mmol · L-1 EDTA,pH 8.0)、20×SSC溶液(3.0 mol · L-1 NaCl,0.3 mol · L-1 C6H5Na3O7)和1.2 mol · L-1 NaCl均为本实验室自行配制。
1.1.4 仪器
各种量程微量移液器、离心机(5804R型)、PCR扩增仪(Mastercycler gradient)购自德国Eppendorf公司;电热鼓风干燥箱(DGX-9053-B1)购自上海福玛实验设备有限公司;冷冻研磨机(SPEX 6850)购自美国SPEX 6850公司;核酸蛋白分析仪(DU640)购自Beckman公司;电泳仪(DYY-6C)购自北京六一仪器厂;凝胶成像仪购自德国Gene Genius公司;生物芯片点样仪(AD3200)购自美国BioDot公司;激光共聚焦扫描仪(GenePix4200A)购自美国Axon公司。
1.2 方法
1.2.1 样品的制备
称取200 g上述试验材料,采用冷冻研磨机将样品研磨成粉状,并于60 ℃烘干3 h。
1.2.2 DNA的提取
[12] 称取100 mg已制备好的样品于2 mL离心管中,加入1.5 mL CTAB提取缓冲液 和10 μL蛋白酶K溶液。60 ℃振荡过夜后13 000 g离心10 min,转移上清液至新的离心管中;加入750 μL 三氯甲烷后用力振荡,13 000 g离心5 min,将上清液转移到新的离心管中;加入2倍体积的CTAB沉淀液,室温静置60 min,13 000 g离心15 min,弃上清液。加入350 μL NaCl溶液将沉淀进行悬浮;再加入350 μL三氯甲烷,涡旋振荡进行混匀,13 000 g离心10 min,将上清液转移到新的离心管中;加入0.6倍体积的异丙醇,室温放置20 min,13 000 g离心10 min,弃上清液。加入500 μL 70%(体积分数)乙醇溶液洗涤沉淀,溶解于100 μL TE溶液中,立即使用或-20 ℃保存备用。
1.2.3 荧光掺入法PCR扩增
PCR反应体系:10×PCR Buffer(Mg2+ free)(0.25 mmol · L-1)2.5 μL,MgCl2(2.0 mmol · L-1)2 μL,Cy5-dNTPs(0.2 mmol · L-1)0.4 μL,正反向引物(10 μmol · L-1)各0.5 μL,Taq DNA聚合酶(5 U · mL-1)0.2 μL,模板DNA(10~20 ng)1 μL,加灭菌ddH2O至25 μL。空白对照为ddH2O代替DNA。扩增反应程序为:95 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 20 s,40个循环;72 ℃ 5 min。扩增结束后,20 g · L-1琼脂糖凝胶电泳分析结果。
1.2.4 芯片的制备
探针用TE缓冲液(pH 8.0)稀释至终浓度为40 μmol · L-1,将10 μL 0.5 g · mL-1 DMSO加入384孔中,再加入等量的探针溶液,轻轻混匀,并按预先设计的探针顺序(图 1),用生物芯片点样仪将探针点至基片上。点样后基片在湿盒中置于37 ℃烘箱里水合12 h,用2 g · L-1 SDS洗液漂洗2 min,去离子水漂洗3次,每次2 min。再放入2 g ·L-1 NaBH4封闭液中封闭15 min,中间静置5 min;去离子水漂洗3次,每次2 min,2 000 r · min-1离心2 min甩干,4 ℃避光保存备用。
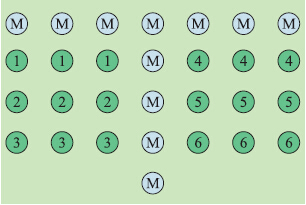 | 图 1 基因芯片探针排布图Fig. 1 Layout of microarray probesM.阳性对照Positive control;1.牛Beef;2.羊Sheep;3.猪Pork;4.马Horse;5.鹿Deer;6.兔Rabbit |
分别取7 μL芯片杂交液(5×SSC,1 g ·L-1 SDS)、6 μL PCR标记产物和1 μL Cy5标记阳性定位探针互补链,混匀后95 ℃变性5 min,立即冰浴5 min。将其按预计排布顺序加入芯片点样区,盖上盖玻片置于杂交盒中50 ℃烘箱中避光杂交2 h。杂交后的芯片依次在洗液Ⅰ(0.3×SSC,0.2 g · L-1 SDS)和洗液Ⅱ(0.06×SSC)中清洗5 min,2 000 r · min-1离心2 min甩干。芯片使用GenePix4200A生物芯片扫描仪扫描,杂交图像与基因芯片背景图对比,对杂交结果进行分析判定。
1.2.6 特异性试验
为了更好地评价本检测方法的特异性,选取牛、羊、猪、马、鹿和兔等6种动物源性DNA模板进行荧光掺入法PCR扩增,分别将扩增产物与芯片进行杂交反应,分析反应结果。
1.2.7 灵敏度试验
1)绝对灵敏度试验:用核酸蛋白分析仪测定抽提样品DNA的质量浓度。将已测定浓度的牛肉和马肉基因组DNA,分别用鸡肉DNA溶液稀释浓度至100 ng · μL-1,取10 μL 100 ng · μL-1 DNA至90 μL鸡肉DNA溶液中,得到10 ng · μL-1的DNA浓度,由此依次类推得到1 000、100、10、1、0.1和0.01 pg · μL-1溶液,用于绝对灵敏度检测。2)取1 g牛肉粉和马肉粉分别加入到9 g鸡肉粉中,充分混合均匀,保证样品在鸡肉粉中均匀散布,制成含有10%(质量分数)牛肉或马肉成分的样品,再取1 g含有10%牛肉或马肉成分的样品加入到9 g鸡肉粉中,充分混合均匀制成含有1%牛肉或马肉成分的样品,按照此方法再依次制成含有0.1%、0.01%、0.001%和0.000 1%牛肉或马肉成分的样品,用于实际灵敏度检测。
1.2.8 基因芯片在市售食品检测中的应用
选用牛肉丸、羊肉卷、牛肉干、马肉肠、酱鹿肉和猪肉松等样品,其样品制备及DNA提取参照1.2.1和1.2.2节,利用已建立的芯片技术分别对上述食品进行检测,以此来验证市售食品标签标识的准确性。
2 结果与分析
2.1 荧光掺入法PCR扩增结果
将牛、羊、猪、马、鹿、兔6种动物源性DNA模板和空白对照进行荧光掺入法PCR扩增,结果如图 2所示:空白对照未出现扩增条带,说明整个PCR反应体系未出现污染;6对引物分别在74、91、127、271、139和195 bp处出现清晰明亮的特异性扩增条带,这表明本试验所建立的PCR反应体系具有特异性强、准确性好等特点,也为下一步的探针特异性杂交打下良好的基础。
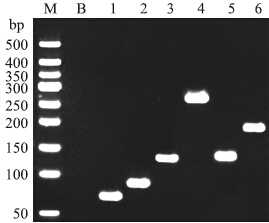 | 图 2 特异性引物动物源性PCR扩增电泳图Fig. 2 Agarose electrophoresis analysis of PCR products by special primersM.50 bp DNA Ladder marker;B.空白对照Blank control(ddH2O);1.猪Pork;2.羊Sheep;3.马Horse;4.牛Beef;5.兔Rabbit;6.鹿Deer |
用特异性引物进行荧光掺入法PCR扩增,按照1.2.5节方法进行杂交与扫描,结果(图 3)显示:阳性质控探针杂交信号正常,6条特异性探针与目的产物分别呈现出明显的杂交信号,可以与背景清晰区分;同一张芯片上其他非目的探针位点则完全与背景一致,没有任何信号;在PCR产物特异性和探针特异性的双重保证下,所有样品都有较好的特异性,没有假阳性结果的出现。表明该芯片技术可以有效、特异地实现6种动物源性成分的种属检测和鉴别;在此基础上,还可进一步开展肉类品种多样本多组分的高通量鉴别体系研究。
 | 图 3 肉类物种的芯片特异性检测结果Fig. 3 Specificity of meat species detection on gene chips1.牛Beef;2.羊Sheep;3.猪Pork;4.马Horse;5.鹿Deer;6.兔Rabbit |
为研究芯片检测的绝对灵敏度和实际灵敏度,选用牛肉和马肉作为芯片检测对象。分别取1.2.7节中不同质量浓度的牛和马DNA溶液5 μL作为PCR反应的模板,使PCR反应体系中的牛和马DNA含量分别为5 000、500、50、5、0.5和0.05 pg。将牛和马PCR扩增产物与芯片进行杂交反应,结果表现出与样品DNA浓度梯度相对应的变化,即特异性结合位点信号随DNA浓度的降低而不断变弱(图 4)。在牛和马DNA浓度为10-6稀释度时,特异性结合位点无杂交信号的产生;而在DNA含量为0.5 pg时,芯片产生较弱的杂交信号。芯片检测牛和马源性成分的绝对灵敏度均可达到0.5 pg。芯片的实际灵敏度可通过检测不同含量的牛肉粉和马肉粉来验证,试验结果表明:当牛肉粉和马肉粉含量为0.000 1%时,芯片上的特异性结合位点无杂交信号产生;而在牛肉粉和马肉粉含量为0.001%时,芯片产生较弱的杂交信号(图 5)。芯片检测牛和马源性成分的实际灵敏度均可达到0.001%。
 |
图 4 基因芯片对牛和马源性成分的绝对灵敏度检测结果Fig. 4 Absolute sensitivity detection result of gene chip method for detecting beef and horse1~6表示牛和马的DNA含量分别为5 000、500、50、5、0.5和0.05 pg。 1-6 indicate 5 000,500,50,5,0.5 and 0.05 pg of beef and horse genomic DNA contents. |
 |
图 5 基因芯片对牛和马源性成分的实际灵敏度检测结果Fig. 5 Practical sensitivity detection result of gene chip method for detecting beef and horse1~6表示牛肉粉和马肉粉的质量分数分别为10%、1%、0.1%、0.01%、0.001%和0.000 1%。 1-6 indicate 10%,1%,0.1%,0.01%,0.001% and 0.000 1% beef and horse powder. |
使用建立的芯片检测方法对市售食品进行检测,以验证该方法的准确性和可靠性。由图 6可知:阳性质控探针杂交信号正常,可以确保芯片检验系统的正常运行,避免假阴性结果的产生。将芯片检测结果与食品标签的标识比对后发现,6种市售食品中仅有牛肉干、马肉肠、酱鹿肉和猪肉松与食品标签的标识相符合;而在牛肉丸中却检测出牛和猪源性成分,在羊肉卷中检测出羊和猪源性成分。研究结果还表明,该方法可有助于解决肉类食品掺假鉴别和食品标签标识的不规范行为。
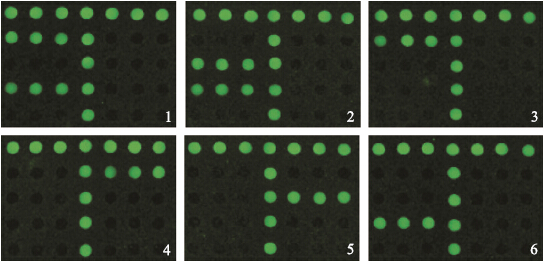 | 图 6 肉类食品的基因芯片检测结果Fig. 6 Detection of meat species in retail meat products using the gene chip method1.牛肉丸Beef meatballs;2.羊肉卷Mutton rolls;3.牛肉干Beef jerky;4.马肉肠Horse sausages;5.酱鹿肉Sauce venison;6.猪肉松Pork floss |
基因芯片技术作为近年发展起来的一种分子生物学技术平台,因其快速、准确、灵敏度高、特异性良好以及高通量等特点被广泛应用于生物学领域。本研究建立了一种运用PCR结合基因芯片技术鉴别牛、羊、猪、马、鹿和兔6种动物源性成分的检测方法,结果表明该方法为肉类食品安全溯源和种类鉴别提供了有效的手段。
基因芯片的特异性是本方法的关键,与所选取的靶基因和筛选的引物和探针密切相关。本试验选取动物mtDNA、cytb基因的保守区域设计物种特异性引物和探针,通过特异性引物和特异性探针的双重筛选作用,避免了基因芯片检测中假阳性结果的出现。基因芯片检测灵敏度也是评价该检测方法是否可行的重要指标之一。基因芯片检测牛肉和马肉成分时绝对灵敏度为0.5 pg,实际检测灵敏度也可达到 0.001%。由于检测技术以及引物和探针的差异性,基因芯片检测灵敏度要高于常规PCR、荧光定量PCR等生物学技术[13, 14],足以满足肉及肉制品中微量动物源性成分检测的需要。
本研究通过对牛、羊、猪、马、鹿、兔6种动物源性成分基因芯片检测方法的初步研究,为进一步拓展多种动物源性成分的高通量检测奠定了基础。该技术大大缩短了检测时间,为进出口检验检疫和食品安全监测提供了高效、快速、准确的检测手段,同时为我国进出口食品、畜产品的快速检测和种类鉴定提供了有效的技术平台,具有广阔的应用前景。
| [1] | Bai H,Wang Z X,Hu R,et al. A 90-day toxicology study of meat from genetically modified sheep overexpressing TLR4 in sprague-dawley rats[J]. PLoS ONE,2015,10(4):e0121636 |
| [2] | Li X P,Yang Y,Bu L,et al. Rosa 26-targeted swine models for stable gene over-expression and Cre-mediated lineage tracing[J]. Cell Research,2014,24(4):501-504 |
| [3] | Lyall J,Irvine R M,Sherman A,et al. Suppression of avian influenza transmission in genetically modified chickens[J]. Science,2011,331(6014):223-226 |
| [4] | 许建香,李宁. 转基因动物生物安全研究与评价[J]. 生物工程学报,2012,28(3):267-281 [Xu J X,Li N. Biosafety assessment of genetically engineered animals:a review[J]. Chin J Biotech,2012,28(3):267-281(in Chinese with English abstract)] |
| [5] | Decock J,Hendrickx W,Thirkettle S,et al. Pleiotropic functions of the tumor- and metastasis-suppressing matrix metalloproteinase-8 in mammary cancer in MMTV-PyMT transgenic mice[J]. Breast Cancer Research,2015,17:38 |
| [6] | Huang Y J,Huang Y,Baldassarre H,et al. Recombinant human butyrylcholinesterase from milk of transgenic animals to protect against organophosphate poisoning[J]. Proc Natl Acad Sci USA,2007,104(34):13603-13608 |
| [7] | Lucocq J M. Efficient quantitative morphological phenotyping of genetically altered organisms using stereology[J]. Transg Res,2007,16(2):133-145 |
| [8] | 朱振营,李风翠,林祥梅,等. 转人乳铁蛋白基因奶牛多重PCR检测方法的研究[J]. 中国畜牧兽医,2011,38(11):73-76 [Zhu Z Y,Li F C,Lin X M,et al. Multiple PCR detection of exogenous gene in hLF transgenic cow[J]. Chinese Animal Husbandry and Veterinary Medicine,2011,38(11):73-76(in Chinese with English abstract)] |
| [9] | 马馨,陈奕冰,张鹏,等. 转基因小鼠的多重PCR快速检测方法的建立[J]. 中国兽医学报,2013,33(9):1403-1406,1416 [Ma X,Chen Y B,Zhang P,et al. The establishment of rapid multiple PCR method for detection of transgenic mice[J]. Chin J Vet Sci,2013,33(9):1403-1406,1416(in Chinese with English abstract)] |
| [10] | 朱振营,林祥梅,刘建,等. 转人溶菌酶基因奶牛多重PCR快速检测方法的建立[J]. 中国畜牧兽医,2011,37(6):40-43 [Zhu Z Y,Lin X M,Liu J,et al. Development of multiplex PCR detection of human lysozyme gene in transgenic cow[J]. Chinese Animal Husbandry and Veterinary Medicine,2011,37(6):40-43(in Chinese with English abstract)] |
| [11] | 台玉磊,韩立强,杨国庆,等. 仔猪组织基因表达中实时定量PCR内参基因的选择[J]. 农业生物技术学报,2010,81(4):732-736 [Tai Y L,Han L Q,Yang G Q,et al. Selection of the reference genes of real-time quantitative PCR in the gene expression of piglet tissues[J]. Journal of Agricultural Biotechnology,2010,81(4):732-736(in Chinese with English abstract)] |
| [12] | 王继英,王彦平,郭建凤,等. 仔猪外周血中内参基因的筛选及细胞因子和受体的表达水平[J]. 中国农业科学,2015,48(7):1437-1444 [Wang J Y,Wang Y P,Guo J F,et al. Selection of reference genes and determination of cytokines and receptor mRNA expression in peripheral blood of piglets[J]. Scientia Agricultura Sinica,2015,48(7):1437-1444(in Chinese with English abstract)] |
| [13] | 岳运锋,吴刚,武玉花,等. 转基因植物中标记基因定性PCR检测方法研究[J]. 中国油料作物学报,2011,33(3):280-289 [Yue Y F,Wu G,Wu Y H,et al. Development of qualitative PCR method targeting marker genes in transgenic plants[J]. Chinese Journal of Oil Crop Sciences,2011,33(3):280-289(in Chinese with English abstract)] |
| [14] | McPherron A C,Lee S J. Double muscling in cattle due to mutations in the myostatin gene[J]. Proc Natl Acad Sci USA,1997,94(23):12457-12461 |
| [15] | Huang X J,Zhang H X,Wang H L,et al. Disruption of the myostatin gene in porcine primary fibroblasts and embryos using Zinc-finger nucleases[J]. Mol Cells,2014,37(4):302-306 |
| [16] | 高飞,唐成程,全龙泉,等. 猪MSTN基因敲除载体的构建及细胞筛选[J]. 中国兽医学报,2013,33(1):142-145 [Gao F,Tang C C,Quan L Q,et al. Construction of MSTN knock-out porcine vector and MSTN knock-out porcine fetal fibroblast[J]. Chin J Vet Sci,2013,33(1):142-145(in Chinese with English abstract)] |
| [17] | 曹随忠,岳成鹤,李西睿,等. 锌指核酸酶技术制备肌肉生长抑制素基因敲除的五指山小型猪成纤维细胞[J]. 遗传,2013,35(6):778-785 [Cao S Z,Yue C H,Li X R,et al. Production of myostatin gene knockout Wuzhishan miniature pig fibroblasts with zinc-finger nucleases[J]. Hereditas,2013,35(6):778-785(in Chinese with English abstract)] |
 2015, Vol. 38
2015, Vol. 38


