文章信息
- 郭玉双, 陈静, 刘筱嘉, 余世洲, 余婧, 林世锋, 邹颉, 付强, 张孝廉, 赵杰宏, 任学良. 2015.
- GUO Yushuang, CHEN Jing, LIU Xiaojia, YU Shizhou, YU Jing, LIN Shifeng, ZOU Jie, FU Qiang, ZHANG Xiaolian, ZHAO Jiehong, REN Xueliang. 2015.
- 烟草中一个LTR类反转录转座子基因的克隆与功能分析
- Isolation and function analysis of a LTR-retrotransposons from tobacco (Nicotiana tabacum)
- 南京农业大学学报, 38(6): 936-942
- Journal of Nanjing Agricultural University, 38(6): 936-942.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.06.010
-
文章历史
- 收稿日期: 2015-03-29
2. 遵义医学院生化 与分子生物学教研室, 贵州 遵义 563009;
3. 番禺出入境检验检疫局, 广东 番禺 510006
2. Department of Biochemistry and Molecular Biology, Zunyi Medical College, Zunyi 563009, China;
3. Panyu Entry-exit Inspection and Quarantine, Panyu 510006, China
反转录转座子(retrotransposon)是真核生物基因组中一类可移动的遗传元件,其转座依赖于反转录酶的反转录反应,以DNA-RNA-DNA方式进行[1]。反转录转座子最早在动物和酵母中发现,在植物基因组中也广泛存在,且占基因组的比例也各不相同。例如,水稻基因中反转录转座子的比例约18%,而玉米中则达到50%~80%,在小麦中高达90%[2]。反转录转座子按其结构可分为两类:长末端重复反转录转座子(long terminal repeats,LTRs)和非长末端重复反转录转座子(non-long terminal repeats,non-LTRs)。LTR在转座子的5′端和3′端有一对同向高度相似的序列,主要包括一些调控元件,通常含有LTR转录需要的启动子和终止子,LTR的转录通常由RNA聚合酶Ⅱ负责,其方向为5′端到3′端。LTR类反转录转座子又可分为Ty1-copia与Ty3-gypsy 2个亚类,它们都编码一个多聚蛋白(polyprotein),随后被蛋白酶切割形成有gag和pol 2个功能多肽。gag蛋白的主要功能是包涵反转录转座时的RNA中间体,而pol编码的蛋白主要是和反转录转座子的生活周期有关[3]。non-LTR根据长度和组成的不同又可分为长散在元件(long interspersed nuclear elements,LINEs)和短散在元件(short interspersed nuclear elements,SINEs)两种类型。LINE在所有植物中都有分布,而SINE仅在被子植物中有分布[4]。
植物反转录转座子在其基因组中具有较多的拷贝,其主要作用是造成染色体的倒位、易位、重复,以及核苷酸序列的缺失、插入等突变,从而对基因组的结构、大小及部分基因功能造成重要影响[5]。
尽管利用生物信息学方法可以对未知功能基因进行预测,但基因的最终功能还是要靠生物学鉴定。随着分子生物学的发展,基因转导、反义技术、RNA干扰、基因敲除、基因芯片技术以及蛋白质组学等方法用于植物基因功能的鉴定。尤其是近年来,VIGS(virus induced gene silencing)作为一种快速的鉴定植物基因功能的方法,越来越受到研究者的重视[6, 7, 8, 9, 10]。
本研究采用同源克隆及RACE的方法,从烟草栽培品种‘贵烟1号’中克隆得到了一个新的反转录转座子,本研究分析了该基因的结构,表达特征,并利用病毒介导的基因沉默在烟草中初步分析了该基因的生物学基本功能,为后期研究该基因调控植物生长发育的机制奠定了基础。
1 材料与方法
1.1 试验材料
供试烟草(Nicotiana tabacum)栽培品种为‘贵烟1号’。
1.2 主要仪器及试剂
BD SMARTTM RACE cDNA Amplification Kit、dNTP、AMV(Reverse Transcriptase XL)、oligo d(T)18、Ribonuclease Inhibitor、DNA Marker DL2000、pMD18-T载体等购自TaKaRa公司;Trans Taq DNA聚合酶HiFi、大肠杆菌(Escherichia coli)DH5α购自北京全式金生物技术有限公司;提取总RNA所用Trizol试剂盒购自Invitrogen公司;氨苄青霉素、DEPC、CaCl2购自上海生工生物工程有限公司;DNA Gel Extraction Kit购自Omega公司,PCR引物由上海英骏生物技术公司合成。
1.3 试验方法
1.3.1 烟草叶片总RNA的提取
取新鲜烟草叶片约0.1 g置于无RNase的研钵中,加入液氮,迅速进行研磨,直至粉末状。按照Invitrogen公司的Trizol试剂说明书,采用Trizol法来提取烟草根部总RNA。将所获得的RNA用凝胶电泳和分光光度计检测浓度和纯度。
1.3.2 cDNA第一链合成参考Protoscript M-MLV First Strand cDNA Synthesis试剂盒,在PCR仪上按以下程序进行反应:37 ℃ 10 min;42 ℃ 1.5 h;99 ℃ 5 min;5 ℃ 5 min。反应结束后取1 μL用作RT-PCR检测,或者保存于-20 ℃冰箱备用。
1.3.3 5′-RACE和3′-RACE1)引物的设计。根据序列检测正确的烟草反转录转座子中间片段,结合RACE试剂盒(Clontech BD SMARTTM RACE cDNA Amplification Kit,TaKaRa,Japan)操作指南中引物设计的基本原则,设计下列特异性引物,引物使用时浓度为10 μmol · L-1,试验中所用的引物见表 1。2)RACE扩增全长基因。 参考Clontech BD SMARTTM RACE试剂盒进行5′-RACE和3′-RACE反应,PCR反应程序为: 95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 5 min,35个循环;72 ℃ 10 min。3)全长基因克隆。对5′-RACE、3′-RACE及中间序列进行拼接获得全长基因,设计全长基因引物QCS和QCAS并通过RT-PCR进行全长基因的克隆验证。4)胶回收、连接、转化和验证。将RT-PCR扩增的产物经8 g · L-1琼脂糖凝胶电泳后按DNA Gel Purlfication Kit试剂盒说明书回收目的片段后,连接到pMDl8-T载体中,然后将其转入DH5α感受态细胞中,挑斑验证正确后送上海英骏生物技术公司测序。
| 引物名称Primer name | 序列(5′→3′)Sequence | 引物目的Purpose of primers |
| 3-GSP-1/3-GSP-2 | GTTGGCACTGTTGCTGGACTA/GCTGACCTATGCAGGCAACCATG | 3′-RACE正向引物Forward primer |
| 5-GSP-1/5-GSP-2 | ACTCCTGGTACTCTTTCACGTCG/CAACAGTCCACGAAGTTCAGAC | 5′-RACE反向引物Reverse primer |
| QCS/QCAS | CCTTCGCAGGTGGTTATGGTG/ATTCGAGCTCGGTACCCGGGG | Ntrt1全长基因克隆Cloning full-length of Ntrt1 |
| UPM | CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT | 通用引物General primer |
| NUP | AAGCAGTGGTAACAACGCAGAGT | 通用引物General primer |
| Q1/Q2 | GCGGGAGTGTATCCAACG/CTTCGCTTCCACCCATTT | 荧光定量检测引物qRT-PCR |
| F1/F2 | GGATCCGTATTTGACAGATGTGGAAGG/TCTAGAAGCAGTGGTATCAACGCA-GAGT | 克隆沉默片段引物,分别引入BamHⅠ/XbaⅠ酶切位点。 Primer for cloning silencing fragment,the underlined is restricting sites. |
序列分析方法采用DNAstar和Vector软件进行,序列比对、氨基酸同源性分析及系统树构建等采用ClustalW和MEGA 4.1软件。
1.3.5 基因沉默载体的构建参照Huang等[10, 11]建立的栽培烟草沉默体系。用PCR扩增Ntrt 1 基因3′-UTR区300 bp的片段,并根据载体的酶切位点在该片段的3端分别添加BamHⅠ和XbaⅠ的酶切位点,将该片段通过BamHⅠ和XbaⅠ双酶切后插入到本试验构建的pBinPLUS-2mDNA1(2mDNA1)中,得到沉默载体2mDNA1-Ntrt1。
1.3.6 病毒接种与沉默效率检测栽培烟草品种‘贵烟1号’盆栽于温室中,温度保持25 ℃,湿度60%。在烟草3~4叶期时进行接种,为了进行病毒诱导的基因沉默测试,所有2mDNA1衍生沉默载体克隆都通过电击法导入农杆菌EHA105,并与含辅助病毒——中国番茄黄化曲叶病毒(tomato yellow leaf curl China virus,TYLCCNV)侵染性克隆pBinPLUS-1.7A的农杆菌等比例混合后注射接种烟草,接种TYLCCNV和空载体的植物为对照。
1.3.7 实时定量RT-PCR检测实时定量RT-PCR(real-time quantitative RT-PCR,qRT-PCR)用STRATAGEN 公司的Mx3000P Real-time PCR系统进行,使用ΔΔCT方法计算在不同处理及不同组织部位基因的表达量,每个试验3次重复[12]。以烟草延伸因子EF-1-α(elongation factor 1-alpha 1 gene)作为实时定量PCR的内参。
2 结果与分析
2.1 5′-RACE和3′-RACE PCR扩增结果
根据前期烟草EST测序获得的273 bp的烟草反转录转座子中间片段(结果未发表),设计2对特异性引物:3-GSP-1、3-GSP-2和5-GSP-1、5-GSP-2,分别用于扩增烟草反转录转座子基因的3′-RACE和5′-RACE反应,先用外侧引物3-GSP-1和5-GSP-1与UPM分别进行扩增,然后以其各自的扩增产物为模板,再用内测引物3-GSP-2和5-GSP-2与NUP进行PCR扩增,分别得到了2 540和2 890 bp左右的目的条带(图 1),测序后全长拼接mRNA全长5 046 bp。图 1所示为该基因3′-RACE和5′-RACE及全长cDNA的克隆及测序结果。
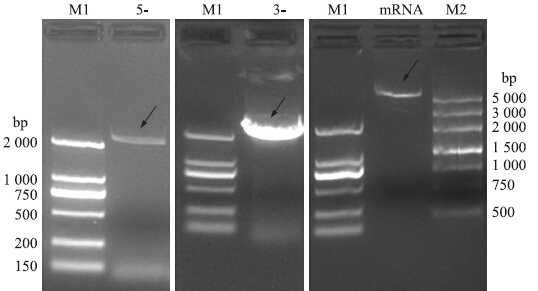 | 图 1 反转录转座子3′-RACE和5′-RACE及全长cDNA的克隆Fig. 1 Cloning of 3′-RACE,5′-RACE and full length of retrotransposonsM1:DL2000 marker;M2:DL5000 marker;3-:3′-RACE;5-:5′-RACE;mRNA:全长cDNA Full length cDNA |
将获得基因全长5 046 bp命名为Ntrt 1 (GenBank登录号:KC899077),该基因编码3个ORF,大小分别为1 014、2 256和825 bp,编码的氨基酸分别为Gag/pol多聚蛋白、反转录转座子的中心蛋白以及一个整合酶,其cDNA序列和氨基酸序列见图 2~图 4。
 | 图 2 OFR1的的核酸与氨基酸序列Fig. 2 The nucleotide and amino acid sequence of the ORF1 |
 | 图 3 ORF2的核酸与氨基酸序列Fig. 3 The nucleotide and amino acid sequence of the ORF2 |
 | 图 4 ORF3的核酸与氨基酸序列Fig. 4 The nucleotide and amino acid sequence of the ORF3 |
在ORF3的N端,存在HX3-7HX23-32CX2C的整合酶的典型锌指结构,说明在扩增的区域中含有整合酶,所以扩增的该段mRNA区域应为ty 3 -gypsy类反转录转座子。
2.3 基因表达分析分别提取烟草的叶片、根、茎中的总RNA,反转录成cDNA,用引物Q1和Q2进行qRT-RCR,分析Ntrt 1 在烟草不同组织中的表达情况,试验结果表明,在检测的组织中,均检测到了该基因的表达,说明该基因的表达没有明显的组织特异性,与叶片相比,根和茎中的表达量相对较低(图 5)。
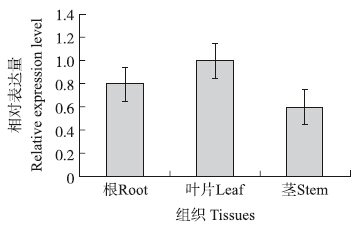 | 图 5 Ntrt 1 基因在烟草不同组织的相对表达水平Fig. 5 Relative expression level of Ntrt 1 in different tobacco tissues |
用F1和F2为引物,通过PCR克隆Ntrt 1 的基因300 bp片段,并插入到在前期构建的沉默载体2mDNA1的多克隆位点中构建成2mDNA1-Ntrt 1 的侵染性克隆pBinPLUS-2mDNA1-Nt-rt1(图 6),取酶切鉴定正确的转化子与辅助病毒中国番茄黄化曲叶病毒TYLCCNV一起通过农杆菌注射接种烟草,接种病毒与空载体的作为对照。
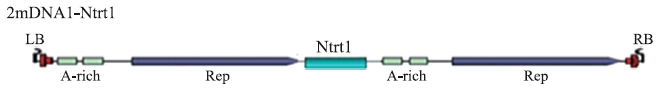 | 图 6 Ntrt 1 基因沉默载体构建示意图Fig. 6 Schematic representation of construct used for silencing Ntrt 1 |
在30 dpi沉默出现最为彻底的时候分别采取沉默植株和接种2mDNA1空载体植株的叶片,用Trizol法抽提这些叶片的总RNA,用Oligo d(T)18引物进行cDNA第一链的合成。利用2mDNA1上插入片段的外侧序列上设计的引物进行qRT-RCR扩增,以保证扩增产物反应是内源基因mRNA的水平。利用qRT-RCR检测Ntrt 1 基因的表达水平,与接种空载体的植株中Ntrt 1 基因的表达量相比,接种沉默载体中目的基因的表达水平显著下调,约为对照载体的15%(图 7)。调查接种沉默载体与对照株高差异,当 Ntrt 1 基因沉默后,烟草发育受到抑制,平均株高为66.6 cm,明显低于对照(89.7 cm)(图 8)。说明烟草Ntrt 1 基因对烟草的发育具有重要作用。
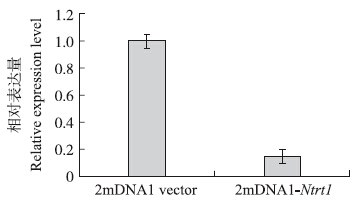 | 图 7 烟草Ntrt 1 基因在接种沉默载体和对照载体植株中的表达水平Fig. 7 Relative expression level of Ntrt 1 in Ntrt 1 silenced plant and control |
 | 图 8 烟草Ntrt 1 基因在接种沉默载体和对照载体植株的株高Fig. 8 Plants height of Ntrt 1 -silenced plant and control |
LTR反转录转座子对于植物功能基因的研究具有重要价值,LTR反转录转座子发挥作用的主要方式是插入到植物基因内部或基因附近,导致该基因的缺失或者破坏该基因的启动子而导致基因的表达量发生变化,从而影响基因的功能[13, 14, 15]。然而,其他的研究表明,反转录转座子可以改变某些蛋白的三维结构从而改变其功能[16]。LTR反转录转座子也可用于基因的克隆,Hirochick[16]在水稻中构建了反转录转座子Tto 1 基因敲除体系,应用该体系,Sato等[17]在水稻中克隆了OSH 15 基因,并证明了该基因的主要功能是控制水稻节间的延伸。
在烟草中,Anca等[18]发现烟草中已知的4个反转录转座子Tnt 1、Tnt2 、Queenti和Tto 1 参与烟草与环境的生物以及非生物胁迫,并证明它们受激发子隐地蛋白(cryptogein)所激发。本研究采用RACE的方法从烟草中克隆得到了一个反转录转座子基因,并对其进行了生物信息学和表达模式分析。为了研究该基因的功能,本研究利用VIGS的方法,该技术操作方便,当代能看到表型,是研究该类基因的理想手段。结果表明Ntrt 1 基因对于烟草的株高有促进作用,且达到极显著水平,这是该类基因调节植物发育的首次报道。本试验对研究植物中反转录转座子的生物学功能尤其是对于调节植物发育的功能研究奠定了重要基础。下一步将在目前的研究基础上,将该基因设计成分子标记,用于分子辅助选择;构建该基因的植物表达载体,转化烟草,在转基因植株中研究其详细的控制烟草株高的机制,为反转录转座子的研究提供新的思路。
| [1] | Noma K,Ohtsubo E,Ohtsubo H. Non-LTR retrotransposons(LINEs)as ubiquitous components of plant genomes[J]. Molecular Genetics and Genomics,1999,261:71-79 |
| [2] | McCarthy E M,Liu J,Gao L,et al. Long terminal repeat retrotransposons of Oryza sativa[J]. Genome Biology,2002,3:1-11 |
| [3] | Macas J,Neumann P. Ogre elements-A distinct group of plant Ty3/gypsy-like retrotransposons[J]. Gene,2007,390:108-116 |
| [4] | Dinesh-Kumar S P,Anandalakshmi R,Marathe R,et al. Virus-induced gene silencing in plants[J]. Methods in Molecular Biology,2003,236:287-293 |
| [5] | Du C,Swigoňová Z,Messing J. Retrotranspositions in orthologous regions of closely related grass species[J]. BMC Evolutionary Biology,2006,6:1-12 |
| [6] | Marchler-Bauer A,Anderson J B,Derbyshire M K,et al. CDD:a conserved domain database for interactive domain family analysis[J]. Nucleic Acids Research,2007,35:237-240 |
| [7] | Matthew L. RNAi for plant functional genomics[J]. Comp Funct Genomics,2004,5:240-244 |
| [8] | Gossele V,Fache I,Meulewaeter F,et al. SVISS-a novel transient gene silencing system for gene function discovery and validation in tobacco plants[J]. Plant Journal,2002,32:859-866 |
| [9] | Miller G,Suzuki N,Ciftci-Yilmaz S,et al. Reactive oxygen species homeostasis and signalling during drought and salinity stresses[J]. Plant Cell and Environment,2010,4:453-467 |
| [10] | Huang C J,Xie Y,Zhou X P. Efficient virus-induced gene silencing in plants using a modified geminivirus DNA1 component[J]. Plant Biotechnology Journal,2009,7:254-265 |
| [11] | Huang C J,Zhang T,Li F F,et al. Development and application of an efficient virus-induced gene silencing system in Nicotiana tabacum using geminivirus alphasatellite[J]. Journal of Zhejiang University:Science B(Biomedicine & Biotechnology),2011,2:83-92 |
| [12] | Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25:402-408 |
| [13] | Leprince A S,Grandbastien M A,Meyer C. Retrotransposons of the Tnt1B family are mobile in Nicotiana plumbaginifolia and can induce alternative splicing of the host gene upon insertion[J]. Plant Molecular Boilogy,2001,47:533-541 |
| [14] | Du C,Swigoňova Z,Messing J. Retrotranspositions in orthologous regions of closely related grass species[J]. BMC Evolutionary Biology,2006,6:62 |
| [15] | Xu Y X,Du J C. Young but not relatively old retrotransposons are preferentially located in gene-rich euchromatic regions in tomato(Solanum lycopersicum)plants[J]. The Plant Journal,2014,80:582-591 |
| [16] | Hirochika H. Activation of tobacco retrotransposons during tissue-culture[J]. EMBO Journal,1993,12:2521-2528 |
| [17] | Sato Y,Sentoku N,Miur Y. Loss-of-function mutations in the rice homeobox gene OSH15 affect the architecture of inter nodes resulting in dwarf plants[J]. EMBO Journal,1999,18:992-1002 |
| [18] | Iulia-Andra A,Fromentin J,Bui Q T,et al. Different tobacco retrotransposons are specifically modulated by the elicitor cryptogein and reactive oxygen species[J]. Journal of Plant Physiology,2014,171:1533-1540 |
 2015, Vol. 38
2015, Vol. 38


