文章信息
- 任昂, 曹鹏飞, 吴凤礼, 李梦娇, 姜爱良, 师亮, 赵明文. 2015.
- REN Ang, CAO Pengfei, WU Fengli, LI Mengjiao, JIANG Ailiang, SHI Liang, ZHAO Mingwen. 2015.
- 药用模式真菌灵芝小G蛋白基因家族的系统性分析
- Systematic characterization of small GTPases gene family in the model medicinal mushroom Ganoderma lucidum
- 南京农业大学学报, 38(6): 923-929
- Journal of Nanjing Agricultural University, 38(6): 923-929.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.06.008
-
文章历史
- 收稿日期: 2015-05-04
灵芝(Ganoderma lucidum)是一种大型担子菌,因其具有多种药理学活性而得到广泛的关注[1]。灵芝作为药用真菌在我国和东南亚国家有着悠久的应用历史。灵芝酸和灵芝多糖是灵芝的主要药用成分,具有抗氧化、抗病毒、抗肿瘤、降血压和镇痛等活性[2, 3]。随着灵芝基因组测序工作的完成以及灵芝转基因过表达技术和基因沉默技术的成功应用,利用分子遗传学实验和生理生化实验共同研究灵芝基础生物学问题成为主要趋势[4, 5, 6]。这些研究将有助于通过基因工程手段定向改良灵芝药用品质,加快灵芝功能基因组学的研究进度,推动灵芝成为大型药用担子菌的模式菌,增加其科研和商业价值[7]。
小G蛋白是真核生物中普遍存在的一类具有GTP酶活性的蛋白家族,因其相对分子质量只有(20~30)×103,相对于G蛋白较小,因此称之为小G蛋白[8, 9]。小G蛋白在细胞信号传导过程中具有重要作用,参与调控真核细胞的生长、发育和次生代谢等生理过程[10, 11, 12]。在前期研究茉莉酸甲酯诱导灵芝转录组变化的过程中,筛选到一个小G蛋白rho基因[13]。灵芝rho可能参与调控灵芝的次生代谢。为了系统地研究灵芝小G蛋白的生物学特性,本论文从灵芝基因组中筛选所有小G蛋白的同源基因,并进一步分析灵芝所有小G蛋白同源蛋白的生物学特性,为深入研究灵芝小G蛋白的功能提供理论基础。
1 材料与方法 1.1 菌株和培养条件灵芝(Ganoderma lucidum)菌株HG,由上海市农业科学院食用菌研究所提供,保存在PDA培养基斜面上。灵芝菌丝体接种于PDB培养基中,摇床150 r · min-1、28 ℃恒温培养7 d。
1.2 灵芝小G蛋白同源基因的筛选利用NCBI蛋白数据库中已知的真菌小G蛋白序列(Saccharomyces cerevisiae DAA10456、P48559、Q02804、P06780、NP_014744、NP_013330和Magnaporthe oryzae EHA49014、EHA57036、XP_003709251、XP_003711370)进行BLASTp检索(E值小于E-05)灵芝基因组数据库(http://www.herbalgenomics.org/galu)[4]。根据小G蛋白的保守功能域,将上述检索到的基因编码蛋白序列进行SMART(http://smart.embl-heidelberg.de/)和Pfam[14],再次确认灵芝小G蛋白同源蛋白的编码基因。
1.3 灵芝小G蛋白同源基因编码蛋白的生物信息学分析[15] 1.3.1 灵芝小G蛋白同源基因及其编码蛋白的基本特性分析将筛选到的灵芝小G蛋白同源基因进行BLASTn比对灵芝基因组数据库,确定同源基因在灵芝染色体上的分布情况以及基因长度和cDNA长度。对灵芝小G蛋白进行蛋白质氨基酸残基数、相对分子质量和等电点的预测(http://www.expasy.ch/tools/pi_tool.html)。将所有预测的灵芝小G蛋白序列BLASTp比对NCBI的Non-redundant protein sequences(nr)数据库,分析灵芝小G蛋白的保守功能域。
1.3.2 灵芝小G蛋白的系统进化树分析利用CLUSTALW对灵芝(G.lucidum)、酿酒酵母(Saccharomyces cerevisiae)、构巢曲霉(Aspergillus nidulans)、新型隐球菌(Crytococcus neoformans)和稻瘟病菌(Magnaporthe oryzae)的小G蛋白序列进行多重比较分析。系统进化树使用MEGA 6.0软件采用Neighbor-joining法进行构建(1 000 bootstrap repetitions)。
1.3.3 灵芝小G蛋白同源蛋白的亚细胞定位预测利用SOFTBERRY网站(http://www.softberry.com/)的ProtComp 9.0软件进行灵芝小G蛋白的亚细胞定位预测分析。
1.4 灵芝小G蛋白同源基因的荧光定量PCR检测 1.4.1 灵芝cDNA制备灵芝新鲜菌丝用去离子水冲洗干净后,液氮充分研磨。灵芝总RNA使用RNAiso plus(TaKaRa,大连)提取。随后使用RNase-free DNaseⅠ消化处理,防止基因组DNA污染。灵芝cDNA合成使用cDNA mixture(TaKaRa,大连)。
1.4.2 灵芝小G蛋白同源基因的荧光定量PCR检测参照荧光定量PCR的方法[16],进行PCR扩增。 荧光定量PCR引物序列见表 1。PCR反应体系为: SYBR GreenⅠMix 10 μL,上、下游引物(10 μmol · L-1)各0.5 μL,cDNA 1.5 μL,ddH2O 7.5 μL,总体积为20 μL。荧光定量PCR反应条件:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 1 min,循环40次;95 ℃,15 s,60 ℃,15 s,60 ℃升温至95 ℃,20 min;95 ℃,15 s,10 ℃,1 min。根据扩增曲线和熔解曲线检测引物的扩增情况。
| 目标基因编号 Target gene ID | 引物方向 Primer direction | 序列 Sequence | 目标基因编号 Target gene ID | 引物方向 Primer direction | 序列 Sequence | |
| GlsG01 | 正向Forward 反向Reverse | 5′-CGGCACCTCAAGAACGA-3′ 5′-TCCAGCCCACAAACCAG-3′ | GlsG16 | 正向Forward 反向Reverse | 5′-ACGATTGAGCGAGGGTT-3′ 5′-CGGACAGGCTATGAGGG-3′ | |
| GlsG02 | 正向Forward 反向Reverse | 5′-GTTGCCGTTCTTGGTGA-3′ 5′-CCTCTTGTCCTGCCGTAT-3′ | GlsG17 | 正向Forward 反向Reverse | 5′-GTTCACGACCTACGACTTGG-3′ 5′-CTCCGCCTTGGACTCTG-3′ | |
| GlsG03 | 正向Forward 反向Reverse | 5′-GTCAACGAGCCGTCCAG-3′ 5′-AGCGGTGTCCCATAAGC-3′ | GlsG18 | 正向Forward 反向Reverse | 5′-TGACGCCCTCCTCCTCG-3′ 5′-CGCTCAGCCACTCAAGAC-3′ | |
| GlsG04 | 正向Forward 反向Reverse | 5′-TAACTACTCGGCGAATGTC-3′ 5′-GGAGAAGCAGATGAGGAAG-3′ | GlsG19 | 正向Forward 反向Reverse | 5′-ACCTCAGCAATCATTTACG-3′ 5′-GCAAGTCCAAGTTTCTCG-3′ | |
| GlsG05 | 正向Forward 反向Reverse | 5′-GGAATATGAACGACTACGGC-3′ 5′-CGGACCTCTTCTATCCACTT-3′ | GlsG20 | 正向Forward 反向Reverse | 5′-CGACCTACAGAGCAGACA-3′ 5′-GGCTCCACCTTGTTACTT-3′ | |
| GlsG06 | 正向Forward 反向Reverse | 5′-CACCTACCAAGCAACGA-3′ 5′-GCTACAGAACAGTCACGAAT-3′ | GlsG21 | 正向Forward 反向Reverse | 5′-TCAACCTGTTTACCACCAA-3′ 5′-CGAGGCAACTGCGTCTGT-3′ | |
| GlsG07 | 正向Forward 反向Reverse | 5′-GACCGACTGGTAACGATG-3′ 5′-TGTGGGCTTGCTTGGAT-3′ | GlsG22 | 正向Forward 反向Reverse | 5′-GCTTCCTATGCCCTAAATT-3′ 5′-GGGTTGCCTGGTGATGT-3′ | |
| GlsG08 | 正向Forward 反向Reverse | 5′-CTTGTGCTGCGTTTCGT-3′ 5′-AGTTGGCATTGCGGTAG-3′ | GlsG23 | 正向Forward 反向Reverse | 5′-GCTGCGGTATGAGATTTGG-3′ 5′-CGAGACTGGATGCCTTTGT-3′ | |
| GlsG09 | 正向Forward 反向Reverse | 5′-GGTCGTTTAACAACATCCG-3′ 5′-CGCTTCTCATTCCAGTCG-3′ | GlsG24 | 正向Forward 反向Reverse | 5′-GGAATATGAACGACTACGGC-3′ 5′-CGGACCTCTTCTATCCACTT-3′ | |
| GlsG10 | 正向Forward 反向Reverse | 5′-TCACGACGGGATACAGGG-3′ 5′-GCGGAAGAAGGCAGAGG-3′ | GlsG25 | 正向Forward 反向Reverse | 5′-AAGGATGGCGATGAGGA-3′ 5′-CGTGGAACACTTCGTTGA-3′ | |
| GlsG11 | 正向Forward 反向Reverse | 5′-CAGCCTCTTGCTCCGATTC-3′ 5′-GCTTGACCTTCTTTCCCTTGA-3′ | GlsG26 | 正向Forward 反向Reverse | 5′-TTAGCACCTTCCACCAAC-3′ 5′-AACGCCTCGTCAACATTA-3′ | |
| GlsG12 | 正向Forward 反向Reverse | 5′-GAGCGGTTTCGTAGCAT-3′ 5′-CTTCTTGAGTTCTGTGGTGAT-3′ | GlsG27 | 正向Forward 反向Reverse | 5′-TCAAGGGCTGGCTACAA-3′ 5′-TGCGACCGAATACTCAAT-3′ | |
| GlsG13 | 正向Forward 反向Reverse | 5′-CAAACGCAGATGACCCG-3′ 5′-TCCTCCTTCGCCCACAG-3′ | GlsG28 | 正向Forward 反向Reverse | 5′-CACAGAGGAGGCGAAGA-3′ 5′-CAGTGAGGATGGTTTGGA-3′ | |
| GlsG14 | 正向Forward 反向Reverse | 5′-GGGTGAATTTGAGAAGAAG-3′ 5′-GTATCCCAGACATTGAAGC-3′ | GlsG29 | 正向Forward 反向Reverse | 5′-CGGAAGAACAGGCAGAA-3′ 5′-GTTGAAGCCCAGCGTAG-3′ | |
| GlsG15 | 正向Forward 反向Reverse | 5′-CGCTCTGGAGGCATTAC-3′ 5′-GACACTCTTTCATTTCCCTAT-3′ | ||||
制备20 g · L-1的琼脂糖凝胶(含溴化乙锭1 μg · mL-1),将荧光定量PCR扩增的产物进行电泳(120 V,30 min),在紫外凝胶成像仪上进行拍照并保存。
2 结果与分析 2.1 灵芝小G蛋白家族成员的预测和基本特性分析根据目前已经报道的真菌小G蛋白进行蛋白同源序列比对,我们在灵芝基因组中一共检索到29个小G蛋白的同源编码基因(表 2)。这些同源蛋白分别命名为GlsG01~GlsG29。灵芝小G蛋白的基因全长大部分分布于800~1 400 bp,cDNA全长大部分分布于450~750 bp,只有GlsG12、GlsG21和GlsG22的基因全长和cDNA全长相对较大。29个小G蛋白编码基因在灵芝12条染色体上的分布数量也存在显著差异。染色体Chr3、Chr1和Chr4上别为存在7、5和4个小G蛋白编码基因,染色体Chr11上没有发现小G蛋白编码基因,其他染色体上存在1~2个小G蛋白编码基因。灵芝小G蛋白同源蛋白的理论等电点主要分布在4.60~7.60之间,只有GlsG02、GlsG12和GlsG15的理论等电点为9.06、8.60和9.34。
| 编号 No. | 基因组序列编号 ID in genome | 基因长度/bp Gene length | 染色体定位 Chromosome location | cDNA长度/bp cDNA length | 氨基酸残基数 Number of amino acid residues | 理论等电点 Theoretical isoelectric point | 蛋白相对分子质量/103 Relative molecular mass |
| GlsG01 | Gl22252 | 1 310 | Chr9 | 711 | 236 | 6.64 | 31.0 |
| GlsG02 | Gl30205 | 865 | Chr3 | 552 | 183 | 9.06 | 20.9 |
| GlsG03 | Gl21897 | 1 177 | Chr6 | 645 | 214 | 7.59 | 24.1 |
| GlsG04 | Gl29462 | 989 | Chr3 | 582 | 193 | 7.59 | 21.3 |
| GlsG05 | Gl23196 | 1 182 | Chr2 | 669 | 222 | 6.44 | 24.2 |
| GlsG06 | Gl28480 | 761 | Chr4 | 528 | 175 | 4.95 | 19.8 |
| GlsG07 | Gl30495 | 960 | Chr3 | 612 | 203 | 4.81 | 22.9 |
| GlsG08 | Gl30695 | 1 018 | Chr3 | 684 | 227 | 7.56 | 24.7 |
| GlsG09 | Gl30095 | 1 262 | Chr3 | 630 | 209 | 5.45 | 23.1 |
| GlsG10 | Gl30427 | 1 359 | Chr3 | 762 | 253 | 6.39 | 27.8 |
| GlsG11 | Gl16439 | 1 058 | Chr12 | 684 | 227 | 5.41 | 24.5 |
| GlsG12 | Gl31439 | 1 469 | Chr1 | 1 146 | 381 | 8.60 | 42.2 |
| GlsG13 | Gl24762 | 1 187 | Chr4 | 735 | 244 | 5.73 | 26.6 |
| GlsG14 | Gl24464 | 1 145 | Chr4 | 645 | 214 | 5.87 | 24.1 |
| GlsG15 | Gl23463 | 934 | Chr8 | 549 | 182 | 9.34 | 20.7 |
| GlsG16 | Gl25221 | 969 | Chr5 | 645 | 214 | 5.83 | 24.5 |
| GlsG17 | Gl24870 | 956 | Chr4 | 591 | 196 | 5.54 | 22.3 |
| GlsG18 | Gl26617 | 859 | Chr1 | 546 | 181 | 6.75 | 20.5 |
| GlsG19 | Gl26705 | 929 | Chr1 | 483 | 160 | 5.09 | 17.8 |
| GlsG20 | Gl26736 | 910 | Chr1 | 564 | 187 | 6.06 | 20.8 |
| GlsG21 | Gl27854 | 1 764 | Chr12 | 1 137 | 378 | 4.68 | 44.9 |
| GlsG22 | Gl23656 | 2 613 | Chr7 | 1 722 | 573 | 5.46 | 66.9 |
| GlsG23 | Gl29920 | 1 106 | Chr3 | 756 | 251 | 4.88 | 26.9 |
| GlsG24 | Gl23194 | 1 182 | Chr2 | 669 | 222 | 6.89 | 24.2 |
| GlsG25 | Gl25179 | 1 171 | Chr5 | 603 | 200 | 6.45 | 23.0 |
| GlsG26 | Gl25859 | 1 070 | Chr8 | 651 | 216 | 5.08 | 24.1 |
| GlsG27 | Gl22843 | 1 010 | Chr10 | 621 | 206 | 4.59 | 22.8 |
| GlsG28 | Gl22829 | 1 247 | Chr10 | 639 | 212 | 5.83 | 23.3 |
| GlsG29 | Gl26542 | 940 | Chr1 | 624 | 207 | 6.84 | 23.4 |
将29个灵芝小G蛋白的同源蛋白与其他真菌中已报道的小G蛋白构建系统进化树,结果如图 1所示。29个灵芝小G蛋白家族成员中有28个分别属于Ras(4个)、Rho(4个)、Rab(12个)、Arf(7个)和Ran(1个)5个亚家族,还有1个GlsG22不在已知的5个亚家族内。根据进化树可以看出,在每一个亚家族内很多分支的引导值较高。这表明:真菌的小G蛋白家族成员结构较为保守,可能来自共同的祖先。
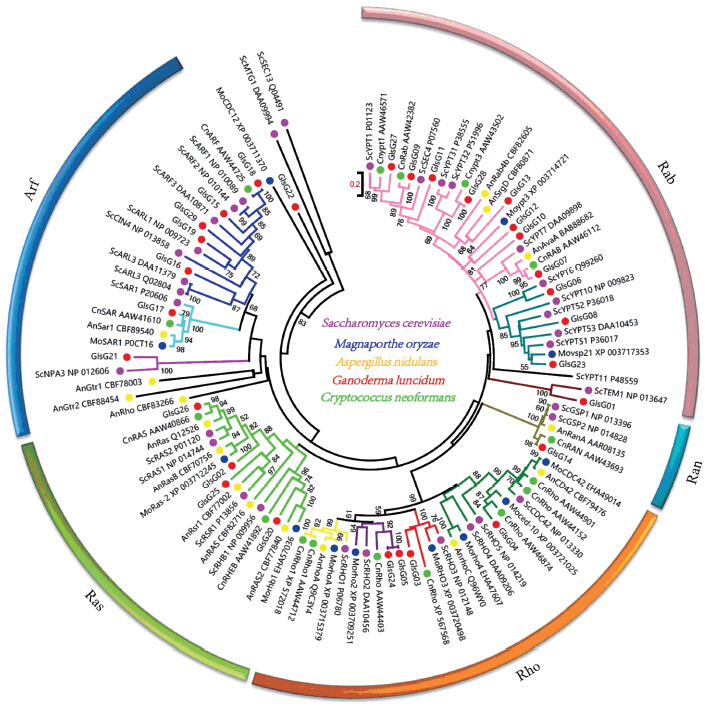 |
图 1 真菌小G蛋白的系统发生树分析Fig. 1 Phylogenetic analysis of putative small GTPases in fungiSc:酿酒酵母;Mo:稻瘟病菌;An:构巢曲霉;Gl:灵芝;Cn:新型隐球菌。蛋白的序列号放在蛋白名称的后面。 Sc:Saccharomyces cerevisiae;Mo:Magnaporthe oryzae;An:Aspergillus nidulans;Gl:Ganoderma lucidum;Cn:Cryptococcus neoformans. The protein IDs are followed with the protein names. |
针对灵芝的小G蛋白家族29个成员进行蛋白结构域分析,结果见图 2。所有灵芝小G蛋白都具有G box结构和SwitchⅠ(Ⅱ)结构域。灵芝小G蛋白GlsG04、GlsG06、GlsG12、GlsG19都含有4个G box,GlsG21含有3个G box,GlsG22含有8个G box,其他小G蛋白均含有5个G box。除了GlsG04、GlsG06和GlsG21外,其他小G蛋白都含有GTP/Mg2+结合位点。GlsG01、GlsG06、GlsG07、GlsG08、GlsG09、GlsG10、GlsG12、GlsG13、GlsG23、GlsG27和GlsG28这11个小G蛋白含有Rab家族元件。这与进化树分析结果一致,这些小G蛋白都属于Rab亚家族。
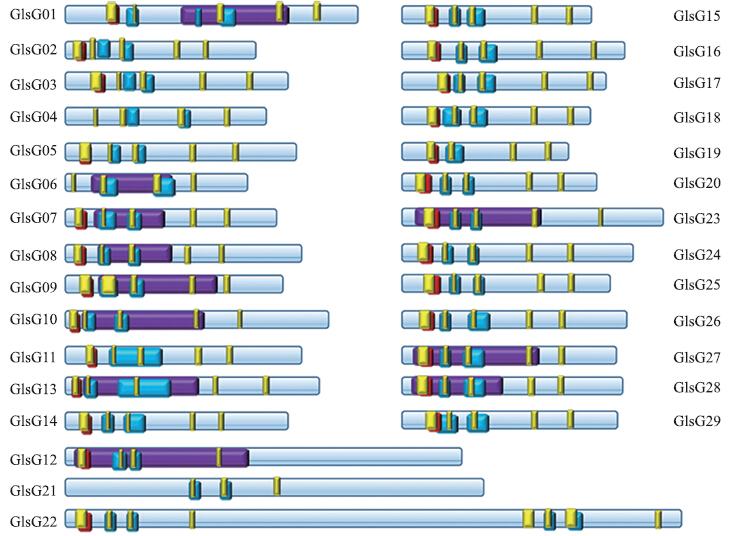 |
图 2 灵芝小G蛋白家族成员的功能域分析Fig. 2 Analysis of functional domains of putative small GTPases in G.lucidum红色盒子代表GTP/Mg2+结合位点;黄色盒子代表G box;蓝色盒子代表SwitchⅠ(Ⅱ)区;紫色盒子代表Rab家族元件(RabF)。 Red boxes represent GTP/Mg2+ binding site;yellow boxes represent G box;blue boxes represent Switch Ⅰ(Ⅱ)region;purple boxes represent Rab family motif(RabF). |
利用softberry网站提供的真菌蛋白亚细胞定位分析功能对灵芝中29个小G蛋白同源基因进行了亚细胞定位分析,结果如表 3所示。灵芝小G蛋白亚细胞定位分布最主要的3个区域是高尔基体、细胞质和质膜。除此之外,GlsG14位于细胞核,GlsG22位于线粒体,GlsG07和GlsG10位于液泡。
| 亚细胞定位 Subcellular localization | 细胞核 Nuclear | 质膜 Plasma membrane | 胞外分泌 Extracellular | 细胞质 Cytoplasmic | 线粒体 Mitochondrial | 内质网(膜) Endoplasmic reticulum (membrane) | 过氧化物 酶体 Peroxisomal | 溶酶体 Lysosomal | 高尔基体 Golgi | 液泡 Vacuolar |
| GlsG01 | 7.5 | 17.8 | 20.0 | 17.4 | 20.4 | 8.7 | 1.9 | 0 | 4.7 | 1.6 |
| GlsG02 | 5.4 | 75.7 | 0 | 6.3 | 0 | 0 | 0.8 | 0 | 11.7 | 0.2 |
| GlsG03 | 21.4 | 33.3 | 0 | 0 | 0 | 0 | 0 | 0 | 45.3 | 0 |
| GlsG04 | 21.5 | 37.3 | 0 | 0 | 0 | 0 | 0 | 0 | 41.2 | 0 |
| GlsG05 | 21.7 | 31.7 | 0 | 0.2 | 0 | 0 | 0 | 0 | 46.4 | 0 |
| GlsG06 | 0 | 25.1 | 0 | 24.1 | 0 | 0 | 0 | 0 | 50.7 | 0.1 |
| GlsG07 | 0 | 23.2 | 0.8 | 4.0 | 0.1 | 0 | 0.1 | 0.2 | 24.9 | 46.7 |
| GlsG08 | 0 | 0 | 0.5 | 0.2 | 0.1 | 0 | 0 | 0 | 98.8 | 0.4 |
| GlsG09 | 3.4 | 26.5 | 0 | 27.0 | 0.3 | 0 | 0 | 0 | 24.5 | 18.2 |
| GlsG10 | 0.2 | 0.6 | 0.8 | 1.4 | 1.8 | 0 | 0 | 0 | 46.3 | 48.9 |
| GlsG11 | 0.3 | 0 | 0 | 9.5 | 0 | 0 | 0 | 0 | 89.9 | 0.3 |
| GlsG12 | 33.0 | 20.2 | 6.8 | 2.6 | 20.5 | 0 | 0.5 | 0 | 16.4 | 0 |
| GlsG13 | 0.2 | 0 | 0.5 | 98.8 | 0 | 0.5 | 0 | 0 | 0 | 0 |
| GlsG14 | 97.4 | 2.5 | 0 | 0.1 | 0 | 0 | 0 | 0 | 0.1 | 0 |
| GlsG15 | 0.5 | 0 | 8.2 | 33.1 | 0 | 0 | 0 | 0.2 | 57.8 | 0.1 |
| GlsG16 | 0 | 0 | 13.2 | 6.1 | 0 | 0 | 0 | 0 | 80.6 | 0 |
| GlsG17 | 0 | 0.7 | 13.9 | 6.0 | 1.4 | 0 | 0 | 0 | 77.9 | 0 |
| GlsG18 | 0 | 0 | 14.1 | 4.1 | 0 | 0 | 0 | 0.2 | 81.5 | 0.1 |
| GlsG19 | 0.1 | 0 | 13.7 | 5.3 | 0 | 0 | 0.1 | 4.4 | 76.3 | 0 |
| GlsG20 | 0 | 22.8 | 1.9 | 32.9 | 0 | 0.6 | 0 | 0 | 40.9 | 0.9 |
| GlsG21 | 0.2 | 0 | 0 | 99.7 | 0 | 0 | 0 | 0 | 0.1 | 0 |
| GlsG22 | 0 | 0 | 0 | 0.4 | 99.3 | 0.1 | 0.1 | 0 | 0 | 0.1 |
| GlsG23 | 0 | 0 | 0 | 45.9 | 0.3 | 1.0 | 0 | 0 | 46.0 | 6.8 |
| GlsG24 | 21.7 | 31.6 | 0 | 0.2 | 0 | 0 | 0 | 0 | 46.4 | 0.1 |
| GlsG25 | 0.2 | 64.2 | 0 | 0 | 0 | 0 | 0 | 0 | 35.6 | 0 |
| GlsG26 | 0 | 46.0 | 0.2 | 41.8 | 0.7 | 0.1 | 0 | 0.1 | 0 | 11.2 |
| GlsG27 | 3.1 | 29.3 | 0 | 23.8 | 0 | 0 | 0 | 0 | 24.6 | 19.2 |
| GlsG28 | 3.4 | 24.0 | 0 | 23.4 | 0.3 | 0 | 0 | 0 | 30.9 | 18.0 |
| GlsG29 | 0.1 | 0 | 10.7 | 7.6 | 0 | 0 | 0.1 | 2.5 | 79.0 | 0 |
针对灵芝29个小G蛋白同源序列设计特异性实时荧光定量PCR引物,利用灵芝cDNA进行实时荧光定量PCR检测。实时荧光定量PCR的扩增曲线结果表明:除了GlsG12没有扩增出PCR产物外,其他小G蛋白同源基因都能够扩增到特异性产物。熔解曲线结果也表明:除了GlsG12没有检测到信号峰外,其他基因的扩增产物都是单一峰型,表明实时荧光定量PCR扩增产物单一。图 3显示了实时荧光定量PCR产物的电泳检测结果。结果表明:GlsG12没有检测到PCR扩增产物,这一结果和实时荧光定量PCR的扩增曲线结果一致。GlsG01~GlsG29均是单一条带,产物大小与预期大小一致。这些结果表明,GlsG12基因可能在灵芝菌丝体中不表达,其他的灵芝小G蛋白同源基因在灵芝菌丝体中都有表达。
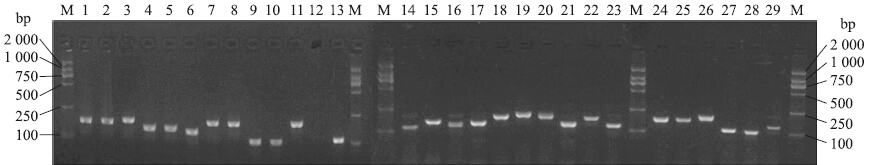 |
图 3 灵芝小G蛋白同源基因的实时荧光定量PCR产物检测Fig. 3 Detection of the real-time quantitative PCR production of small GTPases encoding genes in G.lucidumM:DNA分子质量标准;泳道1~29分别对应灵芝小G蛋白GlsG01~GlsG29的PCR产物。 M:DNA marker;Line 1-29 represent the production of PCR from GlsG01-GlsG29. |
小G蛋白家族在真核细胞中广泛存在,并且家族成员众多,参与调控细胞多种生理功能[8, 9, 11]。相对于动植物中小G蛋白的研究,真菌中报道的相对较少[12]。目前真菌小G蛋白报道最多的物种是酿酒酵母。根据NCBI蛋白数据库中的信息,酿酒酵母中至少存在35个已知的小G蛋白。本研究首次系统地筛选了灵芝中的小G蛋白。灵芝中该家族共有29个成员,其中28个属于已知的5个亚家族中。根据同源比对结果和亚细胞定位分析可以得出,GlsG22是灵芝唯一一个定位于线粒体的小G蛋白。灵芝小G蛋白的进化关系和蛋白结构功能域表明,灵芝小G蛋白家族和其他真菌小G蛋白一致,具有较高的保守性。
随着小G蛋白功能研究的深入,小G蛋白信号通路成为细胞信号转导研究的热点问题。小G蛋白Arf1通过GTP/GDP分子开关功能调控线粒体的形态和功能[11]。在灰霉病菌中,小G蛋白rho3调控了细胞生长、分生孢子形成以及菌株毒力[12]。在研究茉莉酸甲酯调控灵芝酸生物合成的工作中,Ren等[13]通过比较转录组差异筛选到一个小G蛋白编码基因rho,并推测小G蛋白信号通路可能参与了灵芝酸生物合成的调控。本文系统地筛选了灵芝小G蛋白家族的29个成员,为下一步系统性研究小G蛋白信号通路调控灵芝酸的生物合成提供的保障。
大型担子真菌在菌丝生长发育、木质素降解、子实体形成和次生代谢等生理功能上具有很多特性[7, 16]。灵芝作为大型担子真菌的模式菌株,系统地研究其各类家族基因的特性和功能使得解析大型担子真菌的特殊调控机制成为可能。灵芝小G蛋白家族成员不仅数量多,达29个,并且亚细胞定位预测显示这些小G蛋白分布在高尔基体、胞质、细胞膜、细胞核和线粒体等位置。这些结果都暗示:小G蛋白家族成员的功能较为复杂,因此,我们将针对小G蛋白家族在灵芝生理功能上的调控作用开展系统性研究。
| [1] | Hajjaj H,Mace C,Roberts M,et al. Effect of 26-oxygenosterols from Ganoderma lucidum and their activity as cholesterol synthesis inhibitors[J]. Appl Environ Microbiol,2005,71(7):3653-3658 |
| [2] | Joseph S,Sabulal B,George V,et al. Antitumor and anti-inflammatory activities of polysaccharides isolated from Ganoderma lucidum[J]. Acta Pharm,2011,61(3):335-342 |
| [3] | Xu Z,Chen X,Zhong Z,et al. Ganoderma lucidum polysaccharides:immunomodulation and potential anti-tumor activities[J]. Am J Chin Med,2011,39(1):15-27 |
| [4] | Chen S,Xu J,Liu C,et al. Genome sequence of the model medicinal mushroom Ganoderma lucidum[J]. Nature Commun,2012. doi:10.1038/ncomms1923 |
| [5] | Mu D,Shi L,Ren A,et al. The development and application of a multiple gene co-silencing system using endogenous URA3 as a reporter gene in Ganoderma lucidum[J]. PLoS ONE,2012,7(8):e43737 |
| [6] | Shi L,Fang X,Li M,et al. Development of a simple and efficient transformation system for the basidiomycetous medicinal fungus Ganoderma lucidum[J]. World J Microbiol Biotechnol,2012,28(1):283-291 |
| [7] | Mu D,Li C,Zhang X,et al. Functions of the nicotinamide adenine dinucleotide phosphate oxidase family in Ganoderma lucidum:an essential role in ganoderic acid biosynthesis regulation,hyphal branching,fruiting body development,and oxidative-stress resistance[J]. Environ Microbiol,2014,16(6):1709-1728 |
| [8] | Vernoud V,Horton A C,Yang Z,et al. Analysis of the small GTPase gene superfamily of Arabidopsis[J]. Plant Physiol,2003,131(3):1191-1208 |
| [9] | 石佰丽,吕梦燕,赵秋雁. 小GTPase蛋白家族的研究进展[J]. 安徽农业科学,2012,40(5):2566-2569,2573 [Shi B L,Lü M Y,Zhao Q Y. Research progress of the small GTPase family[J]. Journal of Anhui Agricultural Sciences,2012,40(5):2566-2569,2573(in Chinese with English abstract)] |
| [10] | Lickfeld M,Schmitz H P. A network involving Rho-type GTPases,a paxillin and a formin homologue regulates spore length and spore wall integrity in the filamentous fungus Ashbya gossypii[J]. Mol Microbiol,2012,85(3):574-593 |
| [11] | Ackema K B,Hench J,Bckler S,et al. The small GTPase Arf1 modulates mitochondrial morphology and function[J]. EMBO J,2014,33(22):2659-2675 |
| [12] | An B,Li B,Qin G,et al. Function of small GTPase Rho3 in regulating growth,conidiation and virulence of Botrytis cinerea[J]. Fungal Genet Biol,2015,75:46-55 |
| [13] | Ren A,Li M J,Shi L,et al. Profiling and quantifying differential gene transcription provide insights into ganoderic acid biosynthesis in Ganoderma lucidum in response to methyl jasmonate[J]. PLoS ONE,2013,8(6):e65027 |
| [14] | Finn R D,Bateman A,Clements J,et al. The Pfam protein families database[J]. Nucleic Acids Research,2014,42:D222-D230 |
| [15] | 李伟,王龙超,曹金花,等. 大豆基因组CLC同源基因家族的生物信息学分析[J]. 南京农业大学学报,2014,37(3):35-43. doi:10.7685/j.issn.1000-2030.2014.03.105 [Li W,Wang L C,Cao J H,et al. Bioinformatics analysis of CLC homologous genes family in soybean genome[J]. Journal of Nanjing Agricultural University,2014,37(3):35-43(in Chinese with English abstract)] |
| [16] | Ren A,Li X B,Miao Z G,et al. Transcript and metabolite alterations increase ganoderic acid content in Ganoderma lucidum using acetic acid as an inducer[J]. Biotechnol Lett,2014,36(12):2529-2536 |
 2015, Vol. 38
2015, Vol. 38


