文章信息
- 曾文韬, 柴春月, 窦道龙. 2015.
- ZENG Wentao, CHAI Chunyue, DOU Daolong. 2015.
- 适用于大豆实时荧光定量PCR分析的内参基因的筛选和验证
- Selection and validation of reference genes for quantitative RT-PCR analysis in soybean
- 南京农业大学学报, 38(5): 787-795
- Journal of Nanjing Agricultural University, 38(5): 787-795.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.05.013
-
文章历史
- 收稿日期:2014-10-20
2. 南阳师范学院生命科学与技术学院, 河南 南阳 473061
2. College of Life Science and Technology, Nanyang Normal University, Nanyang 473061, China
在诸多检测基因表达水平的方法中,实时荧光定量PCR(real-time quantitative PCR,qRT-PCR)技术以其具有的显著优点如:高灵敏度、较好的重复性、较强特异性以及能实现高通量操作等,被广泛应用于生物及医学研究等领域[1, 2, 3, 4]。也正是这些优点使得它一方面对RNA的质量、反转录效率要求严格,另一方面对引物的特异性、扩增效率以及内参基因的选择等要求严格[5, 6]。因此选择合适的内参基因是精确分析qRT-PCR结果的重要前提[7, 8, 9]。在植物qRT-PCR研究中内参基因通常选自组成性表达的持家基因,主要包括18S核糖体RNA(18S rRNA)、甘油醛-3-磷酸甘油醛脱氢酶基因(GAPDH)、转录延伸因子基因(EF1)、多聚泛素酶基因(UBQ)、泛素连接酶基因(UBI)、肌动蛋白基因(ACT)、α/β微管蛋白基因(TUA/TUB)、亲环蛋白基因(CYP)和组蛋白基因(H2A)等。这些基因是生物体维持生命活动所必需的细胞器骨架的基本组分,或参与生物体的基本生化代谢过程,因此理论上它们能在不同细胞或生理状态下都稳定地表达。但目前已有大量的研究表明,当选择这些传统的持家基因作为内参基因时,在不同植物和不同试验条件下的表达并非持续而且稳定[10, 11, 12, 13]。因此,我们需要针对不同试验条件,筛选出稳定表达的内参基因,为获得可靠试验结果提供保障[14]。
大豆是世界上最重要的一种油料经济作物,也是最大的食用油和蛋白的来源,在农作物生产上扮演着重要的角色。大豆的很多次生代谢物质,如:皂苷类、植酸、寡糖类、致甲状腺肿物、植物雌激素等[15, 16, 17],被广泛应用到化学工业、医药等领域。由大豆疫霉(Phytophthora sojaeKaufmann & Gerdemann)引起的根、茎腐烂病是大豆生产上的毁灭性病害之一,该病害在世界范围内的大豆种植区几乎都可以检测到,严重影响着大豆的产量和质量,每年都造成巨大的经济损失[18]。大豆疫霉归属于卵菌门,其作为土传病害,很多控制真菌的常用方法对其控制效果都不明显。目前控制该病害的主要方法是选育抗病品种,但在生产上持续种植具有单一抗性的品种,其抗性会随着大豆疫霉菌的快速变异而丧失。所以如果能结合植物抗病基因工程策略,研究大豆中的抗病相关基因,对于推动大豆抗疫霉基因工程研究具有重要意义,而筛选稳定的内参基因在大豆抗疫霉相关基因表达研究中起着关键作用。
为此,本研究结合本实验室先前的试验结果[18, 19, 20]及相关文献报道[21, 22]选择了10个(Cons 4、ACT11、TUA、Cons15、ACT20、Cons7、CYP2、Cons6、Tubulin和ELF1 B)候选持家基因,利用qRT-PCR技术系统分析了这10个持家基因在大豆疫霉诱导下的大豆根中以及不同组织(根、茎、叶)中的表达稳定性。结合3个常用的基因稳定性分析统计学软件(GeNorm[23]、NormFinder[24]和BestKeeper[7]),以各个基因的CT值为基础进行综合分析,评估候选持家基因的稳定性,以期获得在相应试验背景下稳定表达的大豆内参基因。
1 材料与方法 1.1 材料供试大豆品种为‘Williams’,大豆疫霉(Phytophthora sojae)菌株是P6497,‘Williams’对大豆疫霉感病,以上材料均由本实验室保存。
1.2 大豆植株预处理将供试的大豆(‘Williams’)种子播种在蛭石培养基中,在25℃、16 h/8 h光周期的温室中培养。待大豆生长7 d左右,2片真叶完全展开后将豆苗从蛭石中拔出,用清水冲洗根部的蛭石,尽量不要伤及根部。将新鲜的大豆根浸泡在大豆疫霉游动孢子悬浮液(浓度为105 mL-1)中进行接种,以清水浸泡作为对照。分别在接种后的0、0.5、1、2、4、8、16和24 h取样,迅速用滤纸吸干水分并剪下根部,保存在液氮中待用。把培养7 d且长势一致的大豆苗从培养基中拔出,用清水冲洗根部的蛭石,分别收集根、茎、叶,置于液氮中保存待用。以上每个时间点、不同组织均随机采样,每个处理3株。
1.3 总RNA的提取与cDNA合成总RNA的提取参照天根公司的RNA simple Total RNA Kit试剂说明书进行操作,用12g· L-1琼脂糖凝胶电泳检测总RNA的完整性。用Eppendorf分光光度计NanoDrop 1000,检测总RNA的浓度和纯度。使用TaKaRa公司的PrimeScript® RT Master Mix Perfect Real Time反转录试剂盒合成cDNA。反转录采用的总RNA量为500 ng,反转录体系为20μL。
1.4 内参基因的选择和引物设计结合本实验室先前的研究结果[19, 20],及相关大豆内参基因研究文献[21, 22],选择了10个内参基因Cons 4、ACT11、TUA、Cons15、ACT20、Cons7、CYP2、Cons6、Tubulin和ELF1 B。通过Primer Premier 5.0软件设计引物(表 1),由南京金斯瑞公司合成。
1.5 实时荧光定量PCRqRT-PCR在ABI Biosystems公司的荧光定量PCR仪7500上完成。操作参照TaKaRa公司的SRBY® Premix Ex TaqTM(Tli RNaseH Plus)的试剂盒说明书进行。分别以各样品cDNA为模板,采用如下反应体系:10μL SYBR® Premix Ex TaqTM,上、下游引物各0.4μL,ROX Reference Dye Ⅱ 0.4μL,模板2μL,用ddH2O定容至20μL。反应程序为:95℃ 30 s;95℃ 5 s,60℃ 34 s,40个循环。每个样品设3个生物学重复,同时设阴性对照,以减小误差。
1.6 制作标准曲线计算各基因扩增效率采用模板浓度梯度法:首先将已合成的cDNA产物稀释6倍作为qRT-PCR扩增的初始浓度,记为50,在此基础上,依次以5倍的稀释量获得4个浓度梯度,分别记为5-1、5-2、5-3、5-4,进行qRT-PCR反应,获得每个基因在不同模板浓度下的CT值。制作以模板的log值为横坐标,以CT值为纵坐标的标准曲线,计算斜率及回归系数。用公式E=(10-1/slope-1)×100计算基因的扩增效率。
1.7 数据分析数据分析采用Excel 2007和3种常用内参稳定性评价软件GeNorm[23]、BestKeeper[24]和NormFinder[7],参照软件的分析结果对各候选内参基因在不同试验条件下的表达稳定性进行分析,获得最稳定的内参基因。
2 结果与分析 2.1 RNA的分离与纯化用12g· L-1琼脂糖凝胶电泳检测所有供试样品的RNA质量,电泳结果(图 1)所示:条带清晰,没有弥散现象,同时28S rRNA条带比18S rRNA条带亮,说明获得的RNA完整性良好,基本没有降解,可用于后续试验研究。用分光光度计检测RNA的纯度和浓度,结果显示:核酸最大吸收波长吸光值(A260)/蛋白和酚类物质最大吸收波长吸光值(A280)介于1.8~2.0之间,核酸最大吸收波长吸光值(A260)/碳水化合物最大吸收波长吸光值(A230)介于2.0~2.2之间,说明RNA纯度较高,符合后续试验要求。
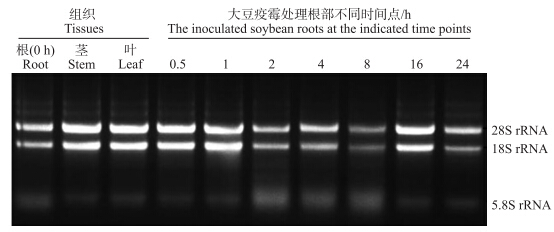
|
图 1苗期大豆不同组织和大豆疫霉侵染根不同时间点样品总RNA琼脂糖凝胶电泳图 Fig. 1 Agarose gel electrophoresis of total RNA extracted from the inoculated soybean roots at the indicated time points and various soybean tissues |
利用合成的10个候选持家基因引物,分别以5个浓度梯度(5倍稀释)的cDNA为模板,进行荧光定量PCR分析,获得各个持家基因在相应浓度下的CT值,制作标准曲线,并计算曲线的斜率。数据分析结果显示:10个候选内参基因的回归系数(R2)不小于0.997,引物扩增效率为87.8%~99.5%,符合荧光定量PCR对引物扩增效率的要求(表 1)。同时用15g·L-1琼脂糖凝胶电泳检测PCR产物,结果(图 2)显示:每对引物获得的产物条带单一,没有非特异性扩增,并且产物的大小与理论一致,说明引物的特异性良好,qRT-PCR反应专一性高,可以用于实时荧光定量PCR反应。
| 基因 Gene | 基因功能 Gene function | GenBank登录号 GenBank accession No. | 引物序列(5′→3′) Primer sequences | 产物大小/bp Product size | 扩增效率/% Amplification efficiency | R2 |
| Tubulin | Beta-tubulin | NM_001252709.1 | F-GGAGTTCACAGAGGCAGAG R-CACTTACGCATCACATAGC | 189 | 96.84 | 0.998 |
| TUA | Tubulin alpha-3 chain-like | XM_006584532.1 | F-ACCATCAAGACTAAGAGGACTG R-AACAAAAAGGAACGAACAATAC | 278 | 93.00 | 0.997 |
| ACT11 | Cytoskeletal structural protein | TC204137 | F-GGTGGTTCTATCTTGGCATC R-CTTTCGCTTCAATAACCCTA | 139 | 96.10 | 0.998 |
| Cons4 | ATP-binding cassette transporter | BU578186 | F-GATCAGCAATTATGCACAACG R-CCGCCACCATTCAGATTATGT | 106 | 87.78 | 0.997 |
| ACT20 | Cytoskeletal structural protein | XM_003531518.2 | F-TCGAGGCACCTAATTCTTGG R-GTGTCTGGATTGGTGGCTCT | 280 | 90.87 | 0.998 |
| Cons15 | CDPK-related protein kinase | AW396185 | F-TAAAGAGCACCATGCCTATCC R-TGGTTATGTGAGCAGATGCAA | 97 | 98.27 | 0.997 |
| Cons7 | Zinc-metallopeptidase | XM_003521127.2 | F-ATGAATGACGGTTCCCATGTA R-GCATTAAGGCAGCTCACTCT | 115 | 93.22 | 0.997 |
| CYP2 | Peptidyl-prolylcis-trans isomerase 1-like | NM_001248294.1 | F-CGGGACCAGTGTGCTTCTTCA R-CCCCTCCACTACAAAGGCTCG | 154 | 97.08 | 0.998 |
| ELF1B | Elongation factor 1-beta 2-like | NM_001249608.1 | F-GTTGAAAAGCCAGGGGACA R-TCTTACCCCTTGAGCGTGG | 102 | 95.95 | 0.998 |
| Cons6 | F-box-like | NM_001254106.2 | F-CTAATGGCAATTGCAGCTCTC R-AGATAGGGAAATGGTGCAGGT | 93 | 99.50 | 0.998 |
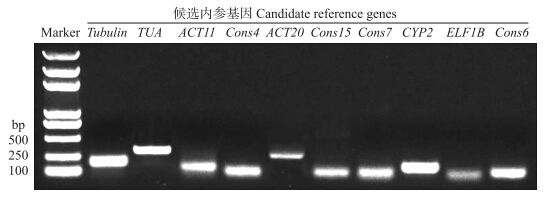
|
图 2 大豆10个候选内参基因的PCR扩增产物琼脂糖凝胶电泳检测 Fig. 2 Agarose gel analysis of 10 candidate reference soybean genes for qRT-PCR amplification |
苗期大豆不同组织和大豆疫霉处理大豆苗根部不同时间点均设3~4个生物学重复,利用CT值估计基因的表达水平,CT值越高,表达丰度越低,同一基因的CT值在不同样品间的变化反映了表达水平的变异[25]。分析发现每个内参基因在不同大豆样品中的表达水平都有一定的变化。如表 2所示:在苗期大豆不同组织样品中,CYP 2基因的CT值为16.97~17.78,是10个基因中表达丰度最高的;其次为TUA、Tubulin和ACT 20,CT值均为16.90~20.96;另外6个基因的CT值为21.32~30.36。在大豆疫霉处理大豆苗根部不同时间点样品中:TUA、ACT 20、ACT11这3个基因的CT值均为18.92~21.84,在不同样品间的CT值相对较低且变化较小;CYP 2基因的CT值为17.74~20.19,在不同样品间CT值相对较低但变化较大;剩余6个基因的CT值为20.72~30.33,相对较高。以上结果表明:内参基因CYP 2在大豆的不同组织中的表达丰度较高,表达水平变异小;其余基因或表达丰度低或表达水平变异大。在大豆疫霉侵染苗期大豆根的不同时间点样品间,ACT 20、ACT11 、TUA这3个基因的表达丰度较高,同时其表达水平变异小;CYP 2基因虽然表达丰度较高,但在不同样品间表达水平变异大;其他基因从CT值可以看出其表达丰度不高或表达水平变异大。此外,这些内参基因在苗期大豆不同组织和大豆疫霉侵染根部不同时间点表达变化的规律性不强,需要进一步借助内参基因分析软件对其稳定性进行评估。
| 组织/疫霉处理时间点 Different tissue or time points | 基因Gene | ||||
| Tubulin | TUA | ACT11 | Cons4 | ACT20 | |
| 根(0 h)Root | 20.83±0.020 | 19.28±0.030 | 19.81±0.032 | 25.99±0.006 | 18.92±0.028 |
| 茎Stem | 18.96±0.045 | 19.98±0.045 | 21.58±0.102 | 27.43±0.018 | 20.96±0.044 |
| 叶Leaf | 17.35±0.110 | 16.90±0.073 | 17.98±0.020 | 24.96±0.036 | 18.50±0.007 |
| 0.5 h | 20.77±0.015 | 19.95±0.017 | 19.22±0.040 | 25.83±0.049 | 21.41±0.034 |
| 1 h | 21.58±0.003 | 20.98±0.015 | 20.33±0.016 | 26.52±0.070 | 19.97±0.033 |
| 2 h | 21.25±0.010 | 20.58±0.022 | 19.56±0.028 | 25.54±0.015 | 20.71±0.157 |
| 4 h | 21.76±0.006 | 20.54±0.009 | 19.53±0.008 | 25.95±0.055 | 21.06±0.058 |
| 8 h | 22.98±0.031 | 21.84±0.058 | 21.24±0.012 | 27.09±0.031 | 20.41±0.084 |
| 16 h | 20.72±0.049 | 19.83±0.026 | 19.10±0.004 | 25.40±0.030 | 19.92±0.067 |
| 24 h | 23.89±0.086 | 20.90±0.047 | 19.95±0.033 | 26.04±0.050 | 19.31±0.058 |
| 组织/疫霉处理时间点 Different tissue or time points | 基因Gene | ||||
| Cons15 | Cons7 | CYP2 | ELF1B | Cons6 | |
| 根(0 h)Root | 28.91±0.071 | 23.94±0.071 | 17.74±0.023 | 21.96±0.030 | 22.97±0.030 |
| 茎Stem | 30.36±0.192 | 26.48±0.067 | 17.78±0.057 | 23.49±0.046 | 24.27±0.016 |
| 叶Leaf | 27.78±0.110 | 22.76±0.041 | 16.97±0.028 | 21.86±0.036 | 21.32±0.011 |
| 0.5 h | 28.49±0.040 | 25.76±0.024 | 18.28±0.057 | 24.57±0.033 | 23.30±0.022 |
| 1 h | 28.00±0.154 | 23.67±0.008 | 19.30±0.024 | 25.55±0.007 | 23.90±0.039 |
| 2 h | 28.78±0.024 | 24.21±0.046 | 18.34±0.022 | 27.54±0.010 | 22.90±0.011 |
| 4 h | 29.67±0.274 | 25.77±0.063 | 20.15±0.049 | 30.33±0.165 | 24.94±0.050 |
| 8 h | 29.43±0.123 | 24.80±0.029 | 20.19±0.037 | 26.88±0.067 | 24.81±0.037 |
| 16 h | 29.02±0.015 | 24.99±0.030 | 19.09±0.050 | 29.11±0.087 | 24.43±0.036 |
| 24 h | 28.14±0.070 | 24.32±0.110 | 19.77±0.171 | 25.19±0.030 | 24.51±0.037 |
为了进一步分析这些内参基因在苗期大豆不同组织和大豆疫霉处理根部不同时间点样品中的表达稳定性,我们借助3个常用的统计学软件BestKeeper、GeNorm和NormFinder,分析10个候选持家基因的CT值,从而明确各个基因的稳定性。
2.4.1 GeNorm软件分析首先将各个基因的CT值转化成相对表达量,数据输入GeNorm软件,软件将计算各个基因在不同试验条件下的平均表达稳定指数(M值),M值的大小反映了基因稳定性的高低。软件默认M值的临界值为0.5,若某个内参基因的M值小于0.5,可以考虑将其作为内参基因。分析结果显示:10个持家基因在不同样品中稳定性有差异。其中在大豆疫霉游动孢子处理的大豆根样品中,10个内参基因的表达稳定性从高到低依次为:Cons 4、ACT11、TUA、Cons15、ACT20、Cons7、CYP2、Cons6、Tubulin、ELF1 B(图 3-A),Cons 4、ACT11 、TUA的M值均小于0.5,表达稳定性较高,可以作为内参基因的选择对象。其中Cons 4和ACT11的M值为0.3,说明这2个基因更适合作为大豆疫霉侵染病程中基因表达分析的内参基因。10个内参基因在苗期大豆不同组织中的表达稳定性从高到低依次为:Cons 4、Cons15、Cons6、ACT20、ACT11、Cons7、TUA、ELF1B、CYP2 、Tubulin(图 3-B)。Cons 4和Cons15的M值为0.064,表达最稳定;除了ELF 1B、CYP2和Tubulin之外,剩余4个基因的M值均小于0.5,说明这4个基因也可以作为研究苗期大豆不同组织基因表达的内参基因。
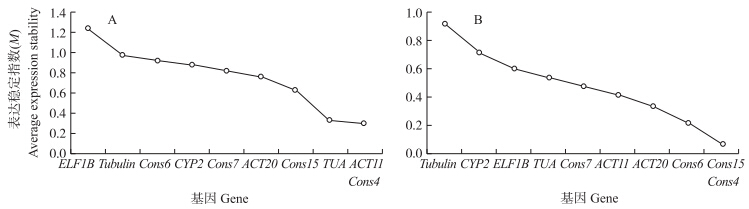
|
图 3 GeNorm软件分析10个候选内参基因在疫霉侵染大豆根(A)和不同组织(B)中的表达稳定性 Fig. 3 The expression stability of ten candidate reference genes at P.sojaeinfection roots(A) and in different tissues(B)analyzed by GeNorm |
GeNorm软件不但可以分析基因的表达稳定性,而且可以分析使用多个内参基因时的配对差异。在同一试验条件下,当有多个内参基因可以选择时,软件会分析其最佳配对变异值Vn/n+1,从而为我们选择最适的内参基因数目提供参考依据。程序默认的基因配对比值为0.15,当有n个内参基因时,比值Vn/n+1<0.15,说明n个内参基因已经能够准确校正目标基因的表达量,此时无需引入第(n+1)个内参基因。从软件分析结果(图 4)可以看出,V2/3(不同组织)=0.099,V2/3(大豆疫霉处理)=0.103,均小于0.15,说明在大豆疫霉侵染的大豆根样品和苗期大豆不同组织样品中,都没有必要引入第3个内参基因来消除差异。因此,在对大豆疫霉处理的大豆根和大豆不同组织样品的qRT-PCR分析中,根据M值以及其最适的基因配对数,最佳的内参基因组合分别为Cons 4/ACT11和Cons4/Cons15。
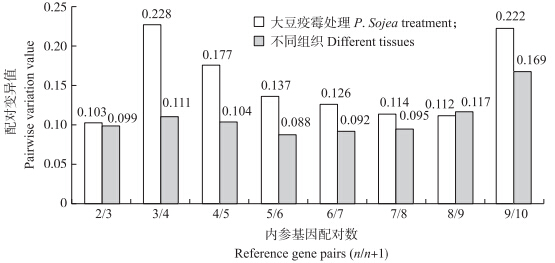
|
图 4 GeNorm软件分析最佳内参基因数目 Fig. 4 Analysis of the optimal numbers of reference genes by GeNorm software |
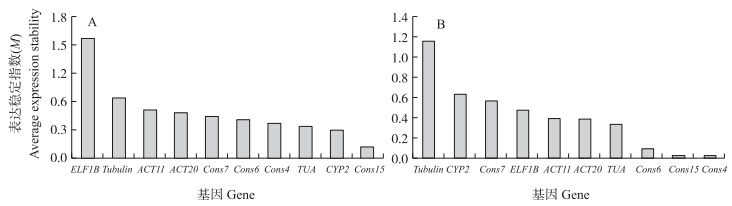
和GeNorm软件类似,NormFinder软件在分析数据之前,需要将基因的CT值转化成相对表达量,软件分析结果会给出基因表达稳定指数M,参考M值的大小来筛选稳定的内参基因,M值越小,基因表达越稳定。由图 5-A可知:在大豆疫霉游动孢子处理的大豆根样品中,基因表达稳定性从高到低依次为:Cons 15、CYP2、TUA、Cons4、Cons6、Cons7、ACT20、ACT11、Tubulin、ELF1B。其中,Cons15的M值为0.133,为最小,表达稳定性最高;其次是CYP 2、TUA、Cons4等。在大豆不同组织的样品中,同样根据10个内参基因的M值(图 5-B),稳定性从高到低依次为:Cons 4、Cons15、Cons6、TUA、ACT20、ACT11、ELF1B、Cons7、CYP2、Tubulin。其中,Cons4和Cons15的M值仅为0.022,表明这2个基因最稳定,其次为Cons 6。
|
图 5 NormFinder软件分析10个候选内参基因在大豆疫霉侵染的大豆根中(A)和不同组织中(B)的表达稳定性 Fig. 5 The expression stability of ten candidate reference genes at P.sojaeinfected roots and in different tissues analyzed by NormFinder |
利用BestKeeper软件直接分析每个候选内参基因在相应试验条件下CT值之间的标准差(SD)和变异系数(CV),其大小反映基因稳定性差异,最终选择SD和CV都较小的基因作为内参基因。其中软件默认SD的临界值为1,当候选内参基因的SD值大于1时,说明该基因的稳定性较差。如表 3所示:在大豆疫霉诱导下大豆根的样品中,Cons 4的SD和CV是10个内参基因中最小的,说明该基因最稳定,其次是ACT 11和TUA,而ELF1 B的SD值大于1,稳定性最差;在大豆的不同组织中,根据以上2个评价指标,10个内参基因中最稳定的基因是CYP 2,其次是ELF1B、Cons4和Cons15,而Cons7、Cons6、TUA、ACT20、ACT11和Tubulin的SD值均大于1,说明这6个基因在苗期大豆的不同组织中稳定性都较差。
| 分析参数 Parameter | 大豆疫霉侵染的大豆根P.sojaeinfected roots | |||||||||
| Tubulin | Cons15 | TUA | Cons7 | ACT11 | CYP2 | Cons4 | ELF1B | ACT20 | Cons6 | |
| GM[CP] | 21.75 | 28.58 | 20.55 | 24.64 | 19.85 | 18.94 | 26.00 | 26.38 | 20.23 | 23.69 |
| AM[CP] | 21.77 | 28.59 | 20.56 | 24.65 | 19.86 | 18.97 | 26.00 | 26.49 | 20.24 | 23.72 |
| Min[CP] | 20.72 | 27.20 | 19.82 | 23.67 | 19.10 | 16.65 | 25.40 | 22.70 | 19.16 | 20.99 |
| Max[CP] | 23.89 | 29.67 | 21.84 | 25.77 | 21.24 | 20.19 | 27.09 | 30.34 | 21.41 | 24.94 |
| SD | 0.83 | 0.63 | 0.52 | 0.68 | 0.50 | 0.91 | 0.41 | 1.98 | 0.65 | 0.99 |
| CV/% | 3.81 | 2.22 | 2.54 | 2.76 | 2.54 | 4.80 | 1.59 | 7.48 | 3.23 | 4.20 |
| 分析参数 Parameter | 苗期大豆不同组织Different tissues of soybean | |||||||||
| Tubulin | Cons15 | TUA | Cons7 | ACT11 | CYP2 | Cons4 | ELF1B | ACT20 | Cons6 | |
| GM[CP] | 18.99 | 29.00 | 18.67 | 24.34 | 19.73 | 17.49 | 26.11 | 22.43 | 19.43 | 22.82 |
| AM[CP] | 19.04 | 29.02 | 18.72 | 24.39 | 19.79 | 17.50 | 26.13 | 22.44 | 19.46 | 22.85 |
| Min[CP] | 17.35 | 27.78 | 16.90 | 22.76 | 17.98 | 16.98 | 24.96 | 21.86 | 18.50 | 21.32 |
| Max[CP] | 20.83 | 30.36 | 19.98 | 26.48 | 21.58 | 17.78 | 27.43 | 23.49 | 20.96 | 24.27 |
| SD | 1.19 | 0.90 | 1.21 | 1.39 | 1.21 | 0.35 | 0.87 | 0.70 | 1.00 | 1.02 |
| CV/% | 6.24 | 3.09 | 6.48 | 5.69 | 6.10 | 2.00 | 3.32 | 3.13 | 5.15 | 4.48 |
| 注:GM:几何平均数GEOMEAN;AM:算术平均数Average mean;CP:置信参数Confidence parameter | ||||||||||
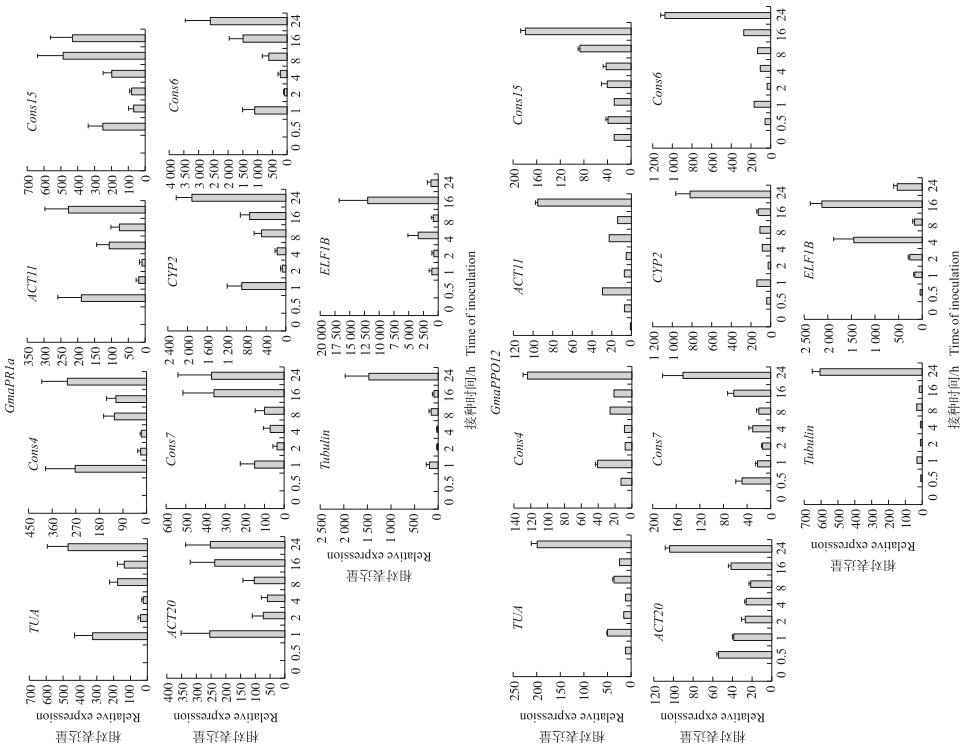
为了进一步验证本试验中10个候选持家基因在分别作为内参基因时,对qRT-PCR结果的不同影响,采用qRT-PCR法分析了大豆中2个已报道的大豆疫霉诱导性基因GmaPPO 12、GmaPR1 a[20]的表达量。分别以10个候选持家基因为内参基因,分析2个大豆疫霉诱导性基因的相对表达量,结果如图 6所示:当以Cons 4、ACT11和TUA为内参基因时,这2个基因均表现出各自一致的疫霉诱导表达趋势,以Cons 15、ACT20、Cons7、CYP2和Cons6为内参基因时,表达趋势与前3个内参基因有所不同,但也可以基本反映基因的表达趋势,以Tubulin和ELF1B为内参基因时,其表达趋势与前几个内参基因明显不同,说明Tubulin和ELF1B这2个基因不适合作大豆疫霉侵染过程中基因表达研究的内参基因。
|
图 6 以不同的持家基因为内参基因分别分析大豆中疫霉诱导性基因GmaPR 1a和GmaPPO12的表达 Fig. 6 The expression of P.sojeainduced expression genes GmaPR1aand GmaPPO 12analyzed using various housekeeping genes as references |
应用qRT-PCR技术研究基因的表达水平,常选用持家基因作为衡量目标基因表达量的参考标准,用稳定表达的持家基因可以校正目标基因的转录效率、起始模板cDNA用量等,避免不同样品制备过程中由于纯度、浓度、操作方法等不同造成的误差,从而为试验结果的准确性提供重要保障[2, 26]。被选用的持家基因作为内参基因时,要求其表达不受外部环境影响,在所有细胞中都能稳定表达,同时其表达水平要尽可能与目标基因一致。然而,在实际应用中,常用的内参基因在不同条件下的表达可能是不稳定的[8],如果不经过试验验证,随意选择一种持家基因作为qRT-PCR分析的内参基因,可能会出现目标基因表达变化的微小差异这种难以发现的现象,甚至会得到完全错误的结论[2, 23]。因此,针对不同试验条件分析和筛选稳定的内参基因是十分重要的。
目前关于大豆不同组织中、不同光周期影响下、不同生长发育过程中以及不同品种内参基因稳定性的研究有一些报道。如Libault等[22]首先应用基因芯片技术,研究了200个大豆持家基因在大豆根瘤形成过程中的表达量变化,进一步研究其中的18个基因在多达130种处理下的样品间表达量变化,最终获得4个在不同条件影响下都能稳定表达的大豆持家基因,并推荐其作为大豆qRT-PCR分析的内参基因。Jian等[21]研究了10个大豆常用内参基因分别在21种处理下的品种中表达量差异,鉴定到3个稳定表达的内参基因。胡瑞波[27]结合文献报道,对7个传统的持家基因,以及7个新报道的持家基因在116份大豆样品间的表达稳定性进行评价,样品处理采用与Jian等类似的方法,其结果表明大多数新报道的持家基因表达稳定性优于传统的持家基因,而来源于同一家族的不同基因其表达稳定性不同。重要的是,这些研究结果均表明,同一持家基因在不同品种间、不同处理下,其表达稳定性是不同的。我们注意到在这些已报道的研究中,几乎没有涉及病原菌诱导条件下的大豆样品,参考这些研究的理论基础和试验结果,本研究挑选了10个在非病原诱导下稳定表达的内参基因,希望寻找到在大豆疫霉侵染过程中,稳定表达的大豆内参基因,为大豆抗疫霉基因工程研究中基因表达量分析提供依据。
本研究选用3个常用的分析内参基因稳定性软件BestKeeper、NormFinder和GeNorm,对10个已报道的大豆内参基因稳定性进行评价。结果表明BestKeeper和GeNorm的评价结果相对一致,而NormFinder的分析结果与前两者有差别,结果导致用这3个软件分析基因的稳定性排序不一致。我们在查阅其他文献时也发现了类似的现象,如在研究二穗短柄草内参基因[28]、亚麻内参基因[29]、柑橘内参基因[30]、黄瓜内参基因[31]时,均出现类似结果,我们推测造成这种差异的根本原因可能是软件的统计学原理不同造成的[32]。虽然3个软件的评价结果有差异,但是在舍去变化较大的数值后,大部分候选持家基因的稳定性较一致。综合软件的分析结果,发现Cons 4、ACT11和TUA在大豆疫霉侵染过程中的大豆根中表达稳定性最好,Cons15、CYP2、Cons4 在苗期大豆不同组织中表达稳定性较好。当根据试验要求,需要同时选择多个内参基因作为校正标准时,可以分别选择相应试验条件下最稳定内参基因中的任意2个基因配合使用即可。而Tubulin和ELF1B在大豆的所有样品中表达稳定性较差,因此不推荐这2个基因作为内参基因使用。我们的研究也进一步证实,持家基因的表达在不同条件下其稳定性是不同的。为了验证这些持家基因在病原侵染进程中的表达稳定性,我们进一步选择了2个已报道的大豆疫霉诱导性基因GmaPPO 12和GmaPR1 a[20],采用qRT-PCR的方法,以这10个候选持家基因作为内参基因,进行疫霉诱导表达分析。从结果来看,以Cons 4、ACT11和TUA为内参基因时,这2个基因均各自表现出一致的疫霉诱导表达趋势,以Cons 15、ACT20、Cons7、CYP2和Cons6为内参基因时,表达趋势有所不同,以Tubulin和ELF1B为内参基因时,其表达趋势与前几个明显不同,说明Tubulin和ELF1B基因不适合作为大豆疫霉侵染大豆病程中基因表达研究的内参基因。
本试验以苗期大豆根、茎、叶,以及苗期大豆根在大豆疫霉诱导过程中不同时间点的材料为研究对象,主要是因为大豆疫霉为土传病害,在大豆与大豆疫霉的互作研究中,我们经常会选用苗期的根、茎、叶为材料开展研究。本研究的结果为苗期大豆抗疫霉基因工程研究中qRT-PCR分析内参基因的选择提供依据。
| [1] | Bustin S A,Benes V,Nolan T,et al. Quantitative real-time RT-PCR a perspective[J]. Journal of Molecular Endocrinology,2005,34(3):597-601 |
| [2] | Huggett J,Dheda K,Bustin S,et al. Real-time RT-PCR normalization;strategies and considerations[J]. Genes and Immunity,2005,6(4):279-284 |
| [3] | Wan H J,Zhao Z G,Qian C T,et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry,2010,399(2):257-261 |
| [4] | Gachon C,Mingam A,Charrier B. Real-time PCR:what relevance to plant studies?[J]. Journal of Experimental Botany,2004,55(402):1445-1454 |
| [5] | Bustin S A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays[J]. Journal of Molecular Endocrinology,2000,25(2):169-193 |
| [6] | VanGuilder H D,Vrana K E,Freeman W M. Twenty-five years of quantitative PCR for gene expression analysis[J]. Biotechniques,2008,44(5):619-626 |
| [7] | Pfaffl M W,Tichopad A,Prgomet C,et al. Determination of stable housekeeping genes,differentially regulated target genes and sample integrity:BestKeeper—Excel-based tool using pair-wise correlations[J]. Biotechnology Letters,2004,26(6):509-515 |
| [8] | Gutierrez L,Mauriat M,Guénin S,et al. The lack of a systematic validation of reference genes:a serious pitfall undervalued in reverse transcription-polymerase chain reaction(RT-PCR)analysis in plants[J]. Plant Biotechnology Journal,2008,6(6):609-618 |
| [9] | Bustin S A,Nolan T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction[J]. Journal of Biomolecular Techniques,2004,15(3):155-166 |
| [10] | Gao Z H,Wei J H,Yang Y,et al. Selection and validation of reference genes for studying stress-related agarwood formation of Aquilaria sinensis[J]. Plant Cell Reports,2012,31(9):1759-1768 |
| [11] | Hu R,Fan C,Li H,et al. Evaluation of putative reference genes for gene expression normalization in soybean by quantitative real-time RT-PCR[J]. BMC Molecular Biology,2009,10:93 |
| [12] | Spinsanti G,Panti C,Lazzeri E,et al. Selection of reference genes for quantitative RT-PCR studies in striped dolphin(Stenella coeruleoalba)skin biopsies[J]. BMC Molecular Biology,2006,7:32 |
| [13] | Radonic A,Thulke S,Mackay I M,et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochemical and Biophysical Research Communications,2004,313(4):856-862 |
| [14] | Zhu X Y,Li X P,Chen W X,et al. Evaluation of new reference genes in papaya for accurate transcript normalization under different experimental conditions[J]. PLoS One,2012,7(8):e44405 |
| [15] | Liener I E. Implications of antinutritional components in soybean foods[J]. Critical Reviews in Food Science Nutrition,1994,34(1):31-67 |
| [16] | Ososki A L,Kennelly E J. Phytoestrogens:a review of the present state of research[J]. Phytotherapy Research,2003,17(8):845-869 |
| [17] | Sakai T,Kogiso M. Soy isoflavones and immunity[J]. The Journal of Medical Investigation,2008,55(3/4):167-173 |
| [18] | Tyler B M. Phytophthora sojae:root rot pathogen of soybean and model oomycete[J]. Molecular Plant Pathology,2007,8(1):1-8 |
| [19] | Guo N,Ye W W,Wu X L,et al. Microarray profiling reveals microRNAs involving soybean resistance to Phytophthora sojae[J]. Genome,2011,54(11):954-958 |
| [20] | Chai C,Lin Y,Shen D,et al. Identification and functional characterization of the soybean GmaPPO12 promoter conferring Phytophthora sojae induced expression[J]. PLoS One,2013,8(6):1-6 |
| [21] | Jian B,Bin L,Yu R B,et al. Validation of internal control for gene expression study in soybean by quantitative real-time PCR[J]. BMC Molecular Biology,2008,9:59 |
| [22] | Libault M,Thibivilliers S,Bilgin D D,et al. Identification of four soybean reference genes for gene expression normalization[J]. The Plant Genome,2008,1:44-54 |
| [23] | Vandesompele J,de Preter K,Pattyn F,et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology,2002,3(7):research0034 |
| [24] | Andersen C L,Jensen J L,Orntoft T F. Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J]. Cancer Research,2004,64(15):5245-5250 |
| [25] | Schefe J H,Lehmann K E,Buschmann I R,et al. Quantitative real-time RT-PCR data analysis:current concepts and the novel"gene expression's CT difference"formula[J]. Journal of Molecular Medicine,2006,84(11):901-910 |
| [26] | Steinau M,Rajeevan M S,Unger E R. DNA and RNA references for qRT-PCR assays in exfoliated cervical cells[J]. The Journal of Molecular Diagnostics,2006,8(1):113-118 |
| [27] | 胡瑞波. 大豆FT/TFL1基因克隆、表达模式及功能分析[D]. 北京:中国农业科学院研究生院,2009:22-38 [Hu R B. Molecular cloning,expression profiles and functional analysis of FT/TFL1 genes in soybean(Glycine max)[D]. Beijing:Graduate School of Chinese Academy of Agriculture Sciences,2009:22-38(in chinese with English abstract)] |
| [28] | Hong S Y,Seo P J,Yang M S,et al. Exploring valid reference genes for gene expression studies in Brachypodium distachyon by real-time PCR[J]. BMC Plant Biology,2008,8:112 |
| [29] | Huis R,Hawkins S,Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax(Linum usitatissimum L.)[J]. BMC Plant Biology,2010,10:71 |
| [30] | Mafra V,Kubo K S,Alves-Ferreira M,et al. Reference genes for accurate transcript normalization in citrus genotypes under different experimental conditions[J]. PLoS One,2012,7(2):e31263 |
| [31] | Wan H,Zhao Z,Qian C,et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry,2010,399(2):257-261 |
| [32] | Hu R B,Qi G,Kong Y Z,et al. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa[J]. BMC Plant Biology,2010,10:145 |
 2015, Vol. 38
2015, Vol. 38


