文章信息
- 吴晶晶, 魏子尧, 杨春艳, 王文艳, 朱星洁, 刘世家, 江玲, 万建民. 2015.
- WU Jingjing, WEI Ziyao, YANG Chunyan, WANG Wenyan, ZHU Xingjie, LIU Shijia, JIANG Ling, WAN Jianmin. 2015.
- 控制水稻抽穗延迟基因OsSET34的鉴定和图位克隆
- Identification and map-based cloning of OsSET34,a gene controling the delay of heading date in rice(Oryza sativa L).
- 南京农业大学学报, 38(5): 704-711
- Journal of Nanjing Agricultural University, 38(5): 704-711.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.05.002
-
文章历史
- 收稿日期:2015-01-28
抽穗期是影响水稻种植地区、季节适应性及产量的重要农艺性状,受多基因调控,遗传基础复杂。目前主要通过构建NILs(near-isogenics lines,近等基因系)、CSSLs(chromosome segment substitution lines,染色体片段置换系)、T-DNA插入突变体以及辐射诱变突变体的定位群体等方法对基因进行定位与克隆。截至2014年7月底,根据Gramene网站(http://www.gramene.org/qtl/)公布的数据,已发现近800个与水稻抽穗期相关的QTL,分布于水稻各染色体上。目前,已对多个水稻抽穗期相关的基因进行了克隆和功能分析。其中,日本Masahiro Yano课题组对水稻抽穗期QTL的定位研究最为广泛和深入,他们利用对光周期敏感的粳稻品种‘日本晴’与籼稻品种‘Kasalath’杂交所得的F2群体及回交后代群体,成功定位出15个水稻抽穗期QTL[1]。利用‘日本晴’与‘越光’的BIL(backcross inbred lines)群体,他们还检测到位于第3条染色体和第6条染色体上的Hd 16与Hd17 [2],并对Hd 1、Hd3a、Hd6 等位点进行了克隆和深入的功能分析。
高等植物的开花是一个复杂的、受多种因素调控的过程,开花转换决定了植物生殖发育的时期和质量,对于植物发育具有非常重要的意义。拟南芥作为长日照模式植物,主要存在4种开花转换途径:光周期途径、自主开花途径、春化途径和生物钟途径[3, 4, 5]。水稻作为短日照模式植物,目前深入研究的开花转换途径主要是光周期途径。近年来,随着水稻抽穗期基因不断被克隆和功能分析,光周期调控网络的模型逐渐清晰,在光周期开花中主要有两条途径:短日照调控途径和长日照调控途径。在短日照条件下,主要有两条途径促进水稻开花:一条途径是与拟南芥同源的途径OsGI-Hd 1-Hd3a,另一条则为水稻特有的Ehd1途径。拟南芥为长日照植物,光周期开花的主要信号通路是GI-CO-FT。但Hd1/CO调控Hd3a/FT 在2种植物中是相反的,在长日照植物拟南芥中起促进作用,而在短日照植物水稻中起抑制作用[6, 7]。Hd 1 是OsGI最重要的下游基因,编码产物包含395个氨基酸,含有锌指结构域,与拟南芥带有锌指结构域的CO同源,能够作用于下游的Hd 3a而促进其表达。Hd1在短日照条件下上调Hd3 a的分子机制与phytochrome B有关。Ehd 1 来源于非洲栽培稻(Oryza glaberrima Steud.)的早抽穗QTL。Doi等[8]证明,在缺乏Hd 1参与情况下,能单独诱导水稻在短日照条件下提早抽穗。在拟南芥中并没有发现Ehd1 的同源基因[9],所以,调控路径也不相同。Ehd 1 编码1个B型响应调控蛋白,通过与DNA结合的GARP结构域进行调控。在短日照条件下,Ehd 1的表达受到抑制,进而抑制开花基因SE5和OsCOL4 ,调控开花。
水稻虽为短日照植物,但在长日照条件下也能完成抽穗,说明一定存在长日照条件下促进水稻开花的因子。研究表明:RFT 1 编码1个含有PBP结构域的蛋白产物[10],与Hd 3a同源性最高,是长日照条件下的开花促进因子。通过对RFT1 的干扰植株研究发现:干扰植株在长日照条件下延迟抽穗大概100 d,在短日照条件下效果却不明显,更进一步证明了RFT 1是水稻长日照条件下的促开花因子。所以,长日照促进开花途径主要由‘成花素’基因RFT1介导,其表达主要受Ehd1诱导。在长日照下促进Ehd1表达的上游基因主要有OsMADS50、OsMADS51、Ehd2、Ehd3和Ehd4 等。尽管国内外许多科学家做了大量研究,但综合而言,对水稻抽穗期调控机制的研究并不十分透彻,仍然有许多未被发掘的控制抽穗期基因,调控网络还需要进一步完善。在本研究中,我们在南京长日照条件下筛选到1个‘南粳35’抽穗期延迟的突变体,利用与籼稻品种‘93-11’配组的F2群体对基因进行了初定位和精细定位,并通过基因预测、测序等方法找到了在长日照和短日照条件下均能促进水稻开花的基因,并对其调控机制进行了分析。
1 材料与方法 1.1 试验材料及田间试验在60Co-γ射线辐射诱变的‘南粳35’M2代中,筛选获得1个抽穗期延迟突变体,经多代自交,其迟抽穗突变表型已稳定遗传。将该突变体与野生型‘南粳35’正反交,对其后代群体进行遗传分析,用与籼稻品种‘93-11’杂交配组获得的遗传分离群体进行基因定位和图位克隆。在南京土桥分别种植亲本、F1和F2,观察并记录抽穗表型及其他农艺性状。所有材料分单株种植,每行10株,行距20 cm,株距15 cm。田间管理同大田生产。
1.2 不同日照长度处理抽穗期相关基因表达分析 2013年6月11日在南京农业大学水稻研究所人工气候室长日照条件下正常播种‘南粳35’和突变体,生长30 d后分别进行长、短日照处理,设置人工长日照条件(14 h光照/10 h黑暗)和人工短日照条件(10 h光照/14 h黑暗),处理7 d后取每个单株的叶片,从光照开始每4 h取样1次,每次随机取3个单株,共取6次,-80 ℃保存。用于抽穗期基因OsSET 34、OsMADS50、OsMADS51和Ehd1 表达分析,所用引物见表 1。| 基因Gene | 引物序列Primer sequence(5′→3′) | |
| OsSET34 | F:AAAATGAAGAGGCAGGGTG | R:CTCCCTCAACAGTCCATTTCTG |
| OsMADS50 | F:AAAGCTGACGCTGATGGTTTG | R:GTTTCGACATCCATGTTGTC |
| OsMADS51 | F:GTTTGCTCTGCTCCTACTC | R:ACTCCTCCTCCAGCATTGAA |
| Ehd1 | F:GAGCAAGTTGCCAGTC | R:CATGCACTCTGAGCCA |
| Ubq | F:AACCAGCTGAGGCCCAAGA | R:ACGATTGATTTAACCAGTCCATGA |
抽穗期以播种日至抽穗日经历的天数表示。调查抽穗期的方法为:以植株第一穗露出叶鞘2~3 cm记为该单株抽穗,从植株开始有孕穗迹象开始调查,每隔2 d调查1次,记录每株的抽穗日期,然后计算从播种到抽穗的天数。
1.4 杂交配组由于在南京长日照条件下突变体的抽穗期太晚以致不能正常结实,这给杂交配组带来不便。因此需要将南京正季突变体的稻桩移栽至海南进行杂交及繁种工作。突变体与分期播种的籼稻品种‘93-11’杂交配组,同时与分期播种的‘南粳35’配正反交组合进行遗传分析。杂交方法为当天下午对突变体进行去雄,然后在柱头及残留的花粉上喷足够多的水,使花药细胞吸水胀破,第2天早上先将吸水胀破的花药弹出,并套上纸袋防止串粉,于上午‘93-11’开花时取正在开花的稻穗,将花粉抖落在袋内,封口,完成杂交。
1.5 DNA提取及分子标记分析对突变体/93-11分离群体的表型进行鉴定,从中挑选出表现迟抽穗的极端个体,挂牌取样,叶片取下后置于-20 ℃保存。参照McCouch等[11]的方法提取水稻叶片总DNA,用于分子标记分析。
根据McCouch等[12]构建的SSR连锁图获得简单序列重复(SSR)标记,对均匀分布于12条染色体上的标记进行多态性筛选,用具有多态性的标记对群体中挑选到的极端个体进行标记分析。将具有突变体带型的单株记为Ⅰ型,具有‘93-11’带型的单株记为Ⅱ型,同时具有突变体和‘93-11’带型的杂合单株记为Ⅲ型。以突变体和‘93-11’的DNA为对照,对30株极端个体进行基因型和表型连锁分析,确定与目的基因连锁的标记后,进一步扩大群体,在初定位基础上利用Primer Premier 5.0软件设计SSR和Indel标记引物(表 2),缩小定位区间实现精细定位。
| 引物名称Primer name | 引物序列Primer sequence(5′→3′) | |
| RM5526 | F:CACATGATCCTCCACCCACTAGC | R:GCCTGGCCTCTCTTATCTGTCTACC |
| RM24001 | F:ACCGGGTCAAATATACTACTCCATCC | R:AGTGTCACGTGTCGAATCAAGAGC |
| SJ-9-3 | F:AAGCGTTGGGTTTCCTC | R:ACGGGAAAGATTGTGGG |
| SJ-9-9 | F:TGGCAGATTGAGGGGC | R:TTATCCTGTAATCAGCAAGAAAG |
| SJ-9-23 | F:ATTAGTTTTTCAGACGGAGAGAG | R:CACGCAACATAGCACAAGG |
| SJ-9-29 | F:GATTGAGCCAACCCCT | R:TCTTGCTACTCTGAAACATTAT |
| SJ-9-35 | F:CAGTTTAGCGATAGGTAGCC | R:CGCCACTTTTTCAAGCATA |
| W5 | F:GACACAACACACACAGAGAA | R:CCCAAGTCCAAACCAGAG |
| W15 | F:GCTCGCTCATCTACAATCTC | R:TCATCTTCATCTCTCCATCC |
| W21 | F:TGGTTTGGACTTGGGCA | R:AAGCAAAGTAAAAGAGACAAGA |
| W24 | F:TAACGCCCTCCTGTCCTT | R:CGCCACTGCTTCTACTCC |
| W25 | F:AGCACCCGACGCCACAG | R:GCACACATACACACAAGAGATA |
在精细定位锁定的目标染色体区域,根据水稻基因组注释数据库信息预测候选基因。设计引物,分别对野生型和突变体的候选基因进行PCR扩增、测序,比对野生型和突变体的序列信息,确定突变位点及对应的候选基因。
1.7 Quantitative RT-PCR分析使用天根公司的RNA提取试剂盒提取RNA。反转录过程参照TaKaRa公司的反转录方法。Quantitative RT-PCR采用BIO-RAD公司的iTaqTM Universal SYBR® Green Supermix,20 μL反应体系为:iTaqTM Universal SYBR® Green Supermix(2×)10 μL,PCR上、下游引物各300~500 nmol · L-1,cDNA 100 ng,ddH2O补齐至20 μL。在Applied Biosystems的ABI PRISM7900HT Real-time PCR仪上进行反应。水稻Ubq基因(LOC_Os03g13170)作为内参。
2 结果与分析 2.1 野生型和突变体的表型分析从2011—2014年,在南京自然长日照条件下,野生型‘南粳35’大约在播种后100 d开始抽穗,突变体则到播种后大约150 d开始抽穗。在人工短日照条件处理下,‘南粳35’和突变体均较自然长日照条件下早抽穗,‘南粳35’较长日照条件下提前8 d抽穗,突变体较长日照条件下提前35 d抽穗。在相同的短日照条件下,突变体仍比野生型迟抽穗23 d(图 1)。说明突变体迟抽穗的表型不仅受日照长度的影响,还受其他因素的影响。我们将此突变体表型命名为activate heading(t)(ah(t))。通过对来自海南收获的‘南粳35’和ah(t)种子进行观察发现:ah(t)种子的粒长、粒宽、粒厚和千粒质量均较野生型低,其中粒厚和千粒质量差异显著(表 3)。
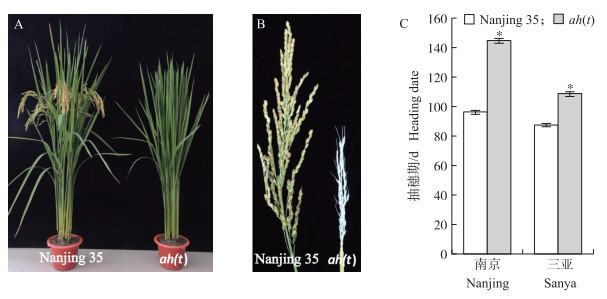 |
图 1 野生型和突变体表型
Fig. 1 The phenotype analysis of wild type and the mutant
A.野生型(左)和突变体ah(t)(右)在南京种植133 d的表型;B.野生型(左)和突变体ah(t)(右)在南京生长133 d的穗发育表型,突变体ah(t)没有抽穗,只有发育的幼穗;C.野生型和突变体ah(t)在南京和三亚抽穗期情况。 A.The phenotypes of wild-type(left)and ah(t)mutant(right)plants grown for 133 d in a paddy field at Nanjing;B.Panicles of wild-type(left)and ah(t)mutant(right)plants grown for 133 d in Nanjing,the ah(t)mutant was not heading at this time and only developed immature panicles;C.Heading date investigation of wild-type and ah(t)mutantplants grown in Nanjing and Sanya. |
| 类型Type | 品种Variety | 粒长/mmGrain length | 粒宽/mmGrain width | 粒厚/mmGrain thickness | 千粒质量/g1 000-grain weight |
| 稻谷Rice grain | 南粳35 Nanjing 35 | 7.45±0.14 | 3.14±0.10 | 2.13±0.04** | 24.78±0.93* |
| ah(t) | 7.39±0.11 | 3.10±0.06 | 1.97±0.03 | 21.73±0.44 | |
| 糙米Brown rice | 南粳35 Nanjing 35 | 5.28±0.12 | 2.80±0.07 | 1.94±0.11** | 19.88±0.80* |
| ah(t) | 5.16±0.24 | 2.75±0.11 | 1.70±0.12 | 17.37±0.21 | |
| 注: *和* *分别表示‘南粳35’与ah(t)在0.05和0.01水平上差异显著。 * and * * mean significant difference at 0.05 and 0.01 levels between‘Nanjing 35’and ah(t),respectively. The same as follows. | |||||
对突变体ah(t)和‘南粳35’进行正反交获得F1,自交获得F2群体。F1在南京自然长日照下播种后大约110 d开始抽穗,‘南粳35’在南京自然长日照下播种后大约100 d开始抽穗,突变体ah(t)在南京自然长日照下播种后大约150 d开始抽穗,不能正常结实。通过对F2群体中的196个单株的抽穗期进行遗传分析发现:抽穗期正常植株与突变体植株分别为157和39,符合3∶1的孟德尔分离规律(χ2=2.454,χ0.052=3.84,n=196),表明突变体ah(t)的突变性状是受1对隐性核基因控制。
2.3 突变体ah(t)的突变基因定位和候选基因分析利用本实验室已有的均匀分布于12条染色体的SSR标记和InDel标记,共500对,在ah(t)和‘93-11’ 间进行多态性分析。共筛选出有多态的标记136对。对从ah(t)与‘93-11’杂交获得的F1及自交获得的F2群体(共300株)中选取抽穗期与ah(t)相近的单株(极端个体)30株,进行标记分析,将目的基因定位于第9染色体长臂SSR标记RM5526和RM24001之间,遗传距离为16.7 cM(图 2-A)。2012年12月,在海南陵水基地种植F2群体700株,从中挑选出75株极端个体用于进一步定位,将目的基因定位在InDel标记SJ-9-5和SJ-9-3之间,物理距离为547 kb(图 2-B)。2013年5月,在南京土桥,种植F2群体10 000株,从中挑选极端个体915株。在前期定位基础上,设计并合成引物,利用交换单株继续缩小区间,并将基因定位到BAC号为OSJNBa0017N10上标记W21和W24之间,物理距离为45 kb(图 2-C)。
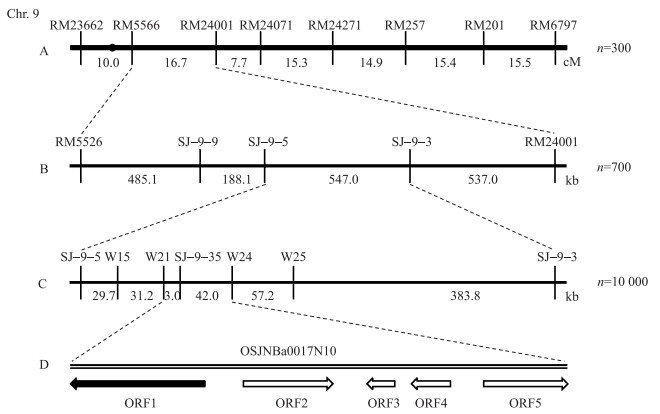 | 图 2 水稻抽穗期基因ah(t)的精细定位 Fig. 2 Fine mapping of ah(t)related heading date in rice |
利用Gramene网站和日本水稻注释计划数据库(Rice Annotation Project Database,RAP-DB,http://rice.plantbiology.msu.edu/index.shtml)对标记W21和W24之间的区段进行基因预测。通过比对目标区域基因预测信息,发现该区段内2个数据库的预测结果不同,前者包含5个开放阅读框(分别编码4个假定蛋白、1个SET domain蛋白),后者仅包含1个开放阅读框(1个SET domain蛋白)(图 2-D)。于是,我们将含SET domain的基因作为候选基因进行分析。根据NCBI提供的‘Nipponbare’该基因的序列设计引物,分别对‘南粳35’和ah(t)的基因编码区进行PCR扩增,并进行测序分析。用Powerblast软件对两者的该基因测序结果进行比对,发现ah(t)较‘南粳35’在编码区存在3个碱基的缺失(图 3-A)。最终导致第288位的组氨酸变为天冬酰胺,289位的精氨酸缺失(图 3-B)。所以,我们认为此基因即为OsSET 34。此外,我们对4个假定蛋白基因进行测序,发现序列没有差异。
 | 图 3 野生型与突变体的核酸和氨基酸序列差异 Fig. 3 Nucleotide and amino acid sequence divergence between wild type and the mutant W.野生型‘南粳35’Wild type‘Nanjing 35’;M.ah(t)突变体ah(t)mutant |
通过查阅文献发现,该基因为SDG 724 [13]的复等位基因。SDG 724 编码组蛋白H3甲基化转移酶,长日照条件下通过调节OsMADS 50和RFT1 位点的H3K36me2/3水平来调控水稻抽穗,其2个突变体分别为在此基因上有79 bp的缺失和115 bp的插入,最终导致此基因功能的变化。而本研究中OsSET 34 只有1个氨基酸发生变化,1个氨基酸缺失。为验证OsSET 34与SDG 724 作用方式是否相同,我们对不同日照长度处理下的‘南粳35’和ah(t)中的抽穗期基因SDG 724 (SET domain group protein)及其他抽穗期相关基因进行了表达分析。结果发现:与抽穗期相关的基因表达没有发生明显变化,与SDG 724 [13]中报道的相似,所以,我们推测OsSET 34的作用模式与SDG 724 相同,是通过调控相关基因的甲基化水平对抽穗期起调控作用(图 4)。
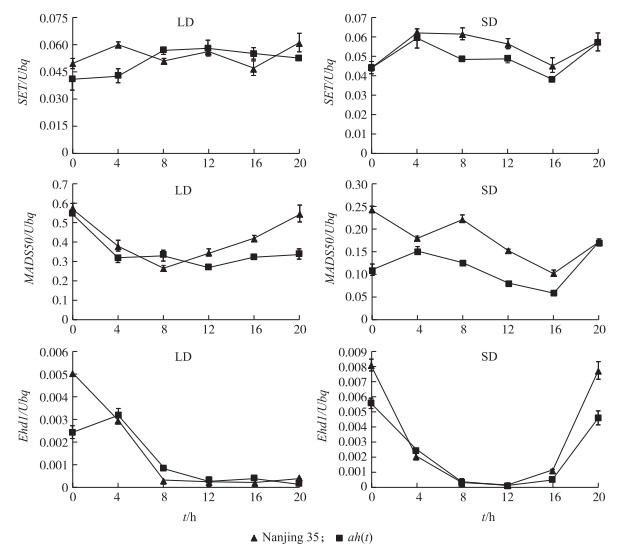 |
图 4 不同日照长度处理后抽穗期相关基因表达情况
Fig. 4 The relative expression of heading date gene in different conditions
LD:长日照,光照/黑暗时间为14 h/10 h;SD:短日照,光照/黑暗时间为10 h/14 h。 LD:Under the condition of long-day,0-14 h light,14-24 h for the night;SD:under the condition of short-day,0-10 h light,10-24 h for the night. |
拟南芥的开花调控路径已研究得较为清楚,在水稻中,越来越多的抽穗期基因也不断地被报道,且多个在水稻光周期调控中起重要作用的基因被克隆,使水稻光周期调控途径逐渐得到完善。水稻Hd 3a(Heading date 3a )基因与拟南芥开花基因FLOWERING LOCUS T(FT)同源[14],在短日照条件下促进抽穗。RFT 1是Hd3 a的同源基因,主要参与长日照条件下水稻的开花调控[15, 16]。水稻中的OsMADS 14和OsMADS15与拟南芥AP1基因同源,短日照条件下Hd3a上调OsMADS14和OsMADS15 的表达[17, 18],长日照条件下,RFT 1上调OsMADS14和OsMADS15 的表达[19]。Hd 1 与拟南芥的CO基因高度同源,在短日照条件下促进抽穗,长日照条件下抑制抽穗[20]。Ehd 1是水稻特有的基因,可在短日照条件下和长日照条件下通过调控Hd3a和RFT1 的表达而调节抽穗[8, 19]。Ehd 4则通过Ehd1来促进Hd3a和RFT1 的表达,从而促进抽穗[21]。而DTH 2可通过独立于Hd1和Ehd1的途径促进Hd3a和RFT1 的表达来促进抽穗[22]。DTH 7在长日照条件下作用于光敏色素PhyB的下游,通过抑制下游Ehd1的表达来下调成花素Hd3a/RFT1 的表达,从而延迟开花[23]。
本研究在‘南粳35’辐射诱变突变体中,鉴定了一个抽穗期延迟的突变体,通过精细定位和图位克隆的方法,克隆到控制水稻抽穗的基因OsSET 34。该基因位于第9染色体长臂OSJNBa0017N10 BAC上,能够促进水稻正常的抽穗开花,基因突变后不能行使正常功能,则导致水稻无论在长日照下还是在短日照下均延迟抽穗。因此,我们推测该基因在促进水稻开花的路径中起重要作用。
通过查阅文献发现,该基因为SDG 724 的复等位基因[13]。SDG 724 编码组蛋白H3甲基化转移酶,长日照条件下通过调节OsMADS 50和RFT1 位点的H3K36me2/3水平来调控水稻抽穗。对不同光照条件下的抽穗期相关基因的表达量分析发现,虽然OsSET 34与SDG 724 的突变情况不同,但两者的作用模式可能是相同的,均是通过调节OsMADS 50和RFT1 的甲基化水平起作用,而不是调控相关基因的表达。OsSET 34基因(SDG 724)从甲基化调控的角度阐述了光周期相关基因对抽穗期的调控作用,为研究光周期调控水稻抽穗期提供了新的研究思路。
致谢: 农业部长江中下游粳稻生物学与遗传育种重点实验室,江苏省现代作物生产协同创新中心给予支持,谨致谢意。
| [1] | Yano M,Kojima S,Takahashi Y,et al. Genetic control of flowering time in rice,a short-day plant[J]. Plant Physiology,2001,127(4):1425-1429 |
| [2] | Matsubara K,Kono I,Hori K,et al. Novel QTLs for photoperiodic flowering revealed by using reciprocal backcross inbred lines from crosses between japonica rice cultivars[J]. Theoretical and Applied Genetics,2008,117(6):935-945 |
| [3] | Levy Y Y,Dean C. The transition to flowering[J]. Plant Cell,1998,10(12):1973-1990 |
| [4] | Mouradov A,Cremer F,Coupland G. Control of flowering time interacting pathways as a basis for diversity[J]. Plant Cell,2002,14(Suppl):S111-S130 |
| [5] | Koornneef M,Alonso-Blanco C,Peeters A J M,et al. Genetic control of flowering time in Arabidopsis[J]. Annu Rev Plant Physiol Plant Mol Biol,1998,49:345-370 |
| [6] | Hayama R,Yokoi S,Tamaki S,et al. Adaptation of photoperiodic control pathways produces short-day flowering in rice[J]. Nature,2003,422(6933):719-722 |
| [7] | Izawa T,Oikawa T,Sugiyama N,et al. Phytochrome mediates the external light signal to repress FT orthologs in photoperiodic flowering of rice[J]. Genes Dev,2002,16(15):2006-2020 |
| [8] | Doi K,Izawa T,Fuse T,et al. Ehd 1 ,a B-type response regulator in rice,confers short-day promotion of flowering and controls FT-like gene expression independently of Hd 1 [J]. Genes Dev,2004,18(8):926-936 |
| [9] | Takahashi Y,Teshima K M,Yokoi S,et al. Variations in Hd 1 proteins,Hd 3 a promoters,and Ehd 1 expression levels contribute to diversity of flowering time in cultivated rice[J]. Proc Natl Acad Sci USA,2009,106(11):4555-4560 |
| [10] | Komiya R,Yokoi S,Shimamoto K,et al. A gene network for long-day flowering activates RFTl encoding a mobile flowering signal in rice[J]. Development,2009,136(20):3443-3450 |
| [11] | McCouch S R,Kochert G,Yu Z H,et al. Molecular mapping of rice chromosomes[J]. Theoretical and Applied Genetics,1988,76:815-829 |
| [12] | McCouch S R,Teytelman L,Xu Y B,et al. Development and mapping of 2240 new SSR markers for rice(Oryza sativa L.)[J]. DNA Res,2002,9(6):199-207 |
| [13] | Sun C H,Fang J,Zhao T L,et al. The histone methyltransferase SDG7 24 mediates H3K36me2/3 deposition at MADS 50 and RFT 1 and promotes flowering in rice[J]. Plant Cell,2012,24(8):3235-3247 |
| [14] | Kojima S,Takahashi Y,Kobayashi Y,et al. Hd 3 a,a rice ortholog of the Arabidopsis FT gene,promotes transition to flowing downstream of Hd 1 under short-day conditions[J]. Plant Cell Physiology,2002,43(10):1096-1105 |
| [15] | Abe M,Kobayashi Y,Yamamoto S,et al. FD,a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex[J]. Science,2005,309(5737):1052-1056 |
| [16] | Wigge P A,Kim M C,Jaeger K E,et al. Integration of spatial and temporal information during floral induction in Arabidopsis[J]. Science,2005,309(5737):1056-1059 |
| [17] | Komiya R,Ikegami A,Tamaki S,et al. Hd 3a and RFT1 are essential for flowering in rice[J]. Development,2008,135(4):767-774 |
| [18] | Furutani I,Sukegawa S,Kyozuka J. Genome-wide analysis of spatial and temporal gene expression in rice panicle development[J]. Plant J,2006,46(3):503-511 |
| [19] | Komiya R,Yokoi S,Shimamoto K. A gene network for long-day flowering activates RFT 1 encoding a mobile flowering signal in rice[J]. Development,2009,136(20):3443-3450 |
| [20] | Yano M,Katayose Y,Ashikari M,et al. Hd 1 ,a major photoperiod sensitivity quantitative trait locus in rice,is closely related to the Arabidopsis flowering time gene CONSTANS[J]. Plant Cell,2000,12(12):2473-2484 |
| [21] | Gao H,Zheng X M,Fei G,et al. Ehd 4 encodes a novel and Oryza-genus-specific regulator of photoperiodic flowering in rice[J]. PLoS Genetics,2013,9(2):e1003281 |
| [22] | Wu W,Zheng X M,Lu G,et al. Association of functional nucleotide polymorphisms at DTH 2 with the northward expansion of rice cultivation in Asia[J]. Proc Natl Acad Sci USA,2013,110(8):2775-2780 |
| [23] | Gao H,Jin M N,Zheng X M,et al. Days to heading 7,a major quantitative locus determining photoperiod sensitivity and regional adaptation in rice[J]. Proc Natl Acad Sci USA,2014,111(46):16337-16342 |
 2015, Vol. 38
2015, Vol. 38


