文章信息
- 阳淑金, 宋爱萍, 何深颖, 朱晓晨, 孙静, 高姣姣, 王银杰, 陈发棣, 蒋甲福. 2015.
- YANG Shujin, SONG Aiping, HE Shenying, ZHU Xiaochen, SUN Jing, GAO Jiaojiao, WANG Yinjie, CHEN Fadi, JIANG Jiafu. 2015.
- CaMV35S启动子在菊花中驱动GUS外源基因的表达分析
- Expression analysis of CaMV 35S promoting GUS exogenous genes in transgenic chrysanthemum
- 南京农业大学学报, 38(4): 554-559
- Journal of Nanjing Agricultural University, 38(4): 554-559.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.04.005
-
文章历史
- 收稿日期:2014-09-10
菊花(Chrysanthemum×morifolium Ramat.)原产中国,是中国十大名花之一,同时也是世界四大切花之一。菊花观赏价值高,近年来在绿化工程中的应用越来越多,因此,急需加大菊花品种选育及创新工作的进度[1, 2]。转基因技术的出现为菊花育种提供了全新的思路和途径。1975年研究者发现了菊花对农杆菌侵染的敏感性,并在菊花上开发了农杆菌介导的遗传转化技术[3, 4]。目前普遍应用的菊花遗传转化体系是农杆菌介导的菊花叶盘外植体转化法。其中,遗传转化载体中多采用CaMV 35S启动子驱动目的基因[5, 6],但CaMV 35S启动子转化菊花具有外源基因表达量低、嵌合体株率高及再生过程中基因沉默等问题[6]。因此,本试验开展了35S和2×35S启动子驱动GUS基因转化菊花‘神马’中表达效率的比较分析,以期为菊花高效转化提供理论指导。
1 材料与方法 1.1 试验材料供试材料为南京农业大学中国菊花种质资源保存中心保存的切花菊品种‘神马’(Chrysanthemum×morifolium Ramat.‘Jinba’)的无菌组织培养苗。大肠杆菌(Escherichia coli)菌株DH5α及根癌农杆菌(Agrobacterium tumefaciens)菌株EHA105由本实验室保存。pCAMBIA1301 Vector(www.cambia.org.au)、pORE R2 Vector(TAIR)由本实验室保存。pORE R2 Vector所含标记基因为新霉素磷酸转移酶基因(NPT Ⅱ )与β-葡萄糖苷酸酶基因(GUS)。pORE R2载体中驱动NPT Ⅱ 的启动子为PENTCUP2(tobacco cryptic constitutive promoter)[7]。
酶和生化试剂:DNA凝胶回收试剂盒(Axygen Agarose Gel DNA Purification Kit Ver.2.0)、 AxyPrep质粒DNA提取试剂盒和X-Gluc均购自Axygen公司;rTaq酶、Prime STARTM HS DNA Polymerase、 TaKaRa Mini Best DNA Fragment Purification Kit Ver 3.0、RNAiso Reagent、T4 DNA连接酶、Reverse Transcriptase M-MLV、RNase Inhibitor、Oligo(dT)18 Primer、dNTP mixture(10和2.5 mmol · L-1)、Recombinant DNase Ⅰ (RNase-free)、DNase Ⅰ 酶和SYBR Green Realtime PCR Master Mix-Plus(QPK-212)均购自TaKaRa公司; Sac Ⅱ酶和NheⅠ 酶购自Fermentas公司。所用引物由上海捷瑞生物公司合成,测序为Invitrogen公司。
1.2 启动子序列的克隆设计35S启动子引物(35S-F:TCCCCGCGGTTTCCCGCCTTCAGTTTAGC;35S-R:CTAGCTAGCGTCAAGAG-TCCCCCGTGTTCTC)、2×35S启动子引物(2×35S-F:TCCCCGCGGAGAGGCGGTTTGCGTATTGG;2×35S-R:CTA- GCTAGCAGAGATAGATTTGTAGAGAGAGACT),用高保真酶Prime STARTM HS DNA Polymerase,以pCAMBIA1301质粒DNA为模板,参照说明书进行PCR扩增反应,琼脂糖凝胶电泳检测产物后回收。
1.3 植物转化载体构建载体pORE R2以Sac Ⅱ和NheⅠ进行双酶切。将1.2节中获得的PCR纯化产物与上述双酶切产物用T4 DNA连接酶连接。连接产物转化大肠杆菌感受态细胞(DH5α),挑取阳性单克隆菌液双酶切验证、测序,获得重组载体pORE R2-35S:GUS和pORE R2-2×35S:GUS。将重组质粒转入菌株(EHA105),琼脂糖凝胶电泳检测。
1.4 转基因菊花再生植株的获得菊花遗传转化参照何俊平[8]的方法,抗生素(Kan,卡那霉素)筛选浓度参照刘鹏等[9]的方法,待生根苗生长60 d后,切茎段扩繁,经炼苗后,移栽到大田中。
1.5 转基因‘神马’抗性株系的RNA水平验证
以RNAiso Reagent提取叶片总RNA,用DNaseⅠ酶去除总RNA中的基因组DNA后,反转录获得 cDNA,以EFLα为内参基因(EFLα-F:TTTTGGTATCTGGTCCTGGAG;EFLα-R:CCATTCAAGCGACAGACTCA) 检测后,分别用GUS引物(GUS-F:ACGGGGAAACTCAGCAAGC;GUS-R:TGAGCGTCGCAGAACATTACAT)进行PCR反应。
将上述cDNA稀释100倍后,按照荧光定量PCR试剂盒SYBR® Green Realtime PCR Master Mix-Plus(QPK-212)的说明书进行实时荧光定量PCR。每个样品重复3次。分析数据得到各个样品的CT值,采用ΔΔCT法进行相对定量,目的基因相对含量=2-ΔΔCT。
参照Jefferson[10]的方法,选取20~35 d苗龄转基因株系上部第1片完全展开叶片,置于X-Gluc染色反应液中过夜染色,乙醇脱色至透明后,观察叶片GUS染色状况。
1.7 数据处理与分析用Excel 2003软件和SPSS 15.0统计软件对数据进行处理,采用单因素方差分析和简单线性相关分析方法对荧光定量PCR数据进行统计学分析。
2 结果与分析 2.1 35S及2×35S驱动GUS基因的植物表达载体构建从pCAMBIA1301 Vector上克隆获得35S启动子、2×35S启动子全长序列。35S启动子序列全长573 bp,2×35S启动子序列全长781 bp,分别插入到pORE R2载体的SacⅡ和NheⅠ位点。pORE R2、pORE R2-35S:GUS和 pORE R2-2×35S:GUS植物表达载体如图 1所示。
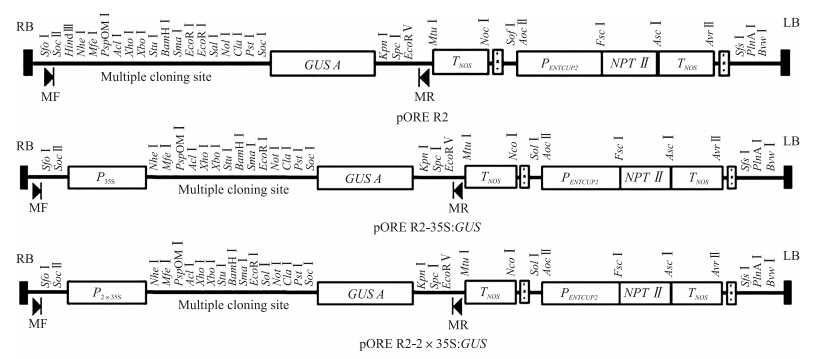 | 图 1 pORE R2载体和35S驱动GUS基因的植物表达载体结构 Fig. 1 The structure of pORE R2 vector and plant expression vector of GUS driven by 35S RB:Right T-DNA border;MF:M13 forward primer binding site;MR:M13 reverse primer binding site;LB:Left T-DNA border |
由图 2-A~C可见:经过Kan筛选,未转化的大多数叶盘长成的愈伤组织在筛选过程中逐渐发生褐化至死亡,未转化细胞生成的芽则会发生芽白化或花叶现象。转化的叶盘只有少数,这些叶盘切口处能分化出绿色抗性小芽。待其长至1.5~2.5 cm时,将抗性芽切下进行生根筛选。生根筛选时(图 2-D、E),若为阴性芽,其在生根筛选培养基中不能顺利生根,即使有根原基发生也不能正常长成根,而芽则会出现顶端枯黄、新叶失绿或白化、叶片不生长或畸形生长、整株萎蔫甚至死亡等状况。若为阳性转化芽,其在生根筛选培养基中能顺利发生根原基并能正常成根,但根有可能短粗;芽也会正常生长,但比在普通培养基上生长慢。阳性株系扩繁后炼苗移栽入大田(图 2-F)。
 | 图 2 转基因菊花植株的获得 Fig. 2 Obtain of transgenic chrysanthemum lines A.转化后叶盘Leaf disc after transformation;B.愈伤筛选Callus on selective medium;C.抗性芽Resistance shoots;D.生根筛选(根)Rooting selection(root);E.生根筛选(芽)Rooting selection(shoots);F.转基因株系(炼苗)Transgenic plants(seedling hardening) |
试验共转化了2 269个叶盘,经继代筛选,多数叶盘逐渐褐化死亡。经生根筛选后共获得216个Kan抗性植株,化学法染色检测GUS表达后发现高表达株系共16个。载体的转化情况如表 1所示,pORE R2-2×35S:GUS转化率为10.8%,且高表达株系数也较多,而pORE R2-35S:GUS转化率为5.26%。
表达载体 Expression vector | 转化叶盘数 Transformed leaf discs | 抗性株数 Resistant plants | 高表达株系 Overexpression lines | 转化率/% Transformation rate |
| pORE R2-2×35S:GUS | 1 193 | 83 | 9 | 10.8 |
| pORE R2-35S:GUS | 1 076 | 133 | 7 | 5.26 |
提取转化‘神马’阳性GUS基因高表达株系的RNA,将RNA反转录获得的cDNA分别用GUS引物及EFLα引物进行PCR反应,检测插入片段大小、cDNA质量以及定量引物设计是否具有高特异性。由图 3可以看出:条带大小与预期大小相符,条带明显,带型稳定,无引物二聚体且无非特异性扩增条带,表明反转录合成的cDNA质量达到标准,引物特异性高,可用于下一步荧光定量PCR试验。
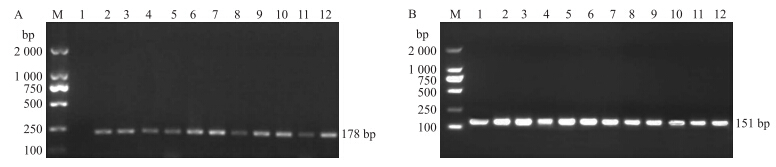 | 图 3 GUS引物(A)和EFLα引物(B)的PCR扩增 Fig. 3 PCR amplification of GUS(A)and EFLα primer(B)to analysis cDNA M.DL2000 marker;1.‘神马’(野生型)‘Jinba’(wild type);2~12.转基因株系Transgenic lines | |
由荧光定量PCR检测结果(图 4)可以看出:2×35S启动子转基因株系中表达水平最高的为E10,35S启动子转基因株系中表达水平最高的为G12,前者为后者的1.69倍。
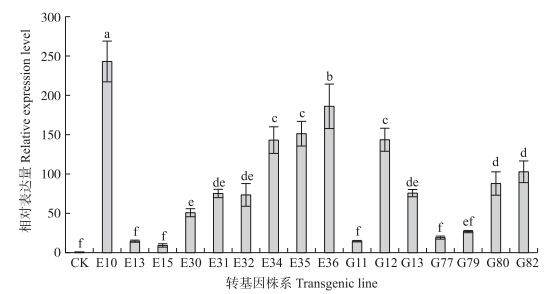 |
图 4 转基因抗性株系及野生型植株中GUS基因的相对表达量 Fig. 4 The relative expression level of GUS gene in the wild type and transgenic lines
1) CK:‘神马’(野生型);E系:pORE R2-2×35S:GUS转基因株系;G系:pORE R2-35S:GUS转基因株系
CK:‘Jinba’(wild type);Line E:Transgenic line of pORE R2-2×35S:GUS;Line G:Transgenic line of pORE R2-35S:GUS. The same as follows. 2)不同小写字母表示差异显著(P<0.05)。Different small letters indicate significant differenc at 0.05 level. | ||
对pORE R2-35S:GUS和pORE R2-2×35S:GUS转基因株系叶片分别进行GUS染色、脱色后,体视显微镜20倍物镜拍照观察叶片GUS染色状况,发现各株系染色深浅与GUS表达量基本一致,2×35S启动子驱动的转基因株系叶片GUS染色效果明显比35S启动子深(图 5)。
 | 图 5 转基因株系的GUS表达检测(标尺=0.05 mm) Fig. 5 Histochemical evaluation of GUS expression in leaf of transgenic lines(bar=0.05 mm) | |||
转基因技术为菊花育种提供了全新的思路和途径。随着转基因技术在菊花遗传转化中的广泛应用,转化遗传效率低成为菊花转基因工程中亟待解决的问题之一[6, 11]。农杆菌介导的菊花遗传转化效率为0~5%[12, 13, 14],而本研究中可以获得9.52%的转化率,这可能与使用PENTCUP 2 驱动NPT Ⅱ 有关。常用于菊花转基因的是CaMV 35S启动子,在使用CaMV 35S启动子驱动GUS时,4-MU从来没有超过492 pmol · mg-1 · min-1[15, 16, 17, 18],而其在烟草中的活跃性几乎是菊花的100倍[19],但原因至今还不清楚。
有研究表明普通的35S启动子若没有翻译增强子,GUS表达量非常低,经过染色肉眼不足以观察到[18];也有报道表明,2×35S和普通的35S启动子在转基因菊花中的GUS表达都很低,且前者不足以获得高表达的转基因株系[20]。然而,本研究中普通的35S启动子就可以使GUS表达出经染色观察到的量,这可能是由于菊花品种不同造成的,因此,菊花转基因选择合适的品种也非常关键。2×35S转化‘神马’的GUS表达量高于普通35S,说明2×35S添加了一段35S重复序列作为增强子的改造启动子,比普通的35S启动子在菊花转基因中具有更好的驱动能力。
本试验中能在菊花中高效转化的载体pORE R2和2×35S启动子可以应用于今后菊花的遗传转化,为今后的菊花基因工程奠定了基础。
| [1] | 洪波,仝征,李邱华,等. 地被菊花Fall Color体细胞胚途径再生、遗传转化及转基因植株的抗寒性检测[J]. 中国农业科学,2006,39(7):1443-1450 [Hong B,Tong Z,Li Q H,et al. Regeneration and transformation through somatic embryogenesis,and determination of cold stress tolerance in ground cover chrysanthemum cv.Fall Color[J]. Scientia Agricultura Sinica,2006,39(7):1443-1450(in Chinese with English abstract)] |
| [2] | 成丽娜,魏倩,Muhammad Imtiaz,等. 转基因育种技术在菊花性状改良中的应用进展[J]. 园艺学报,2013,40(9):1813-1825 [Cheng L N,Wei Q,Imtiaz M,et al. Advances in application of transgenicbreeding technology in the traits improvement of Chrysanthemum[J]. Acta Horticulturae Sinica,2013,40(9):1813-1825(in Chinese with English abstract)] |
| [3] | Teixeira da Silva J A. Chrysanthemum:advances in tissue culture,cryopreservation,postharvest technology,genetics and transgenic biotechnology[J]. Biotechnology Advances,2003,21:715-766 |
| [4] | van Wordragen M F,de Jong J,Huitema H B M,et al. Genetic transformation of Chrysanthemum using wild type Agrobacterium strains:strain and cultivar specificity[J]. Plant Cell Rep,1991,9:505-508 |
| [5] | 洪波,史春凤,张晓娇,等. 菊花观赏性状和农艺性状基因工程改良研究进展[J]. 中国农业科学,2009,42(4):1348-1358 [Hong B,Shi C F,Zhang X J,et al. Advances in research of ornamental and agricultural traits in chrysanthemum by gene engineering[J]. Scientia Agricultura Sinica,2009,42(4):1348-1358(in Chinese with English abstract)] |
| [6] | Teixeira da Silva J A,Shinoyama H,Aida R,et al. Chrysanthemum biotechnology:quo vadis?[J]. Critical Reviews in Plant Sciences,2013,32:21-52 |
| [7] | Coutu C,Brandle J,Brown D,et al. pORE:a modular binary vector series suited for both monocot and dicot plant transformation[J]. Transgenic Research,2007,16:771-781 |
| [8] | 何俊平. 切花菊蚜虫抗性鉴定与机理探讨及LLA转基因研究[D]. 南京:南京农业大学,2010:38-40 [He J P. Studies on aphid resistance identification,resistance related mechanisms and LLA gene transformation of cut chrysanthemum[D]. Nanjing:Nanjing Agricultural University,2010:38-40(in Chinese with English abstract)] |
| [9] | 刘鹏,陈素梅,房伟民,等. 利用卡那霉素筛选菊花转基因植株的方法研究[J]. 南京农业大学学报,2013,36(5):45-50. doi:10.7685/j.issn.1000-2030.2013.05.008 [Liu P,Chen S M,Fang W M,et al. Study on screening method of transgenic chrysanthemum using kanamycin[J]. Journal of Nanjing Agricultural University,2013,36(5):45-50(in Chinese with English abstract)] |
| [10] | Jefferson R A. Assaying chimeric genes in plant the GUS gene fusion system[J]. Plant Molecular Biology Reporter,1987,4:387-405 |
| [11] | Shinoyama H,Aida R,Ichikawa H,et al. Genetic engineering of chrysanthemum(Chrysanthemum morifolium):current progress and perspectives[J]. Plant Biotechnology,2012,29:323-337 |
| [12] | 蒋细旺,包满珠,吴家和,等. 农杆菌介导Cry1Ac基因转化菊花[J]. 园艺学报,2005,32(1):65-69 [Jiang X W,Bao M Z,Wu J H,et al. Transformation of chrysanthemum with a synthetic Cry1Ac gene mediated by Agrobacterium tumefaciens[J]. Acta Horticulturae Sinica,2005,32(1):65-69(in Chinese with English abstract)] |
| [13] | 吴月亮. 根癌农杆菌介导的PEAMT基因转化菊花的研究[D]. 北京:北京林业大学,2006:37-40 [Wu Y L. Studies on genetic transformation of Chrysanthemum morifolium with PEAMT gene mediated by Agrobacterium tumefaciens[D]. Beijing:Beijing Forestry University,2006:37-40(in Chinese with English abstract)] |
| [14] | 魏倩,李超,杨英杰,等. '神马’菊花DREB1A基因的分离及同源遗传转化[J]. 植物生理学报,2011,47(2):153-159 [Wei Q,Li C,Yang Y J,et al. Isolation and homologous genetic transformation of DREB1A in Chrysanthemum cv.'Jinba’[J]. Plant Physiology Journal,2011,47(2):153-159(in Chinese with English abstract)] |
| [15] | Toguri T,Ogawa T,Kakitan M,et al. Agrobacterium-mediated transformation of chrysanthemum(Dendranthema grandiflora)plants with a disease resistance gene(pac1)[J]. Plant Biotechnology,2003,20(2):121-127 |
| [16] | Sherman J M,Moyer J W,Daub M E. A regeneration and agrobacterium-mediated transformation system for genetically diverse Chrysanthemum cultivars[J]. Journal of the American Society for Horticulture Science,1998,123:189-194 |
| [17] | Takatsu Y,Hayashi M,Sakuma F. Transgene inactivation in agrobacterium-mediated chrysanthemum(Dendranthema grandiflorum Ramat.Kitamura)transformants[J]. Plant Biotechnol,2000,17:241-245 |
| [18] | Aida R,Narumi T,Ohtsubo N,et al. Improved translation efficiency in chrysanthemum and torenia with a translational enhancer derived from the tobacco alcohol dehydrogenase gene[J]. Plant Biotechnol,2008,25:69-75 |
| [19] | Outchkourov N S,Peters J,de Jong J,et al. The promoter terminator of chrysanthemum rbcS1 directs very high expression levels in plants[J]. Planta,2003,216:1003-1012 |
| [20] | Annadana S,Mlynárová L,Udayakumar M,et al. The potato Lhca3.St.1 promoter confers high and stable transgene expression in chrysanthemum,in contrast to CaMV-based promoters[J]. Molecular Breeding,2001,8:335-344 |




