文章信息
- 李岩, 徐志胜, 谭国飞, 贾晓玲, 王枫, 熊爱生. 2015.
- LI Yan, XU Zhisheng, TAN Guofei, JIA Xiaoling, WANG Feng, XIONG Aisheng. 2015.
- 芹菜热激转录因子基因AgHSFB2的克隆及不同温度处理下的表达响应
- Cloning and expressional response analysis of AgHSFB2 under different temperature treatments in celery
- 南京农业大学学报, 38(3): 360-368
- Journal of Nanjing Agricultural University, 38(3): 360-368.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.03.002
-
文章历史
- 收稿日期:2014-08-15
芹菜(Apium graveolens)是伞形科芹属1、2年生的草本植物,具有典型的耐寒不耐热的生长特性,较适宜的生长温度在15~20℃左右[1]。研究发现,低于4℃或高于30℃,芹菜种子不易发芽,尤其是环境温度高于25℃时,芹菜会发生早抽苔和外叶糠心现象,严重降低芹菜品质和产量,影响商品性[2]。‘六合黄心芹’是抗性、适应性好的南京市六合区地方品种;‘津南实芹’是抽薹期晚、冬性强的天津南部地方品种;‘文图拉’引自美国,具有冬性强、耐抽薹、抗病性好的特点[3]。3个芹菜品种都具有产量高、品质好的特点,适于保护地、春秋露地栽培及周年生产。其播种期一般在8—10月,但在我国南方频繁的高温气候下,不耐热的芹菜完成周年生产面临巨大挑战
热激转录因子(heat shock transcription factor,HSF)是植物在高温胁迫下起调节作用的主要转录因子,能与热激元件的反向重复序列区域结合,诱导热激蛋白(heat shock proteins,HSPs)发生转录。HSPs作为分子伴侣与其他蛋白质协同作用,缓解高温对植物的伤害[4]。植物热激转录因子HSF家族基因的种类和数目很多,番茄中至少含有18个,拟南芥至少含21个,水稻中至少有25个,大豆中至少有34个HSF基因[5]。番茄、拟南芥、水稻、大豆的HSF家族已被研究[6],但芹菜HSF家族却未被研究。植物的HSF根据结构差异可分为A、B、C三类,研究多集中在A类和B类上,番茄中的HSFA 1、HSFA2、HSFB1 是研究较为深入的HSF基因[6]。拟南芥中含15个A类,5个B类,1个C类HSF基因[5]。HSF家族成员功能虽不尽相同,但结构基本相似,包括DNA结合域DBD、寡聚化结构域HR-A/B、核定位信号NLS、核输出信号NES和C端的转录激活结构域AHA[7]。研究发现,A类HSF在热胁迫下起主要作用,B类HSF对A类有协同作用[7]。蛋白质印迹分析证明拟南芥HSFA2 是严格受热诱导表达的基因,在根、茎、叶、花等组织中表达[8]。43℃高温下,AtHSFA6a过表达可提高拟南芥的耐热性[9]。百合LlHSF 1 在42℃下不同时间段的响应证实LlHSF1 与百合耐热性相关[10]。Zhu等[11]发现大豆GmHSFA1具有组成型表达模式,在大豆不同发育期、不同组织中都稳定表达,高温下GmHSFA1 表达量增加,外源激素处理后表达量下降;常温下,35S-GmHSFA1的转基因植株中GmHSFA1 的表达量高于对照,说明转基因植株更耐高温。目前对植物耐热机制的研究还不够深入,没有充分挖掘高效的耐热基因,从根本上提高植物耐热性
本文以‘六合黄心芹’‘津南实芹’‘文图拉’3个品种为材料,克隆了AgHSFB2 基因并进行了相关的生物信息学分析,对3个芹菜品种植株分别进行4、38和42℃处理,利用实时荧光定量PCR检测该基因的表达响应情况,为进一步了解芹菜耐热机制,培育耐热芹菜新品种提供理论依据 1 材料与方法 1.1 植物材料及其处理
试验所用材料为‘六合黄心芹’‘津南实芹’和‘文图拉’3个芹菜品种的3月龄植株。种子经催芽后,于2014年2月初种植于南京农业大学作物遗传与种质创新国家重点实验室的人工气候室。人工气候室的温度设定为25℃/15℃(昼/夜),光照时间16 h/8 h(光/暗),光照度为3 000 lx
2014年5月初,取3月龄的植株分别进行4℃低温、38℃高温、42℃高温胁迫处理。每个处理6株,处理时间分别为1、2、4、8和24 h。选取植株顶部第3~4片嫩叶,每个处理取3个样品。以同期正常生长于气候室且未经处理的芹菜植株作为对照。同时采取对照芹菜植株的根、茎、叶。所有样品取样后立即放入液氮中,用于提取RNA 1.2 RNA的提取与反转录
RNA的提取采用试剂盒RNA Simple Total RNA Kit(北京Tiangen公司)。DEPC水溶解RNA后,用分光光度计测定D值,通过D260/D280的比值可以判断RNA的质量,通过琼脂糖凝胶电泳检测RNA的完整性。将RNA反转录成cDNA,反转录依据Prime Script RT reagent Kit(大连TaKaRa)说明书进行 1.3 芹菜AgHSFB2 基因的克隆及序列测定
根据本实验室测定的芹菜转录组数据,登录号为SRA109935[12],以拟南芥(Arabidopsis thaliana) HSFB2的氨基酸序列(登录号:NP_201008.2)为检索起点,检索多个HSFB2类的氨基酸序列,拼接出芹菜AgHSFB2 基因序列,设计1对引物,正向为5′-ATGGCTCCGCCTGAGCGCA-3′,反向为5′-TCACACGAGACTCATTCAC-3′。分别以3个芹菜品种的cDNA为模板进行PCR扩增。PCR反应程序为:94℃ 5 min;94℃ 30 s,54℃ 30 s,72℃ 1 min,30个循环;72℃ 10 min。产物经12 g · L-1琼脂糖凝胶电泳后用DNA胶回收试剂盒(杭州爱思进生物技术有限公司)回收,回收产物连接pMD18-T载体(大连TaKaRa)后转化大肠杆菌DH5α(由本实验室保存)筛选,挑取质粒摇菌,菌液经PCR并进行琼脂糖凝胶电泳鉴定后,送到南京金斯瑞生物科技有限公司测序。此过程中涉及的Ex-Taq PCR聚合酶、DL marker 2000等均购自大连TaKaRa公司 1.4 序列分析
其他植物的HSFB2序列来自NCBI数据库,使用BLASTn和BLASTp进行序列比对。利用DNAMAN 6.0分析,比对后用MEGA 5按照邻接法(Neighbor-joining method)构建系统进化树,生成报告图形[13, 14, 15]。使用http://www.expasy.org网站相关软件完成氨基酸序列理化性质分析[16]。使用Swiss-Model建立蛋白质三维空间结构模型[17]。拟南芥HSF家族相应序列来自TAIR(http://www.arabidopsis.org/) 1.5 实时荧光定量PCR
实时定量PCR采用SYBR Premix Ex-Taq(大连TaKaRa公司)并在ABI 7300 Real-time PCR System和7300 System Software进行反应和分析。芹菜actin基因作为内参基因,与目标基因一起扩增。芹菜actin基因引物正向为:5′-CTTCCTGCCATATATGATTGG-3′,反向为:5′-GCCAGCACCTCGATCTTCATG-3′。芹菜AgHSFB2 荧光定量PCR引物正向为5′-GGTTCCAGCAATTATTCCAACAC-3′,反向为5′-TCATCCACCTCATTCCGTTCA-3′。采用20 μL反应体系,反应程序为:95℃ 30 s;95℃ 10 s,58℃ 30 s,40个循环;然后进行熔解曲线分析,起始温度为65℃。对试验结果进行相对表达量分析,算法采用内参基因的ΔCT法[18],表达差异值即2-ΔΔCT,其中ΔCT=CT,目标基因-CT,actin 2 结果与分析 2.1 芹菜AgHSFB2 的克隆及序列分析
以3个芹菜品种的cDNA为模板,PCR扩增后得到长度分别都为900 bp左右的片段。序列测定与分析表明,来源于‘六合黄心芹’‘津南实芹’‘文图拉’的AgHSFB2 均含有1个918 bp的开放阅读框,编码305个氨基酸。DNAMAN 6.0比对显示3个芹菜品种中AgHSFB2 的核苷酸序列相似度高达99.24%,氨基酸序列相似度高达98.69%,有很高的同源性。 3个芹菜品种AgHSFB2的核苷酸差异如下:第74位C/T/T,96位G/G/A,107和108位A/A/T,135位G/G/C,160位G/A/A,208位A/C/A,373位C/A/C,382位C/C/A,500位A/T/A,505位G/A/A,589位G/G/T,685位T/C/T,718位G/C/G(‘六合黄心芹’/‘津南实芹’/‘文图拉’)。对应的编码氨基酸差异如下: 第25位S/L/L,33位D/D/N,37位I/I/F,46位D/D/H,54位S/N/N,70位N/T/N,125位P/H/P,128位P/P/H,169位G/D/D,197位S/S/I,229位L/P/L,240位P/P/R(‘六合黄心芹’/‘津南实芹’/‘文图拉’)
NCBI-CDS分析表明,AgHSFB2在21~114氨基酸位点间有1个保守的HSF-DNA结合域,属于HSF家族(图 1-A)。将芹菜AgHSFB2 基因及编码的氨基酸序列提交到Heatster网站(http://www.cibiv.at/services/hsf/)进行分析[19]。在3个芹菜品种中AgHSFB2都含有相同的结构域:21~114氨基酸位点间存在1个DNA结合域DBD,178~214氨基酸位点间存在寡聚化结构域HR-A/B,254~260氨基酸位点间存在1个调节域(regulatory domain,RD),263~267氨基酸位点间存在核定位信号NLS(图 1-B)。根据NCBI的BLAST结果和Heatster网站注释,‘六合黄心芹’中该基因与HSFB 2、HSFB1、HSFB4、HSFB3和HSFB5 比对的E-value值分别为2.5×10-125、3.2×10-89、9.1×10-72、5.5×10-58和3.6×10-38;‘津南实芹’该基因与HSFB 2、HSFB1、HSFB4、HSFB3和HSFB5 比对的E-value值分别为1.5×10124、7×1089、1.7×1071、7.1×1060和2.4×1037;‘文图拉’该基因与HSFB 2、HSFB1、HSFB4、HSFB3和HSFB5 比对的E-value值分别为6.1×10125、2.5×1089、5×1071、2.1×1057和6.4×1036。E-value值越小,匹配度越高[20]。该基因与HSFB2 有最高匹配
 | 图 1 AgHSFB2序列CDS预测(A)及AgHSFB2的ORF和编码蛋白质的结构域预测(B) Fig. 1 The CDS prediction(A)and the ORF and domains prediction(B)of AgHSFB2 *表示终止密码;下加横线的氨基酸序列代表AgHSFB2的结构域;横线旁的数字表示氨基酸的起止位点;DBD表示DNA结合域;HR-A/B表示寡聚化结构;RD表示调节域;NLS表示核定位信号。* represents the stop codon;the domains of AgHSFB2 are underlined;the numbers beside the lines represent the locations of amino acids;DBD represents the DNA-binding domain;HR-A/B represents the oligomerization domain;RD represents the regulatory domain;NLS represents the nuclear localization signals. |
BLAST同源性检索发现芹菜AgHSFB2与16种植物的HSF蛋白相似性较高。这些植物为可可树(Theobroma cacao,XP_007038632.1)、鹰嘴豆(Cicer arietinum,XP_004511596.1)、甜橙(Citrus sinensis,XP_006477425.1)、黄瓜(Cucumis sativus,XP_004143930.1)、大豆(Glycine max,AFQ93677.1)、苹果(Malus domestica,ADL36731.1)、川桑(Morus notabilis,EXC15958.1)、短花野生稻(Oryza brachyantha,XP_006659656.1)、毛果杨(Populus trichocarpa,XP_002298053.1)、梅(Prunus mume,XP_008234272.1)、蓖麻(Ricinus communis,XP_002510816.1)、番茄(Solanum lycopersicum,XP_004234371.1)、马铃薯(Solanum tuberosum,XP_006353396.1)、华东葡萄(Vitis pseudoreticulata,ADC94861.1)、酿酒葡萄(Vitis vinifera,XP_002283139.1)和拟南芥(Arabidopsis thaliana,NP_201008.2)。其中芹菜AgHSFB2与可可树中HSFB2a的相似性高达53%,与大豆中HSFB2b的相似性为48%。DBD是HSF家族最保守的结构域,对上述植物HSF蛋白的DBD区域进行比对,发现芹菜与上述植物中HSFB2蛋白的相似性高于50%(图 2)。用Protparam分析AgHSFB2蛋白的氨基酸组分及理化性质,结果表明AgHSFB2蛋白的理论相对分子质量为34 048,理论等电点为5.09,酸性氨基酸约占45%,碱性氨基酸约占13%,总平均疏水指数(grand average of hydropathicity,GRAVY)为-0.769,AgHSFB2有一定的亲水性[21]
 | 图 2 芹菜AgHSFB2与其他物种HSFB2类蛋白的DBD序列的比对分析 Fig. 2 Alignment analysis of DBD in sequences of HSFB2 from celery and other plants |
预测蛋白的相关结构需要多种分析软件[22, 23, 24, 25, 26]。采用SignalP 4.0对芹菜AgHSFB2序列进行信号肽预测,结果显示AgHSFB2蛋白序列不存在信号肽也无分泌蛋白。用TMHMM软件对芹菜AgHSFB2序列进行跨膜区预测,结果显示不存在跨膜区。用PredictProtein预测AgHSFB2的亚细胞定位,表明AgHSFB2定位于细胞核。采用Paircoil 2对AgHSFB2氨基酸序列中的卷曲螺旋(coiled-coil)区域进行分析,p值小于0.025的位点所在区域即为卷曲螺旋区域。而结果显示在第170~207个氨基酸残基区域的p值符合,因此存在1个卷曲螺旋区域(图 3)
 | 图 3 Paircoil 2分析芹菜AgHSFB2的卷曲螺旋区域 Fig. 3 Prediction for coiled-coil domain of AgHSFB2 in celery by Paircoil 2 |
拟南芥HSF序列来自TAIR(http://www.arabidopsis.org/)。将芹菜的AgHSFB2与拟南芥HSF家族成员进行多序列比对分析,构建进化树。由图 4可见:‘六合黄心芹’与‘津南实芹’的进化关系更近,二者都是中国地方品种。3个芹菜品种的AgHSFB2高度同源且位于同一进化分支,芹菜AgHSFB2与拟南芥HSFB2a、HSFB2b的进化距离最近,芹菜AgHSFB2属于HSFB2亚族
 | 图 4 芹菜与拟南芥中HSF蛋白序列的系统进化树 Fig. 4 Phylogenetic tree analysis of HSF protein sequences from celery and Arabidopsis thaliana |
对芹菜与上述16种植物中HSFB2类型蛋白比对后构建进化树。结果(图 5)显示:芹菜与茄科的番茄、马铃薯在进化亲缘关系上最近。同属蔷薇科的苹果和梅在进化树的同一分支,二者与桑科的川桑,豆科的大豆进化距离较近,其次与梧桐科的可可树、大戟科的蓖麻、十字花科的拟南芥进化距离较近。同属葡萄科的酿酒葡萄、华东葡萄在进化树的同一分支,一般同科、属的植物进化距离较近,但同属豆科的鹰豆、大豆却不在同一进化分支
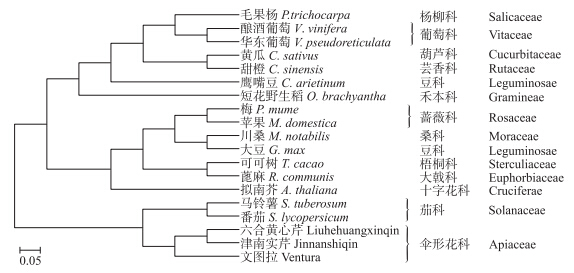 | 图 5 多种植物中类似HSFB2蛋白序列的系统进化树 Fig. 5 Phylogenetic tree of the HSFB2-like proteins from many plants |
利用SWISS-MODEL对3个芹菜品种AgHSFB2蛋白的三级结构进行分析,发现AgHSFB2的3级结构模型中包含3个α螺旋和4个β折叠。‘六合黄心芹’‘津南实芹’和‘文图拉’中AgHSFB2的3级模型均以人(Homo sapiens)的热激转录因子蛋白1(HSF1)的A链(PDB ID为2LDU)为模板构建。该模板分别与‘六合黄心芹’‘津南实芹’和‘文图拉’中AgHSFB2蛋白的108个氨基酸残基进行比对,一致性均大于95%,较为可信(图 6)
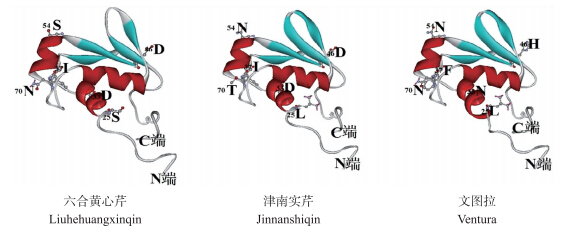 | 图 6 3个芹菜品种AgHSFB2蛋白的3级结构模型预测 Fig. 6 Tertiary structure prediction of AgHSFB2 from three celery cultivars S:丝氨酸Ser;D:天冬氨酸Asp;N:天冬酰胺Asn;I:异亮氨酸Ile;L:亮氨酸Leu;T:苏氨酸Thr;H:组氨酸His;F:苯丙氨酸Phe。氨基酸左边数字代表氨基酸位置。The left number of the amino acid represents the position of the amino acid. |
由图 7-A可见:‘六合黄心芹’AgHSFB2 在根、茎的表达量基本相同,但叶中表达量明显高于根、茎,叶中的表达量均为根和茎的12倍。‘津南实芹’的根、叶中的表达量基本相同,均为茎表达量的5.0倍。‘文图拉’叶中的表达量最高,茎中表达量最低,叶中表达量分别是根和茎的2.5和6.0倍
 | 图 7 AgHSFB2 基因在芹菜不同组织(A)及不同温度(B~D)下的表达量 Fig. 7 Expression level of AgHSFB2 gene in different tissues(A)and under different temperature treatments(B-D)from the three celery cultivars |
4℃低温下,‘六合黄心芹’与‘文图拉’AgHSFB2 表达量均在4 h时达到最大值,分别为对照的14.4和23.6倍。‘津南实芹’AgHSFB2 表达量在2 h处达到最大值,为对照的9.4倍(图 7-B)
38℃处理下,‘六合黄心芹’AgHSFB2 的表达大致呈下调趋势,处理1 h后AgHSFB2 表达量为对照的0.04倍,在4 h处降到最低;‘津南实芹’在1 h时表达量明显下调,但在2 h后迅速上调并达到最大值,在1和2 h处的表达量分别为对照的0.03和23.0倍;‘文图拉’的表达量变化趋势与‘津南实芹’相似,在1和2 h处表达量分别为对照的0.3和40.0倍(图 7-C)
42℃高温下,3个芹菜品种均在1 h处达到最大值。‘六合黄心芹’‘津南实芹’‘文图拉’在1 h的表达量分别为对照的3.1、19.2和15.2倍(图 7-D) 3 讨论
热激转录因子HSF可以调节HSP的表达,位于信号转导途径的下游可以传递热胁迫信号,因此在植物耐热性研究中有重要价值。植物中,HSF不仅参与热激响应调控,还参与其他多种逆境响应[27, 28]。番茄SlHSFB1 正常条件下低水平表达,受热后大量表达[29]。辣椒CaHSFA 2 、玉米ZmHSF、拟南芥AtHSFA 1 类基因都参与多种逆境胁迫的调控[30, 31, 32, 33]。HSF可与其他转录因子协同作用:热胁迫下,番茄SlHSFA2 的表达不仅依赖SlHSFA1 ,还与DREB、MBF等转录因子家族共同参与调控[8]。拟南芥HSFB2亚族可能具有与热激无关的功能,突变拟南芥AtHSFB 2 a的基因位点会影响其营养生长和生殖生长[32]
HSFA亚族的基因已被广泛研究,B亚族研究较少,芹菜转录组测序为克隆HSFB类家族的基因提供了条件。本文分别从3个芹菜品种克隆了1个基因,CDS分析发现该基因属于HSF家族。BLAST比对及进化树分析显示该基因属于B亚族。3个芹菜品种中AgHSFB2 基因和编码的氨基酸序列高度同源,都含有1个编码305个氨基酸的开放阅读框。在植物中,按所含结构域差异可将HSF家族分为A、B、C三类,B亚族基因C-端由于不含有被酸性、芳香族、疏水性氨基酸残基包围的一段AHA序列,无A亚族的转录激活功能[33]。AgHSFB2蛋白包含DBD、HR-A/B、RD和NLS结构域,其中调节域RD不属于HSF家族的基本结构域,该结构域可能有调节基因转录表达的作用。AgHSFB2属于B亚族,不含AHA序列,可能不具备转录活性和激活功能,AgHSFB 2 基因的超表达可能无法直接提高植株的耐热性,但可通过与HSFA类基因的协同作用,激活下游基因,提高植株的耐热性
AgHSFB2的3级结构包含3个α螺旋和4个β折叠,与前人研究结果一致[27, 28]。HSFB亚族HR-A/B 结构域中A区域和B区域之间只有7个氨基酸残基,但A与C类亚族除了这7个氨基酸外分别还有21个、7个氨基酸残基的插入序列。A类亚族连接DBD与HR-A/B的序列含9~39个氨基酸残基,B类则含50~78个,C类含4~49个氨基酸残基[33]。AgHSFB2中DBD与HR-A/B之间的序列有63个氨基酸残基,符合B亚族特征。鹰嘴豆HSFB2 的亚细胞定位于细胞核中[34],芹菜AgHSFB2 也定位于细胞核。芹菜AgHSFB2与拟南芥HSF家族成员构建的进化树显示芹菜AgHSFB2属于B2亚族。最保守的DNA结合域可以作为判断物种间亲缘关系远近的依据[32]。多种植物HSFB2蛋白的进化分析显示芹菜与茄科的番茄和马铃薯在进化亲缘关系上最近。鹰嘴豆、大豆同属豆科,HSFB2蛋白的进化关系却不近,可能与起源地气候条件及后期栽培、驯化过程的差异有关。AgHSFB2 基因的表达存在组织、品种的差异,‘六合黄心芹’叶中的表达量明显高于根、茎;‘津南实芹’根、叶的表达量明显高于茎,‘文图拉’的表达由大到小为叶、根、茎。AgHSFB2 基因的表达在不同温度处理下有品种的差异。不同温度处理下,芹菜AgHSFB2 基因一般在4 h之内出现明显上调:4℃和38℃处理下‘文图拉’均在2 h有明显上调表达;42℃处理下‘津南实芹’和‘文图拉’均在1 h有明显上调表达。AgHSFB2 有利于提高芹菜耐热性,但其在热信号响应机制中所起的作用尚需进一步分析。
| [1] | Lobell D B, Gourdji S M. The influence of climate change on global crop productivity[J]. Plant Physiol, 2012, 160:1686-1697 |
| [2] | 王武台, 古瑜, 韩启厚, 等. 芹菜种质资源亲缘关系的ISSR分析[J]. 中国蔬菜, 2011(8):22-27[Wang W T, Gu Y, Han Q H, et al. ISSR analysis of celery germplasm genetic relationships[J]. China Vegetables, 2011(8):22-27(in Chinese with English abstract)] |
| [3] | 田畅, 王枫, 蒋倩, 等. 芹菜核酸内切酶CELI基因的克隆及其表达分析[J]. 南京农业大学学报, 2014, 37(4):39-45. doi:10.7685/j.issn.1000-2030.2014.04.006[Tian C, Wang F, Jiang Q, et al. Cloning and expression pattern analysis of CELI gene in celery[J]. Journal of Nanjing Agricultural University, 2014, 37(4):39-45(in Chinese with English abstract)] |
| [4] | Akerfelt M, Morimoto R I, Sistonen L. Heat shock factors:integrators of cell stress, development and lifespan[J]. Nat Rev Mol Cell Biol, 2010, 11:545-555 |
| [5] | Nover L, Scharf K D, Gagliardi D, et al. The HSF world:classification and properties of plant heat stress transcription factors[J]. Cell Stress Chaperones, 1996, 1:215-223 |
| [6] | Mishra S K, Tripp J, Winkelhaus S, et al. In the complex family of heat stress transcription factors, HsfA1 has a unique role as master regulator of thermotolerance in tomato[J]. Genes Dev, 2002, 16:1555-1567 |
| [7] | Chan-Schaminet K Y, Baniwal S K, Bublak D, et al. Specific interaction between tomato HsfA1 and HsfA2 creates hetero-oligomeric superactivator complexes for synergistic activation of heat stress gene expression[J]. J Biol Chem, 2009, 284:20848-20857 |
| [8] | Port M, Tripp J, Zielinski D, et al. Role of Hsp17.4-CⅡ as coregulator and cytoplasmic retention factor of tomato heat stress transcription factor HsfA2[J]. Plant Physiol, 2004, 135:1457-1470 |
| [9] | Kotak S, Port M, Ganguli A, et al. Characterization of C-terminal domains of Arabidopsis heat stress transcription factors(HSFs)and identification of a new signature combination of plant class A HSFs with AHA and NES motifs essential for activator function and intracellular localization[J]. Plant J, 2004, 39:98-112 |
| [10] | 易瑾, 罗弦, 曹兴, 等. 百合热激转录因子基因LlHSF1的克隆与表达[J]. 园艺学报, 2012, 39(11):2199-2205[Yi J, Luo X, Cao X, et al. Cloning and expression analysis of LlHSF1 from Lilium longiforum[J]. Acta Horticulturae Sinica, 2012, 39(11):2199-2205(in Chinese with English abstract)] |
| [11] | Zhu B, Ye C, Lü H, et al. Identification and characterization of a novel heat shock transcription factor gene, GmHsfA1, in soybeans(Glycine max)[J]. J Plant Res, 2006, 119:247-256 |
| [12] | Li M Y, Wang F, Jiang Q. Identification of SSRs and differentially expressed genes in two cultivars of celery(Apium graveolens L.)by deep transcriptome sequencing[J].Horticulture Research, 2014, 1(10):1-9 |
| [13] | Altschul S F, Lipman D J. Protein database searches for multiple alignments[J]. Proc Natl Acad Sci USA, 1990, 87:5509-5513 |
| [14] | Tamura K, Peterson D, Peterson N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony methods[J]. Mol Biol Evol, 2001, 28:2731-2739 |
| [15] | Kent W J. BLAT:the BLAST-like alignment tool[J]. Genome Res, 2002, 12:656-664 |
| [16] | Gasteiger E, Gattiker A, Hoogland C, et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research, 2003, 31:3784-3788 |
| [17] | Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22:195-201 |
| [18] | Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucl Acids Res, 2001, 29(9):45 |
| [19] | Scharf K D, Berberich T, Ebersberger I, et al. The plant heat stress transcription factor(HSF)family:structure, function and evolution[J]. Biochim Biophys Acta, 2012, 1819:104-119 |
| [20] | Altschul S F, Koonin E V. Iterated profile searches with PSI-BLAST:a tool for discovery in protein databases[J]. Trends Biochem Sci, 1998, 23(11):444-447 |
| [21] | Banti V, Mafessoni F, Loreti E, et al. The heat-inducible transcription factor HSFA2 enhances anoxia tolerance in Arabidopsis[J]. Plant Physiol, 2010, 152:1471-1483 |
| [22] | Käll L, Krogh A, Sonnhammer E L L. A combined transmembrane topology and signal peptide prediction method[J]. J Mol Biol, 2004, 338(5):1027-1036 |
| [23] | Petersen T N, Brunak S, Nielsen H, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8:785-786 |
| [24] | Krogh A, Larsson B, Von H G, et al. Predicting transmembrane protein topology with a hidden markov model:application to complete genomes[J]. J Mol Biol, 2001, 305:567-580 |
| [25] | Goldberg T, Hamp T, Rost B. LocTree2 predicts localization for all domains of life[J]. Bioinformatics, 2012, 28:458-465 |
| [26] | Mcdonnell A V, Jiang T, Keating A E, et al. Paircoil2:improved prediction of coiled coils from sequence[J]. Bioinformatics, 2006, 22:356-358 |
| [27] | Busch W, Wunderlich M, Schoffl F. Identification of novel heat shock factor-dependent genes and biochemical pathways in Arabidopsis thaliana[J]. Plant J, 2005, 41:1-14 |
| [28] | Guo J K, Wu J, Ji Q, et al. Genome-wide analysis of heat shock transcription factor families in rice and Arodopsis[J]. Genet Genomics, 2006, 354:105-118 |
| [29] | Bharti K, Bharti S, Kumar P, et al. Tomato heat stress transcription factor HsfB1 represents a novel type of general transcription coactivator with a histone-like motif interacting with the plant CREB binding protein ortholog HAC1[J]. Plant Cell, 2004, 16:1521-1535 |
| [30] | Guo M, Yin Y X, Ji J J, et al. Cloning and expression analysis of heat-shock transcription factor gene CaHSFA2 from pepper(Capsicum annuum L.)[J]. Genet Mol Res, 2014, 13(1):1865-1875 |
| [31] | Baniwal S K, Bharti K, Chan K Y, et al. Heat stress response in plants:a complex game with chaperones and more than twenty heat stress transcription factors[J]. J Biosci, 2004, 29:471-487 |
| [32] | Qin F, Kakimoto M, Sakuma Y, et al. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses in Zea mays L.[J]. Plant J, 2007, 50(1):54-69 |
| [33] | Giorno F, Guerriero G, Baric S, et al. Heat shock transcriptional factors in Malus domestica:identification, classification and expression analysis[J]. BMC Genomics, 2012, 13:639 |
| [34] | Alexandre A, Oliveira S. Most heat-tolerant rhizobia show high induction of major chaperone genes upon stress[J]. FEMS Microbiol Ecol, 2011, 75:28-36 |




