文章信息
- 黄菲艺, 唐君, 侯喜林, 马静, 王枫. 2015.
- HUANG Feiyi, TANG Jun, HOU Xilin, MA Jing, WANG Feng. 2015.
- 不结球白菜响应ABA和低温基因WRKY18的克隆及表达分析
- Cloning and expression analysis of WRKY18 gene in non-heading Chinese cabbage under ABA and low temperature treatments
- 南京农业大学学报, 38(2): 189-196
- Journal of Nanjing Agricultural University, 38(2): 189-196.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.02.003
-
文章历史
- 收稿日期:2014-06-19
2. 江苏省中国科学院植物研究所, 江苏 南京 210014
2. Institute of Botany, Jiangsu Province and Chinese Academy of Sciences, Nanjing 210014, China
逆境转录因子是一类重要的参与植物逆境胁迫反应的蛋白家族,依据其结构特征被分为AP2[1]、MYB[2]、NAC[3]、bZIP[4]和WRKY[5]等。其中WRKY类转录因子是最近植物逆境调控分子机制研究中比较活跃的一类,它们广泛参与植物的形态建成、生殖发育、衰老、生物节律调控、生物胁迫和非生物胁迫响应等过程[6]。WRKY转录因子家族成员已在拟南芥[7]、油菜[8]、水稻[9]、湖北海棠[10]等植物中被分离出来,研究主要集中在WRKY参与植物的生物胁迫反应[11, 12]和激素信号转导[13, 14]等方面。
不结球白菜(Brassica campestris ssp.chinensis Makino)是我国长江流域主要绿叶蔬菜之一,并已在全国各地普遍种植,在蔬菜周年生产中占据越来越重要的地位[15]。目前不结球白菜WRKY基因的克隆仅有王彦华等[16]和Wang等[17]的报道。WRKY 18 是WRKY家族中Group Ⅱa中三成员(WRKY 18、WRKY40、WRKY60 )之一[5],具有多种生物学功能,参与生物胁迫、非生物胁迫及ABA信号转导的功能,而目前不结球白菜中尚未有报道。本研究采用电子克隆的方法,获得了不结球白菜WRKY 18 重叠群,并以此为模板从不结球白菜逆境cDNA文库中分离到不结球白菜WRKY 18 基因全长。通过序列分析和同源建模,解析了WRKY 18 基因结构和蛋白质特征,同时利用qRT-PCR技术检测了BcWRKY 18 在低温和ABA处理下的表达变化,旨在为开展不结球白菜抗逆分子机制及WRKY基因对低温和ABA协同响应分子机制的研究提供重要依据。
1 材料与方法 1.1 材料及胁迫处理供试不结球白菜品种为‘苏州青’自交系,由南京农业大学园艺学院不结球白菜课题组提供。种子经双蒸水浸泡4 h后在25 ℃培养过夜,将露白的种子播种于营养土穴盘中,光照培养箱昼夜温度为25 ℃/15 ℃、光周期为12 h/12 h。出苗后浇去离子水,7 d后浇1/2 MS盐溶液(pH 5.8)。待幼苗长至4叶1心时,选择生长良好、发育状态一致的植株进行处理。将幼苗分别置于4 ℃低温光照培养箱和100 μmol · L-1的ABA溶液喷施处理,其他生长条件均一致。分别于1、2、4、6、8、12、16、24、36、48、60、72、84和96 h取样,用液氮速冻后保存于-80 ℃冰箱中备用。
1.2 总RNA提取及逆境cDNA文库构建采用RNA Simple Total RNA Kit总RNA提取试剂盒(TIANGEN,中国)提取试验材料的总RNA,再用15 g · L-1的琼脂糖凝胶电泳检测总RNA的完整性。将不同处理的RNA等量混合,用PrimeScript RT reagent Kit(TaKaRa)将混合的RNA反转录成cDNA构建逆境文库。合成体系的总体积为20 μL,反应条件及过程为:混合RNA和50 μmol · L-1 Oligo(dT)18混液于70 ℃水浴10 min,然后加入M-MLV反转录酶(200 U)、dNTP(10 mmol · L-1)、RNase抑制剂(40 U)(TaKaRa)及灭菌的双蒸水,置于42 ℃水浴60 min,最后于70 ℃水浴变性15 min,冰上冷却2 min后,-70 ℃保存备用。
1.3 电子克隆利用NCBI数据资源,在GenBank的核酸(nr/nt)数据库中检索拟南芥的WRKY 18 基因的序列作为探针,对不结球白菜EST数据库进行BLAST检索,获得与拟南芥WRKY18同源的不结球白菜EST片段,以同源性较高的不结球白菜EST片段再次检索不结球白菜EST数据库,获得多条同源性高的EST片段,然后用DNAMAN(Lynnon Biosoft)进行收尾拼接得到一个重叠群(Contig),然后用不结球白菜EST数据库反复检索Contig,拼接,就可得cDNA全长[18]。
1.4 不结球白菜WRKY 18 基因克隆以电子克隆的不结球白菜WRKY 18 的序列为模板,设计特异引物(表 1),在不结球白菜逆境cDNA文库中进行PCR扩增。PCR反应体系为:cDNA 1.5 μL,LA Taq(5 U · μL-1)0.5 μL,10×LA PCR BufferⅡ(Mg2+Plus)2.5 μL,10 mmol · L-1 dNTP Mixture 1.5 μL,ddH2O 12 μL,引物各1 μL。反应条件为:94 ℃ 4 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。反应产物经12 g · L-1琼脂糖凝胶电泳回收后,连接pMD18-T载体并转化大肠杆菌DH5α,提取质粒经PCR鉴定后测序。引物合成和测序都委托上海英骏生物技术有限公司(Invitrogen)完成。
引物Primer | 序列(5′→3′)Sequence | 用途Usage |
| P1/P2 | TAGAATGGAGGAACTTTTTG/ATCCTTACTCCATTTTAGAG | cDNA全长扩增The full length of cDNA amplification |
| P3/P4 | ATGGACTGTTCTTCCTTTCTTG/TGTTCTAGACTGCTCCATTAAC | BcWRKY18基因ORF扩增 Complete ORF amplification of BcWRKY18 |
| P5/P6 | ACTACTACGCACTACATCAC/CATTCCTTCATCTTCACCTTC | BcWRKY18 qRT-PCR |
| Actin1/Actin2 | GCACCCTGTTCTTCTTACCGAG/AGTAAGGTCACGTCCAGCAAGG | qRT-PCR内参Reference of qRT-PCR |
利用SignalP 4.0、TMHMM及SOSUI对WRKY 18 基因编码蛋白进行信号肽、疏水性及蛋白质跨膜结构分析;利用NucPred、Wolfpsort及Nucleo对WRKY18进行亚细胞定位分析;利用Jalveiw 2.0对WRKY18进行多序列比对,利用SMART进行保守结构域分析;利用MEGA5对WRKY18进行系统发育树分析。利用Phyre对WRKY18二级结构进行预测;利用Swiss-Model和Swiss-Pdb Viewer(http://us.expasy.org/tools/)对WRKY18的三级结构进行同源建模分析和可视化分析。拟南芥、水稻、玉米、油菜等的WRKY氨基酸序列均来自GenBank数据库(http://www.ncbi.nlm.nih.gov/genbank/)。
1.6 实时定量RT-PCR根据BcWRKY 18 基因编码区序列,利用Beacon Designer 7软件进行qRT-PCR引物设计。选取BcWRKY 18 中一段长度为150 bp的片段作为扩增靶片段,扩增上游引物为P5,下游引物为P6(表 1)。qRT-PCR的反应体系为20 μL,按照SYBR Premix Ex TaqTM(TaKaRa)有关说明进行操作,其反应条件如下:95 ℃变性2 min;95 ℃变性20 s、60 ℃退火30 s,40个循环。每个样品重复3次。试验以actin基因作为内参基因(扩增用引物对为Actin1/Actin2)。采用LinRegPCR和Excel 2010软件进行相对定量分析。
2 结果与分析 2.1 电子克隆及BcWRKY 18 基因分离经GenBank数据库BLAST检索,得到1条高相似的EST序列,然后以此序列作为种子序列BLAST检索不结球白菜EST数据库,将检出的与种子序列同源性较高的或有部分重叠的EST序列拼接组装为重叠群(Contig),再以此重叠群序列重复以上BLAST检索过程。反复进行EST重叠群序列的拼接和比对,尽可能获得不结球白菜WRKY 18 的cDNA全长。根据不结球白菜WRKY 18 的cDNA全长设计特异引物P1、P2及P3、P4,以不结球白菜逆境cDNA文库为模板,用P1、P2引物对不结球白菜WRKY 18 的cDNA全长进行PCR扩增,用P3、P4引物对不结球白菜WRKY 18 的cDNA开放阅读框(ORF)进行PCR扩增(图 1)。经克隆测序鉴定,不结球白菜WRKY 18 的cDNA全长为1 080 bp,ORF为978 bp,推测其编码326个氨基酸,相对分子质量约为36.32×103,理论等电点为7.28。
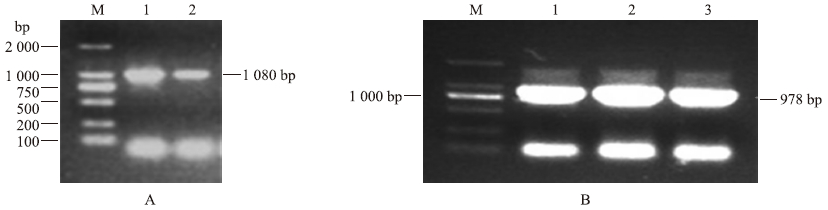 | 图 1 BcWRKY 18 基因的全长(A)和开放阅读框(B)扩增产物 Fig. 1 The full length(A)and open reading frame(B)of BcWRKY 18 amplification products M:DL2000 marker;1、2、3代表重复Number 1,2,3 indicate repeat. |
序列分析表明,BcWRKY 18 编码的氨基酸与其他植物的WRKY蛋白氨基酸有着高度保守的序列,即WRKY结构域(图 2-A),Ishiguro等[19]在甘薯中首次发现该结构域,其可以识别DNA顺式作用元件(cis-acting element)(T)(T)TGAC(C/T)序列即W-box并与之结合。WRKY结构域有60个保守的氨基酸,内含WRKYGQ核心元件及1个锌指蛋白域。根据Eulgem等[5]对植物WRKY转录家族的分类,BcWRKY18转录因子属于Group Ⅱa类,具有1个WRKY结构域和1个C2H2型(C-X4-5-C-X22-23-H-X1-H)锌指结构。通过SMART分析了BcWRKY18氨基酸序列的基序(motif),得出了BcWRKY18的motif模式构型(图 2-B),有卷曲螺旋(coiled coil)在35~65位氨基酸之间,WRKY结构域在201~261位氨基酸残基之间。
 |
图 2 BcWRKY 18 基因核苷酸和氨基酸序列(A)及motifs结构模式图(B)
Fig. 2 Sequences(A)and motifs schematic diagram(B)of BcWRKY 18 gene
翻译起始密码子ATG、终止密码子TGA以及WRKY结构域用方框标出,WRKY保守位点用红色字体标出。不同形状标记分布的motif。 Translation initiation codon ATG,TGA stop codon and WRKY domain are marked with black box,and WRKY conserved sites are showed in red font.The distribution motifs are marked using different shapes. |
利用SOSUI、TMHMM2.0及TMpred分析软件对BcWRKY18进行跨膜区和疏水性分析。SOSUI预测结果为BcWRKY18无信号肽、亲水性蛋白;TMHMM 2.0预测结果为无信号肽和跨膜区;TMpred的预测结果为BcWRKY18为非跨膜蛋白。因此,可以推断BcWRKY18为无信号肽和跨膜区的亲水性蛋白。利用NucPred、Wolfpsort及Nucleo对WRKY18进行亚细胞定位分析。NucPred核定位预测值为0.85,Wolfpsort用KNN 14,细胞核得分为13,Nucleo细胞核定位信号得分为0.79,而非核定位信号得分为0.21。综上可知BcWRKY18亚细胞定位于细胞核,与其具有WRKY结构域的转录因子特征相符。
2.3 WRKY结构多序列比对及BcWRKY18系统进化分析利用BcWRKY18氨基酸序列,通过检索GenBank数据库下载BcWRKY18的同源物[ZmWRKY(玉米)、QsWRKY18(水稻)、BnWRKY18(油菜)、AtWRKY18(拟南芥)、PtWRKY(毛果杨)、GmWRKY(大豆)、MtWRKY(蒺藜苜蓿)、CsWRKY(黄瓜)、SlWRKY(番茄)、SbWRKY(高粱)]的氨基酸序列,用Jalveiw 4.0程序进行WRKY保守结构域联配比对,分析其保守结构域演化模式。结果显示:这些植物的WRKY氨基 酸序列均含有WRKYQK基序和C2H2型锌指,WRKY结构域氨基酸序列变异情况及演化模式见图 3-A。 用MEGA5通过邻接法(N-J)构建WRKY18的系统发育树来分析其系统进化(图 3-B)。从该系统发育树上看,在这些开花植物中的WRKY18可以分为3个大的分支,且BcWRKY18与BnWRKY18和AtWRKY18在进化关系上最近,氨基酸序列相似度分别为87%和73%。在这个系统发育树中,高粱(Sorghum bicolor)、玉米(Zea mays)和水稻(Oryza sativa)在同一个分支,玉米和水稻同为禾本科,进化关系较接近;毛果杨(Populus trichocarpa)、拟南芥(Arabidopsis thaliana)、不结球白菜(Brassica campestris ssp.chinensis Makino)和油菜(Brassica napus)在同一个分支上,不结球白菜和油菜均为十字花科芸薹属,进化关系最为接近,拟南芥也属于十字花科,与这两种植物在进化关系上也较为接近;番茄(Solanum lycopersicum)、大豆(Glycine max)、蒺藜苜蓿(Medicago truncatula)、黄瓜(Cucumis sativus)属于另外一个大的分支,大豆和百脉根均是豆科植物进化关系较近,这与植物系统演化关系类似。
 | 图 3 WRKY18结构域(A)及系统发育进化(B)分析 Fig. 3 Analysis of the amino acid sequence of WRKY18 domains(A)and phylogenetic tree(B) Zm:玉米Zea mays;Os:水稻Oryza sativa;Bc:不结球白菜Brassica campestris ssp.chinensis Makino;Bn:油菜Brassica napus;At:拟南芥Arabidopsis thaliana;Cs:黄瓜Cucumis sativus;Sb:高粱Sorghum bicolor;Pt:毛果杨Populus trichocarpa;Mt:蒺藜苜蓿Medicago truncatula;Sl:番茄Solanum lycopersicum;Gm:大豆Glycine max |
基于BcWRKY18氨基酸序列,利用Phyre2对WRKY18二级结构进行了预测(图 4-A),用Swiss-Model对WRKY18三级结构进行了同源建模分析,通过Molecular Viewer 3D软件实现模型可视化(图 4-B、C、D)。结果表明:BcWRKY18蛋白由无规则卷曲、α螺旋和β折叠构成,C-端WRKY结构域由4个β折叠和β折叠一端的半胱氨酸(Cys)和组氨酸(His)残基包裹的锌离子构成的锌指组成。
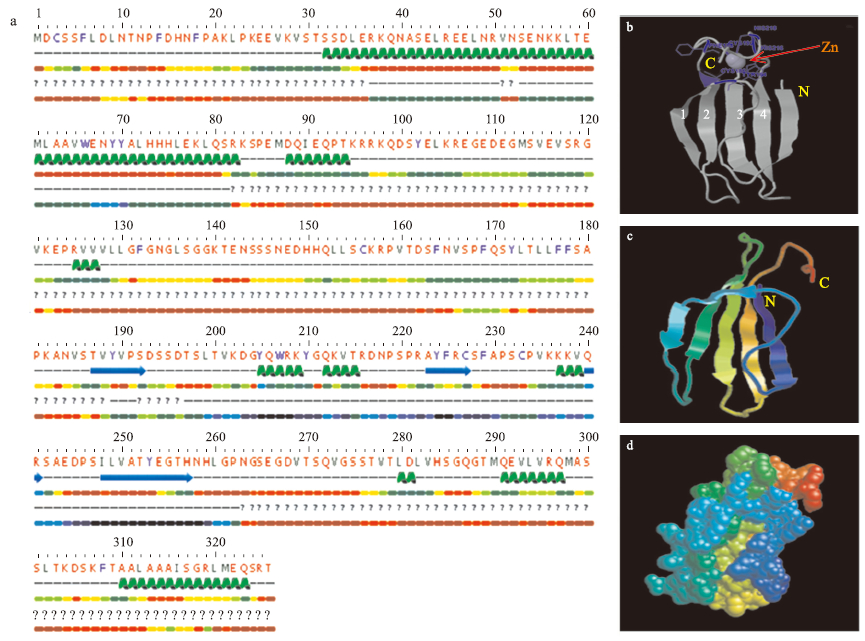 |
图 4 BcWRKY18蛋白质二级及三级结构分析
Fig. 4 Visualization of structure for target BcWRKY18
a.BcWRKY18二级结构示意图,问号代表无规则卷曲,螺旋代表α螺旋,箭头代表β折叠;b.BcWRKY18空间结构示意图,顶部为WRKY结构域,蓝色标出组氨酸和半胱氨酸残基包裹锌离子,1、2、3、4代表C-端的4条β折叠;c.水平旋转90°的BcWRKY18空间结构示意图;d.BcWRKY18的3D分子表面模型示意图,顶部红色和绿色交汇部分为WRKY结构域DNA结合活性区域。 a.Secondary structure and disorder prediction of BcWRKY18,question mark is disorder,green coils indicate α-helix,the blue arrow represents β-sheet;b.3D-superimposition of the structure of BcWRKY18,the top of the structure is WRKY domain,histidine and cysteine residues is marked by blue,and 1,2,3,4 indicate 4 β-strands at C terminal;c.3D-superimposition of the BcWRKY18 structure spins 90° in the anticlockwise direction,which displayed using colored cartons;d.BcWRKY18 molecular surface structure,the DNA binding active center of WRKY domain located on the top of the red and green overlapping areas. |
由图 5可见:BcWRKY 18 基因在低温诱导下表达量在1~8 h时开始快速升高然后处于一个较平稳表达的过程,8 h后逐渐升高,12~48 h缓慢升高,60 h达到最高值后降低,72 h后处于较平稳状态;在100 μmol · L-1 ABA处理下也呈相似的规律,1~2 h升高并处于一个较高的水平,4 h后降低并处于一个平稳的较低的状态,12 h后升高,12~24 h较为平稳,36 h后升高,到48 h达到最高,84 h后开始降低。BcWRKY 18 在低温和ABA处理下具有快速升高—下降—再升高—再下降的表达模式。这表明低温和ABA都能诱导BcWRKY 18表达,且BcWRKY 18 对ABA的胁迫响应更敏感;而相似的表达模式则表明,BcWRKY 18 对ABA和低温存在协同共表达现象。
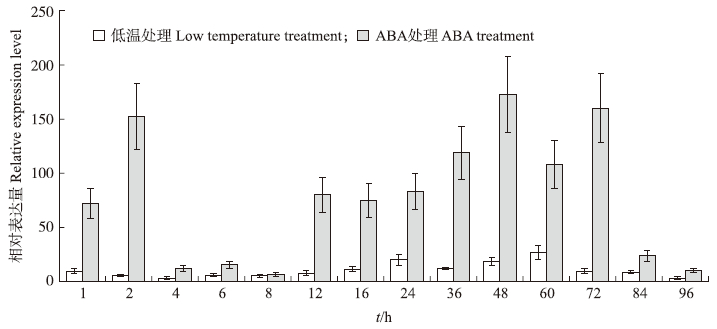 | 图 5 BcWRKY 18 在ABA和低温处理下的相对表达量 Fig. 5 The relative expression level of BcWRKY 18 under ABA and low temperature treatment |
WRKY转录因子家族是一类植物特有的超级基因家族,广泛参与植物的生长发育和生理过程。本研究中,我们采用3种不同算法的软件分别对BcWRKY18信号肽、跨膜区及亚细胞定位等进行预测。通过比较分析,3种分析软件(SignalP 4.0、TMHMM及SOSUI)得到一致的结果,由此确定预测结果的准确性,这比单一一种算法预测的结果可靠性高。基于序列信息和同源性等特征采用多种预测软件进行同一生物学预测分析,不但可以校正不同预测算法的差异,更能很好检出阳性结果,这是生物数据分析必须要考虑的。BcWRKY18无信号肽、跨膜区,亚细胞定位于细胞核,这与其为转录因子功能特征相符,更能说明预测分析的准确性。我们通过同源建模的方式建立了BcWRKY18的空间结构,形象直观地展示了其空间构象和功能域构成,为进一步研究BcWRKY18核蛋白的功能创造条件。WRKY18的基序(motif)变异和WRKY18在植物群体中进化关系,WRKY蛋白功能结构的保守性和植物系统进化相一致,亲缘关系相近的物种WRKY蛋白进化关系近,系统进化树分析能聚在同一个分支内[20]。
WRKY基因在植物体内并非组成型表达,而是受外界环境的激发而诱导表达,如病原物及其诱发因子、防卫反应信号分子如水杨酸及其功能类似物、环境胁迫(如低温、高温、高盐)和创伤均能诱导WRKY基因的表达[21]。以前报道WRKY 18多参与生物胁迫调节,如在拟南芥中AtWRKY 18 受损伤诱导参与植物防御反应[22],ABA能迅速上调AtWRKY 18 ,然后下调[23],而低温对AtWRKY 18 的调控还未见报道。本试验发现,在ABA处理中,BcWRKY 18表达量降低比AtWRKY18 提前,且12 h有一个再上调的变化。这种表达变化的差异,暗示了BcWRKY 18可能具有和AtWRKY18 不同的功能。WRKY蛋白能结合自身启动子中的W-box从而调控基因本身的表达[24]。BcWRKY 18 在低温和ABA处理下表达量迅速升高后又降低,与BcWRKY18蛋白和自身启动子中的W-box相结合从而调控自身表达有关。而随后表达量的升高,可能是因为存在其他转录调节因子的竞争或者干扰,抑制了BcWRKY18对自身的调控。虽然在低温和ABA处理下不结球白菜BcWRKY18是否能结合自身启动子中的W-box来调节自身表达以及与顺式作用元件是否存在竞争或拮抗作用还需要进一步研究,但这一结果加深了我们对WRKY 18 参与植物逆境调控的认识,将为不结球白菜WRKY基因参与低温逆境分子调控机制及WRKY转录因子协同参与低温和ABA响应机制研究提供重要数据和科学依据。
| [1] | Xu Z S, Chen M, Li L C, et al. Functions and application of the AP2/ERF transcription factor family in crop improvement[J]. Journal of Integrative Plant Biology, 2011, 53(7):570-585 |
| [2] | Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana[J]. Current Opinion in Plant Biology, 2001, 4(5):447-456 |
| [3] | Nuruzzaman M, Manimekalai R, Sharoni A M, et al. Genome-wide analysis of NAC transcription factor family in rice[J]. Gene, 2010, 465(1):30-44 |
| [4] | Kang S G, Price J, Lin P C, et al. The Arabidopsis bZIP1 transcription factor is involved in sugar signaling, protein networking, and DNA binding[J]. Molecular Plant, 2010, 3(2):361-373 |
| [5] | Eulgem T, Rushton P J, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J]. Trends in Plant Science, 2000, 5(5):199-206 |
| [6] | Rushton D L, Tripathi P, Rabara R C, et al. WRKY transcription factors:key components in abscisic acid signalling[J]. Plant Biotechnology Journal, 2012, 10(1):2-11 |
| [7] | Wang Q, Wang M, Zhang X, et al. WRKY gene family evolution in Arabidopsis thaliana[J]. Genetica, 2011, 139(8):973-983 |
| [8] | Yang B, Jiang Y, Rahman M H, et al. Identification and expression analysis of WRKY transcription factor genes in canola(Brassica napus L.)in response to fungal pathogens and hormone treatments[J]. BMC Plant Biology, 2009, 9(1):68 |
| [9] | Jang J Y, Hwang D J. The WRKY superfamily of rice transcription factors[J]. The Plant Pathology Journal, 2010, 26(2):110-114 |
| [10] | 罗昌国, 乔玉山, 渠慎春, 等. 湖北海棠MhWRKY42基因克隆及表达分析[J]. 南京农业大学学报, 2013, 36(3):29-35. doi:10.7685/j.issn.1000-2030.2013.03.005 [Luo C G, Qiao Y S, Qu S C, et al. Cloning and expression of MhWRKY42 gene from Malus hupehensis[J]. Journal of Nanjing Agricultural University, 2013, 36(3):29-35(in Chinese with English abstract)] |
| [11] | Eulgem T, Somssich I E. Networks of WRKY transcription factors in defense signaling[J]. Current Opinion in Plant Biology, 2007, 10(4):366-371 |
| [12] | Pandey S P, Somssich I E. The role of WRKY transcription factors in plant immunity[J]. Plant Physiology, 2009, 150(4):1648-1655 |
| [13] | Zhang Z L, Xie Z, Zou X, et al. A rice WRKY gene encodes a transcriptional repressor of the gibberellin signaling pathway in aleurone cells[J]. Plant Physiology, 2004, 134(4):1500-1513 |
| [14] | Ramamoorthy R, Jiang S Y, Kumar N, et al. A comprehensive transcriptional profiling of the WRKY gene family in rice under various abiotic and phytohormone treatments[J]. Plant and Cell Physiology, 2008, 49(6):865-879 |
| [15] | 侯喜林. 不结球白菜育种研究新进展[J]. 南京农业大学学报, 2003, 26(4):111-115. doi:10.7685/j.issn.1000-2030.2003.04.026 [Hou X L. Advances in breeding of non-heading Chinese cabbage[J]. Journal of Nanjing Agricultural University, 2003, 26(4):111-115(in Chinese with English abstract)] |
| [16] | 王彦华, 侯喜林, 申书兴. 白菜WRKY转录因子cDNA全长的克隆及分析[J]. 农业生物技术学报, 2007, 15(5):810-815 [Wang Y H, Hou X L, Shen S X. Cloning and characterization of a full-length cDNA of WRKY transcription factor gene in Brassica campestris ssp.chinensis[J]. Journal of Agricultural Biotechnology, 2007, 15(5):810-815(in Chinese with English abstract)] |
| [17] | Wang F, Hou X, Tang J, et al. A novel cold-inducible gene from Pak-choi(Brassica campestris ssp.chinensis), BcWRKY46, enhances the cold, salt and dehydration stress tolerance in transgenic tobacco[J]. Molecular Biology Reports, 2012, 39(4):4553-4564 |
| [18] | Gill R W, Sanseau P. Rapid in silico cloning of genes using expressed sequence tags(ESTs)[J]. Biotechnology Annual Review, 2000, 5:25-44 |
| [19] | Ishiguro S, Nakamura K. Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5' upstream regions of genes coding for sporamin and β-amylase from sweet potato[J]. Molecular and General Genetics, 1994, 244(6):563-571 |
| [20] | Agarwal P, Reddy M P, Chikara J. WRKY:its structure, evolutionary relationship, DNA-binding selectivity, role in stress tolerance and development of plants[J]. Molecular Biology Reports, 2011, 38(6):3883-3896 |
| [21] | Rushton P J, Somssich I E, Ringler P, et al. WRKY transcription factors[J]. Trends in Plant Science, 2010, 15(5):247-258 |
| [22] | Pandey S P, Roccaro M, Schön M, et al. Transcriptional reprogramming regulated by WRKY18 and WRKY40 facilitates powdery mildew infection of Arabidopsis[J]. The Plant Journal, 2010, 64(6):912-923 |
| [23] | Chen H, Lai Z, Shi J, et al. Roles of arabidopsis WRKY18, WRKY40 and WRKY60 transcription factors in plant responses to abscisic acid and abiotic stress[J]. BMC Plant Biology, 2010, 10(1):281 |
| [24] | Eulgem T, Rushton P J, Schmelzer E, et al. Early nuclear events in plant defence signalling:rapid gene activation by WRKY transcription factors[J]. The EMBO Journal, 1999, 18(17):4689-4699 |




