文章信息
- 张守科, 方林鑫, 刘亚宁, 王毅, 张威, 舒金平, 张亚波, 汪阳东, 王浩杰.
- Zhang Shouke, Fang Linxin, Liu Yaning, Wang Yi, Zhang Wei, Shu Jinping, Zhang Yabo, Wang Yangdong, Wang Haojie.
- 茶籽象ATP合成酶基因在不同海拔选择压力下的遗传分化及结构变异
- Genetic Differentiation and Structural Variation of ATP Synthase Gene of Curculio chinensis (Coleptera: Curculionidae) under Selection Pressure at Different Altitudes
- 林业科学, 2019, 55(6): 65-73.
- Scientia Silvae Sinicae, 2019, 55(6): 65-73.
- DOI: 10.11707/j.1001-7488.20190608
-
文章历史
- 收稿日期:2018-07-10
- 修回日期:2018-09-18
-
作者相关文章
2. 林木遗传育种国家重点实验室 北京 100091;
3. 南京林业大学 南京 210037;
4. 青田县林业局 青田 323900
2. State Key Laboratory of Tree Genetics and Breeding, Chinese Academy of Forestry Beijing 100091;
3. Nanjing Forestry University Nanjing 210037;
4. Qingtian Forestry Bureau Qingtian 323900
生境改变在昆虫种群遗传分化中起到至关重要的作用(Sanford et al., 2011; Fan et al., 2014;Maixner et al., 2014)。气候变化、地质事件或者人为活动等因素都会对种群遗传变异产生一定影响,适应环境的变异得以保留,最终形成对特定环境适应的性状(Kassen et al., 2004;Fan et al.,2014;Miyakawa et al., 2018)。具有专食特性的植食性昆虫遗传分化受到寄主影响明显,当寄主植物迁移(或分布发生变化)时,昆虫长时间形成的种群分布格局随之被打破,遗传结构相应变化(Zhang et al., 2018)。在迁飞能力强的物种遗传多样性和种群系统发育学研究中发现,地理阻隔是分化出现的主要因素(Meng et al., 2010;Fan et al., 2014;Ohwaki et al., 2018);而针对一些飞行能力弱的鞘翅目昆虫,寄主植物是降低昆虫基因交流的重要原因(Schluter,2009;Xue et al., 2010; 2014;Nakadai et al., 2016)。
近年来,昆虫适应环境变化的分子机理研究成为热点,随着测序技术的不断进步,线粒体全基因组比较分析的实现进一步推动昆虫系统发育研究的快速发展。线粒体作为生命有机体主要的供能单元,其基因组结构变异与其生态环境的适应性进化密切相关(Wang et al., 2010;2011)。当前,鞘翅目群体遗传学、DNA条形码开发以及进化遗传学等长时间进化尺度的研究,主要集中在相对保守的线粒体蛋白编码基因COI、COII、ND等区域(聂瑞娥等,2014)。而ATP合成酶基因因其相对进化、对环境变化敏感的特性(Peng et al., 2012),较多应用于种下阶元生命体对生存环境的适应性进化研究中(Gu et al., 2012;2016;Zhang et al., 2017)。
茶籽象(Curculio chinensis)隶属于鞘翅目(Coleoptera)象甲科(Curculionidae),主要分布在地理环境复杂的长江以南及云贵高原地区(何立红等,2014;李志文等,2015),成虫将卵产进茶果,幼虫孵化后以茶果仁为食,产生毁灭性危害,是我国油茶(Camellia oleifera)重要的种实害虫(舒金平等,2013;何立红等,2104;李苗苗等,2016; 2017)。茶籽象飞行能力弱,寄主依存度高,是研究适应环境进化的良好材料。笔者结合发表的鞘翅目昆虫线粒体全基因组,比较分析了茶籽象线粒体全基因组,发现线粒体13个编码蛋白中ATP合成酶基因ATP6、ATP8基因片段变异显著(Zhang et al., 2019)。另外,云贵高原地区分布的茶籽象种群与其他长江以南地区的种群ATP合成酶基因变异较大,这些变异是否因环境压力所致,茶籽象在适应性进化过程中海拔差异是否起到主导作用等问题均不明晰。本文通过分析不同海拔区域茶籽象种群ATP合成酶基因遗传及结构的变异特征,试图揭示茶籽象线粒体基因对环境选择的响应机制,同时也为茶籽象的高效防控提供理论依据。
1 材料与方法 1.1 茶籽象样品采集2015年10月于浙江(遂昌、松阳、云和、丽水)、江西、湖北(麻城)、湖南(益阳)、河南(新乡、信阳)、重庆(彭水)、贵州、云南(腾冲、广南)、广西(百色)和广东(广州、韶关)等16个地点采集到119个茶籽象老熟幼虫样品(表 1)。样品保存在实验室-80 ℃的无水乙醇中备用。
|
|
使用Tianamp Genomic DNA Kit (Tiangen, Shanghai, China)试剂盒提取总DNA。提取前以样本针扎虫体的方式(Juradorivera et al., 2009),加入蛋白酶K隔夜消化,既保持虫体完整又可提取足够的总DNA量(Xue et al., 2014)。引物采用根据茶籽象线粒体全基因组(NCBI序列号:MG728094)ATP基因区段设计的特异性引物F:5′-AATGCT CAGAAATCTGTGGATCAAA-3′,R:5′-ATGCTATGTT TATTCGTGACGGGA-3′,序列长度为528 bp。PCR扩增采用50 μL反应体系,按比例混合好后,向PCR管里加入48 μL混合液(包括Mix、ddH2O和引物),然后加入DNA模板2 μL,简短离心混匀。放入PCR扩增仪,开始扩增。PCR扩增条件为:95 ℃预变性5 min循环1次;95 ℃变性1 min,57 ℃退火1min 28 s,72 ℃延伸2 min,循环48次;72 ℃延伸10 min循环1次;4 ℃保存。凝胶成像检测条带,合格后送样测序。引物及测序工作均由杭州尚亚生物完成。
1.3 数据分析 1.3.1 遗传多样性分析将获取的119条约为528 bp基因序列导入Mega 6.0进行比对分析,获得500 bp的完整序列,并计算个体间遗传距离。利用Excel软件计算个样本间海拔差。遗传多样性分析:利用DnaSP 5.0分析软件(Librado et al., 2009)计算多态位点数量(number of polymorphic sites, S),单倍型数量(number of haplotypes, H),单倍型多样度(haplotype diversity, Hd),单倍型多样性标准偏差(standard deviation of haplotype diversity, k),核苷酸多态性(nucleotide diversity, π),平均核苷酸差异数(average number of nucleotide differences, K)。使用DnaSP 5.0分析软件和TCS version 1.21软件包(Clement et al., 2010;Rousset et al., 2017)筛选单倍型及每个单倍型包含个体数量,并将单倍型序列上传NCBI获取序列号(MH560360—MH560391)。分子变异分析及中性检验分析采用Arlguin3.0软件包,同时使用DnaSP 5.0分析软件分析Tajima’s D值(D)及Fu’s Fs(Fs)值加以矫正。
1.3.2 发育树构建系统发育分析前,利用DNMBE 5软件在GTR模型下获取了序列碱基替换饱和性(Xia, 2018),在满足建树的条件下,利用jModelTest软件(Posada et al., 2008)获取构建ML树的最优模型HKY+I。从NCBI经过blast选取KY057374栎实象(Curculio davidi)和KX087269欧洲栗象(Curculio elephas)作为外群。本研究系统发育树的构建采用RAxML网页版分析工具(www.phylo.org)及MRBAYES version 3.152软件包(Ronquist et al., 2003)。
1.3.3 环境选择压力及ATP8基因分析利用EasyCodeML分析软件(Gao et al., 2018)对系统发育树分支进行选择压力分析。ATP合成酶基因编码氨基酸序列结构比较利用氨基酸序列比对分析在线工具(http://meme-suite.org.)分析ATP合成酶基因包含的ATP8片段编码氨基酸序列结构变异。用ArcGIS软件、AI及CorelDRAW等软件作图。
2 结果与分析 2.1 茶籽象种群遗传多样性分析共获得119个有效的茶籽象样本序列,其中:A+T占比高达79.07%,与线粒体DNA高A+T占比规律一致;H为32,S为140,Hd为0.858,k为0.023,π为0.050 5,K为22.774,整体上遗传分化明显(表 1)。各地理种群中,彭水种群具有最高的单倍型个数及核酸多样性(H=9,π=0.050 3);百色、广南、遂昌、松阳及江西具有较高的核酸多样性(π > 0.02)和单倍型多样性(Hd > 0.6);麻城、丽水、腾冲及新乡具有较低的核酸多样性(π < 0.001)和单倍型多样性(Hd < 0.5);百色、广州、云和种群处于中等水平(表 1)。32个单倍型中有共享单倍型12个,最大的单倍型为H05,包含36个个体,由10个地理种群共享;其次为H07,包含22个个体,由6个地理种群共享,其余为H01、H04、H06、H08、H10、H11、H12、H25、H31、H32,由2~7个个体共享。20个独享单倍型分布于遂昌、广州、广南、江西等地理种群中,其中江西种群单倍型数量最多(表 2、图 3)。
|
|
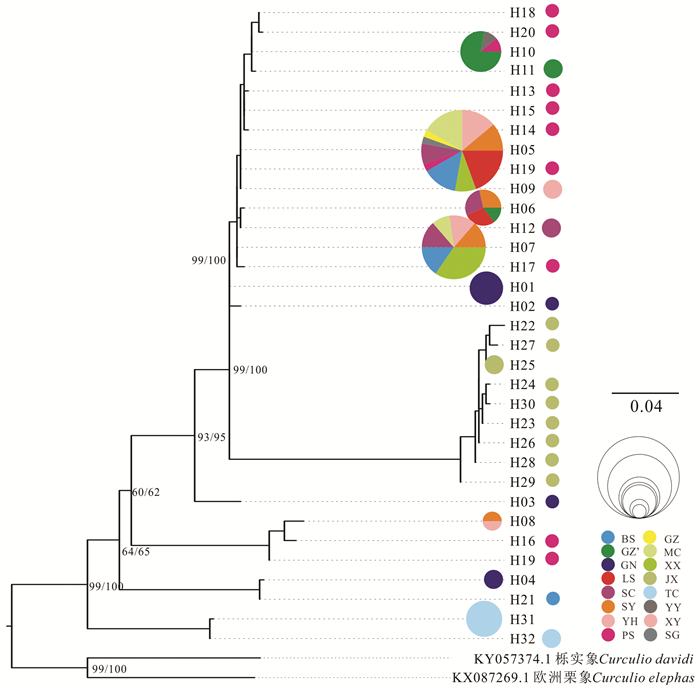
|
图 3 32个单倍型构建的ML/BI系统发育树以及每个单倍型的分享情况及包含的个体数 Fig. 3 The ML/BI phylogenetic trees constructed by 32 haplotypes and the number of individuals involved in each haplotype 圆的面积表示单倍型包含个体数,1个颜色代表 1个地理种群。 The area of the circle in the figure represents a haplotype containing the number of individuals, and a color represents a geographic population. |
在序列用于系统发育研究之前,需进行核酸序列替换饱和性计算,本研究使用的ATP基因片段500 bp的饱和替代计算结果为ISS=0.144 8,小于ISS.c=0.370 4(图 1),可用于系统发育分析(Xia et al., 2018)。利用茶籽象32条单倍型序列构建ML系统发育树及贝叶斯树,树形完全一致,仅节点支持率有所差异,32个单倍型可以归类为7个分支,分支聚类并没有显现地理种群分化规律,各地理种群穿插分布于各支,其中仅江西种群及腾冲种群可分别聚为一支(图 3)。
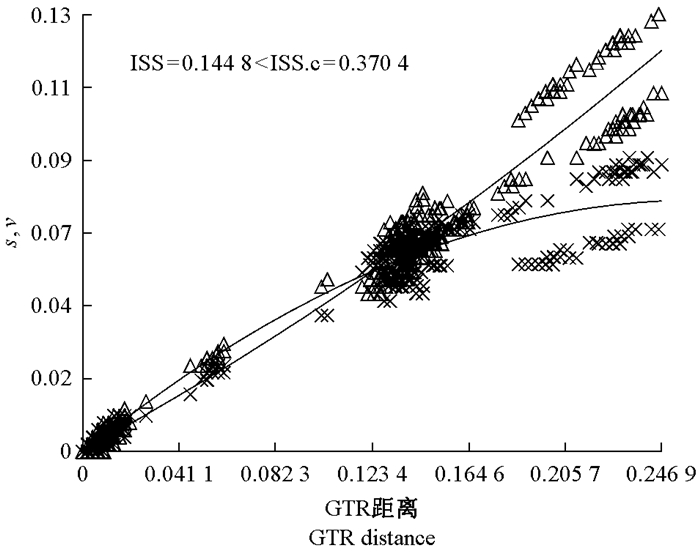
|
图 1 ATP合成酶基因序列替换饱和性检验 Fig. 1 Saturation plots for ATP synthase gene s表示转换率,v表示颠换率。 The plot showed uncorrected pairwise divergences in transitions (s) and transversions (v) against divergences calculated using the GTR model. |
利用茶籽象119个ATP序列构建ML系统发育树,系统发育树可分为2个大支(Lineage 1、Lineage 2),其中Lineage 1中绝大部分个体为江西、百色、贵州、广南、腾冲、彭水等云贵高原地区地理种群,Lineage 2中包含剩余个体,主要为长江中下游低海拔区域的地理种群(图 2A)。基于遗传距离与采集点海拔之间的聚类分析表明所采集样本可明显分为高、低海拔的2个类群,与系统发育树结果一致(图 2B)。AMOVA遗传分化指数分析表明,2个分支已经存在显著分化(Fst=0.374,P < 0.001),且62.598%的变异来自于新划分的类群间(表 2)。
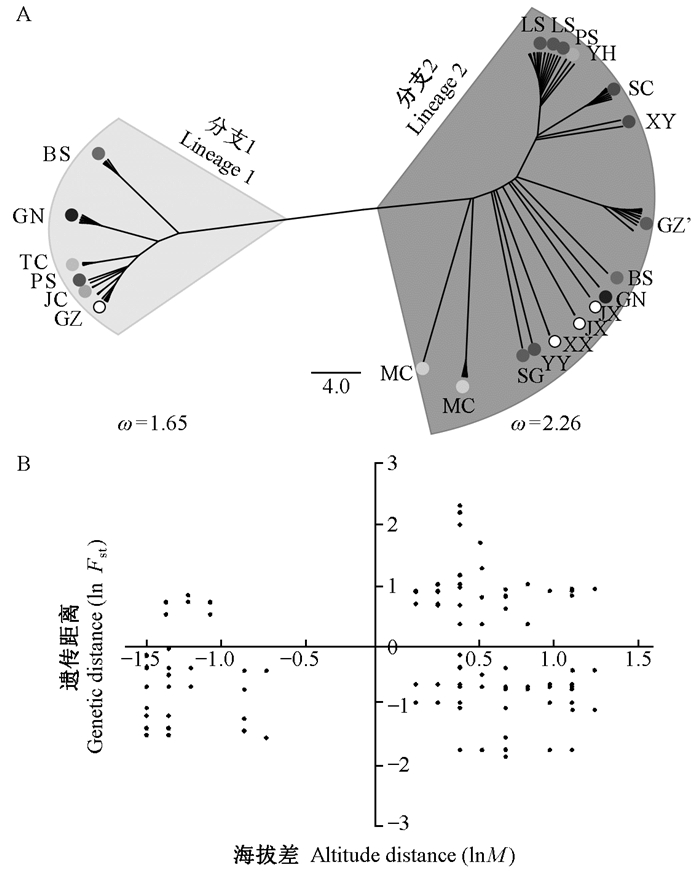
|
图 2 ATP合成酶基因ML树(A)及估算海拔对ATP基因选择压ω值(B)的影响 Fig. 2 ML tree based on ATP synthase gene (A), the estimate of altitude influence on the ω (selective compression value) and clustering analysis (B) based on genetic distance and altitude M.样本采集地间的海拔高度差Altitude difference between two sample sites;Fst.样本间遗传距离Genetic distance between two samples。数据均利用自然对数(ln)进行标准化处理Data were normalized using Napierian logarithm (ln) transforms. |
对茶籽象种群动态分析基于Tajima’s D值及Fu’s Fs值的检验,整体上茶籽象种群处于一个群体扩张过程但不显著,且近期没有发生扩张(Tajima’s D=-0.888 14,P>0.05;Fu’s Fs=5.088),广东地理种群与整体具有相同的扩张趋势;百色、松阳及云和种群历史上发生显著的扩张,但近期没有发生扩张(Tajima’s D为负值,P < 0.05;Fu’s Fs为正值);彭水、遂昌种群近期种群发生过扩张(Tajima’s D为正值,P>0.05;Fu’s Fs为负值);江西种群近期远期都有扩张,但扩张不明显(Tajima’s D为负值,P>0.05;Fu’s Fs为负值);腾冲、新乡种群Tajima’s D、Fu’s Fs为正值,种群近期远期都未发生扩张(表 1)。
根据AMOVA分析可将茶籽象划分为2个大的地理类群(Lineage 1:云贵高原高海拔类群;Lineage 2:长江中下游低海拔类群),选择压力分析结果显示2个类群受到的环境选择压力均为正选择,其中,Lineage 1的选择系数ω1=1.65,LRT P < 0.001;Lineage 2为ω2=2.26,LRT P < 0.001(表 3)。这说明ATP基因在2个地理区域的进化都是有利于个体适应环境,群体中出现能够提高个体生存力及生殖能力的突变,个体将可留下更多的后代,而突变基因最终在整个群体中扩散。ω(Lineage 1)<ω(Lineage 2)表明云贵高原种群人为扰动少,受到的环境压力相对其他扰动大地区较小。
|
|
利用线粒体全基因组与获得的基因序列比对分析后,发现Lineage 1整体上50个位点的ATP8基因编码的氨基酸序列包含27个保守位点,Lineage 2整体上ATP8基因编码的氨基酸序列包含36个保守位点(图 4 A);根据氨基酸功能统计分析,得出各类氨基酸对应2个分支使用率为脂肪质类氨基酸(23.8%,26.2%)、芳香族类氨基酸(23.8%,25.56%)、含硫氨基酸(14.18%,11.3%)、碱性氨基酸(11.3%,12.42%)、酸性氨基酸(13.7%,10.26%)、脂肪族羟基氨基酸类(8.41%,8.49%)、tRNA合成酶Ⅰ(51.2%,49.52%)、tRNA合成酶Ⅱ(48.8%,50.48%)(图 4B)。
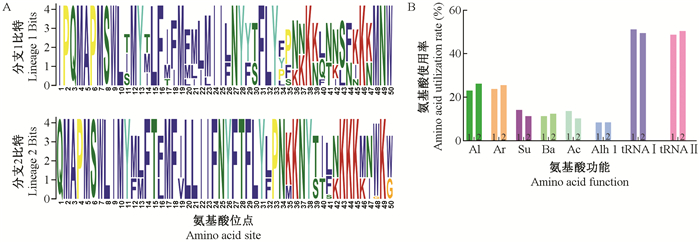
|
图 4 ATP8基因编码氨基酸序列保守性及氨基酸使用率差异分析 Fig. 4 Analysis on the conserved degree and difference of amino acid utilization rate of ATP8 amino acid sequence Al.脂肪质类氨基酸Aliphatic;Ar.芳香族类氨基酸Aromatic;Su.含硫氨基酸Sulphur;Ba.碱性氨基酸Basic; Ac.酸性氨基酸Acidic; Alh.脂肪族羟基氨基酸类Aliphatic hydroxyl; tRNAs Ⅰ. tRNA合成酶Ⅰ tRNA synthetase class Ⅰ; tRNAs Ⅱ. tRNA合成酶Ⅱ tRNA synthetase class Ⅱ. |
本研究基于茶籽象ATP合成酶基因进行的不同地理种群对茶籽象物种遗传多样性和种群系统发育学分析结果与Zhang等(2018)利用COI基因及EF1-a基因的研究结果基本一致,这说明ATP基因具有一定的保守性,可以作为种下阶元遗传分化的研究。对ATP基因进行的单倍型分析结果发现,各地理种群中,除腾冲种群和江西种群外并未形成明显的局域种群,在不同海拔地区同时具有高的核酸多样性及单倍型多样性种群(表 1),存在多个共享单倍型,最大的共享单倍型由10个地理种群共享(图 3),说明这些地理种群存在广泛的基因交流。同时对32个单倍型的系统发育分析结果进一步证明了中国南方茶籽象种群遗传分化水平低,不存在明显的局域种群(图 3)。昆虫的遗传分化与生境息息相关,生存环境、寄主以及人为活动干扰等都可能影响昆虫的遗传分化(Drummond et al., 2010)。茶籽象飞行能力低,无迁飞习性,人为扰动是各地理种群间基因交流产生的最重要因素(Zhang et al., 2018)。不同地理种群的茶籽象随着种苗运输等人为活动进入新的生境中定殖,这个过程是一个复杂的适应性进化过程,昆虫会对不同的环境条件采取不同的进化对策(Schluter,2009)。昆虫具有极高的环境适应能力,r对策种可以产生大量个体及变异,迅速适应并占领生态位,种群在短时间内快速恢复。本文对16个地理种群进行的种群动态分析表明,仅腾冲、新乡种群在历史上或者近期未发生过种群扩张,其余14个地理种群都有不同程度的扩张,但程度不明显(表 1),说明茶籽象种群已经处于一个相对稳定的状态。
研究表明,线粒体通过氧化磷酸化过程(OXPHOS)产生大量ATP,为生命活动提供能量。昆虫能否顺利适应环境,线粒体ATP的合成效率往往起到重要作用(Guderley et al., 2002;Guderley, 2004)。ATP的合成效率往往决定着动物能否适应高海拔、低温、不适宜寄主等不利于生存的环境条件(Guderley,2004),这一过程中与ATP合成密切相关的ATP合成酶基因的适应性进化影响显著。近年来随着比较基因组学研究的不断发展,对昆虫线粒体全基因组适应性进化研究不断深入,线粒体基因对环境影响的应答也有所报道(Peng et al., 2012;张棋麟等,2014)。Zhang等(2017)对青藏高原不同海拔的草原毛虫(Gynaephora alpherakii和G. menyuanensis)13个蛋白基因表达量与环境的相关性及基因结构分析发现ATP8基因存在着明显差异。Peng等(2012)对2种山羊(Pseudois nayaur和P. schaeferi)ATP合成酶基因环境选择压力分析发现,不同环境下的ω比率(dN/dS)相差高达16倍。本研究中,依据ATP基因和样本海拔信息,16个茶籽象地理种群可聚类为高、低海拔2个显著分化的大支(图 2A、表 2、表 3),且分化来自2个大支之间,ω比率(dN/dS)低海拔分支约为高海拔分支的1.4倍,证明茶籽象在不同的环境条件下的对策完全不同且海拔对茶籽象进化的影响不可忽视。
昆虫线粒体中13条蛋白编码基因(PCGs)中主要有ND、CtyB、COX和ATP合成酶基因参与OXPHOS过程(Wallace,2007),mtDNA基因组编码的氨基酸序列变异会直接影响ATP合成过程,进而影响生命体对环境的适应(Bai et al., 2004;Gu et al., 2012)。研究表明半胱氨酸(Vevea et al., 2013;2014)、苏氨酸等在线粒体氨基酸序列的占比增加而增强螺旋结构-螺旋作用这一过程中起到至关重要的作用,可以增加蛋白稳定性,从而提高氧结合效率以及氧化呼吸效率,是对高海拔环境的一种适应(Hassanin et al., 2009;Yu-Rong et al.,2010;Wang et al., 2011)。本研究中,高、低海拔区域茶籽象种群的ATP8基因的编码氨基酸序列存在多个差异位点,且氨基酸使用率存在明显差异,其中高海拔地区含硫氨基酸类、酸性氨基酸类占比明显增加(图 4),进一步证实了ATP合成酶基因的变异可能是对海拔等环境因素变化的响应。昆虫对环境的适应性分子机制是当前研究热点之一,通过改变环境观测昆虫分子水平的反应,对于昆虫的监测及防控具有重要指导意义。围绕茶籽象线粒体基因组重要的编码蛋白基因(ND、COX、ATP等基因)对环境的适应性进化分析,可以明确昆虫对环境选择的响应,为合理种植油茶,利用环境因素有效规避茶籽象种群暴发产生的经济损失具有重要意义,也可为茶籽象的高效环保型防控提供理论依据。
4 结论茶籽象是我国重要的油茶虫害,每年造成巨大的经济损失。本研究将ATP合成酶基因引入茶籽象群体遗传学分析中,并证实了ATP合成酶基因存在对海拔的适应性进化,这为揭示昆虫线粒体基因对环境选择的响应机制提供了新的信息。
何立红, 李志文, 刘劲军, 等. 2014. 油茶象危害与小果油茶果实特征的相关性. 林业科学, 50(12): 151-155. (He L H, Li Z W, Liu J J, et al. 2014. Correlation between damage of Curculio chinensis and fruit traits of Camellia meiocarpa. Chinese Silvae Sinicae, 50(12): 151-155. [in Chinese]) |
李苗苗, 舒金平, 张威, 等. 2017. 茶籽象危害与不同品种油茶果实物理性状的关系. 林业科学研究, 30(2): 232-237. (Li M M, Shu J P, Zhang W, et al. 2017. Relationship between Curculio chinensis damage and physical characteristics of cones among Camellia oleifera varieties. Forest Research, 30(2): 232-237. [in Chinese]) |
李苗苗, 张威, 吕军美, 等. 2016. 茶籽象为害对油茶果产量和茶油品质的影响. 植物保护, 42(5): 65-68. (Li M M, Zhang W, Lü J M, et al. 2016. Effects of the damage caused by Curculio chinensis on fruit production of camellia tree and nutritional value of camellia seed oil. Plant Protection, 42(5): 65-68. DOI:10.3969/j.issn.0529-1542.2016.05.010 [in Chinese]) |
李志文, 何立红, 夏姣, 等. 2015. 油茶象幼虫虫龄的划分. 昆虫学报, 58(2): 181-189. (Li Z W, He L H, Xia J, et al. 2015. Determination of larval instars of the camellia weevil, Curculio chinensis (Coleoptera: Curculionidae). Acta Entomologica Sinica, 58(2): 181-189. [in Chinese]) |
聂瑞娥, 杨星科. 2014. 鞘翅目昆虫线粒体基因组研究进展. 昆虫学报, 57(7): 860-868. (Nie R E, Yang X K. 2014. Research progress in mitochondrial genomes of Coleoptera. Acta Entomologica Sinica, 57(7): 860-868. [in Chinese]) |
舒金平, 滕莹, 刘剑, 等. 2013. 油茶采前落果原因初步分析. 中国植保导刊, 33(1): 9-14. (Shu J P, Teng Y, Liu J, et al. 2013. Preliminary analysis on the causes of pre-harvest fruit drop in Camellia oleifera. China Plant Protection, 33(1): 9-14. DOI:10.3969/j.issn.1672-6820.2013.01.002 [in Chinese]) |
张棋麟. 2014.两种草原毛虫的比较线粒体基因组学研究.兰州: 兰州大学硕士学位论文. (Zhang Q L. 2014. Comparative analysis of mitochondrial genomes between two Gynaephora species (Lepidoptera: Lymantriidae). Lanzhou: MS thesis of Lanzhou University.[in Chinese]) |
Bai Y, Hu P, Park J S, et al. 2004. Genetic and functional analysis of mitochondrial DNA-encoded complex I genes. Annals of the New York Academy of Sciences, 1011(1): 272. DOI:10.1196/annals.1293.026 |
Clement M, Posada D, Crandall K A. 2010. TCS: a computer program to estimate gene genealogies. Molecular Ecology, 9(10): 1657-1659. |
Drummond C S, Xue H J, Yoder J B, et al. 2010. Host-associated divergence and incipient speciation in the yucca moth Prodoxus coloradensis (Lepidoptera: Prodoxidae) on three species of host plants. Heredity, 105(2): 183-196. DOI:10.1038/hdy.2009.154 |
Fan Z, Jiang G F, Liu Y X, et al. 2014. Population explosion in the Yellow-Spined Bamboo Locust Ceracris kiangsu and inferences for the impact of human activity. PLoS One, 9(3): e89873. DOI:10.1371/journal.pone.0089873 |
Gao F, Chen J. 2018. EasyCodeML: an interactive visual tool for CodeML analysis. Available from https://github.com/BioEasy/EasyCodeML.
|
Gu M, Dong X, Shi L, et al. 2012. Differences in mtDNA whole sequence between Tibetan and Han populations suggesting adaptive selection to high altitude. Gene, 496: 37. DOI:10.1016/j.gene.2011.12.016 |
Gu P, Liu W, Yao Y F, et al. 2016. Evidence of adaptive evolution of alpine pheasants to high-altitude environment from mitogenomic perspective. Mitochondrial DNA, 27(1): 455. DOI:10.3109/19401736.2014.900667 |
Guderley H. 2004. Metabolic responses to low temperature in fish muscle. Biological Reviews, 79(2): 409-427. DOI:10.1017/S1464793103006328 |
Guderley H, St-Pierre J. 2002. Going with the flow or life in the fast lane: contrasting mitochondrial responses to thermal change. Journal of Experimental Biology, 205(15): 2237-2249. |
Hassanin A, Ropiquet A, Couloux A, et al. 2009. Evolution of the mitochondrial genome in mammals living at high altitude: new insights from a study of the tribe Caprini (Bovidae, Antilopinae). Journal of Molecular Evolution, 68(4): 293-310. DOI:10.1007/s00239-009-9208-7 |
Juradorivera J A, Vogler A P, Reid C A M, et al. 2009. DNA barcoding insect-host plant associations. Proceedings of the Royal Society B: Biological Sciences, 276(1657): 639-648. DOI:10.1098/rspb.2008.1264 |
Kassen R, Rainey P B. 2004. The ecology and genetics of microbial diversity. Annual Review of Microbiology, 58(1): 207-231. DOI:10.1146/annurev.micro.58.030603.123654 |
Librado P, Rozas J. 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
Maixner M, Albert A, Johannesen J. 2014. Survival relative to new and ancestral host plants, phytoplasma infection, and genetic constitution in host races of a polyphagous insect disease vector. Ecology & Evolution, 4(15): 3082-3092. |
Meng X F, Shi M, Chen X X. 2010. Population genetic structure of Chilo suppressalis (Walker) (Lepidoptera: Crambidae): strong subdivision in China inferred from microsatellite markers and mtDNA gene sequences. Molecular Ecology, 17(12): 2880-2897. |
Miyakawa M, Hosoi M, Ai K, et al. 2018. Genetic variations and phylogeography of the swallowtail butterfly Papilio machaon on the Japanese Islands. Entomological Science, 21(3): 248-259. DOI:10.1111/ens.12302 |
Nakadai R, Kawakita A. 2016. Phylogenetic test of speciation by host shift in leaf cone moths (Caloptilia) feeding on maples (Acer). Ecology & Evolution, 6(14): 4958-4970. |
Ohwaki A, Hayami S I, Kitahara M, et al. 2018. The role of linear mown firebreaks in conserving butterfly diversity: Effects of adjacent vegetation and management. Entomological Science, 21(1): 112-123. DOI:10.1111/ens.12289 |
Peng Q, Tang L, Tan S, et al. 2012. Mitogenomic analysis of the genus Pseudois: evidence of adaptive evolution of morphological variation in the ATP synthase genes. Mitochondrion, 12(5): 500-505. DOI:10.1016/j.mito.2012.07.107 |
Posada D. 2008. jModelTest: phylogenetic model averaging. Molecular Biology and Evolution, 25(7): 1253-1256. DOI:10.1093/molbev/msn083 |
Ronquist F, Huelsenbeck J P. 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics, 19(12): 1572-1574. DOI:10.1093/bioinformatics/btg180 |
Rousset F, Gouy A, Martinez-Almoyna C, et al. 2017. The summary-likelihood method and its implementation in the infusion package. Molecular Ecology Resources, 17(1): 110. DOI:10.1111/men.2017.17.issue-1 |
Sanford E, Kelly M W. 2011. Local adaptation in marine invertebrates. Annual Review of Marine Science, 3(1): 509-535. DOI:10.1146/annurev-marine-120709-142756 |
Schluter D. 2009. Evidence for ecological speciation and its alternative. Science, 323(5915): 737-741. DOI:10.1126/science.1160006 |
Vevea J D, Jason D, Liza A., et al. 2014. Inheritance of the fittest mitochondria in yeast. Trends in Cell Biology, 24(1): 53-60. |
Vevea J D, Wolken D M, Swayne T C, et al. 2013. Ratiometric biosensors that measure mitochondrial redox state and ATP in living yeast cells. Journal of Visualized Experiments Jove, (77): 50633. |
Wallace D C. 2007. Why do we still have a maternally inherited mitochondrial DNA? Insights from evolutionary medicine. Annual Review of Biochemistry, 76(76): 781-821. |
Wang Z, Shen X, Liu B, et al. 2010. Phylogeographical analyses of domestic and wild yaks based on mitochondrial DNA: new data and reappraisal. Journal of Biogeography, 37(12): 2332-2344. DOI:10.1111/jbi.2010.37.issue-12 |
Wang Z, Yonezawa T, Liu B, et al. 2011. Domestication relaxed selective constraints on the yak mitochondrial genome. Molecular Biology & Evolution, 28(5): 1553-1556. |
Xia X. 2018. DAMBE7: New and improved tools for data analysis in molecular biology and evolution. Molecular Biology & Evolution, 35(6): 1550-1552. |
Xue H J, Li W Z, Yang X K. 2010. Genetic analysis of feeding preference in two related species of Altica (Coleoptera: Chrysomelidae: Alticinae). Ecological Entomology, 34(1): 74-80. |
Xue H J, Li W Z, Yang X K. 2014. Assortative mating between two sympatric closely-related specialists: inferred from molecular phylogenetic analysis and behavioral data. Scientific Reports, 4: 5436. |
Yu-Rong D U, Guo S C, Wang Z F, et al. 2010. Demographic history of the Tibetan antelope Pantholops hodgsoni (chiru). Journal of Systematics and Evolution, 48(6): 490-496. DOI:10.1111/jse.2010.48.issue-6 |
Zhang Q L, Zhang L, Zhao T X, et al. 2017. Gene sequence variations and expression patterns of mitochondrial genes are associated with the adaptive evolution of two Gynaephora species (Lepidoptera: Lymantriinae) living in different high-elevation environments. Gene, 610: 148-155. DOI:10.1016/j.gene.2017.02.014 |
Zhang S K, Shu J P, Wang Y D, et al. 2019. The complete mitochondrial genomes of two sibling species of camellia weevils (Coleoptera: Curculionidae) and patterns of Curculionini speciation. Scientific Reports, (9): 3412. |
Zhang S K, Shu J P, Xue H J, et al. 2018. Genetic diversity in the camellia weevil, Curculio chinensis Chevrolat (Coleptera: Curculionidae) and inferences for the impact of host plant and human activity. Entomological Science, 21(4): 447-460. DOI:10.1111/ens.12329 |
 2019, Vol. 55
2019, Vol. 55

