文章信息
- 刘道凤, 王霞, 代银, 杨建峰, 马婧, 李名扬, 眭顺照.
- Liu Daofeng, Wang Xia, Dai Yin, Yang Jianfeng, Ma Jing, Li Mingyang, Sui Shunzhao.
- 蜡梅转录因子CpTAF10基因的克隆及功能分析
- Cloning and Function Analysis of CpTAF10 from Wintersweet (Chimonanthus praecox)
- 林业科学, 2019, 55(6): 176-183.
- Scientia Silvae Sinicae, 2019, 55(6): 176-183.
- DOI: 10.11707/j.1001-7488.20190621
-
文章历史
- 收稿日期:2018-06-28
- 修回日期:2018-08-15
-
作者相关文章
在植物的转录调控网络中, 转录因子作为表达网络的关键元件参与调控了大量基因的表达,对植物的生长发育及外界环境的反应等方面都起着重要的调节作用。而通用转录因子是基因转录所必需的作用于核心启动子上的蛋白质辅助因子,在细胞中普遍存在,又称为基本转录因子。已被鉴定的通用转录因子有TFⅡA、TFⅡB、TFⅡD、TFⅡE、TFⅡF、TFⅡH等(Muller, 2001)。其中,TFⅡD(transcription factor ⅡD)是由TATA结合蛋白(TATA-binding protein,TBP)和一些TBP相关因子(TBP associated factors,TAFs)等构成的多亚基复合物。在真核生物的转录过程中,由TFⅡD复合物首先识别并结合启动子的TATA框序列,而起始转录复合物和其他普通转录因子(如TFⅡA、TFⅡB等)再结合到该位置上,最后与RNA聚合酶Ⅱ(RNA Pol Ⅱ)结合并启动基因的转录。因此,TFⅡD在调控RNA Pol Ⅱ转录机制的启动过程中发挥着中枢作用(Cler et al., 2009;Papai et al., 2011)。TAFs是真核生物识别转录启动子TATA框的TFⅡD转录因子的重要组成部分,在它与TATA结合蛋白结合以后生成不同种类的基本转录因子,随后在RNA聚合酶的参与下起始转录及调控(潘建伟等,1998)。目前,在动物和微生物细胞中对TAFs的研究较多,而人和果蝇(Drosophilidae)细胞中最早鉴定获得了TAFs(Dynlacht et al., 1991; Tanese et al., 1991)。研究发现鉴定获得的TAF家族成员序列在不同物种间具有很高的序列保守性,并与多个重要的生长发育过程相关,且可以识别特异的启动子序列,参与特异基因的表达(Shen et al., 2003;郝国礼等,2014)。因此,TAFs是一个具备转录起始识别和调控的独立的蛋白家族,且家族各成员均有其功能独特性(Hiller et al., 2001)。
目前,在植物中关于TAFs基因功能的研究仍然较少。在拟南芥(Arabidopsis thaliana)中,已经鉴定获得了整个TAFs家族基因,TAFs基因的表达检测发现其广泛参与了植物的生长发育过程(Lago et al., 2004)。例如,拟南芥中T-DNA插入突变发现TAF6基因调控了拟南芥花粉管的生长(Lago et al., 2005)。通过对拟南芥taf5突变体的研究,发现TAF5基因影响了花序分生及花粉的发育,是植株生长发育所必需的基因(Mougiou et al., 2012)。TAF10是形成通用转录因子TFⅡD的TAFs家族成员之一。研究发现TAF10基因参与调节了植物形态相关基因的表达并影响了植株的盐胁迫耐性。在黄花菊(Flaveria trinervia)cDNA文库中分离出了TAF10同源基因ftTAF10,通过Northern杂交和原位杂交分析发现ftTAF10基因的表达存在着组织特异性;将ftTAF10基因在拟南芥中的过量表达引起了花序异常和叶片的变形(Furumoto et al., 2005)。拟南芥耐盐突变体stg1中突变的STG1基因编码TAF10蛋白,过表达AtTAF10基因提高了拟南芥种子发芽过程中的盐胁迫耐性,而缺失突变体对盐胁迫更敏感(Gao et al., 2006)。进一步的研究发现,拟南芥TAF10基因在侧根、莲座叶及大部分花器官中选择性表达,taf10突变体影响了植株分生组织与叶片发育(Tamada et al., 2007)。
蜡梅(Chimonanthus praecox)是我国特有的名贵花木,有着悠久的栽培历史。蜡梅因其生长周期长、根系发达,具有较强的综合抗逆性,如耐荫、耐寒、耐旱等,广泛地应用于城乡园林建设。随着蜡梅花cDNA文库(Sui et al., 2012)和转录组数据库的构建(Liu et al., 2014),为蜡梅的分子生物学研究奠定了一定的基础。本研究以蜡梅转录组数据库中获得的蜡梅TAFs家族基因序列,克隆得到CpTAF10基因的cDNA序列,并对其编码蛋白进行基本特征分析及与同源蛋白进行进化关系分析,研究该基因在蜡梅不同组织、不同花期及低温、高温、盐胁迫和ABA处理下的表达特性。同时,通过在拟南芥中过表达CpTAF10基因分析转基因拟南芥胁迫耐性的变化,丰富对植物TAFs基因的功能及在转录调控过程中作用机制的认识,为解析蜡梅的抗逆性形成的转录调节机制及利用基因工程技术提高植物的胁迫耐性提供新的理论依据。
1 材料与方法 1.1 试验材料素心蜡梅(Chimonanthus praecox ‘Luteus’)生长在西南大学校园内。胁迫处理所用的蜡梅幼苗为长势一致的六叶期实生植株。拟南芥野生型为哥伦比亚型(Columbia ecotype)。试验材料均放置在人工气候箱培养,光周期为16 h光照和8 h黑暗,光照强度为20 000 lx。蜡梅苗培养温度为昼25 ℃/夜20 ℃,湿度为85%;拟南芥培养温度为(22±2)℃,湿度为60%~70%。
1.2 蜡梅CpTAF10基因全长cDNA的获得蜡梅组织的总RNA提取按照RNAprep pure植物总RNA提取试剂盒(天根,北京)说明书进行。以提取的总RNA为模板,用PrimeScript RT-PCR Kit反转录试剂盒(TaKaRa公司)合成cDNA第1链。根据蜡梅转录组数据库中获得的基因序列,设计引物(表 1),进行基因克隆和序列测序验证。
|
|
利用DNAStar 7.0软件包对该序列进行开放阅读框预测,将其编码的蛋白序列在NCBI上进行BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对,分析序列同源性。利用ProtParam(http://web.expasy.org/protparam/)和SignalP(http://www.cbs.dtu.dk/services/SignalP)分别进行蛋白序列特征分析和蛋白序列中信号肽的剪切位点预测。TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)预测编码蛋白的跨膜区段和在膜上的取向;利用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)和PSORT Ⅱ Prediction软件(http://psort.hgc.jp/form.html)分别预测蛋白质的二级结构和亚细胞定位。用BioEdit软件以Cluster W进行同源蛋白的多序列比对,并利用MEGA7.0软件采用邻接法(Neighbor-Joining, NJ)构建系统进化树。
1.4 CpTAF10基因表达特性分析在蜡梅幼苗植株上分别取根、茎、子叶及嫩叶;在生长良好的蜡梅成年植株上取成熟叶片,并在开花期时分别取不同花发育时期(萌动期、花蕾期、露瓣期、初开期、盛开期和衰老期)的花朵(Sui et al., 2012)。每个样本均取样3个生物学重复。选取长势一致的六叶期蜡梅实生苗,分别进行高温(42 ℃)、低温(4 ℃)、高盐(1 mol· L-1 NaCl)和ABA(50 μmol · L-1)胁迫处理,在处理0、0.25、1、6、12 h时分别取蜡梅苗相同部位的叶片,每个时间点设置3个生物学重复。采集到的样本立即用液氮速冻后保存到-80 ℃冰箱中,用于总RNA的提取。以蜡梅Actin-b和Tublin作为内参基因(表 1),采用SYBR Green荧光染料法,用CFX96(Bio-Rad)实时荧光定量PCR(real-time quantitative PCR)仪对CpTAF10基因在蜡梅不同组织、不同花期及低温、高温、盐胁迫和ABA处理的蜡梅叶片中表达量进行实时荧光定量PCR分析,按照SsoFastTM EvaGreen Supermix试剂盒(Bio-Rad)说明书配制反应体系,每个样品设3次技术重复。使用Bio-Rad Manager TM(Version 1.1)软件对试验数据进行分析,采用2-ΔΔCT法计算获得CpTAF10基因的相对表达量(Livak et al., 2001)。
1.5 CpTAF10基因植物过表达载体的构建根据植物过表达载体pCAMBIA2301和CpTAF10基因特征,设计引物(表 1)在CpTAF10基因5′端和3′端分别添加Xba I和Sac I限制性内切酶位点。利用双酶切和T4连接酶连接,构建CpTAF10基因过表达载体,命名为pCAMBIA2301-CpTAF10,采用电击法将质粒转入根癌农杆菌(Agrobacterium tumefaciens)菌株EHA105感受态细胞。
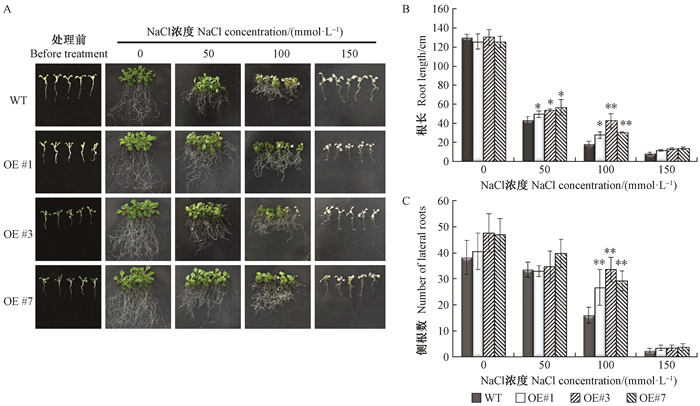
1.6 CpTAF10基因转化拟南芥及盐胁迫耐性分析选取健壮盛花期拟南芥,采用农杆菌花序侵染法将目的基因转入拟南芥。收获侵染植株种子,用NaClO溶液消毒后播种于含25 mg·L-1潮霉素(hygromycin)的MS培养基上进行筛选培养,得到若干不同株系,并对各株系的T1、T2代种子继续筛选培养,直至获得T3代转基因纯合株系。分别取相同苗期的野生型和过表达CpTAF10基因的转基因拟南芥叶片,提取RNA,反转录后,用检测CpTAF10基因表达的特异引物(表 1)进行荧光定量PCR分析,鉴定转基因株系中基因的表达情况。取野生型和转基因拟南芥T3代种子,消毒后分别播种于含有0、50、100、150 mmol·L-1 NaCl的MS培养基上,春化后放置于人工培养箱中培养,6天后统计种子萌发率,试验重复3次。取野生型和转基因拟南芥T3代种子播种后生长1周的幼苗各30株,10株为1次重复,分别平行放置于含有0、50、100、150 mmol·L-1 NaCl的MS培养基上,竖直培养10天后观察植株生根情况,统计根长和侧根数目,并进行拍照(Nikon,D7000)。
2 结果与分析 2.1 CpTAF10克隆子的获得及其序列特征根据研究室已构建的蜡梅转录组数据库中获得的基因序列,在目的基因cDNA序列最大开放阅读框(open reading frame)两侧序列设计引物进行基因克隆和测序验证,测序结果与数据库中获得的序列一致,获得基因全长cDNA序列。利用NCBI蛋白质数据库进行Blastp比对,结果表明该基因编码的蛋白与其他植物中的TAF10高度相似,将其命名为CpTAF10。CpTAF10基因cDNA序列为712 bp,包含405 bp的开放阅读框(ORF)。
CpTAF10开放阅读框编码的蛋白理论分子量为15.21 kDa,由134个氨基酸组成,其中含有40个疏水氨基酸(hydrophobic amino acids),占氨基酸总数的29.85%,35个极性氨基酸(polar amino acids),占总数的26.11%。预测的等电点pI值为5.19。用SOPMA软件对CpTAF10蛋白的二级结构预测结果显示该蛋白的二级结构由α螺旋(α-helix,47.01%)、延伸链(extended strand,5.22%)和大量的随机卷曲(random coil,47.76%)组成。信号肽预测和跨膜结构分析结果显示CpTAF10蛋白无明显的信号肽和跨膜结构。蛋白质亚细胞定位是分析和研究蛋白质功能的重要方面,利用PSORT Ⅱ Prediction软件分析CpTAF10蛋白的亚细胞定位,发现该蛋白主要定位于细胞核和细胞质中,分别占47.8%和34.8%。
2.2 CpTAF10编码蛋白多序列比对和进化树分析将蜡梅CpTAF10蛋白序列与其他植物同源序列比较发现,该序列与葡萄(Vitis vinifera)、大豆(Glycine max)、黄瓜(Cucumis sativus)、可可(Theobroma cacao)、拟南芥及黄花菊中的TAF10蛋白序列高度同源,同源性分别为85.29%、81.62%、80.15%、79.41%、78.68%和70.59%,说明该基因在植物中序列较为保守。利用Clustal W方法进行多重比对(图 1),保守结构域分析显示,CpTAF10蛋白属于TAF10同源蛋白,并含有组蛋白折叠(histone-fold motif)结构域。将CpTAF10编码蛋白序列与人、动物、双子叶和单子叶植物中的同源序列构建进化树,结果显示,蜡梅CpTAF10属于双子叶植物一支,且与葡萄亲缘关系最近(图 2)。
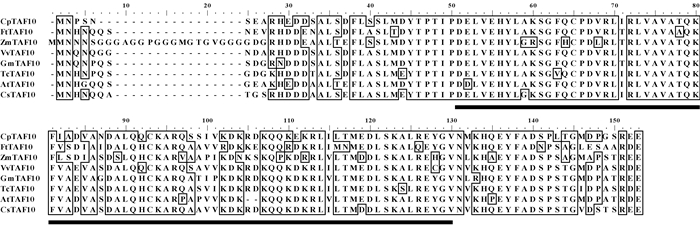
|
图 1 蜡梅CpTAF10蛋白序列与其他植物同源蛋白序列比对 Fig. 1 Multiple alignments of homologus proteins with high similarity sequences to CpTAF10 in wintersweet Cp:蜡梅Chimonanthus praecox; Ft:黄花菊Flaveria trinervia; Zm:玉米Zea mays; Vv:葡萄Vitis vinifera; Gm:大豆Glycine max; Tc:可可Theobroma cacao; At:拟南芥Arabidopsis thaliana; Cs:黄瓜Cucumis sativus.下划线表示组氨酸折叠结构域。Underling indicates the region of the histon-fold motif (Gangloff et al., 2001). |
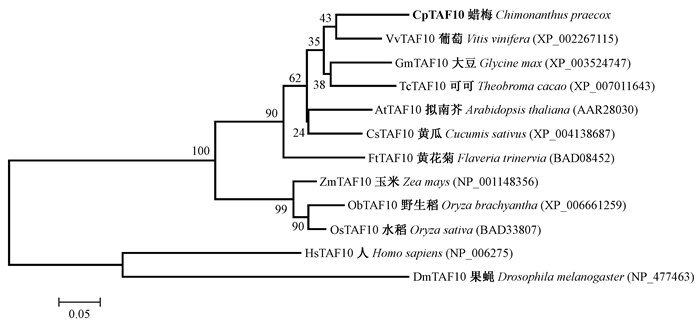
|
图 2 CpTAF10蛋白的进化树分析 Fig. 2 The phylogenetic tree analysis of CpTAF10 protein |
设计基因表达检测特异引物,采用实时荧光定量PCR来检测基因的表达特性。结果发现,CpTAF10基因在蜡梅的根、茎、子叶、幼叶、成熟叶和花6个不同组织中均有不同程度的表达,其中,在成熟叶中的表达量最高,其次是在子叶中(图 3A)。CpTAF10在蜡梅花朵的不同花期中,呈现出波动的表达模式,在萌动期表达量最低,而在衰老期表达量最高(图 3B)。分别检测低温、高温、盐胁迫及ABA处理不同时间(0.25、1、6、12 h)对蜡梅叶片中CpTAF10基因表达的影响,结果发现,在低温、盐胁迫和ABA处理的蜡梅叶片中均能被诱导表达,但其表达变化各不相同(图 4)。盐胁迫处理后,CpTAF10基因的转录水平显著高于其他胁迫处理,处理0.25 h时CpTAF10基因的表达量就快速升高为盐胁迫处理前相对表达量的6倍左右。在低温胁迫处理下,CpTAF10基因的相对表达量在处理0.25,1,6 h时均有显著诱导升高;而在高温胁迫处理下CpTAF10基因无显著诱导表达。在ABA处理0.25,1,6,12 h时,CpTAF10基因的相对表达量均有显著诱导表达,在0.25 h时相对表达量最高,达到了处理前的5倍左右(图 4)。
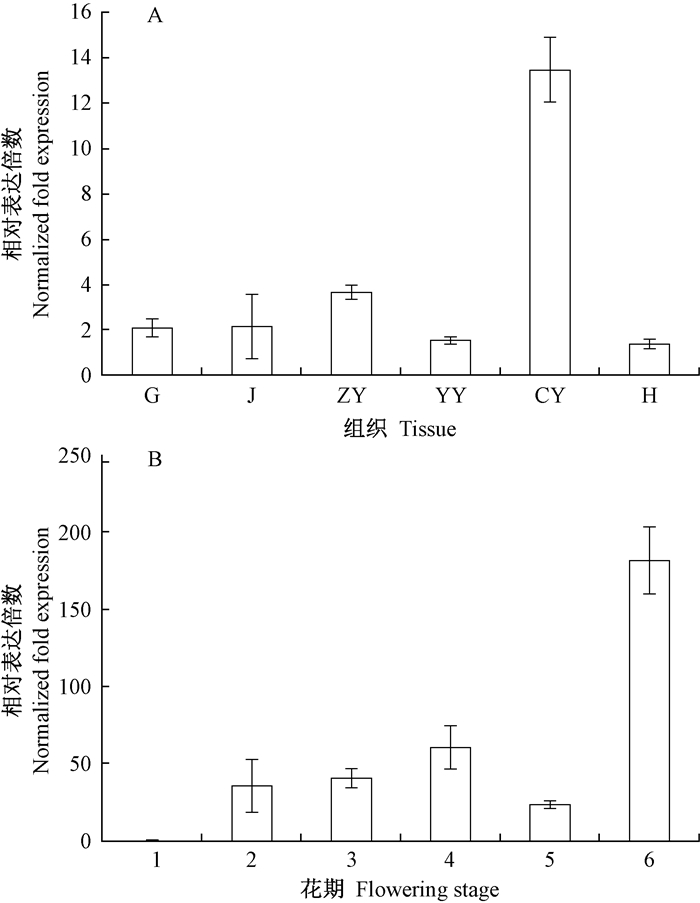
|
图 3 CpTAF10基因在蜡梅不同组织和不同花期中的表达 Fig. 3 Expression analysis of CpTAF10 in various tissues and different flowering stages of wintersweet G:根; J:茎; ZY:子叶; YY:幼叶; CY:成熟叶; H:花。1:萌动期; 2:花蕾期; 3:露瓣期; 4:初开期; 5:盛开期; 6:衰老期。 G: Roots; J: Stems; ZY: Cotyledons; YY: Young leaves; CY: Mature leaves; H: Flower. 1: Sprout period; 2: Flower-bud period; 3: Display-petal period; 4: Initiating bloom period; 5: Bloom period; 6: Wither period. |
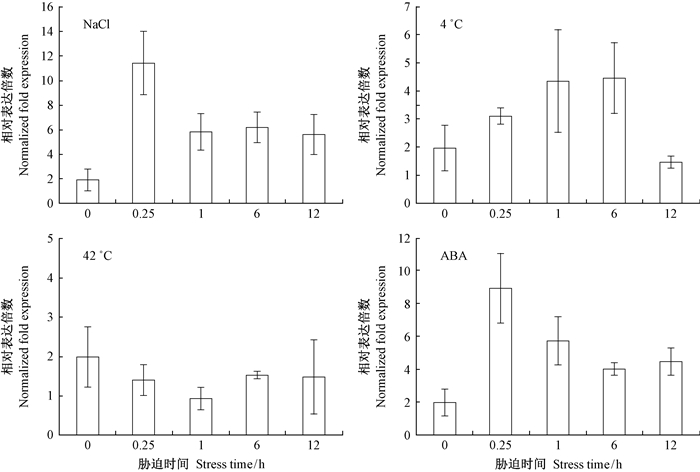
|
图 4 CpTAF10基因在不同胁迫处理下的表达分析 Fig. 4 Expression analysis of CpTAF10 under different stress treatment |
将拟南芥野生型(WT)和过表达CpTAF10基因的转基因株系T3代种子分别播种到NaCl浓度为0、50、100、150 mmol·L-1的MS培养基上,分别观察种子的萌发及存活情况。结果发现,在MS培养基上,WT和转基因种子之间在萌发率上无明显差异,而随着NaCl浓度的升高,过表达CpTAF10基因的转基因拟南芥表现出较强的盐胁迫耐性。在NaCl浓度为100 mmol·L-1的MS培养基上,转基因拟南芥的存活率为80.4%~92.7%,而对照的存活率为44.1%(图 5)。同时,将野生型和转基因拟南芥植株在不同盐浓度处理的MS培养基中竖直培养,10天后观察根长和侧根的生长情况。结果发现随着盐浓度的提高,野生型拟南芥植株的主根和侧根生长均表现为受到盐胁迫抑制,而过表达CpTAF10基因的转基因株系的主根和侧根与野生型相比均表现出了一定的生长优势(图 6A)。统计分析结果显示,转基因拟南芥株系的根长和侧根数在100 mmol·L-1的盐胁迫下与野生型拟南芥存在显著性差异(图 6B, C),说明过表达CpTAF10在一定程度上能够提高拟南芥植株对盐胁迫的耐受性。

|
图 5 不同浓度NaCl处理后拟南芥种子的存活率 Fig. 5 The survival rate of Arabidopsis thaliana seeds under different concentrations of NaCl treatment |
|
图 6 不同浓度NaCl处理后拟南芥生根情况观察 Fig. 6 Observation on root growth condition under different concentrations of NaCl treatment |
转录水平的调控是基因表达过程中最重要的环节,也是植物对逆境胁迫产生应答的关键步骤。然而,对基因转录起始起重要作用的基础转录机制方面的研究仍然十分有限,对通用转录因子的研究还远远落后于哺乳动物、蝇和酵母(Albright et al., 2000; Srivastava et al., 2015)。目前,已经从不同的植物中克隆获得了TAFs家族基因并进行了相关的功能分析,结果表明植物TAFs可能特异地参与转录的激活和调控基因的表达(Moraga et al., 2015)。本试验中根据蜡梅转录组数据库中的基因序列,克隆得到TAF家族同源基因,其编码的蛋白与其他植物的TAF10蛋白序列具有较高的同源性,且包含的一个组蛋白折叠(HFD)结构是通用转录因子TFⅡD的一个重要结构基序。对蜡梅CpTAF10基因编码蛋白进行了亚细胞定位预测分析,表明该蛋白主要分布在细胞核和细胞质中。黄花菊中ftTAF10基因在洋葱(Allium cepa)下表皮细胞中特异定位于细胞核中,符合作为通用转录因子的特性(Furumoto et al., 2005)。
CpTAF10基因在蜡梅的根、茎、子叶、幼叶及花中均有不同程度的表达,其中,在成熟叶片中的表达量最高。在拟南芥中AtTAF10基因在根、莲座叶和花序中表达(Lago et al., 2004),而黄花菊ftTAF10基因在根和茎中表达量比叶片中高,且优先在维管组织中表达(Furumoto et al., 2005)。CpTAF10在蜡梅花朵的不同花期中,从蕾期到初开期的表达量显著诱导升高,相对表达量较高。蜡梅花朵开放期正值冬季逆境环境,此时花朵开放过程中CpTAF10基因的高水平表达,可能是蜡梅抵抗逆境的一种保护机制,CpTAF10基因可能参与了启动相关调控网络来增强花器官的抗逆性,而在衰老期基因表达量最高,推测可能与蜡梅花朵衰老脱落的调控有相关性。此外,CpTAF10基因在高温、低温、盐胁迫和ABA处理后的蜡梅叶片中的表达模式各不相同。低温、盐胁迫和外源ABA处理可不同程度诱导蜡梅幼苗叶片中CpTAF10基因的表达。TAF10基因的表达模式与可能参与的转录起始功能相关,说明CpTAF10可能参与蜡梅逆境胁迫的应答调控,并有可能在脱落酸信号转导途径中发挥作用,参与逆境胁迫的响应,但其调控的具体分子机理仍有待进一步研究。
环境胁迫常常引起植物分子和生理水平上的变化。植物在高盐环境下的低发芽率是对盐胁迫的适应性表现,因此统计种子的发芽率是分析盐胁迫应答的一种有效且快速的方法(Verslues et al., 2006)。将CpTAF10基因在野生型拟南芥中过量表达,发现转基因拟南芥种子在盐胁迫处理下的发芽率显著高于野生型种子的发芽率。此外,在盐胁迫下,转基因拟南芥叶片鲜绿,根长和侧根数明显大于野生型拟南芥。根长和侧根数表明了植株获得水源的能力和生长优势,在受到盐胁迫时,转基因拟南芥的根长和侧根数与野生型存在显著性差异,并表现出一定的生长优势,说明转基因拟南芥的盐胁迫耐性强于野生型,蜡梅CpTAF10基因在拟南芥中过表达能在一定程度上增强拟南芥的盐胁迫耐性。这一结果与拟南芥中同源基因AtTAF10的功能相似,过量表达AtTAF10基因可以提高拟南芥种子发芽过程中的耐盐性,说明该基因在影响植物盐胁迫耐性方面发挥了一定的作用(Gao et al., 2006)。
植物通过调节基因表达模式来响应非生物胁迫,进而调节一系列生化过程以减少逆境胁迫造成的细胞损伤。而转录起始复合体、SAGA和/或TFⅡD成员的表达变化在植物的发育进程和环境胁迫应答中,可能影响到一系列基因在转录水平上的表达,从而影响植物的胁迫耐性。CpTAF10基因过表达对与盐胁迫耐性调控相关基因表达谱的影响还有待试验的进一步验证。总之,本文中对于蜡梅CpTAF10基因的分离和鉴定仅仅是这些TAFs在植物中功能分析的一部分,其提高植株盐胁迫耐性的分子机制需要进一步的深入研究。
4 结论从蜡梅中克隆获得了CpTAF10基因,该基因在蜡梅不同组织中均有不同程度的表达,其中,在成熟叶中的表达量最高。在蜡梅花朵的不同花期中,CpTAF10基因在衰老期表达量最高。在低温、盐胁迫和ABA处理的蜡梅叶片中均能被不同程度地诱导表达。在拟南芥中过表达CpTAF10基因提高了盐胁迫下拟南芥种子的萌发率,相对于野生型植株,转基因植株的主根和侧根在盐胁迫下均表现出一定的生长优势。初步表明蜡梅CpTAF10基因可能参与蜡梅逆境胁迫耐性的分子调控,并能在一定程度上增强转基因拟南芥的盐胁迫耐性。
郝国礼, 于海泉. 2014. TFⅡD在配子发生和早期胚胎发育过程中的作用. 中国细胞生物学学报, 36(4): 521-529. (Hao G L, Yu H Q. 2014. The role of TFⅡD in gametogenesis and early embryonic development. Chinese Journal of Cell Biology, 36(4): 521-529. [in Chinese]) |
潘建伟, 赵章杏, 朱睦元, 等. 1998. TBP的联结因子TAFs的结构与功能. 遗传, 20(4): 39-41. (Pan J W, Zhao Z X, Zhu M Y, et al. 1998. Structure and functions of TBP-associated factors TAFs. Hereditas (Beijing), 20(4): 39-41. [in Chinese]) |
Albright S R, Tjian R. 2000. TAFs revisited:more data reveal new twists and confirm old ideas. Gene, 242(1): 1-13. |
Cler E, Papai G, Schultz P, et al. 2009. Recent advances in understanding the structure and function of general transcription factor TFⅡD. Cellular and Molecular Life Sciences, 66(13): 2123-2134. DOI:10.1007/s00018-009-0009-3 |
Dynlacht B D, Hoey T, Tjian R. 1991. Isolation of co-activators associated with the TATA-binding protein that mediate transcriptional activation. Cell, 66: 563-576. DOI:10.1016/0092-8674(81)90019-2 |
Furumoto T, Tamada Y, Izumida A, et al. 2005. Abundant expression in vascular tissue of plant TAF10, an orthologous gene for TATA box-binding protein-associated factor 10, in Flaveria trinervia and abnormal morphology of Arabidopsis thaliana transformants on its overexpression. Plant & Cell Physiology, 46(1): 108-117. |
Gangloff Y G, Romier C, Thuault S, et al. 2001. The histone fold is a key structural motif of transcription factor TFⅡD. Trends in Biochemical Sciences, 26(4): 250-257. DOI:10.1016/S0968-0004(00)01741-2 |
Gao X, Ren F, Lu Y T. 2006. The Arabidopsis mutant stg1 identifies a function for TBP-associated factor 10 in plant osmotic stress adaptation. Plant Cell Physiologist, 47(9): 1285-1294. DOI:10.1093/pcp/pcj099 |
Hiller M A, Lin T Y, Wood C, et al. 2001. Developmental regulation of transcription by a tissue-specific TAF homolog. Genes Development, 15: 1021-1030. DOI:10.1101/gad.869101 |
Lago C, Clerici E, Dreni L, et al. 2005. The Arabidopsis TFⅡD factor AtTAF6 controls pollen tube growth. Developmental Biology, 285(1): 91-100. DOI:10.1016/j.ydbio.2005.06.006 |
Lago C, Clerici E, Mizzi L, et al. 2004. TBP-associated factors in Arabidopsis. Gene, 342(2): 231-241. DOI:10.1016/j.gene.2004.08.023 |
Liu D F, Sui S Z, Ma J, et al. 2014. Transcriptomic analysis of flower development in wintersweet (Chimonanthus praecox). PLoS ONE, 9(1): e86976. DOI:10.1371/journal.pone.0086976 |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
Moraga F, Aquea F. 2015. Composition of the SAGA complex in plants and its role in controlling gene expression in response to abiotic stresses. Frontiers in Plant Science: 6. DOI:10.3389/fpls.2015.00865 |
Mougiou N, Poulios S, Kaldis A, et al. 2012. Arabidopsis thaliana TBP-associated factor 5 is essential for plant growth and development. Molecular Breeding, 30(1): 355-366. DOI:10.1007/s11032-011-9626-2 |
Muller C W. 2001. Transcription factors global and detailed views. Current Opinion Structural Biology, 11(1): 26-32. DOI:10.1016/S0959-440X(00)00163-9 |
Papai G, Weil P A, Schultz P. 2011. New insights into the function of transcription factor TFⅡD from recent structural studies. Current Opinion in Genetics & Development, 21(2): 219-224. |
Shen W C, Bhaumik S R, Causton H C, et al. 2003. Systematic analysis of essential yeast TAFs in genome-wide transcription and preinitiation complex assembly. European Molecular Biology Organization, 22(13): 3395-3402. DOI:10.1093/emboj/cdg336 |
Srivastava R, Rai K M, Pandey B, et al. 2015. Spt-Ada-Gcn5-Acetyltransferase (SAGA) complex in plants:genome wide identification, evolutionary conservation and functional determination. PLoS One, 10(8): e0134709. DOI:10.1371/journal.pone.0134709 |
Sui S Z, Luo J H, Ma J, et al. 2012. Generation and analysis of expressed sequence tags from Chimonanthus praecox (Wintersweet) flowers for discovering stress-responsive and floral development related genes. Comparative and Functional Genomics. DOI:10.1155/2012/134596 |
Tamada Y, Nakamori K, Nakatani H, et al. 2007. Temporary expression of the TAF10 gene and its requirement for normal development of Arabidopsis thaliana. Plant & Cell Physiology, 48(1): 134-146. |
Tanese N, Pugh B F, Tjian R. 1991. Coactivators for a proline-rich activator purified from the multisubunit human TFⅡD complex. Genes Development, 5: 2212-2224. DOI:10.1101/gad.5.12a.2212 |
Verslues P E, Agarwal M, Katiyar-Agarwal S, et al. 2006. Methods and concepts in quantifying resistance to drought, salt and freezing, abiotic stresses that affect plant water status. Plant Journal, 45(4): 523-539. DOI:10.1111/tpj.2006.45.issue-4 |
 2019, Vol. 55
2019, Vol. 55

