文章信息
- 马秋月, 戴晓港, 陈赢男, 张得芳, 廖卓毅, 李淑娴
- Ma Qiuyue, Dai Xiaogang, Chen Yingnan, Zhang Defang, Liao Zhuoyi, Li Shuxian
- 枣基因组的微卫星特征
- Characterization of Microsatellites in the Genome of Ziziphus jujuba
- 林业科学, 2013, 49(12): 81-87
- Scientia Silvae Sinicae, 2013, 49(12): 81-87.
- DOI: 10.11707/j.1001-7488.20131212
-
文章历史
- 收稿日期:2013-02-04
- 修回日期:2013-03-23
-
作者相关文章
微卫星(microsatellite),又称简单序列重复(simple sequence repeat,SSR),其侧翼区序列比较保守,核心区以1~6个碱基组成串联重复序列。微卫星普遍存在于真核生物和原核生物基因组中(Mrazek et al.,2007;Tóth et al.,2000),甚至在病毒基因组中(杨纪青等,2010),并且在编码区和非编码区都有分布(Li et al.,2004)。以往的研究还发现,SSR在非编码区的含量要比在编码区的含量多(Ellegren,2004;Qian et al.,2013)。微卫星在群体和不同个体间通常表现出很高的变异性,且按照孟德尔方式分离,具有多态性丰富、共显性遗传等特点,现已在基因组指纹图谱的构建、基因定位和克隆以及品种鉴定和进化研究等领域被广泛应用(Selkoe et al.,2006;Beheregaray et al.,2003)。SSR具有影响转录、基因调节、蛋白质功能以及基因组构建等功能(Kashi et al.,2006;Lawson et al.,2006)。
近年来,随着基因组测序技术的发展,越来越多的物种进行了大规模测序并对物种的基因组进行了微卫星分布特征分析(Tóth et al.,2000;Katti et al.,2001;Kong et al.,2005;Cruz et al.,2005),从而为在全基因组层面上分析基因组中SSR的分布、丰度等特性提供了可能。对不同物种中微卫星序列分析的结果表明,虽然微卫星广泛分布于真核、原核生物甚至病毒基因组中,但微卫星序列在不同物种的分布存在较大的差异,而且不同物种基因组间微卫星密度、碱基组成以及突变率也存在着较大差异(Oliveira et al.,2006)。研究微卫星序列在物种基因组中的丰度及其特征还有助于了解物种基因组的结构和进化(张学勇等,2000)。
枣(Ziziphus jujuba),是我国北方分布较为广泛的经济林树种,对气候、土壤的适应能力很强,且耐干旱、瘠薄,在76°—124°E,23°—42.5°N内的平原、沙地、盐碱地及高原地带均有分布。枣在中国被广泛地应用于食品、药材、园林绿化等诸多领域。近年来国内外学者利用SSR(麻利颖等,2012)、 RAPD(智福君等,2009)、 AFLP(王永康等,2007)等分子标记技术对枣的品种分类、鉴别以及遗传多样性方面开展了研究,但枣种属特异性的标记资源及基因组序列信息还比较匮乏,枣基因组水平上微卫星特征分析研究还未见报道。目前,苗木市场混乱,存在以假乱真现象,给苗农造成不必要的经济损失,已有很多研究者应用SSR分子标记技术对同一属种及不同品种间进行分子鉴定(张红莲等,2010;黄平等,2012)。本研究利用Roche-454 FLX高通量测序平台对枣进行低覆盖度基因组测序,并进行微卫星序列特征和组成分析,以期展开枣基因组微卫星特征分析研究,进而为我国枣品种鉴别以及遗传研究提供标记和序列资源,减少枣农因品种不明而带来的经济损失。
1 材料与方法 1.1 供试材料2012年春天在南京林业大学校园内采集新萌发的1株金丝小枣(Z. jujuba‘Jinsixiaozao’)的幼叶作为枣基因组测序的材料,采后立即置于便携式冰盒中带回实验室,置-80 ℃超低温冰箱中备用。
1.2 DNA提取基因组DNA的提取和提纯采用DNeasy PlantMini Kit(50)(上海玉博生物科技有限公司)的植物DNA提取试剂盒,具体为:取3~5片幼嫩叶片约1 g于2 mL的冻存管中,按照试剂盒的标准程序以及史洁等(2012)的方法操作,提取的DNA置于-20 ℃下保存备用。
1.3 枣基因组测序文库构建及测序利用Roche GS Rapid Library Preparation Kit构建基因组文库,取2 μg基因组DNA用TE稀释到100 μL,加入500 μL Nebulization buffer,混匀后在0.2 MPa的氮气压力下打断1 min,回收打断后的DNA,进行末端修复加A,连接接头后用AMPurebeads去除小片段。再用TBS-380荧光分光光度计对文库进行定量,根据定量结果用TE将文库稀释成1×10 7 molecules·μL-1,-20 ℃保存。用Agilent-2100生物分析仪测定文库质量,保证文库的长度在900~1 200 bp之间,小于350 bp的片段不超过总量的10%。采用emPCR试剂盒将文库中的DNA片段连接到珠子上,1个珠子连接1条DNA,然后进行油包水的PCR扩增,PCR扩增完成后回收含有DNA的珠子,并连接测序引物,用颗粒计数仪数取200万珠子用于上机测序。采用Roche-454 GS FLX测序仪进行测序,将PTP板分为2个区,测序为200个循环。测序后仪器将图像文件转换为sff文件,再利用Roche Newbler 2.8软件进行序列拼接。
1.4 分析方法采用Misa(www. pgrc. ipkgatersleben. de/misa)程序查找1~6碱基重复的微卫星,其中参数设置为:单碱基重复最短为10个重复,二碱基重复最短为6个重复,三碱基重复最短为4个重复,四碱基重复最短为3个重复,五碱基重复最短为3个重复,六碱基重复最短为2个重复。不同重复单元微卫星密度(D)的计算按照史洁等(2012)的方法。侧翼序列的查找通过自编Perl程序,在已设计好的引物中随机挑取100对,分别对这100对引物的核心序列SSR的左侧上游100 nt序列和右侧下游100 nt序列(其中上游或下游序列少于100 nt的SSR被剔除掉)进行GC含量分析,应用Bio-Edit 6.0.7软件对基因组侧翼序列比对分析,其中Expectation Value(E)设置为1.0E-3。
2 结果与分析 2.1 测序及拼接结果利用Roche-454 FLX高通量测序后,共测得203.3 Mb的枣基因组序列,片段总数为203 271 297 bp,平均读长为360 bp,通过RocheNewbler 2.7软件进行序列拼接和组装,产生266 870个组装序列及293 458个单一序列数(singleton assembled)。其中大于500 bp的contig有8 448个,contig的最大长度为84 716 bp,部分拼接数据见表 1。
|
|
Weber(1990)按照微卫星重复序列结构的不同,将其分为完整型SSR(perfect SSR)、不完整型SSR(imperfect SSR)以及复合型SSR(compound SSR)。完整型SSR一般是由1种串联重复序列以不间断的重复方式构成的单一重复类型的微卫星;不完整型SSR是指2个或2个以上的同种重复序列被3个或3个以下的非重复碱基分隔开;复合型SSR指2个或2个以上的串联核心序列被3个或者3个以上连续的非重复碱基所间隔,但这种连续性的核心序列重复数不得少于5。本研究对枣基因组中1~6碱基重复完整型和复合型进行分析,共获得15 036条枣微卫星重复序列,其中部分微卫星信息见表 2。
|
|
在枣基因组SSR数据库中,六碱基重复单元的SSR含量最多,约占总数的40.1%,之后为复合碱基(18.0%)、单碱基(17.1%)、四碱基(8.1%)、二碱基(7.5%)、三碱基(7.0%)、五碱基(2.2%)(表 3)。每种碱基重复单元包含不同种类的重复碱基,其中单碱基微卫星由2种不同的重复碱基组成,二碱基、三碱基、四碱基、五碱基、六碱基微卫星分别由12,53,133,100,1 814种组成,复合型碱基微卫星由2 707种不同重复组成。
|
|
对枣部分基因组覆盖度的序列进行微卫星查找,从总长为8 333 952 bp的8 448个重叠中发现15 036个微卫星。从微卫星的分布密度来看,枣基因组中平均每554.3 bp出现1个微卫星。由于不同重复单元微卫星数量差异较大,所以分布密度变化也很大(表 3)。
2.4 优势重复单元碱基在枣基因组微卫星中的组成对枣不同类型重复单元微卫星中各重复单元数量的变化情况进行统计发现:单碱基重复微卫星中,A/T为最主要的重复单元,占99.1%。在二碱基重复类型中,AT/TA重复的数量最多,共506个,占73.26%;其次为AG/TC,66个,占10.20%。在10种三碱基重复类型中,AAT/ATT数量最多,共504个,占48.0%;其次为AAG/CTT(23.9%)和ATC/ATG(10.5%),其他重复碱基则相对较少。四碱基重复类型共有60种,AAAT/ATTT数量最多,共632个(51.9%),其次为AATT/TTAA(13.1%)、AAAG/CTTT(12.2%),这3种类型约占四碱基重复类型总量的80%。78种五碱基重复类型中,AAAAT/ATTTT的数量最多,共157个(46.9%),其次为AAAAG/CTTTT(18.2%)。325种六碱基重复类型中,AAAAAT/TAAAAA数量较多,共663个(11.0%),其次为AAAAAG/CTTTTT,共413个(6.8%)。
上述分析结果还表明,枣基因组中最丰富的微卫星类型是六碱基重复,主要优势序列为AAAAAT/TAAAAA、 AAAAAG/CTTTTT;五碱基重复丰度最低,主要优势序列为AAAAT/TAAAA、AAAAG/CTTTT。同样在二、三、四、五碱基重复类型中,(AN)n、(AAN)n、(AAAN)n和(AAAAN)n分别比其他碱基组成的微卫星数量大些。这些优势重复拷贝类型均富含A和T碱基。但在枣基因组的不同碱基重复类型中,(GC)n含量仅占0.18%,在三碱基中发现1个(CGG)5和1个(GGC)4,四碱基中发现1个(GCCG)3以及五碱基的1个(GGCGG)3。
2.5 枣基因组微卫星长度分布及变异分析本研究中枣微卫星的平均长度为26.6 bp,最长的为577 bp,最短的为10 bp(图 1)。从图 1中可以看出,微卫星长度为12 bp所占比例最多(54.02%),长度大于20 bp的微卫星仅占23.02%对不同重复微卫星类型中的重复长度进行统计分析以进一步了解不同长度重复单元微卫星长度的变异情况(图 2)。微卫星长度的变异情况表示微卫星获得或失去重复单元速率的快慢程度。图 2的结果表明:二碱基重复微卫星重复单元的变化次数最高,单碱基重复微卫星次之,变异程度最低的为五碱基重复微卫星,仅有3种不同微卫星变化长度。每种重复微卫星类型都随着重复长度的递增,微卫星丰度呈递减趋势,即微卫星长度越长,微卫星丰度越低。
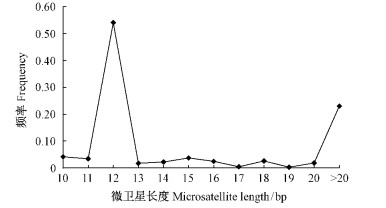 |
图 1 枣不同长度微卫星出现频率
Fig. 1 Frequency of microsatellites in different lengths
|
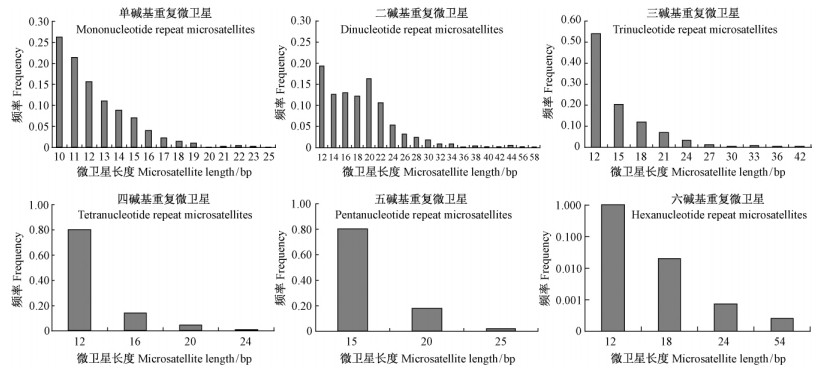 |
图 2 不同长度重复单元微卫星变异情况
Fig. 2 Length diversification of the microsatellites
柱状图中每一条形对应不同长度微卫星。 In above chart,each column corresponds to microsatellites in particular length. |
从已设计好的引物中随机挑取100对,分别对这些引物的核心序列SSR的左侧上游100 nt序列和右侧下游100 nt进行GC含量分析,对满足条件的76个SSR进行分析发现,左侧与右侧序列GC含量相差不大,左侧GC总平均含量占33.2%(16% ~53%),右侧占33.3%(17%~51%)。利用Bio-Edit6.0.7软件(Hall,1999)对成对的侧翼序列与枣树所测基因组序列进行比对分析发现,左侧序列与右侧序列查询覆盖率达到100%,左侧序列平均期望值为8.96E-5(2.00E-7~5.00E-4),最大得分50.1,最小得分38.2,匹配率为100%;右侧序列平均期望值为0.14E-7(2.00E-8~8.00E-4),最大得分54,最小得分38.2,最大匹配率100%,最小匹配率95.65%。在分析的100对引物中,86对引物为单拷贝,且SSR区域只存在于侧翼序列之间,所以约有86%的引物能够特异扩增出含有相应SSR序列的位点。而另有14对引物为多拷贝,这些侧翼序列在非SSR区域也同时存在,这些引物会扩增出不含SSR序列的位点。
3 讨论 3.1 枣优势重复碱基类型分析本研究利用Roche-454 FLX高通量测序平台对枣进行了低覆盖度基因组测序,并利用Misa软件在枣全基因组总长为8 333 952 bp的8 448个重叠中发现了15 036个微卫星。从微卫星的分布密度来看,平均每554.3 bp出现1个微卫星,分布密度为1 804.2 Mb-1。对测得的基因组分析发现:六碱基重复微卫星出现的频率(40.1%)明显高于其他类型,之后依次为复合碱基(18.0%)、单碱基(17.1%)、四碱基(8.1%)、二碱基(7.5%)、三碱基(7.0%)、五碱基(2.2%)。史洁等(2012)研究发现,二碱基重复微卫星为油茶(Camellia spp.)基因组的最优重复类型。郑燕等(2011)对水稻(Oryzasativa)、高粱(Sorghum bicolor)、二穗短柄草(Brachypodium distachyon)、玉米(Zea mays)4种禾本科植物进行基因组微卫星序列分析发现,水稻、高粱的优势重复碱基类型为三碱基重复,而二穗短柄草和玉米则为六碱基重复微卫星,与本研究中枣的优势碱基重复类型相同。由此可见微卫星优势类型在不同物种间的分布有较大差异。不同物种的基因组大小不同,碱基组成含量不同,因而微卫星丰度也会不同。有研究报道基因组越小,单碱基重复序列越多,随着物种基因组的增大,其他类型微卫星逐渐占主导地位(Karaoglu et al.,2005)。
3.2 枣优势重复单元碱基组成分析Tóth等(2000)认为微卫星的形成可能与DNA复制滑脱、核酸交替以及重组不平衡有一定的关系;Biet等(1999)认为微卫星重复序列,如CA、 GA、 GT等重复可以通过影响DNA的结构而影响DNA重组。因此微卫星中的重复单元碱基组成在很大程度上会影响生物的生命活动。本研究中,枣不同重复碱基类型优势重复单元的共同特点是富含AT碱基(单碱基重复微卫星占99.1% 、二碱基占73.26% 、三碱基占48.0% 、四碱基占51.9% 、五碱基占46.9% 、六碱基占11.0%)。以往的研究发现,不同植物中优势重复单元有所不同: Echt等(1997)比较北美乔松(Pinus strobus)和火炬松(P. taeda)基因组微卫星时发现,(AC)n、(AG)n、(AAT)n、(ATC)n是最丰富的4种重复单元;油茶基因组中单、三、四碱基重复类型中AT碱基为最优重复单元(史洁等,2012)。大多数植物中(AG)n含量要比(AC)n高(Akkaya et al.,1992;Wang et al.,1994),在枣基因组微卫星的分析中亦如此,(AG)n所占比例约为(AC)n的2倍,但在水稻和玉米中却截然相反(Wu et al.,1993;Panaud et al.,1995)。枣基因组的不同碱基重复类型中,(GC)n含量仅占0.18%,在三碱基中发现1个(CGG)5和1个(GGC)4,四碱基中发现1个(GCCG)3,五碱基也有1个(GGCGG)3。Katti等(2001)、 Tóth等(2000)的研究也发现GC两碱基的重复拷贝类别在所研究的生物基因组中的含量也很少,有研究者对脊椎动物基因组分析认为可能是由于基因组DNA中CpG岛的甲基化造成的(Schorderet et al.,1992),甲基化的胞苷酸C很容易经过脱氨基作用转变成胸腺嘧啶T,同时基因组DNA中CpG的甲基化会成为突变的热点,而且少量的GC又是维持DNA热力学稳定性所必需的,这可能是导致GC含量偏少的原因。
造成不同植物基因组中不同重复碱基类型及重复单元偏好性的原因除了与不同物种间的真实微卫星信息差异有关外,可能还与不同微卫星查找工具中的参数设置有一定关系(阎毛毛等,2011),另外所测基因组的覆盖度的深浅也会影响微卫星的分析。
3.3 枣不同长度重复单元微卫星长度变异情况分析本文对枣不同重复类型碱基长度的变异情况分析发现,随着微卫星重复单元长度的不断增加,其丰度普遍呈递减的趋势(二碱基变异频率除外)。本研究中枣微卫星的平均长度为26.6 bp,最长的为577 bp,最短的为10 bp。从图 1中可以看出,微卫星长度为12 bp所占比例最多(54.02%),长度大于20 bp的微卫星仅占23.02%。由于不同类型微卫星序列获得或失去重复单元的速率可以从序列长度的变化情况得以反映,所以这一特征与微卫星位点的多态性有密切关系。Samadi等(1998)的模拟分析认为,重复单元长度变化与选择压力有关,重复单元长度越长,所受的选择压力越大,拷贝数就越少,因此基因组中长度较短的微卫星变异速率较快,而较长的重复单元变异速率较慢,相对较为稳定。本研究中六碱基为枣最优势微卫星,所占比例最多,但值得关注的是二碱基微卫星重复单元次数的变化显著高于其他重复类型,这在一定程度上说明二碱基在整个枣基因组中变异最为活跃。
3.4 枣基因组微卫星侧翼序列分析对枣基因组微卫星侧翼序列的分析发现,左侧与右侧序列GC含量相差不大,左侧GC总平均含量占33.2%(16%~53%),右侧占33.3%(17% ~51%),这在一定程度上也说明基因组中对AT的偏好性。同时利用Bio-Edit 6.0.7软件对随机挑选的100对引物序列与枣树所测基因组序列进行比对分析,发现左侧序列与右侧序列查询覆盖率均达到100%。但这100对引物中有86对具有特异性(即SSR区域只存在于特定的2个侧翼序列之间),发现非SSR区域也可以同时存在这样的2个侧翼序列,因此如果依据这样的侧翼序列设计引物,所得到的PCR扩增产物中就会有非SSR产物,因此在利用SSR进行基因定位时,要考虑产物的假阳性可能(詹少华等,2010)。Jeffreys等(1985)认为SSR是高等生物在减数分裂过程中由于DNA滑动复制以及不对等交换所产生的。这只能解释高度保守的SSR侧翼序列的重复基元所具有的多态性,对于在非SSR区域存在SSR侧翼序列或者SSR具有多种侧翼序列的现象无法解释,说明SSR的产生也可以通过易位等方式产生。因此在常规的SSR引物设计中,缺乏对SSR侧翼序列保守性的分析,而通过常规试验来进行筛选,工作耗时又存在一定的误差。本研究中利用序列比对分析,可以为后期引物的开发提供一种可靠而快速的方法。
3.5 微卫星技术在枣研究中的应用前景微卫星序列对基因的功能有重要影响,序列特征是了解不同物种基因组差异的重要指标,并且微卫星是生物基因组中变异频率最快的序列,含有微卫星的基因比不含微卫星的基因更容易产生突变,因此,虽然目前本研究中所得到的枣基因组SSR序列得到应用,但对以后该物种微卫星标记资源的开发以及物种进化、遗传多样性等方面的研究将提供重要信息资源。本研究还将对开发枣多态性高的微卫星标记提供帮助,从而为枣品种的分子鉴别打下基础。
| [1] |
黄平,崔鹏娇,郑勇奇,等.2012.基于SSR标记月季品种鉴定及遗传关系分析.林业科学,48(10): 55-62.( 1) 1)
|
| [2] |
麻丽颖,孔德仓,刘华波,等.2012.36份枣品种SSR指纹图谱的构建.园艺学报,39(4): 647-654.( 1) 1)
|
| [3] |
史洁,尹佟明,管宏伟,等.2012.油茶基因组微卫星特征分析.南京林业大学学报:自然科学版,36(2): 47-51.( 4) 4)
|
| [4] |
王永康,田建保,王永勤,等.2007.枣树品种品系的AFLP分析.果树学报,24(2): 146-150.( 1) 1)
|
| [5] |
阎毛毛,戴晓港,李淑娴,等.2011.松树、杨树及桉树表达基因序列微卫星比对分析.基因组学与应用生物学,30(1): 103-109.( 1) 1)
|
| [6] |
杨纪青,杨硕,杨莉,等.2010.Tomato leaf curl Bangladesh病毒完整基因组上微卫星分布.中国农学通报,26(24): 20-27.( 1) 1)
|
| [7] |
詹少华,盛新颖,樊洪泓,等.2010.大豆二核苷酸SSR侧翼序列保守型分析.大豆科学,29(2): 195-198.( 1) 1)
|
| [8] |
张红莲,李火根,胥猛,等.2010.鹅掌楸属种及杂种的SSR分子鉴定.林业科学,46(1): 36-39.( 1) 1)
|
| [9] |
张学勇,李大勇.2000.小麦及其近亲基因组中的DNA重复序列研究进展.中国农业科学,33(5): 14-24.( 1) 1)
|
| [10] |
郑燕,张耿,吴为人.2011.禾本科植物微卫星序列的特征分析和比较.基因组学与应用生物学,30(5): 513-520.( 1) 1)
|
| [11] |
智福君,贾彦丽,梁海永,等.2009.利用RAPD技术进行枣树的品种鉴定.华北农学报,24(增刊): 110-114.( 1) 1)
|
| [12] |
Akkaya M S,Bhagwat A A,Cregan P B.1992.Length polymorphisms of simple sequence repeat DNA in soybean.Genetics,132(4): 1131-1139.( 1) 1)
|
| [13] |
Beheregaray L B,Ciofi C,Geist D,et al.2003.Genes record a prehistoric volcano eruption in the Galpagos.Science,302(5642): 75.( 1) 1)
|
| [14] |
Biet E,Sun J S,Dutreix M.1999.Conserved sequence preference in DNA binding among recombination proteins: an effect of ssDNA secondary structure.Nucleic Acids Research,27(2): 596-600.( 1) 1)
|
| [15] |
Cruz F,Pérez M,Presa P.2005.Distribution and abundance of microsatellites in the genome of bivalves.Gene,346: 241-247.( 1) 1)
|
| [16] |
Echt C S,May-Marquardt P.1997.Survey of microsatellite DNA in pine.Genome,40: 9-17.( 1) 1)
|
| [17] |
Ellegren H.2004.Microsatellites: simple sequences with complex evolution.Nature Reviews Genetics,5: 435-445.( 1) 1)
|
| [18] |
Hall T A.1999.BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT.Nucleic Acids Symp Ser,41: 95-98.( 1) 1)
|
| [19] |
Jeffreys A J,Wilson V,Thein S L.1985.Hypervariable 'minisatellite' regions in human DNA.Nature,314(7): 67-73.( 1) 1)
|
| [20] |
Karaoglu H,Lee C M Y,Meyer W.2005.Survey of simple sequence repeats in completed fungal genomes.Molecular Biology and Evolution,(22): 639-649.( 1) 1)
|
| [21] |
Kashi Y,King D,Soller M.2006.Simple sequence repeats as a source of quantitative genetic variation.Trends in Genetics,13(2): 74-78.( 1) 1)
|
| [22] |
Katti M V,Ranjekar P K,Gupta V S.2001.Differential distribution of simple sequence repeats in eukaryotic genome sequences.Molecular Biology and Evolution,18(7): 1161-1167.( 2) 2)
|
| [23] |
Kong J,Gao H.2005.Analysis of tandem repeats in the genome of Chinese shrimp Fenneropenaeus chinensis.Chinese Science Bulletin,50(14): 1462-1469.( 1) 1)
|
| [24] |
Lawson M J,Zhang L.2006.Patterns of SSR distribution in the Arabidopsis thaliana and rice genomes.Genome Biology,7: R14.( 1) 1)
|
| [25] |
Li C Y,Korol A B,Fahima T,et al.2004.Microsatellites within genes: structure,function,and evolution.Molecular Biology and Evolution,21: 991-1007.( 1) 1)
|
| [26] |
Mrazek J,Guo X,Shah A.2007.Simple sequence repeats in prokaryotic genomes.PNAS,104: 8472-8477.( 1) 1)
|
| [27] |
Oliveira E J,Pádua J G,Zucchi M I,et al.2006.Origin,evolution and genome distribution of microsatellites.Genetics and Molecular Biology,29(2): 294-307.( 1) 1)
|
| [28] |
Panaud O,Chen X,McCouch S R.1995.Frequency of microsatellite sequences in rice(Oryza sativa L.).Genome,38: 1170-1176.( 1) 1)
|
| [29] |
Qian J,Xu H B,Song J Y,et al.2013.Genome-wide analysis of simple sequence repeats in the model medicinal mushroom Ganoderma lucidum.Gene,512: 331-336.( 1) 1)
|
| [30] |
Samadi S,Artiguebielle E,Estoup A,et al.1998.Density and variability of dinucleotide microsatellites in the parthenogenetic polyploid snail Melanoides tuberculata.Molecular Ecology,7(9): 1233-1236.( 1) 1)
|
| [31] |
Schorderet D F,Gartler S M.1992.Analysis of CpG suppression in methylated and nonmethylated species.PNAS,89(3): 957-961.( 1) 1)
|
| [32] |
Selkoe K A,Toonen R J.2006.Microsatellites for ecologists: a practical guide to using and evaluating microsatellite markers.Ecology Letters,9(5): 615-629.( 1) 1)
|
| [33] |
Tóth G,Gáspári Z,Jurka J.2000.Microsatellites in different eukaryotic genomes: survey and analysis.Genome Research,10(7): 967-981.( 4) 4)
|
| [34] |
Wang Z,Weber J L,Tanksley S D.1994.Surey of plant short tandem DNA repeats.Theoretical and Applied Genetics,88(1): 1-6.( 1) 1)
|
| [35] |
Weber J L.1990.Informativeness of human(dC-dA)n·(dG-dT)n polymorphisms.Genomics,7(4): 524-530.( 1) 1)
|
| [36] |
Wu K S,Tanksly S D.1993.Abundance,polymorphism and genetic mapping of microsatellites in rice.Molecular and Genetics,241: 225-235.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

