文章信息
- 徐莉莉, 童再康, 林二培, 黄华宏
- Xu Lili, Tong Zaikang, Lin Erpei, Huang Huahong
- 杉木苯丙氨酸解氨酶基因ClPAL的克隆与表达分析
- Isolation and Expression of ClPAL Genes in Chinese Fir (Cunninghamia lanceolata)
- 林业科学, 2013, 49(12): 64-72
- Scientia Silvae Sinicae, 2013, 49(12): 64-72.
- DOI: 10.11707/j.1001-7488.20131210
-
文章历史
- 收稿日期:2013-02-05
- 修回日期:2013-09-11
-
作者相关文章
木质素(lignin)是苯丙烷衍生物的单体构成的聚合物,占木材干质量的15%~35%(Zhong et al.,2000),对木材的形成和品质具有重要影响。因此,木质素的生物合成已是当前木材形成分子机制解析的主要内容之一。木质素的合成须经历苯丙烷类途径和木质素单体特异途径。近十几年来,随着分子生物学的快速发展,许多参与其中的酶编码基因被不断地分离和克隆出来(Boerjan et al.,2003;Li et al.,2006)。苯丙氨酸解氨酶(phenylalanineammonia-lyase,PAL)催化苯丙氨酸脱氨生成肉桂酸,即木质素、黄酮类和水杨酸盐等的前体,它是植物苯丙烷类途径的第1个限速酶,是主代谢和次生代谢间的重要调控位点。迄今为止,多个来自拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)、水稻(Oryza sativa)、北美短叶松(Pinus banksiana)、火炬松(Pinus taeda)和欧洲颤杨(Populus tremuloides)等植物的PAL编码基因已被克隆鉴定(Cochrane et al.,2004;Nagai et al.,1994;Minami et al.,1989;Butland et al.,1998;Whetten et al.,1992;Bagal et al.,2012;Kao et al.,2002)。植物PAL基因家族一般由2~6个成员组成,且不同成员表现出不同的表达模式和代谢功能。拟南芥基因组中含有4个PAL,突变体分析发现这4个基因存在一定的功能冗余,其中PAL1在参与苯丙烷类化合物合成方面比PAL2具有更重要的作用,值得注意的是pal1 pal2双缺突变体和pal1 pal2 pal3 pal4四缺突变体花序茎中木质素含量分别仅为野生型的25%和30%左右(Rohde et al.,2004;Huang et al.,2010)。研究发现来自欧洲颤杨的PtrePAL1主要在茎、叶、根的积累单宁的非木质化细胞中表达,且随木质化程度增加而下降;PtrePAL2主要在茎的木质化细胞和由非木质化细胞组成的根尖中表达(Kao et al.,2002)。可见,PAL活性高低也是影响木质素含量的关键因子之一。
杉木(Cunninghamia lanceolata)是我国重要的针叶速生用材树种,现阶段材性的改良是其主要育种目标之一。然而,在材性育种方面国内育种者虽以杉木木材密度、色泽等性状改良为目标开展了一些常规育种工作,但涉及木材性状形成分子机制的研究相对较少。如,以独干杉(Cunninghamia lanceolata var. duganshan)为材料建立相关的cDNA文库和EST数据库,并对扩展蛋白基因ClEXPA1和ClEXPA2进行克隆和异源转化烟草的研究(王桂凤等,2007;Wang et al.,2007;2011);利用高通量测序技术建立含8万余条序列的杉木EST数据库,并对参与纤维素和木质素合成的部分基因进行初步的表达分析(Huang et al.,2012)。因此,本研究以杉木速生无性系B057为材料,分离ClPAL基因序列,并分析其在不同组织和非生物胁迫处理条件下的表达特性,探讨其在木质素合成中的功能,以期为后续以材性改良为目标的分子辅助育种提供重要基因资源。
1 材料与方法 1.1 植物材料试验材料为杉木速生无性系B057,栽植在浙江农林大学实验苗圃,共10种器官或组织被采集,取样时间为4月初至6月底。其中雌球花(femalecone,FC)、雄球花(male cone,MC)取自树冠上部枝条,根取自同一无性系的水培植株。叶为成熟针叶。茎取自树冠上部的当年生枝条,分为2段,分别标记为Stem1和Stem2。经解剖观测,Stem1的木质化区域很少,可认为是非木质化茎,Stem2为木质化茎,具体形态见图 1。在1年生茎段的皮层一侧刮取韧皮部,而剥皮后在木质部一侧刮取厚2~3 mm的木质部。另外,在11月截取同一无性系6年生植株胸径部位主干,依据生长年轮分别取边材(sapwood,SW)和中间材(intermediate wood,IW)。所有样品采集后立即放入液氮中冻存备用。
 |
图 1 试验中使用的植物材料
Fig. 1 Plant materials in the experiments
标尺 Scale A: 100 mm; B-D,G,H: 10 mm; E,F: 60 μm. A: B057 无性系; B: 雌雄球花; C: 根; D: 当年生枝条(1: 未木质化; 2: 木质化); E,F: 未木质化的茎和木质化的茎的横切片; G: 皮和木质部; H: 针叶。 A: Chinese Fir clone B057; B: Male and female cone; C: Root; D: Current-year branches(1: Non-lignified stem; 2: Lignified stem); E,F: Transverse sections of non-lignified and lignified stem; G: Bark and xylem; H: Leaf. |
茎段的激素处理参照Karpinska等(2004)的方法。在6月底采集树冠上部的当年生枝条,将粗细相对一致的木质化茎段截成8 cm段,直立放入50 mL旋盖离心管中(每离心管放1段)。将不同的处理溶液加入离心管,使浸没茎段,并真空渗透处理12 h。处理溶液分别为含500 μmol·L-1吲哚-3-乙酸(IAA)的1/2MS、含5 μmol·L-1赤霉素3(GA3)的1/2 MS和含0.5 μmol·L-1油菜素内酯(BR)的1/2MS,以1/2MS溶液为对照。每种处理(含对照)重复3次,结束后分别刮取韧皮部和木质部,并立即放入液氮中冻存。
1.3 应压木诱导处理采用拉弯处理诱导应压木,选取生长相对一致的杉木无性系植株,将其拉弯与垂直方向成45°角。在处理1,6和10天时,截取拉弯部位主干,分别刮取应压区和对应区木质部,对应样品记为CW1d,OW1d,CW6d,OW6d,CW10d和OW10d,并迅速放入液氮中冻存,以未处理的直立植株为对照(CK)。每次截取6株(其中3株为对照)。
1.4 总RNA提取及cDNA第1链的合成用PureLinkTM Plant RNA Reagent(上海英骏生物技术有限公司)提取样品总RNA。具体提取过程按照试剂盒说明书进行。按PrimeScriptTM RTReagent Kit(TaKaRa)说明将杉木总RNA逆转录合成cDNA第1链,作为模板待用。
1.5 ClPAL序列的分离在杉木转录组测序基础上,设计5'和3' RACE引物。按照SMARTTM RACE cDNA Amplification Kit(Clontech)说明书进行RACE反应。将得到的目的片段连接在PMD® 19-T Simple Vector(TaKaRa)上,转化大肠杆菌(Escherichia coli)DH5α,筛选阳性克隆后送华大基因公司测序。同时,为验证序列的正确性,在基因的5' UTR和3' UTR设计引物,进行PCR和克隆测序。
1.6 生物信息学分析序列比对应用NCBI数据库中的BLASTX,利用DNASTAR软件包进行开放阅读框(open readingframe,ORF)确定,氨基酸序列的推测,以及蛋白质的分子质量、等电点的预测。运用InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)数据库推测蛋白质结构域。蛋白质跨膜区预测、信号肽和二级结构分析分别采用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/),SignalP 4.0 Server(http://www.cbs.dtu.dk/services/SignalP/)和SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa.automat.pl?page=/NPSA/npsa_sopma.html)工具进行。利用MEGA 4.0的邻接(Neighbor Joining,NJ)算法构建PAL蛋白的系统进化树(Tamura et al.,2007)。
1.7 基因表达模式分析采用荧光定量PCR分析PAL基因在各样品中的相对表达量。使用购自TaKaRa公司的定量PCR专用的PrimeScript® Reagent Kit和SYBR® PremixEx TaqTM(Tli RNaseH Plus)试剂盒,持家基因Actin作为内参。
2 结果与分析 2.1 3个ClPAL基因的克隆及序列分析在转录组测序(Huang et al.,2012)的基础上,3个ClPAL基因被成功克隆,相应GenBank登录号分别为: JQ904042,JQ904043,JQ904044。3个克隆基因的全长在2 418~2 689 bp间变化,对应ORF皆大于2 110 bp,ORF序列间的一致性在59%~64%间变化,而与对应最高相似度的火炬松PtPAL序列间的一致性则在73%~76%间变化,说明克隆到的3个ClPAL是该家族的不同成员,而非等位变异或同一基因的不同拷贝。推测的3个ClPALs蛋白分别由709,710和709个氨基酸组成,分子质量为77.18~77.50 kDa,理论等电点(pI)分别为6.09,6.35和6.22。氨基酸序列间的一致性变幅为59%~67%,而它们与相应具最高相似度的火炬松PtPAL氨基酸序列间的一致性达68%~77%(表 1)。
|
|
通过与来自拟南芥、毛果杨(Populus trichocarpa)和火炬松等植物中已鉴定的PAL进行氨基酸序列比对和基序分析,发现3个ClPAL蛋白与来自其他植物的PAL蛋白一样都含有PAL-HAL(histidine ammonia-lyase)和PAL功能域,且这2个功能域均含有该类酶的1个酶活性中心基序,即G-[STG]-[LIVM]-[STG]-[AC]-S-G-[DH]-L-x-P-L-[SA]-x(2)- [SAV](图 2)。PAL蛋白的酶活性中心还含有1个高度保守的三肽Ala-Ser-Gly,它能通过环化作用和消除2分子水生成亲电子辅基3,5-二氢-5-次甲基-4H-咪唑-4-酮(MIO)(Ritter et al.,2004)。
 |
图 2 ClPALs与同源基因的编码区氨基酸序列比对
Fig. 2 Alignment of the deduced amino acid sequences of ORF of ClPALs and homologous genes
At: 拟南芥 Arabidopsis thaliana; Nt: 烟草 Nicotiana tabacum; Ptr: 毛果杨 Populus trichocarpa; Pm: 马尾松 Pinus massoniana; Pt: 火炬松 Pinus taeda.圆点 Circle: MIO; 下划线 Underline: 酶活中心 Enzymatic activity center. |
为了分析ClPALs与其他物种之间的系统进化关系,将来自8种裸子植物、 7种单子叶植物和7种双子叶植物的共33个全长PAL氨基酸序列进行比对和构建系统进化树(图 3)。从拓扑结构看,构建的进化树可明显分为3个主分支,即来自裸子植物、单子叶植物和双子叶植物的PAL分别聚类为一支,暗示在裸子植物与被子植物或单子叶植物与双子叶植物分化之前,多数PAL可能已进化产生了不同的类型。在裸子植物分支中,ClPAL1与来自银杏(Ginkgo biloba)的PAL序列最为相似,并与火炬松PtPAL1、马尾松(Pinus massoniana)PmPAL、海岸松(Pinus pinaster)PpPAL1聚为一个亚组;ClPAL2与PtPAL3亲缘关系最近,并进一步与松叶蕨(Psilotum nudum)PnPAL聚为一个亚组;而ClPAL3则与PtPAL4、 PtPAL5聚类在一起。
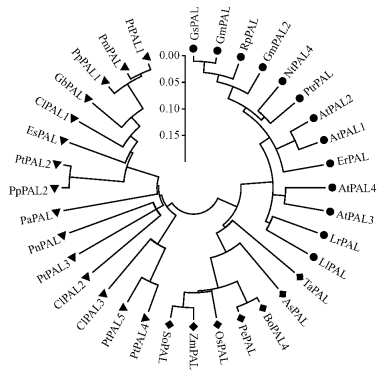 |
图 3 基于不同物种ClPAL氨基酸序列构建的分子系统树
Fig. 3 Phylogenetic tree derived from the alignment of the deduced amino acid sequences of different species
At: 拟南芥 Arabidopsis thaliana; Nt: 烟草 Nicotiana tabacum; Gs: 野大豆 Glycine soja; Gm: 大豆 Glycine max; Er: 大叶桉 Eucalyptus robusta; Ll: 麝香百合 Lilium longiflorum; Lr: 石蒜 Lycoris radiata; Ptr: 毛果杨 Populus trichocarpa; Rp: 刺槐 Robinia pseudoacacia; Gb: 银杏 Ginkgo biloba; Pm: 马尾松 Pinus massoniana; Pp: 海岸松 Pinus pinaster; Pt: 火炬松 Pinus taeda; Es: 草麻黄 Ephedra sinica; Pn: 松叶蕨 Psilotum nudum; Pa: 欧洲蕨 Pteridium aquilinum; Cl: 杉木 Cunninghamia lanceolata; Zm: 玉米 Zea mays; As: 燕麦 Avena sativa; Os: 水稻 Oryza sativa; Bo: 绿竹 Bambusa oldhamii; Pe: 毛竹 Phyllostachys edulis; So: 甘蔗 Saccharum officinarum; Ta: 小麦 Triticum aestivum.▲: 裸子植物 Gymnosperm; ●: 双子叶植物 Dicotyledon; ◆: 单子叶植物 Monocotyledon. |
首先利用RT-PCR、定量PCR产物的融解曲线分析和克隆测序等方法,对设计的3对ClPAL定量PCR引物的特异性和正确性进行验证,结果发现扩增特异且产物大小、序列正确,可用于后续定量PCR分析。图 4A为检测的ClPAL基因在6种不同器官中的表达模式(以叶做参照)。ClPAL1在6个不同的器官中都有表达,木质化茎中的表达量最高,其次是在针叶、雌球花和根中。ClPAL2基因在针叶中的表达丰度最高,其次是雌球花,在茎、雄球花和根中的表达量皆小于0.1。ClPAL3在木质化茎中的表达丰度最高,在针叶、雄球花和根中的表达量中等,在雌球花和非木质化茎中的表达量最低。为了进一步研究ClPAL基因与木质素生物合成之间的关系,取木质化茎的皮层和木质部进行基因表达量的对比分析,结果见图 4B。ClPAL1在木质部的表达量是皮层的10.1倍以上;而ClPAL2主要在皮层表达,其表达量约是木质部的8.3倍。值得注意的是,ClPAL3在木质部中的表达量接近皮层的120倍,可能主要在木质化组织中表达。以来自6年生主干的边材和中间材(心边过渡区)RNA为模板的定量PCR结果(图 4C)表明,ClPAL1,ClPAL2与ClPAL3的表达模式相似,都表现为中间材中的表达量明显高于边材中的相应数值,相应的差异倍数分别为20.7,3.6和144.7。
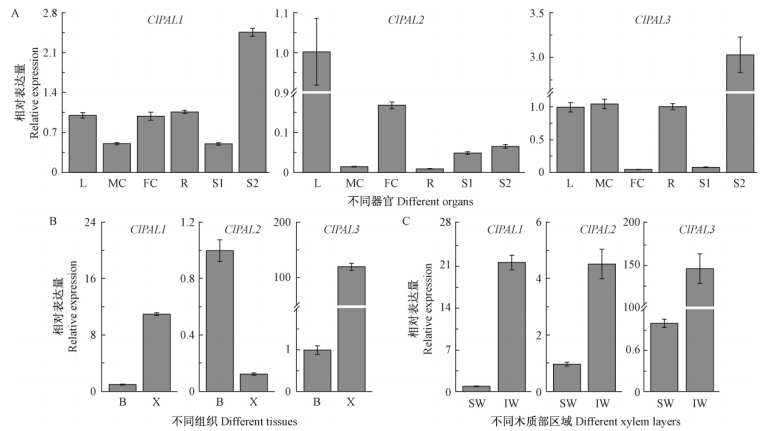 |
图 4 不同器官和组织中ClPALs基因的相对表达量
Fig. 4 Relative expression levels of ClPALs in different organs and tissues
(A) L: 针叶 Leaf; MC: 雄球花 Male cone; FC: 雌球花 Female cone; R: 根 Root; S1: 未木质化的茎 Non-lignified stem; S2: 木质化的茎 Lignified stem. (B) B: 树皮 Bark; X: 木质部 Xylem. (C) SW: 边材 Sapwood; IW: 中间材 Intermediate wood. |
吲哚乙酸(IAA)、赤霉素(GA)和油菜素内酯(BR)等是调节植物生理反应的活性物质,在形成层的生长和次生维管组织的形成中具有重要作用(Ursache et al.,2012)。因此,本研究分析了IAA、GA3和BR对木质部中ClPALs基因表达的影响(图 5)。经IAA、 GA3和BR处理12 h后,ClPAL1的表达受到了不同程度的诱导,其中BR处理后最为明显,表达量提高了2.1倍以上;同样ClPAL3的表达量也得到了提高,且受IAA和BR的诱导均非常明显,相应数值是对照的3.1和4.5倍。然而,对于ClPAL2基因,经这3种激素处理后,其表达则受到相对弱的抑制,与对照的平均差值在0.18~0.32间变化。
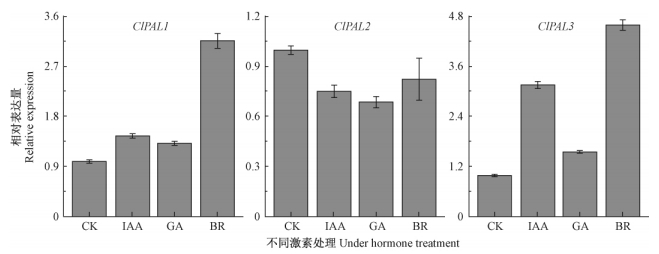 |
图 5 激素处理后木质部中ClPALs基因的相对表达量
Fig. 5 Relative expression levels of ClPALs in the xylems after treatment with different hormones
|
应力木是指在外力作用下造成的弯曲树干或树枝试图恢复到原来位置,形成的解剖、物理力学性质明显不同的木材(IAWA Committee,1989)。在裸子植物中,应力木通常出现在倾斜树干或树枝的下部,被称为应压木。相比正常木,应压木的管胞壁厚、管胞长度短(Onaka,1949;Wardrop et al.,1950)、微纤丝角大且木质素含量高(Wilson et al.,1977;Timell,1986)。为了探讨ClPALs在应压木形成中的作用,试验中应用荧光定量PCR分析其在拉弯处理不同时间应压区和对应区木质部中的表达变化(图 6)。ClPAL1在应压区木质部的表达量表现为先下降后上升,拉弯处理6天时为对照的0.5倍,第10天的相应数值是对照的1.5倍;而在对应区木质部中的表达呈现下调,处理第10天后的表达量为0.5。这能部分解释杉木应压木和对应木中木质素含量的变化趋势。在拉弯处理过程中,ClPAL2在应压区木质部中表现为上调表达,第10天的表达量为对照的2.0倍;而在对应区木质部中,仅第10天的表达量与对照间有明显差异,为1.7。另外,经拉弯处理后,ClPAL3的表达量变化不是很明显,与对照的相应差值变幅仅在0.2~0.4间。
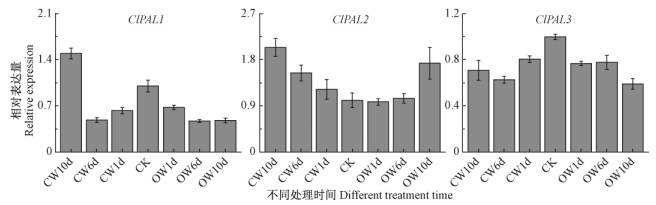 |
图 6 应压木形成中ClPALs的相对表达量
Fig. 6 Relative expression levels of ClPALs during the formation of compression wood
CW1d,CW6d 和 CW10d 分别指拉弯处理 1,6 和 10天的应压木; OW1d,OW6d 和 OW10d 分别指拉弯处理 1,6 和 10 天的对应木; CK 则指未处理的直立木。 The compression woods after the bending of one-day,six-day and ten-day were individually designated by CW1d,CW6d and CW10d,and the opposite woods were accordingly designated by OW1d,OW6d and OW10d. CK means the normal wood. |
PAL基因是植物苯丙烷类物质生物合成的重要调控位点,多以基因家族形式存在。拟南芥基因组中含4个PAL(Huang et al.,2010),近来完成测序的黄瓜(Cucumis sativus)基因组中则有7个PAL成员(Shang et al.,2012)。火炬松是裸子植物中具较好序列信息积累的树种,目前已鉴定出5个PAL成员(Bagal et al.,2012)。在前期转录组测序的基础上,本试验成功分离到3个ClPAL的全长cDNA序列,且对应氨基酸序列皆含有植物PAL蛋白所具有的特征结构域。Bagal等(2012)研究认为在不同进化阶段发生基因重复事件导致产生了5个PtPAL基因,且来自早期基因重复的PAL成员往往具有不同的功能,而近期基因重复产生的PAL成员在功能上表现冗余。从进化树分析结果看,3个ClPAL分别与来自早期基因重复的不同PtPAL聚类在一起,暗示它们也可能来自不同时期发生的基因重复事件,且具不同功能。不同PAL基因的产物具有不同的功能,参与不同苯丙烷类合成的分支途径,且催化形成相应的前体,因此,其表达模式也往往不尽相同。Hamberger等(2007)研究发现毛果杨的PtrPAL2主要在正在发育的木材组织中表达,与木质素的合成有关;PtrPAL1则主要参与缩合单宁的合成。在本试验中,虽然3个ClPAL基因在不同的器官和组织中皆有表达,但仍表现出一定的组织特异性。ClPAL1和ClPAL3在木质化茎中优势表达,相应表达量是非木质化茎的6.5倍以上,且在木质化茎中的表达部位均集中在木质部区域;ClPAL2主要在富含韧皮部的针叶和皮层中高丰度表达。这些结果说明ClPAL1和ClPAL3可能主要参与了杉木木材组织的木质化过程。IAA、 GA3和BR能促进木质部细胞的分化,与纤维素、木质素和半纤维素等细胞壁成分间必然有直接或间接的联系。IAA被认为能提高次生木质部细胞的木质化程度,并促进管状分子的分化(Sitbon et al.,1999;Demura et al.,1993)。Israelsson等(2003)通过转基因提高了杂交杨中的GA水平,并发现转化植株中木纤维的数目和长度明显增加。Hossain等(2012)应用RNAi使得拟南芥的BR生物合成受阻,导致转化植株花序茎中的木质素含量比野生型下降了38%,且S/G木质素单体比例更低。在本试验中,经此IAA、 GA3和BR处理12 h后,ClPAL1和ClPAL3在木质部中的表达模式相似,均受到了诱导,尤其是BR的诱导效应最为显著,进一步表明这2个基因可能都在杉木木质素的生物合成中发挥重要作用。然而,在杉木应压木的诱导形成中,ClPAL1的表达呈现出与木质素变化相对一致的模式,而ClPAL3的表达却无明显变化。可见,这2个基因仍可能存在不同的调控机制。
中间材是心材和边材之间存在的窄过渡区,是心材不断形成的区域。试验中发现木质部优势表达的ClPAL1和ClPAL3在中间材中的表达量均高于边材20倍以上。而最近的研究报道,在杉木边心材转变过程中,木质素含量和分子结构发生了变化,心材细胞壁木质素含量高于边材,且木质素交联程度高于边材(宋坤霖,2012)。因此,ClPAL1和ClPAL3对于杉木木质素的这种变化可能有重要作用。值得注意的是,主要在韧皮部表达的ClPAL2在中间材中的表达丰度也显著高于边材。中间材所含薄壁射线细胞能不断合成各种次生代谢物,如单宁、类黄酮、木脂体类等,并随着死亡能分散到邻近细胞和空腔,使心材具有特殊色泽(Hillis,1996;Pallardy,2008)。Beritognol等(2002)研究发现,参与苯丙烷途径的查尔酮合酶(chalcone synthase,CHS)、 PAL在秋季黑核桃(Juglans nigra)中间材中的表达量均明显高于其他区域的木质部,认为与心材中酚类物质的积累有关。可见,该基因是否参与单宁、类黄酮等次生代谢物的合成,与杉木心材色泽的加深有关,值得深入研究。
| [1] |
宋坤霖. 2012. 杉木边心材转变过程中细胞壁结构与性能的变化. 北京: 中国林业科学研究院硕士学位论文.( 1) 1)
|
| [2] |
王桂凤, 高 燕, 杨立伟, 等. 2007. 杉木木材形成过程特异表达基因的鉴定与分析. 遗传, 29(4): 483-489. ( 1) 1)
|
| [3] |
Bagal U R, Leebens-Mack J H, Lorenz W W, et al. 2012. The phenylalanine ammonia lyase (PAL) gene family shows a gymnosperm-specific lineage. BMC Genomics, 13(Suppl 3): S1.( 2) 2)
|
| [4] |
Beritognolo I, Magel E, Abdel-Latif A, et al. 2002. Expression of genes encoding chalcone synthase, flavanone 3-hydroxylase and dihydroflavonol 4-reductase correlates with flavanol accumulation during heartwood formation in Juglans nigra. Tree Physiology, 22(5): 291-300.( 1) 1)
|
| [5] |
Boerjan W, Ralph J, Baucher M. 2003. Lignin biosynthesis. Annual Review of Plant Physiology, 54(10): 519-546.( 1) 1)
|
| [6] |
Butland S L, Chow M L, Ellis B E. 1998. A diverse family of phenylalanine ammonia-lyase genes expressed in pine trees and cell cultures. Plant Molecular Biology, 37(1):15-24.( 1) 1)
|
| [7] |
Cochrane F C, Davin L B, Lewis N G. 2004. The Arabidopsis phenylalanine ammonia lyase gene family: kinetic characterization of the four PAL isoforms. Phytochemistry, 65(11):1557-1564.( 1) 1)
|
| [8] |
Demura T, Fukuda H. 1993. Molecular cloning and characterization of cDNAs associated with tracheary element differentiation in cultured zinnia cells. Plant Physiology, 103(2): 815-821.( 1) 1)
|
| [9] |
Hamberger B, Ellis M, Friedmann M, et al. 2007. Genome-wide analyses of phenylpropanoid-related genes in Populus trichocarpa, Arabidopsis thaliana, and Oryza sativa: the Populus lignin toolbox and conservation and diversification of angiosperm gene families. Canadian Journal of Botany, 85(12):1182-1201.( 1) 1)
|
| [10] |
Hillis W E. 1996. Formation of robinetin crystals in vessels of Intsia species. IAWA Journal, 17(4): 405-419. ( 1) 1)
|
| [11] |
Hossain Z, McGarvey B, Amyot L, et al. 2012. DIMINUTO1 affects the lignin profile and secondary cell wall formation in Arabidopsis. Planta, 235(3): 485-498. ( 1) 1)
|
| [12] |
Huang Huahong, Xu Lili, Tong Zaikang, et al. 2012. De novo characterization of the Chinese fir (Cunninghamia lanceolata) transcriptome and analysis of candidate genes involved in cellulose and lignin biosynthesis. BMC Genomics, 13(1): 648.( 2) 2)
|
| [13] |
Huang Junli, Gu Min, Lai Zhibing, et al. 2010. Functional analysis of the Arabidopsis PAL gene family in plant growth, development, and response to environmental stress. Plant Physiology, 153(4): 1526-1538.( 2) 2)
|
| [14] |
IAWA Committee. 1989. IAWA list of microscopic features for hardwood identification. IAWA Bulletin, 10(3): 219-332.( 1) 1)
|
| [15] |
Israelsson M, Eriksson M E, Hertzberg M, et al. 2003. Changes in gene expression in the wood-forming tissue of transgenic hybrid aspen with increased secondary growth. Plant Molecular Biology, 52(4): 893-903.( 1) 1)
|
| [16] |
Kao Y Y, Harding S A, Tsai C J. 2002. Differential expression of two distinct phenylalanine ammonia-lyase genes in condensed tannin-accumulating and lignifying cells of quaking aspen. Plant Physiology, 130(2): 796-807.( 2) 2)
|
| [17] |
Karpinska B, Karlsson M, Srivastava M, et al. 2004. MYB transcription factors are differentially expressed and regulated during secondary vascular tissue development in hybrid aspen. Plant Molecular Biology, 56(2):255-70.( 1) 1)
|
| [18] |
Li Laigeng, Lu Shanfa, Chiang V. 2006. A genomic and molecular view of wood formation. Critical Reviews in Plant Sciences, 25(3): 215-233.( 1) 1)
|
| [19] |
Minami E, Ozeki Y, Matsuoka M, et al. 1989. Structure and some characterization of the gene for phenylalanine ammonia-lyase from rice plants. European Journal of Biochemistry, 185(1):19-25.( 1) 1)
|
| [20] |
Nagai N, Kitauchi F, Shimosaka M, et al. 1994. Cloning and sequencing of a full-length cDNA coding for phenylalanine ammonia-lyase from tobacco cell culture. Plant Physiology, 104(3): 1091-1092.( 1) 1)
|
| [21] |
Onaka F. 1949. Studies on compression and tension wood. Wood Research, 24(1):1-88.( 1) 1)
|
| [22] |
Pallardy S G. 2008. Physiology of woody plants. London: Academic Press, 454.( 1) 1)
|
| [23] |
Ritter H, Schulz G E. 2004. Structural basis for the entrance into the phenylpropanoid metabolism catalyzed by phenylalanine ammonia-lyase. Plant Cell, 16(12): 3426 -3436.( 1) 1)
|
| [24] |
Rohde A, Morreel K, Ralph J, et al. 2004. Molecular phenotyping of the pal1 and pal2 mutants of Arabidopsis thaliana reveals far-reaching consequences on phenylpropanoid, amino acid, and carbohydrate metabolism. Plant Cell, 16(10): 2749-2771.( 1) 1)
|
| [25] |
Shang Qingmao, Li Liang, Dong Chunjuan. 2012. Multiple tandem duplication of the phenylalanine ammonia-lyase genes in Cucumis sativus L. Planta, 236(4):1093-1105.( 1) 1)
|
| [26] |
Sitbon F, Hennion S, Little C H A, et al. 1999. Enhanced ethylene production and peroxidase activity in IAA-overproducing transgenic tobacco plants is associated with increased lignin content and altered lignin composition. Plant Science, 141(2): 165-173.( 1) 1)
|
| [27] |
Tamura K, Dudley J, Nei M, et al. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4. 0. Molecular Biology and Evolution, 24(8): 1596 -1599.( 1) 1)
|
| [28] |
Timell T. 1986. Compression wood in gymnosperms: Vol. 1-3. Berlin: Springer-Verlag, 1339-2150.( 1) 1)
|
| [29] |
Ursache R, Nieminen K, Helariutta Y. 2012. Genetic and hormonal regulation of cambial development. Physiologia Plantarum, 147(1): 36-45.( 1) 1)
|
| [30] | Wang Guifeng, Gao Yan, Wang Jinjun, et al. 2011. Overexpression of two cambium-abundant Chinese fir (Cunninghamia lanceolata) α-expansin genes ClEXPA 1 and ClEXPA 2 affect growth and development in transgenic tobacco and increase the amount of cellulose in stem cell walls. Plant Biotechnology Journal, 9(4): 486-502. |
| [31] |
Wang Guifeng, Gao Yan, Yang Liwei, et al. 2007. Identification and analysis of differentially expressed genes in differentiating xylem of Chinese fir (Cunninghamia lanceolata) by suppression subtractive hybridization. Genome, 50(12): 1141-1155.( 1) 1)
|
| [32] |
Wardrop A B, Dadswell H E. 1950. The nature of reaction woodⅡ: the cell wall organization wood tracheid. Australian Journal of Scientific Research, 3(1): 1-13.( 1) 1)
|
| [33] |
Whetten R, Sederoff R. 1992. Phenylalanine ammonia-lyase from loblolly pine: Purification of the enzyme and isolation of complementary DNA clones. Plant Physiology, 98(1): 380-386.( 1) 1)
|
| [34] |
Wilson B F, Archer R R. 1977. Reaction wood: induction and mechanical action. Annual Review of Plant Physiology, 28(6): 23-43.( 1) 1)
|
| [35] |
Zhong Ruiqin, Morrison W H, Himmelsbach D S, et al. 2000. Essential role of caffeoyl coenzyme A O-methyltransferase in lignin biosynthesis in woody poplar plants. Plant Physiology, 124(2): 563-577.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

