文章信息
- 王京京, 童再康, 黄程前, 程龙军
- Wang Jingjing, Tong Zaikang, Huang Chengqian, Cheng Longjun
- 巨桉EgrCBF1和EgrCBF2基因的克隆和胁迫响应表达分析
- Cloning of EgrCBF1 and EgrCBF2 Genes from Eucalyptus grandis and their Expression under Stresses
- 林业科学, 2012, 48(10): 41-48.
- Scientia Silvae Sinicae, 2012, 48(10): 41-48.
-
文章历史
- 收稿日期:2012-02-17
- 修回日期:2012-03-19
-
作者相关文章
巨桉(Eucalyptus grandis)由于生长速度快,适应性强,被广泛作为经济林木种植。然而由于它的冷敏感性,分布区域受到限制;同时,由于近年来气候的异常,使巨桉生产遭受冷冻危害的频率也在不断提高。巨桉的抗冷研究一直是桉树育种的一个重要方向,但以前主要局限在常规杂交育种方面,抗冷分子机制的研究十分缺乏,大大限制了分子辅助育种措施在其抗逆育种方面的应用。
近年来的研究表明,在植物对抗低温胁迫时,一个CBF(CRT/DRE-binding factor)基因家族在发挥重要的作用(Stockinger et al., 1997;Zhou et al., 2011)。CBF/DREB属于AP2类转录因子。这类转录因子广泛存在于拟南芥(Arabidopsis thaliana)、玉米(Zea mays)(Qin et al., 2004)、水稻(Oryza sativa)(Dobouzet et al., 2003)、小麦(Triticum aestivum)(Jaglo et al., 2001)以及马铃薯(Solanum tuberosum)(Zhang et al., 2004)等植物中,该基因家族的成员都含有60个左右氨基酸残基组成的非常保守的DNA结合区即AP2结构域。位于AP2/ERF结构域上游还含有典型的DREB1型转录因子核定位信号(NLS)区:KR/KPAGRT/KKFRETRHP,CBF转录激活因子进入细胞核的过程受该区域的控制;位于AP2/ERF结构域下游还含有DSA盒,这些区域决定了CBF转录因子的功能与特性(Qin et al., 2004)。当植物在低温等非生物逆境因子胁迫下,CBF的AP2区域能特异地与下游基因启动子中的CRT/DRE元件相互作用,启动下游与抗逆性相关的功能基因的表达,如COR,LTI,RD和KIN等基因,参与植物提高对寒冷等逆境胁迫抵御能力的过程(Cook et al., 2004;Fowler et al., 2002;Ingram et al., 1996;Pearce,1999)。
目前,拟南芥中发现了4个CBF基因,其中AtCBF1,AtCBF2,AtCBF3参与植株冷胁迫信号的传递,受低温诱导表达,将HOS1,ICE1,MYB15等传递来的低温信息传递给下游参与冷胁迫反应的功能基因COR等(Cook et al., 2004;Chinnusamy et al., 2003)。CBF基因可以快速强烈地响应低温逆境。拟南芥植株在2.5 ℃低温的诱导下,CBF转录物储量迅速增加,并在随后几个小时内继续增加,随后又呈现逐步减少的趋势,而在2 h后,含有CRT/DRE元件的COR基因的转录物才开始积累(Imar et al., 1996;Shinwari et al., 1998),说明,CBF在冷胁迫响应中扮演的是敏感的信号传递角色。同时,这些基因在具体功能上又有所不同,研究表明它们不但参与植物的抗冷胁迫,同时还可能参与ABA、干旱和高盐逆境的响应。例如AtCBF1,AtCBF2,AtCBF3都受冷胁迫诱导,但AtCBF2又对AtCBF1,AtCBF3有一定抑制作用,AtCBF4目前发现它与植物干旱胁迫有关系,同时与冷胁迫相关的下游基因RD29A互作参与冷胁迫反应,而且受ABA的调控(Novillo et al., 2004;Haake et al., 2002)。葡萄(Vitis vinifera)中的VvCBF4既受冷胁迫诱导,同时对盐胁迫也有响应(Xiao et al., 2008)。这些都表明CBF基因家族的成员功能多样而复杂,同一种植物中不同成员之间具有不同分工,不同植物中即使同源性很高的成员之间在功能上也可能存在较大差异。
CBF途径在植物抵抗低温等逆境胁迫中具有非常重要的作用,对于巨桉这种冷敏感植物,了解CBF基因在其抗冷和其他逆境因子中的作用,对巨桉抗逆分子辅助育种具有非常重要的意义。因此,对巨桉CBF基因的克隆,以及它们对低温等逆境因子的响应研究,是深入研究这些基因功能并在将来育种工作中加以利用的基础。本研究利用从巨按中克隆到的2个CBF基因EgrCBF1和EgrCBF2,详细分析了这2个基因在不同低温条件下,以及低温下不同处理时间时的表达情况,揭示了它们在低温条件下被诱导表达的模式。同时,还针对这2个基因对ABA、干旱以及盐胁迫响应做了初步的研究,探讨这2个基因对其他逆境条件的反应,初步阐释了它们的抗逆功能。为对它们功能的进一步研究打下了坚实的基础。
1 材料与方法 1.1 材料巨桉材料为福建永安林业公司提供的优良无性系幼苗,苗龄为4个月,取自于浙江农林大学桉树良种繁育中心苗圃。
克隆用大肠杆菌(Escherichia coli) DH5α为本实验室保存;T4 DNA连接酶、Taq DNA聚合酶、克隆载体pGEM-T Easy Vector为Promega公司产品;RT-PCR试剂盒为Invitrogen公司产品;引物由南京金斯瑞生物工程技术服务有限公司合成;RACE试剂盒为ClonTech产品;SYBR荧光染料为TaKaRa公司产品。
1.2 目的基因的克隆与序列分析取4 ℃条件下处理2 h的巨桉茎叶组织提取总RNA,用于基因克隆。RNA提取用改进的CTAB法(侯夫云等,2008)。称取150 mg样品, 在液氮中研成粉末转入到65 ℃水浴的700 μL CTAB(2%)提取液中, 充分混匀。65 ℃水浴30 min后冷却至室温。加等体积(700 μL)氯仿:异戊醇(24:1), 混匀, 4 ℃下以10 000×g离心10 min。小心吸取上清液, 重复上述步骤, 直至中间层无白色絮状沉淀。吸取上清液, 加1/4体积的LiCl(10 mol·L-1), 混匀后于4 ℃沉淀过夜, 然后10 000×g离心20 min。弃上清, 加入500 μL SSTE(1 mol·L-1 NaCl,0.5%SDS,1 mmol·L-1 EDTA, pH8.0,10 mmol·L-1 Tris-HCl, pH8.0)溶解沉淀, 加入等体积的氯仿:异戊醇(24:1), 混匀, 4 ℃下10 000×g离心10 min;取上清, 加入2倍体积无水乙醇-70 ℃沉淀至少30 min。4 ℃下12 000×g离心20 min。弃上清, 75%的乙醇洗涤2次, 吹干沉淀, 30 μL DEPC处理的水溶解沉淀, 得到RNA样品。电泳检测后,将提取的RNA逆转录成cDNA作为模板待用。
本研究对从GenBank数据库获得的CBF基因的编码序列通过ClustalX进行多序列比对,找出保守区,设计兼并性引物CBF-F和CBF-R(表 1),以cDNA为模板进行PCR扩增,获得的片段连入T载体进行克隆,经过检测后,将阳性克隆送至南京金斯瑞生物工程技术服务有限公司测序。
|
|
基因5′和3′端序列的获得利用SMARTTMRACE cDNA Amplification Kit(ClonTech)来进行,根据RT-PCR片段的序列测定结果,设计、合成RACE-PCR所需的特异引物GSP1和GSP2,结合试剂盒提供的通用引物UPM(表 1),克隆cDNA的5′和3′端序列。具体步骤按试剂盒说明书进行,PCR产物回收后经T载体克隆、测序,并与已克隆的CBF基因片段进行拼接,获得全长cDNA序列。根据拼接得到的基因全长cDNA设计引物(表 1),再经PCR扩增,凝胶电泳分析、测序,验证所获基因全长序列的准确性。
1.3 EgrCBF基因的生物信息学分析利用推测的蛋白质序列在http://au.expasy.org/tools/pi_tool.html上用其Compute pI/MW工具分析EgrCBF1和EgrCBF2等电点(pI);在http://www.bioinfo.tsinghua.edu.cn/SubLoc/进行蛋白质的亚细胞定位分析;利用蛋白质保守结构域推测工具SMART(http://smart.embl-heidelberg.de)分析EgrCBF1和EgrCBF2蛋白的结构;分析巨桉EgrCBF1和EgrCBF2蛋白与其他植物中同源蛋白序列的同源性时,分别以EgrCBF1和EgrCBF2的蛋白序列在NCBI(http://www.ncbi.nlm.nih.gov/blast)进行tblastn比对,搜寻与它们同源性较高的其他植物中CBF蛋白,然后,用ClustalX软件对不同植物的CBF蛋白质序列与巨桉的CBF蛋白质序列进行多序列联配,并用MEGA4和PHYLIP软件构建不同植物的CBF蛋白进化树,分析它们之间的进化关系。
1.4 EgrCBF基因组织特异性表达分析采用半定量RT-PCR,根据EgrCBF1,EgrCBF2以及作为内参基因的Egr18SRNA基因全长设计半定量RT-PCR引物(表 2)。按照1.2中方法提取正常生长的巨桉叶、茎、根组织中的RNA,反转录成cDNA。以获得的2个EgrCBF的cDNA为模板进行PCR扩增,对该基因的组织表达特异性进行分析。
|
|
其中一组处理将长势一致的桉树无性系幼苗分别放于25 ℃黑暗条件作为对照,其他幼苗在0,2,4,6,8 ℃条件下(控温冰箱中)进行低温黑暗处理,处理时间为2 h;将另外一组长势相同的同一桉树无性系幼苗放于4 ℃黑暗条件下分别处理2,4,8,24,48 h。处理后采处理植株的叶片,提取总RNA,并进行逆转录、待用。每一处理均重复3次。
1.6 ABA、干旱和盐处理对于ABA和盐处理,参考Kayal等(2006)的方法进行。从桉树幼苗上随机采取健康、完整叶片,浸于100 μmol·L-1ABA(DMSO作为ABA溶剂,终浓度为10%),以叶片浸于10%的DMSO溶液中2 h作为ABA处理的对照;高盐处理则以巨桉叶片浸于200 mmol·L-1 NaCl溶液中2 h,蒸馏水处理作为对照;对于干旱处理,将随机采取的桉树叶片置于开口的空培养瓶中2 h。将浸于蒸馏水中2 h作为盐和干旱的对照。取处理后的植株叶片提取总RNA,进行逆转录,待用。每一处理均重复3次。
1.7 实时荧光定量RT-PCR根据已获得的EgrCBF1和EgrCBF2 cDNA全长设计qRT-PCR引物,仍以Egr18SRNA基因作为内参基因(表 2),进行qRT-PCR。PCR反应体系10 μL,包括SYBR Premix Ex Taq 5 μL,上、下游引物各0.2 μL,cDNA模板1 μL,反应于Bio-Rad CFX96荧光定量PCR仪上进行。所有生物学重复样品均做3次重复,反应条件为:95 ℃预变性3 min;95 ℃ 10 s,65 ℃ 40 s,40个循环。定量结果数据用SPSS V13.0进行统计分析,依据相对定量计算公式2-ΔΔCt计算基因的相对表达倍数,并用GraphPad Prism 5进行作图。
2 结果与分析 2.1 EgrCBF1和EgrCBF2基因的克隆通过测序获得1个约500 bp的片段,RACE后经过拼接、验证、测序,得到2条CBF的全长cDNA序列,全长分别为1 062 bp和1 203 bp,分别含有1个663 bp和591 bp的完整开放阅读框,分别命名为EgrCBF1和EgrCBF2。经过Blast(http://www.ncbi.nlm.nih.gov/blast)比对,结果表明EgrCBF1基因与冈尼桉EguCBF1a基因的同源性达到95%,EgrCBF2基因与冈尼桉(Eucalyptus gunnii)EguCBF1d基因的同源性达到98%,证明这2条序列也属于CBF基因家族成员。
2.2 EgrCBF1和EgrCBF2蛋白的序列分析EgrCBF1和EgrCBF2分别编码220个氨基酸和196个氨基酸。预测其分子量分别为24.5 ku和21.6 ku,等电点pI分别为8.64和6.04。蛋白结构分析表明:EgrCBF1和EgrCBF2与其他植物中的CBF蛋白一样也具有AP2结构域(图 1),以及CBF蛋白特征基序PKKAGRKKFETRHP和DSAWR,它们分别位于AP2结构域的上游和下游;同时,在54—64残基之间有一高度保守区,大部分是碱性氨基酸,为核定位区,蛋白质亚细胞定位预测也表明二者均属于核蛋白。

|
图 1 巨桉CBF蛋白序列与其他植物同源蛋白序列的比对 Fig.1 Multiple alignment of CBF in E. grandis and its homologous proteins from other plants AtCBF1, AtCBF2, AtCBF3, AtCBF4:拟南芥Arabidopsis thaliana(AF076155,AF076155, AF076155,EF523160);LeCBF1, LeCBF2, LeCBF3:番茄Lycopersicon esculentum (AY497899);StCBF3:马铃薯Solanum tuberosum(EU365384);BpCBF1:欧洲白桦Betula pendula(EF530205);EguCBF1a, EguCBF1b, EguCBF1c, EguCBF1d:冈尼桉Eucalyptus gunnii(DQ241820,DQ241821,EU794855, EU794856);EgCBF1:蓝桉Eucalyptus globulus(DQ523829).黑色方框表示AP2结构域;黑色下划线表示CBF特征基序。 AP2 domain is boxed and the CBF signature motif is marked with a line. |
氨基酸序列比对分析表明EgrCBF1和EgrCBF2与冈尼桉、蓝桉(Eucalyptus globulus)CBF蛋白的同源性都达到78%以上。与冈尼桉中的EguCBF1a蛋白的同源性分别达到91%和80%。进化分析(图 2)表明,CBF家族的基因大致分为3个亚家族,而EgrCBF1和EgrCBF2与冈尼桉中的EguCBF1a,EguCBF1b,EguCBF1c,EguCBF1d,蓝桉中的EgCBF1和欧洲白桦中的BpCBF1同属于一个亚家族,它们均为木本植物。
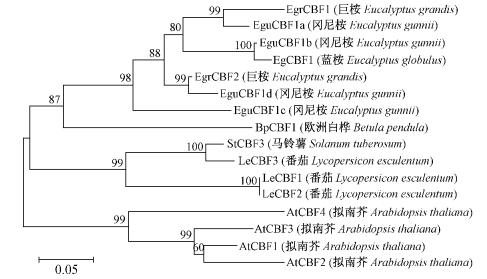
|
图 2 植物CBF蛋白的系统发育树 Fig.2 Phylogenetic tree of CBF protein in different plants |
半定量RT-PCR分析的结果(图 3)表明,正常条件下,EgrCBF1和EgrCBF2基因在叶、茎、根中都有表达,EgrCBF1主要在叶和根中表达,在茎中表达量较低;而EgrCBF2在叶、茎、根中表达量没有明显差异。
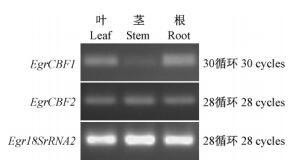
|
图 3 巨桉CBF基因在叶、茎、根中的表达 Fig.3 The expression of CBF genes in leaf, stem and root in E. grandis |
从图 4可以看到,在正常条件下,EgrCBF2的表达量高于EgrCBF1,为EgrCBF1的8.63倍。在不同的低温处理条件下,2个基因的表达都明显受到低温的诱导,EgrCBF1和EgrCBF2基因都在2 ℃时诱导表达量达到最高水平,相对表达量分别是对照水平的32.4倍和40.2倍;随着温度的继续降低,EgrCBF1和EgrCBF2基因的表达又开始呈现下降的趋势,其中EgrCBF1下降幅度更大,仅为2 ℃条件下的0.28倍。
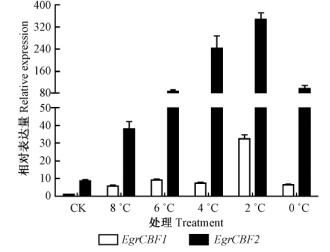
|
图 4 不同低温胁迫下EgrCBF1和EgrCBF2基因相对表达差异 Fig.4 Relative expression of EgrCBF1 and EgrCBF2 under different cold stress |
在低温胁迫下,CBF基因表达都具有瞬时表达特性。受冷胁迫影响后,短时间内即被强烈诱导表达,但随时间的延续诱导表达程度减弱。EgrCBF1和EgrCBF2也都具有这种特性,从图 5可以看到:EgrCBF1和EgrCBF2基因在4 ℃不同时间条件下,都能被显著诱导表达,在2 h时,表达被诱导上调达到最高水平,表达量分别为对照的6.63倍和40.8倍;而后EgrCBF1在24 h前基本稳定地诱导表达,48 h时下降到24 h时的0.32倍,EgrCBF2在2 h后即开始下降,8 h后下降趋势趋缓。这种现象可能跟基因表达产物存在一定的反馈抑制作用有关。
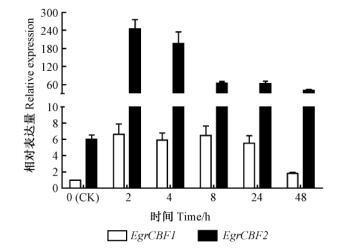
|
图 5 4 ℃不同时间下EgrCBF1和EgrCBF2基因相对表达差异 Fig.5 Relative expression of EgrCBF1 and EgrCBF2 under different time of 4 ℃ stress |
从图 6可以看到,EgrCBF1在100 μmol·L-1 ABA处理下,表达有所上调,上调幅度为对照的1.89倍;而EgrCBF2基因的表达与对照相比,变化没有达到显著水平。ABA参与很多逆境响应的分子机制,有些植物中的CBF基因同样受ABA的诱导,暗示某些CBF基因作用的发挥可能会依赖于ABA(Zhou et al., 2011)。但从EgrCBF1和EgrCBF2对ABA的反应来看,EgrCBF1可能与ABA的作用有关,而EgrCBF2基因作用的发挥极可能是不依赖于ABA的。
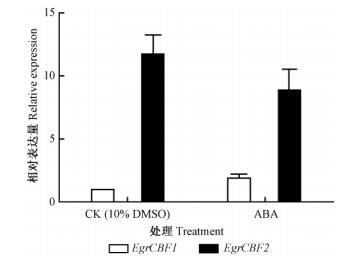
|
图 6 ABA胁迫下EgrCBF1和EgrCBF2基因相对表达差异 Fig.6 Relative expression of EgrCBF1 and EgrCBF2 under ABA stress |
CBF基因除了在冷胁迫逆境和ABA控制的逆境响应中发挥作用外,干旱和盐胁迫也可能改变它们的表达模式,进而在相应的逆境中发挥作用。从图 7可以看到,EgrCBF2的表达在干旱、盐胁迫下,均被诱导强烈上调,上调倍数分别为7.35和6.02。说明该基因可能同时参与了巨桉冷、干旱和盐等逆境响应的调控。而EgrCBF1在干旱条件下被微弱诱导上调,为对照的1.76倍;在盐胁迫条件下的变化与对照相比没有达到显著水平。由此可见,EgrCBF1,EgrCBF2在应对不同逆境响应过程中,发挥的功能也有所不同。
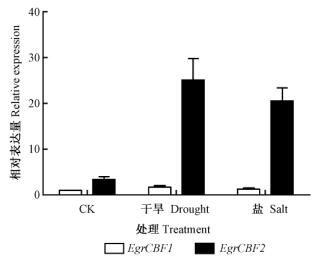
|
图 7 干旱、盐胁迫下EgrCBF1和EgrCBF2基因相对表达差异 Fig.7 Relative expression of EgrCBF1 and EgrCBF2 under drought and salt stress |
植物中的CBF是参与众多非生物逆境,尤其是冷胁迫逆境的一个重要转录因子。它在植物的抗冷分子机制中形成一个独特的CBF通路发挥作用(杨家森等,2006)。本研究通过RACE技术从巨桉中克隆到2个CBF基因,EgrCBF1和EgrCBF2。2个基因与其他木本植物中克隆到的CBF基因编码的蛋白比较,具有很高相似性,表明这2个基因均属于典型的CBF基因,而且它们都具有明确的CBF蛋白序列和结构特征,具有核定位区以及与冷胁迫下游基因COR等启动子的CRT/DRE结合的AP2功能基序。但这2个基因编码的蛋白在氨基酸数目和组成上有明显差异,属于2个完全不同的基因。
在桉树正常生长条件下,EgrCBF2在叶、茎、根不同器官中都有表达,表达量也没有明显差异;而EgrCBF1在茎中的表达量相对叶和根较低。由于这2个基因具有不同的蛋白序列组成,可能导致它们的功能也有所差异,因而在组织表达模式上有所不同。对巨桉进行低温处理后,不同低温条件下,2个基因的表达都有明显的诱导效应,随温度降低,表达量持续上升,在2 ℃时达到最高,0 ℃时又有所下降,这可能与更低温度条件下,细胞伤害导致的基因调控紊乱有关。2个基因相比,无论正常条件还是低温处理,EgrCBF2表达量都要高于EgrCBF1。4 ℃低温不同时间处理下,2个基因也都表现出随处理时间的延长,表达先升后降的趋势,但EgrCBF1诱导表达下降的时间要迟于EgrCBF2,在24 h后才开始下降,而EgrCBF1在2 h即达到最高水平随后就表现出下降的趋势。这种受低温诱导且随时间延长诱导水平先升后降的趋势与大部分植物中CBF基因的表达模式是相同的,拟南芥中除AtCBF4外,AtCBF1,AtCBF2,AtCBF3都有这种现象(Gilmour et al., 1998;Medina et al., 1999;Haake et al., 2002),冈尼桉中EguCBF1a和EguCBF1b同样有这种表达规律(Kayal et al., 2006),这说明EgrCBF1和EgrCBF2应该是巨桉CBF抗冷反应通路中的2个重要成员,担负着传递冷胁迫信号给下游功能基因的重要作用。另外,2个基因在表达模式上和低温诱导表现上也有一定的差异,暗示这2个基因在具体功能上可能存在一定不同。
本研究表明,EgrCBF1和EgrCBF2不但在冷胁迫诱导的表达方面具有不同表现,对其他逆境因子也有不同的反应。EgrCBF1在100 μmol·L-1 ABA处理下,有一定的上调效应,尽管目前大部分研究认为,CBF冷诱导反应通路是独立于ABA途径的,但现在已经有发现某些CBF成员可能与这2种途径都有互作,参与植物的抗逆反应。AtCBF4和冈尼桉中的EguCBF1a和EguCBF1b都不同程度受ABA诱导表达(Haake et al., 2002;Kayal et al., 2006),因此关于CBF抗冷胁迫通路与ABA途径的关系仍需要进一步的研究加以明确。本研究中的EgrCBF1很可能类似于冈尼桉中的EguCBF1a和EguCBF1b基因,同时参与巨桉抗逆分子机制中的ABA途径和CBF途径。
尽管植物中的CBF基因主要参与冷胁迫响应分子机制作用,但已有的CBF基因功能研究证明,提高CBF基因的表达,不但可以提高植物抵抗冷胁迫的能力,往往也能增强植物对干旱、盐胁迫抵抗的功能。AtCBF1,AtCBF3超表达的拟南芥植株抗低温、干旱和高盐的能力都得到增强(Jaglo-Ottosen et al., 1998);Ito等(2006)将OsCBF基因转入水稻后, 水稻的抗寒性、抗旱性、抗盐性都得到了提高;大麦(Hordeum vulgare)中CBF基因HvCBF4转入水稻中,玉米的ZmCBF转入拟南芥中,都能提高植株对冷、旱和盐的抵抗能力(Oh et al., 2007;Qin et al., 2004)。另外,CBF基因的表达也不仅仅受低温的影响,干旱、高盐等逆境因子同样影响它们的表达,冈尼桉中的EguCBF1a和EguCBF1b都受高盐的诱导(Kayal et al., 2006),AtCBF4则同时受高盐和干旱逆境的诱导(Xiao et al., 2008)。这些都表明CBF可能不仅在植物冷逆境中发挥作用,其可能会受多种逆境因子的影响,并能启动多个逆境反应调控途径,参与植物的抗逆响应。本研究中的EgrCBF1和EgrCBF2在干旱条件下都被诱导,尤其是EgrCBF2,相对于对照,干旱条件下上调强烈。暗示其在干旱胁迫信号的传递中同样可能发挥着重要的作用。在高盐条件下,EgrCBF1的表达变化相对于对照没有达到显著水平,而EgrCBF2同样被强烈诱导,表明在盐胁迫方面,EgrCBF2也可能发挥重要作用。
以上结果表明,本研究中克隆的2个基因尽管都是巨桉冷胁迫反应中的重要的CBF成员,但他们的功能可能更为复杂。EgrCBF1可能参与了ABA和冷胁迫反应机制的互作;而EgrCBF2在抗逆方面可能具有多效性,既参与了巨桉冷胁迫响应的调控,在干旱、高盐逆境响应的分子机制中也发挥着重要的作用。对它们功能的深入研究,将对理解巨桉抵抗寒冷等逆境胁迫的分子机制具有重要意义,这将是下一步研究的主要方向。
| [] | 侯夫云, 赵兵, 赵宝杰, 等. 2008. 利用改良的CTAB-LiCl法提取甘薯块根RNA. 山东农业科学(9): 90–92. |
| [] | 杨家森, 张洪涛, 李新国, 等. 2006. 拟南芥CBF冷反应通路. 植物生理学通讯, 42(1): 152–161. |
| [] | Chinnusamy V, Ohta M, Kanrar S, et al. 2003. ICEl:a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis. Genes&Development, 17(60): 1043–1054. |
| [] | Cook D, Fowler S, Fiehn O, et al. 2004. A prominent role for the CBF cold response pathway in configuring the low-temperature metabolome of Arabidopsis. Proc Natl Acad Sci USA, 101(42): 15243–15248. DOI:10.1073/pnas.0406069101 |
| [] | Dobouzet J, Sakuma Y, Ito Y, et al. 2003. OsDREB genes in rice, Oryza sativa L., encode transcription activators that function in drought-, high-salt-and cold-responsive gene expression. Plant J, 33(4): 751–763. DOI:10.1046/j.1365-313X.2003.01661.x |
| [] | Fowler S, Thomashow M F. 2002. Arabidopsis transcriptome profiling indicates that multiple regulatory pathways are activated during cold acclimation in addition to the CBF cold response pathway. The Plant Cell, 14(8): 1675–1690. DOI:10.1105/tpc.003483 |
| [] | Gilmour S J, Zarka D G, Stockinger E J, et al. 1998. Low temperature regulation of the Arabidopsis CBF family of AP2 transcriptional activators as an early step in cold-induced COR gene expression. Plant J, 16(4): 433–442. DOI:10.1046/j.1365-313x.1998.00310.x |
| [] | Haake V, Cook D, Riechmann J L, et al. 2002. Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis. Plant Physio, 130(2): 639–648. DOI:10.1104/pp.006478 |
| [] | Imar R, Chang L, Ohta A, et al. 1996. A lea-class gene of tomato congers salt and freezing tolerance when expression in Saccharaomyces cerebisive. Gene, 170: 243–248. DOI:10.1016/0378-1119(95)00868-3 |
| [] | Ingram J, Bartels D. 1996. The molecular basis for dehydration tolerance in plants. Annu Rev Plant Physiol&Plant Mol Biol, 47: 377–403. |
| [] | Ito Y, Katsura K, Maruyama K, et al. 2006. Functional analysis of rice DREB1/CBF type transcription factors involved in cold-responsive gene expression in transgenic rice. Plant Cell Physiology, 47(1): 141–153. DOI:10.1093/pcp/pci230 |
| [] | Jaglo K R, Kleff S, Amundsen K L, et al. 2001. Components of the Arabidopsis C-repeat/dehydration-responsive element binding factor cold-response pathway are conserved in Brassica napus and other plant species. Plant Physiology, 127(3): 910–917. DOI:10.1104/pp.010548 |
| [] | Jaglo-Ottosen K R, Gilmour S J, Zarka D G, et al. 1998. Arabidopsis CBF1 overexpression induces COR genes and enhances freezing tolerance. Science, 280(5360): 104–106. DOI:10.1126/science.280.5360.104 |
| [] | Kayal E W, Navarro M, Marque G, et al. 2006. Expression profile of CBF-like transcriptional factor genes from Eucalyptus in response to cold. Journal of Experimental Botany, 57(10): 2455–2496. DOI:10.1093/jxb/erl019 |
| [] | Medina J, Bargues M, Terol J, et al. 1999. The Arabidopsis CBF gene family is composed of three genes encoding AP2 domain-containing proteins whose expression is regulated by low temperature but not by abscisic acid or dehydration. Plant Physiol, 119(2): 463–470. DOI:10.1104/pp.119.2.463 |
| [] | Novillo F, Alonso J M, Ecker J R, et al. 2004. CBF2/DREBlC is a negative regulator of CBFI/DREBlB and CBF3/DREBlA expression and plays a central role in stress tolerance in Arabidopsis. Proc Natl Acad Sci USA, 101(11): 3985–3990. DOI:10.1073/pnas.0303029101 |
| [] | Oh S J, Kwon C W, Choi D W, et al. 2007. Expression of barley HvCBF4 enhances tolerance to abiotic stress in transgenic rice. Plant Biotech J, 5(5): 646–656. DOI:10.1111/pbi.2007.5.issue-5 |
| [] | Pearce R. 1999. Molecular analysis of acclimation to cold. Plant Growth Regul, 29(12): 47–76. |
| [] | Qin F, Sakuma Y, Li J, et al. 2004. Cloning and functional analysis of a novel DREB1/CBF1 transcription factor involved in cold-responsive gene expression in Zea mays L. Plant Cell Physio, 45(8): 1042–1052. DOI:10.1093/pcp/pch118 |
| [] | Shinwari Z K, Nakashima K, Miura S, et al. 1998. An Arabidopsis gene family encoding DRE/CRT binding proteins involved in low-temperature-responsive gene expression. Biochem Bioph Res Comm, 250(1): 161–170. DOI:10.1006/bbrc.1998.9267 |
| [] | Stockinger E J, Gilmour S J, Thomashow M F. 1997. Arabidopsis thaliana CBF1 encodes an domain containing transcriptional activator that stimulates transcription in response to low temperature and water deficit. Proc Natl Acad Sci USA, 94(3): 1035–1040. DOI:10.1073/pnas.94.3.1035 |
| [] | Xiao H G, Tattersall E A R, Siddiqua M K, et al. 2008. CBF4 is a unique member of the CBF transcription factor family of Vitis vinifera and Vitis riparia. Plant Cell Env, 31(1): 1–10. |
| [] | Zhang X, Fowler S G, Cheng H, et al. 2004. Freezing-sensitive tomato has a functional CBF cold response pathway, but a CBF regulator that differs from that of freezing-tolerant Arabidopsis. Plant J, 39(6): 905–919. DOI:10.1111/tpj.2004.39.issue-6 |
| [] | Zhou M Q, Shen C, Wu L H, et al. 2011. CBF-dependent signaling pathway:A key responder to low temperature stress in plants. Cri Rev Bio, 31(2): 186–192. DOI:10.3109/07388551.2010.505910 |
 2012, Vol. 48
2012, Vol. 48