文章信息
- 洑香香, 赵虎, 王玉
- Fu Xiangxiang, Zhao Hu, Wang Yu
- 松属近缘种形态和分子鉴定及其亲缘关系探讨
- Species Identification and Genetic Relationship Assessment of Pinus (Sect. Pinus) Related Species Based on Morphological and Molecular Markers
- 林业科学, 2011, 47(10): 51-58.
- Scientia Silvae Sinicae, 2011, 47(10): 51-58.
-
文章历史
- 收稿日期:2010-12-15
- 修回日期:2011-01-26
-
作者相关文章
松属(Pinus)树种分布于北半球,我国有22种,其中大部分为重要的造林树种,如马尾松(P. massoniana)、黑松(P. thunbergii)、云南松(P. yunnanensis)等。国内外研究均表明松属树种表型上存在广泛的变异,因此传统种的划分主要根据形态特征进行分类。但形态特征在一些近缘种甚至属间种的区分上并不十分可靠(Morgenstern et al., 1964; Manley, 1971),主要表现在:一些种天然分布上存在重叠区域,而生殖上不存在隔离,同时种内的遗传变异较大(Fowler et al., 1988),如黄山松(P. hwangshanensis)和马尾松(邢有华等,1992);而有些种间杂种分布于2个亲本的中间区域,如高山松(P. densata)是云南松与油松(P. tabulaeformis)的杂交种,位于亲本都不能生长的青藏东南缘2 700~4 200 m的高海拔地带(Rieseberg, 1997),其形态和解剖性状介于2个亲本之间(Fu et al., 1999);还有些松树种内群体形态性状的变异较大,在不同的立地条件下,形态上产生显著的变异,如云南松在高山贫瘠的地区,主干不明显,形成“地盘松”,而在云南南部温湿度较高的地区形成“细叶云南松”(虞泓,1996);还有的种间性状差异仅在一定的生长发育阶段才表现出来(Donoghue et al., 1992)。这些都限制了表型性状在种质鉴定上的可靠性(Gordon, 1976; Fowler et al., 1988)。
分子标记为遗传背景一致的种质鉴别提供了成熟可靠的技术。研究结果表明,基因的进化可以反映物种的进化,并且不同的基因由于变异频率不同,从而在不同分类等级上具有一定的保守性(Chase et al., 1993; Johnson et al., 1995; 汪小全等, 1997)。同时由于生殖隔离、地理隔离或生态位上的差异,不同种在进化过程中固定了不同的基因,其中一些被固定的基因在种的水平上具有高度的保守性,这些差异可以通过DNA指纹技术被发现(Furman et al., 1997)。利用分子标记对种质进行鉴别,要求获得的标记位点在进化上具有一定的保守性,同时具有一定的变异速率,即在种间具有多态性而在种内具有高度的保守性。在过去的二十多年间,DNA标记为植物分类和种质鉴定提供了有效而又准确的依据(Furman et al., 1997; Sun et al., 2005; Ross-Davis et al., 2008; Koc et al., 2009)。
在各类分子标记中,微卫星标记(simple sequence repeat, SSR)具有共显性遗传、重复性与稳定性好、多态性高等优点,缺点是需设计特异性引物,引物开发成本高;而ISSR(inter-simple sequence repeat)技术是在SSR基础上开发的,大部分ISSR标记为显性标记,既具SSR标记的优点,又克服了其缺点。二者的稳定性优于RAPD(random amplified polymorphic DNA),在处理大批量的样品时比AFLPs(amplified fragment length polymorphisms)更经济,因此SSR和ISSR标记目前广泛应用于近缘种及杂种的种质鉴定及种质的遗传多样性评价(Hao et al., 2002; Balasaravanan et al., 2005;吴学尉等,2009;张红莲等,2010)。
松属造林树种在生产中主要采用播种育苗或直播造林。一些亲缘关系较近的种,如油松组树种,种间种内的表型多样性使得种质鉴定困难,其种子和幼苗在表型上更是难以区分,这给林业生产带来很大的困扰。近年来随着林权制度改革和林业经济的发展,对松属种苗的需求增加,种苗的调拨频繁,真假混杂的种子和苗木对林业生产的影响深远。因此亟待开发一套实用、快速、准确的松属种子和幼苗的鉴别方法。本研究利用种子和幼苗的形态特征结合分子标记方法(ISSR和SSR)对松属(油松组)近缘种进行早期鉴定,为林业生产中树种的正确使用提供科学依据。
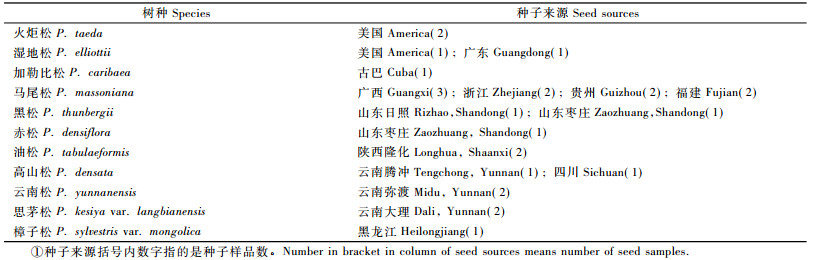
1 材料与方法 1.1 材料 1.1.1 植物材料11个松属(油松组)树种的种子来源见表 1。种子在25 ℃光照培养箱中萌发至子叶脱壳,每个种批收集30株新鲜幼苗,于-20 ℃冰箱中保存待用。
|
|
SSR引物分别来自松属树种的262对核SSR和14对叶绿体SSR(cp SSR),包括黑松(Vendramin et al., 1996)、火炬松(Elsik et al., 2000; Zhou et al., 2002)、北美乔松(P. strobus)(Echt et al., 1996)、海岸松(P. pinaster)和地中海松(P. halepensis)(González et al., 2002)、小干松(P. contorta var. latifolia)(Hicks et al., 1998)等。ISSR引物序列是加拿大哥伦比亚大学(UBC)设计并公布的100条单引物(http://www.biotech.ubc.ca/services/naps/primers/Primers.pdf)。
1.2 试验方法 1.2.1 形态性状观察与测定根据GB 2772-1999《林木种子检验规程》测定种子的千粒质量,记录种子的形状、颜色、幼苗下胚轴颜色,萌发结束时随机测定30株幼苗子叶数和苗高。
1.2.2 分子检测用2%CTAB和微量提取法(Dellaporta et al., 1983)提取幼苗基因组总DNA,每个种批随机取样30株分别提取个体DNA,并分别制备种源和种的混合基因池。1%琼脂糖凝胶电泳检测并稀释备用。
利用分群法(bulked segregate analysis,BSA)(Michelmore et al., 1996)筛选SSR和ISSR引物。
SSR扩增体系(10 μL)为:20~50 ng的DNA模板,dNTP各0.2 mmol·L-1,2.5 mmol·L-1 Mg2+,正反引物各0.5 μmol·L-1,0.25 U Taq酶(TaKaRa,TaqTM)和1×Buffer(10 mmol·L-1 Tris-Cl;50 mmol·L-1 KCl)。扩增采用touchdown PCR程序:94 ℃ 5 min;95 ℃ 30 s、58 ℃ 30 s(0.5 ℃↓/cycle至50 ℃)、70 ℃ 30 s,16 cycles;当退火温度降至50 ℃时,再运行24循环;70 ℃ 5 min。扩增产物用6%变性聚丙烯酰胺凝胶电泳分离,银染检测。
ISSR扩增体系(25 μL)为:20 ng DNA,dNTP 0.25 mmol·L-1,Taq酶0.5 U,1×Buffer,2.0 mmol·L-1 Mg2+,0.8 μmol·L-1引物。扩增程序为:94 ℃ 7 min;94 ℃ 30 s,56 ℃ 45 s,72 ℃ 2 min,40个循环;72 ℃ 7 min。1%琼脂糖凝胶电泳检测。
1.3 数据分析统计清晰、再现性强的电泳条带,并按1/0形式进行数据转换。采用NTSYS 2.1(Rohlf,2000)软件计算SM相似指数并进行聚类UPGMA (unweighted pair group method arithmetic averages)分析。
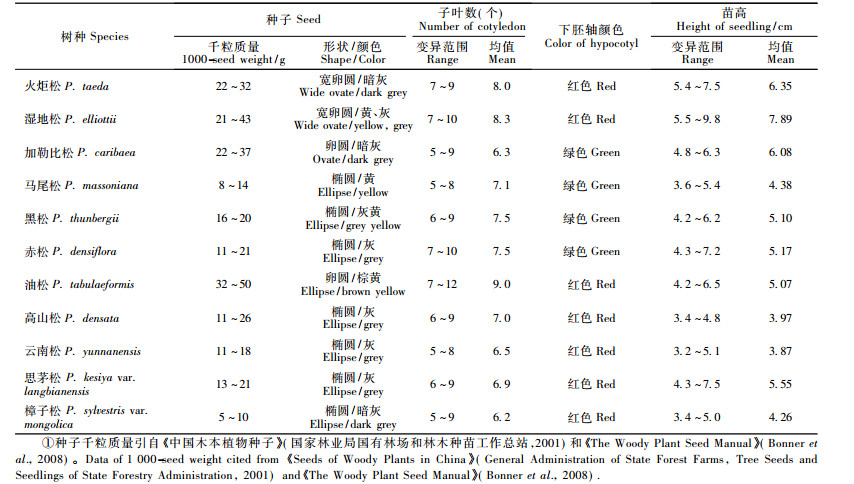
2 结果与分析 2.1 种子形态和幼苗表型性状的特异性11个树种种子及幼苗的形态特征描述见表 2。油松组树种的种子从形态上很难区分。从表 2可知,只有加勒比松(P. caribaea)和油松种子的形状和颜色较易分辨,可以作为鉴定的依据;而其他树种基本形状为椭圆形,颜色有黄、灰、黑色,或者一个种批内混杂着几种颜色,或同种不同种源间颜色差异也十分显著,因此难以作为鉴定依据。
|
|
种子千粒质量是衡量种子大小的重要指标,尽管不同种源和不同年份的种批千粒质量差异较大,但其变异不足以影响种间差异。从表 2可知,油松、火炬松、湿地松和加勒比松的千粒质量均在20 g以上,其他树种则在20 g以下。由此可根据千粒质量(20 g为界)把11个松属树种分为2大类:大粒类种子包括油松、火炬松、湿地松和加勒比松;小粒类种子有马尾松、黑松、云南松、高山松、思茅松(P. kesiya var. langbianensis)、赤松(P. densiflora)和樟子松(P. sylvestris var. mongolica)。
从幼苗的特性看,各种间差异主要体现在胚轴的颜色、子叶数及苗高。
松属树种种子萌发初期幼苗下胚轴颜色存在差异。观察发现下胚轴颜色为绿色的树种有加勒比松、马尾松、黑松和赤松;其余的均表现为红色,并在整个萌发期间颜色不变。
与被子植物不同的是裸子植物的子叶数在种内并不是一定的,种内个体间变异很大。观察表明,油松的子叶数目变异范围和平均值最大,分别达到7~12和9.0,可明显区别于其他种;其次是火炬松和湿地松,其均值都达到8.0以上。而且,各树种不同子叶数个体所占的比例也有明显差异(图 1A)。
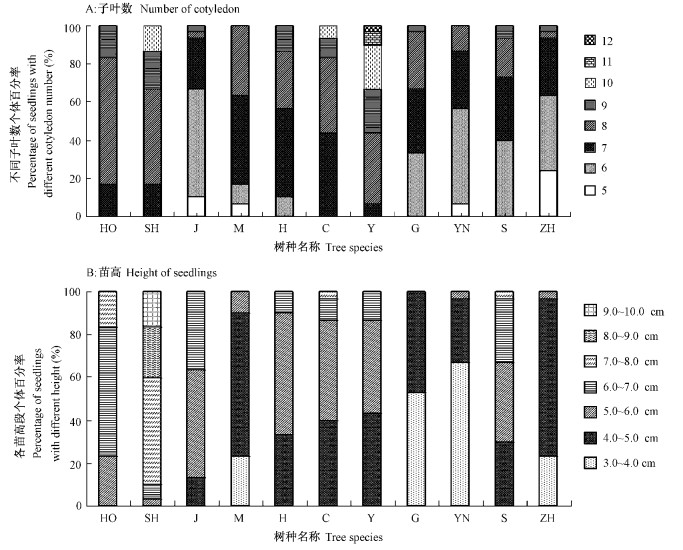
|
图 1 各树种不同子叶数和苗高植株的数量分布 Figure 1 Percentage of seedlings with different number of cotyledon and height HO:火炬松 P. taeda; SH:湿地松 P. elliottii; J:加勒比松 P. caribaea;M:马尾松 P. massoniana;H:黑松 P. thunbergii;C:赤松 P. densiflora;Y:油松 P. tabulaeformis;G:高山松 P. densata;YN:云南松 P. yunnanensis;S:思茅松 P. kesiya var. langbianensis;ZH:樟子松 P. sylvestris var. mongolica.下同。The same below. |
幼苗高度在种间也差异明显,在种内不同种源间则受到种子千粒质量的影响。从表 2中可以看出,尽管种内个体间的变异很大,但种群的平均水平在种间存在明显差异。火炬松、湿地松和加勒比松(均值分别为6.35,7.89,6.08 cm),明显高于油松(均值为5.07 cm);而分布于高海拔地区的云南松和高山松的苗高平均值分别为3.97 cm和3.87 cm,明显区别于其他树种(表 2)。各树种的苗高分布情况差异也较大(图 1B),如火炬松和湿地松苗高达6 cm以上的个体达70%以上,其群体明显高于其他种;其他树种有80%左右的植株苗高在4.0~6.0 cm之间。
综合种子和幼苗形态,可将11种松属树种中的油松、加勒比松鉴定出来。油松种子大、椭圆形/棕黄色、子叶数平均高达9个,但其苗高在大粒类种子中最小;加勒比松种子大、三角形、暗灰色,大粒类种子中只有其幼苗的下胚轴呈绿色。
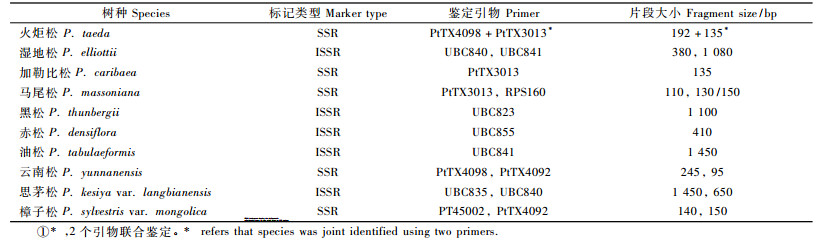
2.2 分子检测 2.2.1 引物筛选利用分群法(BSA)筛选获得种内保守、种间有变异的5对SSR和5条ISSR引物(表 3)。
|
|
根据种质鉴定的要求,只对在种内保守而在种间有变异的特异性引物进行扩增鉴定(图 2,图 3)。利用5对SSR引物和5条ISSR引物对混合基因池和个体基因组的扩增检测,其中有9个树种获得了特异性扩增产物,火炬松需2个引物的2个特异性条带可联合鉴定出来,仅有高山松没有获得特异性产物(表 4)。
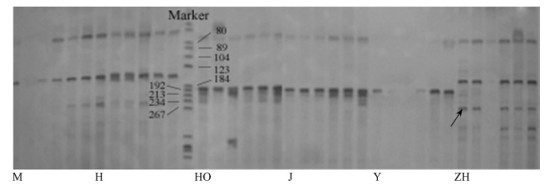
|
图 2 SSR引物PtTX4098鉴定部分松属树种 Figure 2 Pinus species identified using SSR primer PtTX4098 每6个泳道为同一树种的6个个体。 Every 6 lanes were 6 seedlings from same species. |
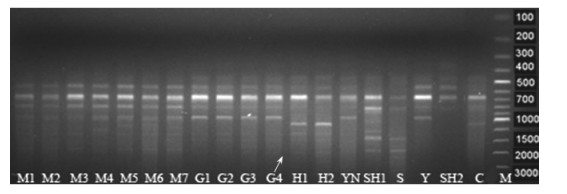
|
图 3 ISSR引物UBC-855对部分松属树种的鉴定 Figure 3 Pinus species identified using ISSR primer UBC-855 每个泳道为1个种源的混合基因池。 Each lane refers mixed gene pool from one provenance of same species. |
|
|
通过5对SSR引物对所有材料的扩增产物分析,共获得48个等位基因,其中种内保守、种间多态性位点有41个,多态性比例为85.4%。利用NTSYS 2.0计算10个松属树种间的相似系数,用UPGMA法对其进行系统聚类(图 4)分析,在相似系数0.60处将10个树种分为3个类群。从图 4中可知,马尾松和赤松聚为第1个类群;亲缘关系较近的云南松、思茅松、高山松、油松聚成一小类,和另一小类即黑松、樟子松聚成第2个类群,它与第1类群关系较近又相对独立;第3类群为火炬松和加勒比松,它与前2个类群亲缘关系较远,其相似系数仅为0.42。

|
图 4 基于SSR标记构建的10个松属树种亲缘关系 Figure 4 UPGMA dendrogram showing the phylogenetic relationship among 10 pine trees based on SSR markers |
松属(油松组)大部分树种为我国的主要造林树种,具有很高的生态和经济价值,如马尾松、油松、湿地松等。但由于松属树种进化历史相同且伴随广泛的基因交流,近缘种的短期隔离不足以产生基因库的显著分化(Bucci et al., 1998),因而许多种间的系统学关系模糊不清。松属许多种的种子、幼苗和成年树的形态特征高度相似;细胞水平上,松属(油松组)树种具有相同的染色体数目2n=24和极其相似的核型(吴若菁等,1994),因而无论从形态还是细胞水平上进行种质鉴定都存在一定的困难。
形态鉴定之难在于种间的差异较小,或者由于种间分布区重叠,种间发生着不同程度的基因渐渗杂交(虞泓等,2000;Li et al., 2010),从而使得种间的分类界限模糊,分类鉴定困难;同时有些树种分布范围广,种内常因种源、立地条件、不同年份气象因子而产生更大的差异(虞泓等,2000;Rehfeldt, 1991),这也使得一些种或变异类型的分类学地位长时间处于混乱状态,如高山松和巴山松(P. henryi)(杨雪等,2007)。本研究发现,11个松属树种的种子和幼苗形态存在一些差异,结合种子形状、颜色、千粒质量和幼苗子叶数、下胚轴颜色、苗高,仅可把油松、加勒比松鉴定出来,其余的树种由于没有特异的形态特征,难以鉴定。
鉴于形态鉴定的困难,利用分子技术从基因组水平上进行鉴定被广泛接受,如RAPD可区分马尾松和黄山松,AFLP鉴定油松、云南松、黄山松和黑松(李淑娴等,2003)。但这些研究所涉及的树种少,同时没有真正找到种特有性引物或种特异性位点来进行快速有效的鉴定。
利用SSR标记鉴定近缘种及杂种具准确、快速、简单的优点,现已被广泛接受(张红莲等,2010),但其引物大部分来源于近缘种。SSR引物的通用性取决于近缘分类群之间微卫星侧翼序列的保守性和进化过程中微卫星的稳定性。遗传距离近的物种SSR引物的通用性更高,近90%的亚属间转移都获得成功,但其通用率的高低仍是较大的困惑。González-Martínez等(2004)研究发现油松组的通用率为36%~59%,稍高于亚组Pineae和Pnaster 32%~45%;Hodgetts等(2001)利用白云杉(Picea glauca)的5对引物验证在属内的通用率较高,但在松属中无扩增产物。本研究结果表明,尽管来自于松属树种的SSR引物在油松组的通用率不高,但仍有5对引物符合种内保守种间存在变异的要求、同时在10个树种中获得扩增产物(其中湿地松由于在种内变异大,研究时放弃了该树种),并产生9个种特异性位点,可有效鉴定出马尾松、火炬松、加勒比松、云南松、樟子松;结合5个ISSR引物扩增出的7个种特异性位点,可鉴定出湿地松、黑松、赤松、思茅松、油松。综合2种标记方法,可把11个松属树种中的10个鉴定出来,仅有高山松没有获得种特异性位点,这可能与高山松复杂的遗传背景有关。Song等(2002;2003)通过对父系遗传的叶绿体DNA和母系遗传的线粒体DNA的对比分析,发现高山松有些居群的起源是以油松为母本云南松为父本的组合,有的则正好相反;或是由于高山松与同一分布区内云南松和思茅松发生了基因渐渗杂交所致(虞泓等,2000),其种子的形状、颜色、千粒质量和苗高与云南松相似,而幼苗的子叶数则介于油松和云南松之间。因此从分子水平上难以鉴定。
3.2 松属(油松组)树种亲缘关系探讨随着分子生物学的发展,DNA序列变异越来越多地应用于研究物种的进化关系,因为它直接反映了遗传物质的变异。利用cpDNA和mtDNA进行系统发育的研究结果和传统的形态分类基本一致(Eckert et al., 2006; Geada López et al., 2002;卢孟柱等,1999)。
本研究首次利用松属树种开发的SSR引物,对松属(油松组)树种进行了系统发育的研究。利用5对SSR引物获得的41个多态性位点构建的10个树种的系统进化关系与传统的分类地位比较一致。其中云南松、思茅松、高山松和油松表现出亲缘关系很近,聚为一小类。来自形态学、解剖学、等位酶、cpDNA的证据都表明高山松是云南松和油松的天然杂种(管中天,1981;Wang et al., 1990a; 1990b; 1994);大量研究表明云南松、油松、高山松和思茅松的遗传距离很小(虞泓等,2000;卢孟柱等,1999;Eckert et al., 2006),说明其遗传分化并不明显,其原因在于它们在地理位置上的相互交错,且存在着广泛的基因渗入。从10个松属种的系统发育关系来看,马尾松和赤松占据中间类型,它们可能是几个近缘种中分化较早的原始类型,但进化缓慢;而进化较快的油松类群则是近1 000万年内物种扩散、生态隔离的结果(Eckert et al., 2006);火炬松和加勒比松成为独立的类群,与二者起源于北美东南部密切相关,也与Eckert等(2006)的研究结果一致。但本文的研究结果显示赤松和马尾松亲缘关系较近,而与樟子松的关系较远,这与Eckert等(2006)的结果存在差异。研究认为樟子松为欧洲赤松分布至远东的地理变种,因此赤松与樟子松的关系应更近。这可能是由于本研究的种子只来源于山东,而其主要分布区则在东北地区,因而结果存在一定的偏差;同时分子标记类型也可能产生一定的影响。
用于研究生物系统进化的DNA序列大都为相对比较保守的cpDNA,mtDNA和nrDNA序列,而SSR较少用于种的亲缘关系探讨,一是它在种内就存在很高的变异,二是在种间的通用率较低。本研究通过大量筛选,仅获得5对SSR引物,但分析结果与文献报道结果基本一致,表明种内保守种间有变异的SSR引物可应用于系统进化分析。但需要注意的是每个种要尽可能涵盖所有的地理种源,进行全面的居群遗传学研究,在了解种间基因交流及物种的生态隔离机制之后,才能正确阐明松属(油松组)树种的系统进化规律和亲缘关系。
GB 2772-1999. 林木种子检验规程. 国家质量技术监督局.
|
管中天. 1981. 四川松杉类植物分布的基本特征[J]. 植物分类学报, 19(4): 393-407. |
国家林业局国有林场和林木种苗工作总站. 2001. 中国木本植物种子[M]. 北京: 中国林业出版社: 171.
|
李淑娴, 尹佟明, 邹惠渝, 等. 2003. 利用分子标记技术对林木近缘种进行遗传鉴别的研究[J]. 林业科学, 39(3): 129-135. DOI:10.11707/j.1001-7488.20030321 |
卢孟柱, SzmidtA E. 1999. 松属线粒体基因序列变异研究[J]. 林业科学, 35(4): 14-20. |
汪小全, 洪德元. 1997. 植物分子系统学近五年的研究进展概况[J]. 植物分类学报, 35(5): 465-480. |
吴若菁, 卢芳作. 1994. 松属51个种间的核型进化初探[J]. 福建林学院学报, 14(1): 73-76. |
吴学尉, 崔光芬, 吴丽芳, 等. 2009. 百合杂交后代ISSR鉴定[J]. 园艺学报, 36(5): 749-754. |
杨雪, 刘占林. 2007. 巴山松分类学地位研究—基于cpSSR和AFLP分子标记. www.paper.edu.cn/index.php/default/releasepaper/200704-155.
|
邢有华, 方永鑫, 吴根荣. 1992. 安徽大别山和黄山松天然杂交的初步研究[J]. 安徽林业科技, (4): 5-9. |
虞泓. 1996. 云南松遗传多样性与进化研究. 云南大学博士学位论文.
|
虞泓, 葛颂, 黄瑞复, 等. 2000. 云南松及其近缘种的遗传变异与亲缘关系[J]. 植物学报, 42(1): 107-110. |
张红莲, 李火根, 胥猛, 等. 2010. 鹅掌楸属种及杂种的SSR分子鉴定[J]. 林业科学, 46(1): 36-39. DOI:10.11707/j.1001-7488.20100106 |
Balasaravanan T, Chezhian P, Kamalakannan R, et al. 2005. Determination of inter-and intra-species genetic relationships among six Eucalyptus species based on inter-simple sequence repeats (ISSR)[J]. Tree Physiology, 25(10): 1295-1302. DOI:10.1093/treephys/25.10.1295 |
Bonner F T, Karrfalt R P. 2008. The woody plant seed manual[J]. Washington DC: USDA, Forest Service, Agric Handbk 727: 833. |
Bucci G, Anzidei M, Madaghiele A, et al. 1998. Detection of haplotypic variation and natural hybridization in halepensis-complex pine species using chloroplast simple sequence repeat (SSR) markers[J]. Molecular Ecology, 7(12): 1633-1643. DOI:10.1046/j.1365-294x.1998.00466.x |
Chase M W, Soltis D E, Olmstead R G, et al. 1993. Phylogenetics of seed plants: an analysis of nucleotide sequences from the plastid gene rbcL[J]. Annals of the Missouri Botanical Garden, 80(3): 528-580. DOI:10.2307/2399846 |
Dellaporta S, Wood J, Hicks J. 1983. A plant DNA mini-preparation: Version Ⅱ[J]. Plant Molecular Biology Reporter, 1(4): 19. DOI:10.1007/BF02712670 |
Donoghue M J, Sanderson M J. 1992. The suitability of molecular and morphological evidence in reconstructing plant phylogeny//Soltis P S, Soltis D E, Doyle J J. Molecular systematics of plants. New York: Chapman and Hall, 340-368.
|
Echt C S, May-Marquardt P, Hseih M, et al. 1996. Characterization of microsatellite markers in eastern white pine[J]. Genome, 39(6): 1102-1108. DOI:10.1139/g96-138 |
Eckert A J, Hall B D. 2006. Phylogeny, historical biogeography, and patterns of diversification for Pinus (Pinaceae): Phylogenetic tests of fossil-based hypotheses[J]. Molecular Phylogenetics and Evolution, 40(1): 166-182. DOI:10.1016/j.ympev.2006.03.009 |
Elsik C G, Minihan V T, Hall S E, et al. 2000. Low-copy microsatellite markers for Pinus taeda L[J]. Genome, 43(3): 550-555. DOI:10.1139/g00-002 |
Fowler D P, Park Y S, Gordon A G. 1988. Genetic variation of red spruce in the Maritimes[J]. Canadian Journal of Forest Research, 18(6): 703-709. DOI:10.1139/x88-107 |
Furman B J, Grattapaglia D, Dvorak W S, et al. 1997. Analysis of genetic relationships of central American and Mexican pines using RAPD markers that distinguish species[J]. Molecular Ecology, 6(4): 321-331. DOI:10.1046/j.1365-294X.1997.00183.x |
Fu L K, Li N, Mill R R. 1999. Pinus// Wu Z Y, Raven P H. Flora of China. Science Press, Beijing; Missouri Botanical Garden Press, St Louis, 11-25.
|
Geada López G, Kamiya K, Harada K. 2002. Phylogenetic relationships of Diploxylon pines (subgenus Pinus) based on plastid sequence data[J]. International Journal of Plant Sciences, 163(5): 737-747. DOI:10.1086/342213 |
Gordon A G. 1976. The taxonomy and gentics of Picea rubens and its relationship to Picea mariana[J]. Canadian Journal of Botany, 54(9): 781-813. DOI:10.1139/b76-084 |
González-Martínez S C, Gerber S, Cervera M T, et al. 2002. Seed gene flow and fine-scale structure in a Mediterrantean pine (Pinus pinaster Ait.) using nuclear microsatellite markers[J]. Theoretical and Applied Genetics, 104(8): 1290-1297. |
González-Martínez S C, Robledo-Arnuncd J J, Collada C, et al. 2004. Cross-amplification and sequence variation of microsatellite loci in Eurasian hard pines[J]. Theoretical and Applied Genetics, 109(1): 103-111. DOI:10.1007/s00122-004-1596-x |
Hao G, Lee D H, Lee J S, et al. 2002. A study of taxonomical relationships among species of Korean Allium sect.Sacculiferum (Alliaceae) and related species using inter-simple sequence repeat (ISSR) markers[J]. Botanical Bulletin of Academia Sinica, 43(1): 63-68. |
Hicks M, Adams D, O′Keefe S, et al. 1998. The development of RAPD and microsatellite markers in lodgepole pine (Pinus contorta var.latifolia)[J]. Genome, 41(6): 797-805. DOI:10.1139/g98-084 |
Hodgetts R B, Aleksiuk M A, Brown A, et al. 2001. Development of microsatellite markers for white spruce (Picea glauca) and related species[J]. Theoretical and Applied Genetics, 102(8): 1252-1258. DOI:10.1007/s00122-001-0546-0 |
Johnson L A, Soltis D E. 1995. Phylogenetic inference in Saxifragaceae sensu stricto and Gilia (Polemoniaceae) using matK sequences[J]. Annals of the Missouri Botanical Garden, 82: 149-175. DOI:10.2307/2399875 |
Koc A, Akbulut M, Orhan E, et al. 2009. Identification of Turkish and standard apple rootstocks by morphological and molecular markers[J]. Genetic and Molecular Research, 8(2): 420-425. DOI:10.4238/vol8-2gmr600 |
Li S X, Chen Y, Gao H D, et al. 2010. Potential chromosomal introgression barriers revealed by linkage analysis in a hybrid of Pinus massoniana and P. hwangshanensis. BMC Plant Biology. http: //www.biomedcentral.com/1471-2229/10/37.
|
Manley S A M. 1971. Identification of red, black and hybrid spruce[M]. Canada: Canadian Forest Service Publ: 1301.
|
Michelmore R W, Paran I, Kesseli R V. 1991. Identification of markers linked to disease resistant gene by bulked segregation population[J]. Proceedings of National Academy Sciences, 88(21): 9828-9832. DOI:10.1073/pnas.88.21.9828 |
Morgenstern E K, Farrar J L. 1964. Introgressive hybridization in red spruce and black spruce[J]. Technical Report No. 4. Toronto, Ontario: Faculty of Forestry, University of Toronto: 1-46. |
Rehfeldt E G. 1991. A model of genetic variation pattern of Pinus ponderosa and its application in gene resources management[J]. Canadian Journal of Forest Research, 21(10): 1491-1500. DOI:10.1139/x91-209 |
Rieseberg L H. 1997. Hybrid origins of plant species[J]. Annual Review of Ecology and Systematics, 28(1): 359-389. DOI:10.1146/annurev.ecolsys.28.1.359 |
Rohlf F J. 2000. NTSYS-PC, numerical taxonomy and multivariate analysis system[M]. Version 2.10e. New York, USA: Exeter Software, Setauket.
|
Ross-Davis A, Huang Z, McKenna J, et al. 2008. Morphological and molecular methods to identify butternut (Juglans cinerea) and butternut hybrids: relevance to butternut conservation[J]. Tree Physiology, 28(7): 1127-1133. DOI:10.1093/treephys/28.7.1127 |
Song B H, Wang X Q, Wang X R, et al. 2002. Maternal lineages of Pinus densata, a diploid hybrid[J]. Molecular Ecology, 11(6): 1057-1063. DOI:10.1046/j.1365-294X.2002.01502.x |
Song B H, Wang X Q, Wang X R, et al. 2003. Cytoplasmic composition in Pinus densata and population establishment of the diploid hybrid pine[J]. Molecular Ecology, 12(11): 2995-3001. DOI:10.1046/j.1365-294X.2003.01962.x |
Sun J W, Jin D M, Zhou C J, et al. 2005. Identification of Porphyra lines (Rhodophyta) by AFLP-DNA fingerprinting and molecular markers[J]. Plant Molecular Biology Reporter, 23(3): 251-262. DOI:10.1007/BF02772755 |
Vendramin G G, Lelli L, Rossi P, et al. 1996. A set of primers for the amplification of 20 chloroplast microsatellites in Pinaceae[J]. Molecular Ecology, 5(4): 595-598. DOI:10.1111/j.1365-294X.1996.tb00353.x |
Wang X R, Szmidt A E, Lewandowski A, et al. 1990a. Evolutionary analysis of Pinus densata Masters, a putative tertiary hybrid.1. Allozyme variation[J]. Theoretical and Applied Genetics, 80(5): 635-640. |
Wang X R, Szmidt A E. 1990b. Evolutionary analysis of Pinus densata (Masters), a putative tertiary hybrid.2.A study using species specific chloroplast DNA markers[J]. Theoretical and Applied Genetics, 80(5): 641-647. |
Wang X R, Szmidt A E. 1994. Hybridization and chloroplast DNA variation in a Pinus species complex from Asia[J]. Evolution, 48(4): 1020-1031. DOI:10.1111/evo.1994.48.issue-4 |
Zhou Y, Bui T, Auckland L D, et al. 2002. Undermethylated DNA as a source of microsatellites from a conifer genome[J]. Genome, 45(1): 91-99. DOI:10.1139/g01-119 |
 2011, Vol. 47
2011, Vol. 47