文章信息
- 潘炜, 田佳星, 杜庆章, 张志毅, 张德强
- Pan Wei, Tian Jiaxing, Du Qingzhang, Zhang Zhiyi, Zhang Deqiang
- 毛白杨蔗糖合酶基因PtSUS1的克隆、达及单核苷酸多态性分析
- Isolation, Expression and Single Nucleotide Polymorphisms Analysis of Sucrose Synthase Gene (PtSUS1) from Populus tomentosa
- 林业科学, 2011, 47(3): 52-58.
- Scientia Silvae Sinicae, 2011, 47(3): 52-58.
-
文章历史
- 收稿日期:2010-11-23
- 修回日期:2011-01-14
-
作者相关文章
为应对人类面临的全球性能源危机和温室效应的日益加剧,新能源研发已成为世界各国优先发展的战略,纤维素生物量是最丰富的可再生资源,通过生物转化成乙醇可替代石油作液体燃料,有望解决能源短缺问题(Demirbas,2005)。而森林作为最重要的陆地生态系统,为人类生存提供了约80%的纤维质生物量(Demirbas,2005)。在树木中,纤维素含量的多少将直接影响到发酵产物乙醇的转化率和制浆造纸过程中纸浆得率(Chiang et al., 1988; Sassner et al., 2005)。为此,研究者对纤维素的生物合成过程进行了广泛研究,认为其主要由纤维素合酶复合体催化,且高度有序、多基因协同调控来完成(Joshi et al., 2004)。期间,树木将光合作用所固定的碳不可逆地汇积在主干的初生及次生细胞壁中,而蔗糖则是这一过程的主要碳源。在树木生长发育过程中,蔗糖合酶(sucrose synthase,SUS; EC 2.4.1.13)是蔗糖代谢的关键酶,该酶可逆地催化:蔗糖+ UDP⇌果糖+ UDP-葡萄糖,但在植物中它更多地催化蔗糖分解,由此形成的UDP-葡萄糖则是纤维素合酶催化合成纤维素的最直接的前体(Joshi et al., 2004)。因此,SUS就成为调控树木木材纤维素生物合成过程中碳循环的限制酶,那么在树木中克隆SUS基因并检测其表达模式,是深入研究纤维素生物合成过程,并对其进行遗传操作的基础。
而先前对SUS基因结构与功能的研究主要集中于模式植物及主要农作物上,并在这些物种中检测到了多个SUS等位酶,认为在植物中SUS属于多基因家族(Bieniawska et al., 2007; Hirose et al., 2008)。例如在模式植物拟南芥(Arabidopsis thaliana)中SUS家族包括6个成员,依据这些成员的基因组结构、表达模式及进化关系将其分为3个亚家族(Bieniawska et al., 2007)。在水稻基因组中也检测到6个SUS成员,其中4个具有组织及发育时期特异性表达(Hirose et al., 2008)。正义或反义表达SUS基因成员的转基因植株表明,SUS成员在植物纤维素合成及细胞壁形成中具有特异的调控作用。如在陆地棉(Gossypium hirsutum)中反义表达SUS基因导致纤维素结晶区含量显著减少,且阻止了棉纤维合成的起始及延长(Ruan et al., 2003)。而最近,Coleman等(2009)将陆地棉SUS基因导入杂交杨(Populus alba×grandidentata)中,转基因植株不仅显著提高了木材次生细胞壁纤维素的含量,还改变了细胞壁的超微结构。上述研究显示,植物SUS家族不同成员具有不同的结构与表达模式,是植物木材纤维素生物合成的主要限制因子。
鉴于树木纤维素对林产工业经济价值的重要性,与农作物相比,在树木中克隆SUS基因成员并研究其表达模式应具有更重要的应用价值,但在树木中还未见到关于该基因分离、表达及单核苷酸多态性(single nucleotide polymorphisms,简称SNPs)研究的报道。为此,本研究从毛白杨(P.tomentosa)成熟木质部cDNA中分离出一SUS基因,并对其进行了组织特异性表达、单核苷酸多态性及进化分析。研究结果为分离其他树种SUS成员以及进行该基因内SNP辅助树木材性育种提供了重要基础。
1 材料与方法 1.1 植物材料用于PtSUS1基因内SNP多样性分析的材料取自全国毛白杨基因库中能够最大程度地反映毛白杨天然分布范围的40株基因型个体(徐煲铧等,2009); 而用于RNA提取的材料取自1年生毛白杨无性系‘鲁毛50’盆栽苗。
1.2 总DNA的提取总DNA的提取按DNeasy Plant Mini Kits (Qiagen,Inc.,Valencia,CA,USA)描述的方法。
1.3 RNA提取和cDNA的合成毛白杨不同组织材料总RNA提取和cDNA合成按Zhang等(2010a)描述的方法进行。
1.4 PtSUS1基因检测为了分离在毛白杨成熟木质部特异表达的基因,作者所在课题组构建了毛白杨成熟木质部cDNA文库,并对ESTs克隆进行了随机测序及片段拼接,同源比较分析后获得了一全长SUS1基因的cDNA序列。
1.5 引物设计和PCR扩增根据所得的PtSUS1 cDNA序列,在其可能的开放阅读框两端设计1对寡聚核苷酸引物:PtSUS1F: 5′-GCGTTCTGCGTGTTCGTTGTGATTTGTGTT-3′; PtSUS1 R: 5′-CTCAGCAGCCGAAGTGCAAGCAATGCCGC-3′。
应用25 μL DNA聚合酶链式反应(PCR)体系,以毛白杨成熟木质部cDNA为模板,加入2.5 μL 10×buffer,1.8 μL 25 mmol·L-1 MgCl2,1 μL 10 mmol·L-1 dNTP,Taq DNA聚合酶1.0 U(以上试剂购自Promega公司),10 μmol·L-1正向和反向引物各1 μL,加适量双蒸水至25 μL。于94 ℃,3 min →(93℃,30 s →65 ℃,30 s→ 72℃,1 min),40个循环→ 72 ℃,5 min热循环条件下,扩增出长约2 700 bp的cDNA片段。
1.6 PCR产物的克隆、测序及计算机分析将PCR扩增得到的目的基因片段回收后连接于pGEM-T Easy上。连接产物转化大肠杆菌DH5ɑ,筛选阳性克隆,送公司测序。应用DNAMAN6.0软件和CLUSTALX软件包程序推导出氨基酸序列并进行NCBI检索分析,然后分别估算和预测推导蛋白质的分子量和等电点。
1.7 Realtime PCR检测PtSUS1基因在不同组织和器官中的表达模式采用ABI Primer Express 3.0软件在PtSUS1基因的3′未翻译区域(3′ Un-translated region,3′ UTR)设计能够扩增长度约为56 bp cDNA片段的Realtime PCR引物,其序列如下:PtSUS1RTF: 5′-GGAGGAAACACGGGTTGGTT-3′; PtSUS1RTR: 5′-GTGAAATGAACATTATTCTGGTT-3′。
以先前通过基因芯片表达分析获得的在杨树不同组织与器官相对稳定表达的肌动蛋白基因Actin作为内参,引物序列为ActF: 5′-CTC,CAT,CAT,GAA,ATG,CGA,TG-3′; ActR: 5′-TTG,GGG,CTA,GTG,CTG,AGA,TT-3′。定量PCR反应按照Zhang等(2010b)描述的方法进行,最后用该基因在不同组织和器官中的扩增量与内参Actin基因扩增量的比值,即相对表达量分析其表达模式。
1.8 PtSUS1基因的SNP多样性分析依据已克隆的PtSUS1基因的核苷酸序列设计基因特异的引物,以选取的40株个体的总DNA分别为模板进行PCR扩增; 将PCR扩增产物进行琼脂糖凝胶电泳分离,回收、纯化目的片段后与PGEM-T Easy载体连接,转化后挑取阳性单克隆进行序列测定,然后将每一基因片段的核苷酸序列拼接成完整的基因序列。在此基础上,组合利用MEGA4.0和DnaSP4.50.4软件对每一基因的40个序列进行分析,标出SNP位点、计算SNP频率及多样性指数; 分析同义突变、错义突变和无义突变情况。
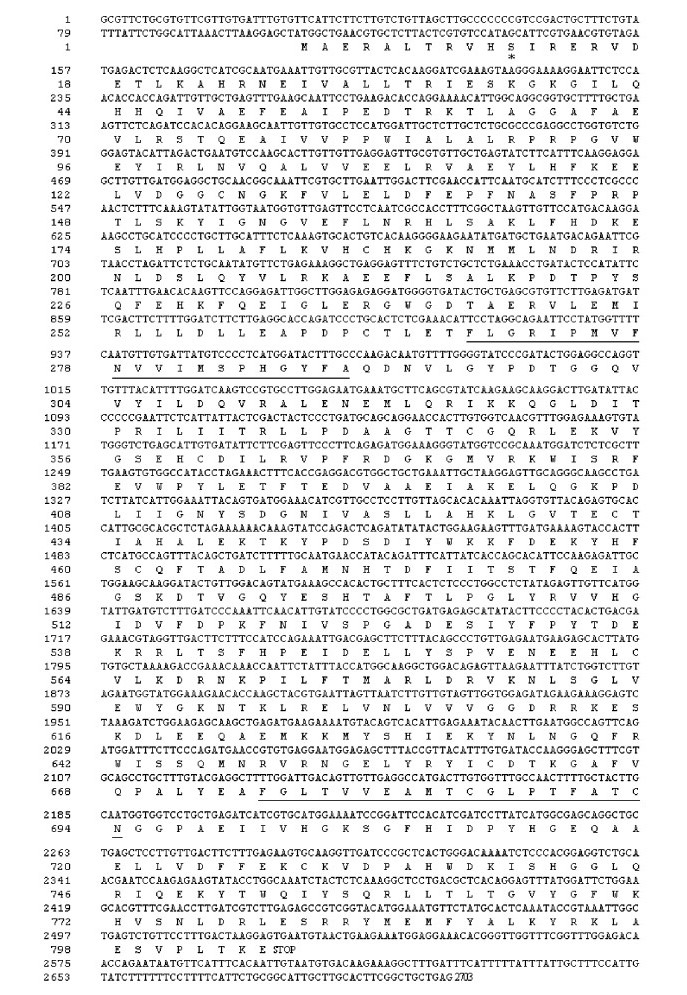
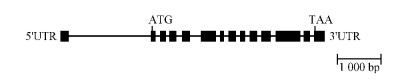
2 结果与讨论 2.1 毛白杨PtSUS1基因的克隆及其结构特征分析在前期对毛白杨成熟木质部cDNA文库中ESTs克隆随机测序及拼接的基础上,获得了一全长SUS1基因的cDNA序列,并利用基因特异的引物,以毛白杨成熟木质部总cDNA为模板进行PCR扩增。扩增产物经0.8%琼脂糖凝胶电泳分离,在大小约2 700 bp处扩增出1条明亮的特异条带。将扩增的特异片段回收、纯化并与pGEM-T Easy载体连接后克隆测序。结果表明,克隆的PtSUS1 cDNA总长2 703 bp,分别包含长度为106 bp与179 bp的5′ UTR及3′ UTR(图 1); 在PtSUS1序列中,基因内部含有完整的开放阅读框架,大小为2 415 bp,可编码长度为805个氨基酸残基的蛋白质,并将其进行GenBank提交和注册,其序列接收号为GU559727。运用DNAMAN6.0估算推导的PtSUS1蛋白质的分子量约为92.3 ku,其等电点为6.69。为分析该基因结构,克隆了PtSUS1的基因组DNA序列,与cDNA比较显示,该基因编码区域被12个内含子所分割,每一内含子长度为84~249 bp(图 2)。在该基因的5′ UTR区域,即在起始密码子ATG上游第5个核苷酸处存在1个长度为1 893 bp的内含子。因此,在该基因的转录单位共含有13个内含子,所有内含子的5′供体及3′受体信号均遵循内含子的“GT-AG”剪切位点法则。
|
图 1 毛白杨PtSUS1基因核苷酸及其推测的蛋白质序列 Figure 1 Nucleotide and deduced amino acid sequences of PtSUS1 * :蛋白磷酸化位点丝氨酸Protein phosphorylation site of Ser; 下划线Underline:跨膜结构域Transmembrane domain. |
|
图 2 毛白杨PtSUS1基因的结构示意 Figure 2 The schematic structure of the PtSUS1 |
将PtSUS1基因序列进行NCBI检索,结果初步表明PtSUS1是毛白杨中首次克隆的SUS基因。与先前在模式植物与主要农作物中克隆的SUS基因的核苷酸及其蛋白质序列同源性比较显示,PtSUS1与双子叶植物如拟南芥、番茄(Lycopersicon esculentum)及单子叶植物如水稻(Oryza sativa)与玉米(Zea mays)的SUS的核苷酸及蛋白质氨基酸序列高度同源,即核苷酸同源性分别为71.1%~76.4%,氨基酸同源性为74.1%~85.7%。分析蛋白质一级结构可见,与其他植物的SUS相似,PtSUS1含有植物SUS所具有的一切保守结构域,如在N端存在糖代谢关键酶家族保守的蛋白磷酸化位点Ser11,主要作用是调控磷酸化丝氨酸与靶蛋白结构域间的互作(Winter et al., 2000) (图 1); 在其蛋白质氨基酸序列内还含有2个高度保守的跨膜结构域,1个在N端(位于氨基酸序列的269—289),1个在C端(位于675—694),其主要功能被认为是在蔗糖生物合成过程中,参与蛋白质间的互作(Winter et al., 2000) (图 1)。在PtSUS1氨基酸序列的第8—547和558—737区域分别存在蔗糖合酶及糖基转移酶功能结构域,被认为行使蔗糖合成及糖基化合物转移功能(Baud et al., 2004) (图 1)。
2.2 SUS蛋白在被子植物中的进化为了分析毛白杨PtSUS1与其他物种SUS蛋白在被子植物中的系统发育进化关系,对在NCBI注册的双子叶植物、单子叶植物及原核生物中11个物种共35个SUS成员的蛋白氨基酸序列进行了序列排列,后利用MEGA 4.0软件(Tamura et al., 2007)得到了这些SUS蛋白的系统进化树(图 3)。由图 3建立的进化树可见,所有的被子植物SUS蛋白成员形成了一独立家族,而细菌SUS蛋白聚在同一分支,且位于植物蛋白家族进化分支的基部。这一结果表明,所有的被子植物SUS基因家族成员均属于古代起源类型,推测约在20~30亿年前起源于原核生物(Lunn,2002)。以细菌为类外群,植物SUS蛋白家族可明显地分为3个亚家族,即类型Ⅰ,Ⅱ,Ⅲ。其中,Ⅰ与Ⅲ又进一步分别向双子叶植物及单子叶植物方向发生分歧,而Ⅱ仅为双子叶植物组成,且每一分支都有较高的统计支持度(图 3)。由图 3显示的SUS系统进化关系可推测,最初产生的被子植物SUS蛋白成员伴随着基因组内染色体的复制经历了一系列SUS蛋白的复制,其中在被子植物中第1次基因复制事件,即产生了Ⅰ,Ⅱ,Ⅲ亚家族成员,发生在双子叶植物与单子叶植物分歧之前,约在2亿年前(Mitchell-Olds et al., 2002)。随后在Ⅰ,Ⅱ及Ⅲ亚家族内发生的基因复制事件中,有些成员出现在双子叶或单子叶内物种分化之前(3个亚家族中的AtSUSs / PsSUSs,StSUSs / LeSUSs及OsSUSs / ZmSUSs成员),而有些则出现在物种分化之后(亚分支中的某些SUS成员)。有趣的是,在3个亚家族中,Ⅰ包含了被子植物SUS家族成员的78.0%,Ⅱ与Ⅲ成员则少于22.0%,这一结果建议,Ⅰ,Ⅱ与Ⅲ是以不同的进化速度形成了现在的被子植物SUS成员。
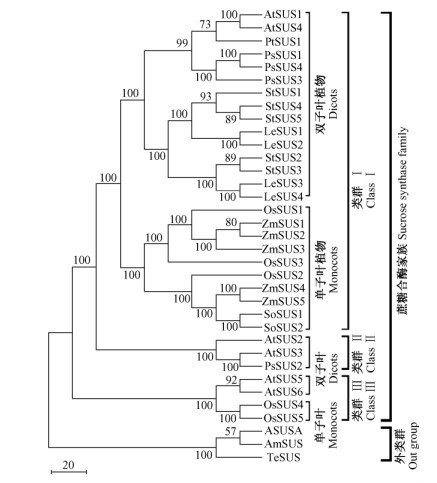
|
图 3 PtSUS1蛋白与其他物种SUS的系统发育进化树 Figure 3 The phylogenetic tree of PtSUS1 with SUSs of the other species At:拟南芥Arabidopsis thaliana; Ps:豌豆Pisum sativum; St:马铃薯Solanum tuberosum; Le:番茄Lycopersicon esculentum; Os:水稻Oryza sativa; Zm:玉米Zea mays; So:甘蔗Saccharum officinarum; Pt:毛白杨Populus tomentosa; A:项圈藻Anabaena; Te:耐热菌Thermosynechococcus elongates; Am:海藻Acaryochloris marina. |
为了检测PtSUS1基因在杨树不同组织与器官中的表达模式,利用该基因3′ UTR内设计的Realtime-PCR引物,对该基因进行了杨树组织器官特异性表达分析(图 4)。由图 4可见,PtSUS1基因在杨树根、茎、叶片和顶端分生组织中均有表达,但其表达模式不同:在成熟木质部表达丰度最高(0.907 2),而在成熟叶片、根部及正在发育的木质部表达量中等,在顶端分生组织、嫩叶、形成层组织及韧皮部表达丰度最低。比较分析显示,PtSUS1基因在杨树次生微管组织如成熟木质部和正在发育中木质部的表达量约为在形成层中的6~13倍。先前在陆地棉纤维发育及纤维素生物合成过程中也检测到了GhSUS(与杨树PtSUS1高度同源)的高丰度表达(Ruan et al., 2003),而最近有研究者将GhSUS在杨树中过量表达,结果不仅提高了转基因植株次生细胞壁纤维素的含量,还改变了次生壁的超微结构(Coleman et al., 2009)。这些研究结果都表明了植物SUS同源基因参与了次生木质部的发育过程和次生细胞壁的加厚,显示了该基因在木质纤维木材形成过程中的重要调控作用。
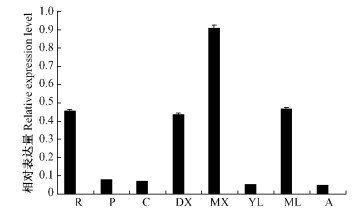
|
图 4 PtSUS1基因的组织表达谱分析 Figure 4 Relative transcript abundance of PtSUS1 in different tissues and organs R:根Root; P:树干韧皮部Phloem of stem; C:树干形成层Cambium of stem; DX:树干正在发育的木质部Developing xylem of stem; MX:树干成熟木质部Mature xylem of stem; YL:嫩叶Young leaf; ML:成熟叶Mature leaf; A:顶端分生组织Apex. |
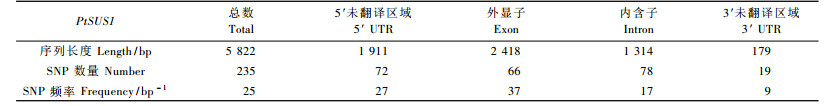
检测PtSUS1基因在自然群体中的SNP多样性不仅是进行“基于候选基因内SNP连锁不平衡作图”的基础,也是获知该基因是否在种内演化过程中遭受自然选择影响的关键。为此,以选取能够最大范围覆盖毛白杨生长区域的40株基因型个体为材料,分别对包含长度为1 911 bp的5′ UTR、2 418 bp的外显子、1 314 bp的内含子和179 bp的3′ UTR等共5 822 bp的核苷酸序列进行了测序及SNP变异分析(表 1)。由表 1可见,在PtSUS1基因的5′ UTR、外显子、内含子和3′ UTR分别检测到了72,66,78和19个SNPs,其出现频率分别为1 /27 bp,1 /37 bp,1 /17 bp和1 /9 bp。因此,在PtSUS1基因内部共检测到235个SNPs,平均频率为1 /25 bp(表 1)。由SNP在PtSUS1基因内部出现的频率可知,在毛白杨自然繁衍过程中,该基因不同区域的保守性不同,而在编码区域,其核苷酸变异程度最低,显示了PtSUS1基因在进化过程中编码区域受到了较强的选择压(Suha et al., 2005)。
|
|
为了检测PtSUS1编码区内核苷酸位点的改变是否影响了其编码氨基酸的序列,在分析SNP多样性的基础上进行了氨基酸的多态性分析。分别对PtSUS1编码区内的66个SNPs进行了同义突变、错义突变和无义突变分析(表 2)。在PtSUS1编码区内的66个SNPs中,55个属于同义突变,10个为错义突变,1个为无义突变。在错义突变中,有8个位于密码子的第1个核苷酸位置上,2个位于密码子的第2个位置上。其中,6个属于罕见SNPs,4个为常见SNPs,这些常见SNPs对于“基于候选基因内SNP的连锁不平衡作图”是非常有用的。因此,进一步分析了它们的氨基酸变异,如位于外显子5中的常见错义突变,其密码子由原来的ACT突变为GCT,导致对应的苏氨酸(Thr)突变为丙氨酸(Ala); 在外显子7中检测到2个常见错义突变位点,均位于密码子的第2个核苷酸位置上,其密码子分别由GCA突变为GTA,AAA突变为AGA,分别导致编码的丙氨酸突变为缬氨酸(Val),赖氨酸(Lys)突变为精氨酸(Arg); 在外显子10中检测到1个常见错义突变,密码子由GAG突变为CAG,导致相应谷氨酸(Glu)突变为谷氨酰胺(Gln)。因此,在毛白杨PtSUS1编码区内部发生的非同义突变与同义突变的比率为0.20 < 1,由这一结果可推测在该基因进化过程中,纯化选择对其非同义位点起了非常重要的作用(Fu et al., 1993)。
|
|
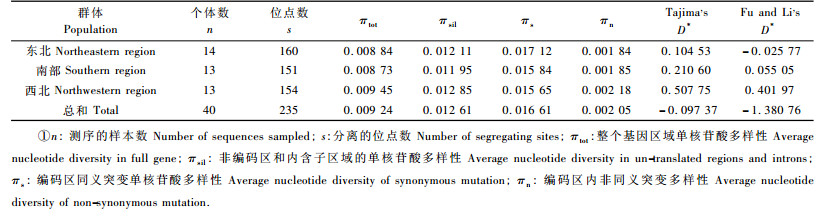
毛白杨为我国的乡土树种,在长期的物种演化过程中,形成了3个相对独立的气候与地理区域,但对于区域内及区域间的SNP遗传多样性及遗传分化还未见报道。为此,对其分别进行了单核苷酸多样性、Tajima's D* (Tajima,1989)和Fu and Li's D* (Fu et al., 1993)的中性检验(表 3)。对于检测的SNP多样性指数如πtot,πsil,πs和πn在3个群体间虽有不同但显示了相似的结果,表明在毛白杨进化过程中3个区域受到了相似的选择压。总体上看,利用Tajima's D*和Fu and Li's D*进行的中性检验结果表明,D*均为负值,这显示了在整个群体内,PtSUS1在进化过程中存在过剩的罕见SNP,特别在东北群体内存在较多的低频率SNPs。对于3个群体来说,均为πn < πs,即非同义突变与同义突变的比率 < 1,这一结果显示了在毛白杨物种演化过程中,是纯化选择,而不是自然选择对PtSUS1基因内同义SNP位点起了主要的进化驱动作用。
|
|
1) 利用RT-PCR方法,首次从毛白杨成熟木质部cDNA文库中分离出PtSUS1全长cDNA及其基因组DNA片段,并进行了测序和序列分析,结果表明克隆的PtSUS1基因内部含有完整的开放阅读框架,大小为2 415 bp,可编码长度为805个氨基酸残基的蛋白质。推导的PtSUS1蛋白质一级结构显示,PtSUS1具有植物蔗糖合酶一切保守结构域,即在N端存在糖代谢关键酶家族保守的蛋白磷酸化位点Ser11,在第8—547和558—737区域分别存在蔗糖合酶及糖基转移酶功能结构域,且在其蛋白质氨基酸序列内含有2个高度保守的跨膜结构域,1个在N端(位于氨基酸序列的269—289),1个在C端(位于675—694)。
2) 组织特异性Realtime-PCR检测表明,PtSUS1基因在杨树根、茎、叶片和顶端分生组织中均有表达,但其表达模式不同:PtSUS1在成熟木质部表达丰度最高,在成熟叶片、根部和正在发育的木质部表达丰度中等,在韧皮部和形成层有少量表达,在嫩叶与顶端分生组织中表达丰度最低。比较显示,PtSUS1基因在次生微管组织如成熟木质部和正在发育中木质部的表达量约为在形成层中的6~13倍,由此推测,PtSUS1参与了木质部的发育过程和次生细胞壁的加厚,在木质纤维木材形成过程中具有重要调控作用。
3) 在毛白杨自然群体内,PtSUS1基因内存在丰富的SNP变异,多样性水平π高达0.009 24、SNP频率为1/25 bp,而该基因的编码区核苷酸变异程度最低,显示了PtSUS1基因在进化过程中编码区域受到了较强的选择压; 在编码区内,非同义突变与同义突变的比率 < 1,可推测,在毛白杨物种演化过程中,纯化选择是PtSUS1基因内同义SNP位点主要的进化驱动力。
徐煲铧, 杨晓慧, 李百炼, 等. 2009. 毛白杨纤维素合酶基因PtCesA4的克隆、表达及单核苷酸多态性分析[J]. 林业科学, 45(5): 1-10. DOI:10.11707/j.1001-7488.20090501 |
Baud S, Vaultier M N, Rochat C. 2004. Structure and expression profile of the sucrose synthase multigene family in Arabidopsis[J]. J Exp Bot, 55(396): 397-409. DOI:10.1093/jxb/erh047 |
Bieniawska Z, Barratt D H P, Garlick A P, et al. 2007. Analysis of the sucrose synthase gene family in Arabidopsis[J]. Plant J, 49(5): 810-828. DOI:10.1111/tpj.2007.49.issue-5 |
Chiang V L, Puumala R J, Takeuchi H, et al. 1988. Comparison of softwood and hardwood kraft pulping[J]. Tappi J, 71(9): 173-176. |
Coleman H D, Yan J, Mansfield S D. 2009. Sucrose synthase affects carbon partitioning to increase cellulose production and altered cell wall ultrastructure[J]. Proc Natl Acad Sci U S A, 106(31): 13118-13123. DOI:10.1073/pnas.0900188106 |
Demirbas A. 2005. Bioethanol from cellulosic materials: a renewable motor fuel from biomass[J]. Energy Sources, 27(4): 327-337. DOI:10.1080/00908310390266643 |
Fu Yunxin, Li Wenhsiung. 1993. Statistical tests of neutrality of mutatons[J]. Genetics, 133(3): 693-709. |
Hirose T, Scofield G N, Terao T. 2008. An expression analysis profile for the entire sucrose synthase gene family in rice[J]. Plant Science, 174(5): 534-543. DOI:10.1016/j.plantsci.2008.02.009 |
Joshi C P, Bhandari S, Ranjan P, et al. 2004. Genomics of cellulose biosynthesis in poplars[J]. New Phytologist, 164(1): 53-61. DOI:10.1111/j.1469-8137.2004.01155.x |
Lunn J E. 2002. Evolution of sucrose synthesis[J]. Plant Physiol, 128(4): 1490-1500. DOI:10.1104/pp.010898 |
Mitchell-Olds T, Clauss M J. 2002. Plant evolutionary genomics[J]. Curr Opin Plant Biol, 5(1): 74-79. DOI:10.1016/S1369-5266(01)00231-X |
Ruan Y L, Llewellyn D J, Furbank R T. 2003. Suppression of sucrose synthase gene expression represses cotton fiber cell initiation, elongation, and seed development[J]. Plant Cell, 15(4): 952-964. DOI:10.1105/tpc.010108 |
Sassner P, Galbe M, Zacchi G. 2005. Steam pretreatment of Salix with and without SO2 impregnation for production of bioethanol[J]. Appl Biochem Biotech, 124(1-3): 1101-1118. DOI:10.1385/ABAB:124:1-3 |
Suha Y, Vijg J. 2005. SNP discovery in associating genetic variation with human disease phenotypes[J]. Mutation Research, 573(1/2): 41-53. |
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 123(3): 585-595. |
Tamura K, Dudley J, Nei M, et al. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
Winter H, Huber S C. 2000. Regulation of sucrose metabolism in higher plants: localization and regulation of activity of key enzymes[J]. Critical Reviews in Biochemistry and Molecular Biology, 35(4): 253-289. DOI:10.1080/10409230008984165 |
Zhang Deqiang, Du Qingzhang, Xu Baohua, et al. 2010a. The actin multigene family in Populus: organization, expression and phylogenetic analysis[J]. Mol Genet Genomics, 284(2): 105-119. DOI:10.1007/s00438-010-0552-5 |
Zhang Deqiang, Yang Xiaohui, Zhang Zhiyi, et al. 2010b. Expression and nucleotide diversity of the poplar COBL gene[J]. Tree Genetics and Genomes, 6(2): 331-344. DOI:10.1007/s11295-009-0252-7 |
 2011, Vol. 47
2011, Vol. 47