文章信息
- 李海波, 吴学谦, 魏海龙, 付立忠, 吴庆其.
- Li Haibo, Wu Xueqian, Wei Hailong, Fu Lizhong, Wu Qingqi.
- 几种森林大型真菌纯培养菌种的RAPD及ITS分子标记鉴定
- Identification of Pure Culture for Macrofungi with RAPD and ITS Molecular Markers
- 林业科学, 2007, 43(12): 94-100.
- Scientia Silvae Sinicae, 2007, 43(12): 94-100.
-
文章历史
- 收稿日期:2006-06-14
-
作者相关文章
2. 浙江益圣菌物发展有限公司 丽水 323000
2. Lishui Edible Fungal Reasearch and Development Center Lishui 323000
外生菌根(Ectomycorrhizae)是外生菌根真菌(ectomycorrhizal fungi, ECMF)与植物根部之间形成的具有特定形态结构和功能的一种共生体。据统计,高等真菌有10目、30科、81属、535种可与280种树木形成外生菌根(于富强等,2002)。外生菌根的形成不仅可以提高寄主植物对矿质元素,特别是磷和其他微量元素的吸收功能,还具有提高植物抗逆能力、苗木生长量和造林成活率的作用(花晓梅,2001)。外生菌根菌不仅是形成菌根的菌种资源,也是食用菌中的重要资源。在我国已知的500余种食用菌中,可形成外生菌根的有352种(卯晓岚,1988)。许多珍稀、名贵的食用菌如鸡油菌(Cantharellus cibarius)、松乳菇(Lactarius deliciosus)、黑孢块菌(Tuber melanosporum)、松口蘑(Tricholoma matsutake)、美味牛肝菌(Boletus edulis)等均属外生菌根菌。
外生菌根菌长期以来一直是根据子实体的形态解剖特征来进行菌种的分类鉴定(黄亦存等,1992)。但对于一些不产生子实体或尚未找到子实体的外生菌根菌则无法进行。此外,象木腐菌一样采用经典的柯赫法则(Koch's Postulate)对外生菌根菌纯培养菌种进行真伪鉴定,回接试验的执行难度很大。因此,对外生菌根菌及其纯培养的分类鉴定需借助现代分子生物学的技术手段,即开展DNA亲菌鉴定。
与真菌表型特征相比,遗传物质DNA实际上更能客观和真实地反映其系统发育特性和亲缘关系,且不受环境条件变化的影响,在菌种鉴定和系统分类上同样可作为重要的依据。建立在PCR基础上的RAPD分子标记以其对模板DNA的用量及纯度要求不高、操作简单、经济快捷等优点在大型真菌尤其是食药用菌的分类鉴定、种质资源评估以及遗传多样性和亲缘关系分析上得到了较为广泛的应用(李继东等,2002)。利用RAPD指纹技术对外生菌根菌组织分离菌株进行亲菌鉴定在鳞盖红菇(Russula sp.)、点柄乳牛肝菌(Suillus granulatus)、细裂硬皮马勃(Scleroderma areolatum)和大红菇(Russula rubra)上有报道(孙文波等,2000;曾东方等,2001)。ITS(rDNA内转录间隔区,internal transcribed spacer)是核糖体DNA中的非编码转录间隔区,ITS作为分子标记主要是通过DNA序列测定和PCR-RFLP分析的方法用于属内种间或种内群体的系统学研究。根据文献报道,ITS已成功用于对松茸(Tricholoma matsutake)、正红菇(Russula vinos)、松乳菇、致命鹅膏菌(Amanita exitialis)、乳牛肝菌(Suillus bovinus)等外生菌根菌纯培养菌种的鉴定(沙涛等,2004;王桂文等,2004;熊涛等,2006;Zhang等,2005;佟丽华等,2005)。
在前期的资源调查中,笔者共采集到了38种源自浙江丽水地区的外生菌根菌子实体,并明确了其主要的共生树种(李海波等,2005)。其中,彩色豆马勃(Pisolithus tinctorius)和缘盖牛肝菌(Boletus appendiculatus)主要与马尾松(Pinus massoniana)、黄山松(P. taiwanensis)和赤松(P. densiflora)等松属树种共生;正红菇(Russula vinosa)主要与甜楮(Castanopsis eyrei)、栲属(Castanopsis)等壳斗科树种共生,也可与红椎(Castanopsis hystrix)形成外生菌根;白乳菇(Lactarius piperatus)可与蒙古栎(Quercus mongolicus)等栎属(Quercus)树种形成外生菌根。本文即以上述4种外生菌根菌子实体及其组织分离菌株和1对木腐菌香杯伞(Clitocybe odora)的子实体及其组织分离菌株作为研究材料,采用RAPD结合ITS2种DNA分子标记对子实体及其组织分离菌株的基因组DNA进行比较分析,通过RAPD分子数据统计、聚类分析明确分离菌株与其对应子实体间的亲缘关系,通过ITS测序比较二者的rDNA ITS区段的碱基序列,以最终明确子实体组织分离菌株是否为其纯培养菌种。
1 材料与方法 1.1 试验材料 1.1.1 子实体及分离菌株有4对外生菌根菌、1对木腐菌子实体及其分离菌株,分离菌株为当日采集的子实体组织分离培养物(表 1)。组织分离及培养采用MMN培养基(赵志鹏等,1989)。
|
|
PCR扩增所用试剂、DNA纯化试剂盒和DNA凝胶回收试剂盒均购自杭州博日科技有限公司。RAP D分析所用的10碱基S系列随机引物和rDNA ITS分析所用的ITS5/ITS4引物(White et al ., 1990)购自上海生工公司。
1.2 试验方法 1.2.1 子实体与菌丝体基因组DNA的提取及检测子实体的菌盖部分(含菌褶)来提取基因组DNA;组织分离菌株先从MMN培养基试管斜面转接到MMN培养基平皿培养,待菌丝长满平皿后,刮取平皿上的菌丝体来提取基因组DNA。基因组DNA的提取参照曾大兴(2003)的SDS-CTAB法,并使用杭州博日公司的DNA纯化试剂盒对提取的DNA进行纯化。纯化的基因组DNA先用1.5%的琼脂糖凝胶电泳定性检测,再用DNA/RNA紫外分光光度计(GeneQuant Pro,GE Healthcare公司)定量检测。
1.2.2 RAPD-PCR分析RAPD-PCR分析的扩增体系为:10×PCR Buffer 2 μL,10 mmol·L-1 dNTPs 2 μL,25 mmol·L-1 MgCl2 2 μL,5 U·μL-1 Taq DNA酶0.3 μL,0.2 μmmol·5L-1随机引物3 μL,模板DNA 3 μL(51 ng),ddH2O 7.7 μL,反应总体积为20 μL。扩增反应在杭州博日(Bioer)公司的TC-XP型扩增仪上进行,94 ℃预变性6 min后,随即进入92 ℃变性40 s、38 ℃退火40 s、72 ℃延伸2 min的循环,共进行30个循环,最后于72 ℃补平7 min,终止温度为4 ℃。以5对供试材料中的子实体DNA作为模板,通过对随机挑选的165个随机引物进行RAPD分析,从中筛选出了96个对供试材料的DNA能进行有效扩增且稳定性较好的随机引物(表 2)用于进一步分析。
|
|
ITS-PCR的扩增体系为:10×PCR Buffer 2.5 μL,10 mmol·L-1 dNTPs 2 μL,25 mmol·L-1 MgCl2 2μL,5 U·μL-1 Taq DNA酶0.3 μL,10 μmol ·L-1 ITS5/ITS4引物各1 μL,17 ng·μL-1模板DNA 3 μL,ddH2O14.2 μL,反应总体积为25 μL。扩增反应在杭州博日公司的TC-XP型扩增仪上进行,94 ℃预变性1 min后;92 ℃变性15 s,61 ℃退火15 s,72 ℃延伸1 min,共30个循环;最后于72 ℃补平5 min,终止温度为4 ℃。
1.2.4 PCR产物的检测、扩增图谱的摄取和片段长度估算采用1.5%的琼脂糖凝胶电泳检测RAPD-PCR和ITS-PCR扩增产物,采用上海培清科技有限公司的JS-380A自动凝胶图像分析仪摄取扩增图谱,采用Quantity One 1-D Analysis Softw are(Version 4.4,美国Bio-Rad公司)估算扩增产物中的片段长度。
1.2.5 rDNA ITS片段的回收、克隆及测序采用杭州博日(Bioer)公司的Biospin凝胶回收Kit,从1.5%的琼脂糖凝胶中回收鹅膏菌的ITS片段,并用GeneQuant Pro DNA/RNA紫外分光光度计(GeneQuant Pro,英国GE Healthca re公司)检测浓度。回收产物用大连宝生物工程公司的pMD-18T Vector连接,转化于大肠杆菌(E.coli)DH 5α菌株感受态细胞,经蓝白斑筛选和PCR鉴定后,每个样品均随机挑选3个阳性单克隆交付大连宝生物工程公司完成测序。
1.3 数据统计与分析方法将子实体与分离菌株的RAPD-PCR图谱中相对分子质量不同的DNA条带作为多态性状,有性状记为1,无性状记为0,转换为数字矩阵。用NTSYSpc2.10e版软件包(Exeter Software:Se tauket,NY,USA)中的Simqual程序计算子实体与对应分离菌株间的Dice相似系数,UP GMA法聚类分析,构建亲缘关系树状图。
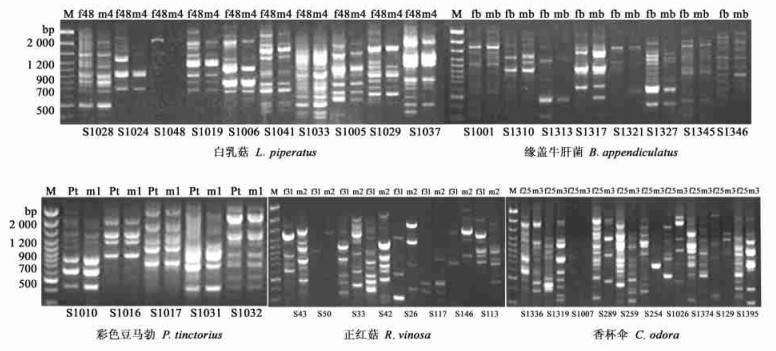
2 结果与分析 2.1 子实体及其分离菌株的RAPD多态性分析用筛选出的96个随机引物对5对供试材料进行RAPD多态性分析。结果显示,同一引物对5对供试材料以及不同引物对同一对供试材料的扩增效果均存在显著的不同,所产生的DNA片段数目、DNA片段大小以及多态性也不尽相同(图 1)。统计和片段大小测定表明:每一引物对每对供试材料扩增的条带数介于1~21条,一般为5~15条,DNA片段的大小介于0.2~3.1 kb,96个引物对5对供试材料扩增所产生的DNA片段均在200条以上。RAPD扩增所产生的多态片段数量在5对供试材料间的差异极其显著,最低的有1条,最高的达到15条。所有这些数据用于对5对供试材料进行相似性分析,构建亲缘关系树状图。
|
图 1 子实体及其组织分离菌株的RAPD-PCR图谱 Fig. 1 RAPD amplification patterns of the fruitbody and their tissue isolates M. 100bp DNA ladder.下同The same as below;泳道上方代表子实体及其组织分离菌株On top of lanes is the fruitbody and their tissue isolate s;泳道下方的S数值为随机引物编号In the bottom of lanes, the number of random primers were indicated. |
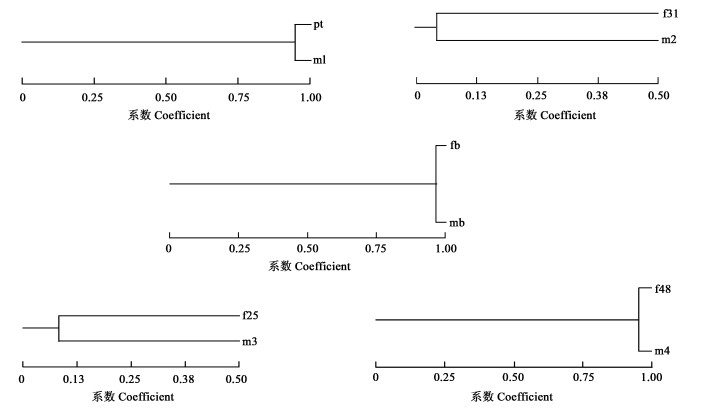
依照1.3的方法,将对5对供试材料分析获得的RAPD-PCR图谱转换为数字矩阵,应用NTSYSp c2.10e版软件包中的Simqual程序计算子实体与对应分离菌株间的Dice相似系数,用UP GMA法进行聚类分析,生成供试子实体与其分离菌株的亲缘关系树状图(图 2)。结果显示,彩色豆马勃子实体及其分离菌株(Pt-m1)、白乳菇子实体及其分离菌株(f48-m4)以及缘盖牛肝菌子实体及其分离菌株(fb-mb)分别在0.949、0.953以及0.969的Dice相似系数水平上聚为一类,而正红菇子实体及其分离菌株(f31-m2)和香杯伞子实体及其分离菌株(f25-m3)仅在0.04和0. 08的Dice相似系数水平上才聚为一类。
|
图 2 菌株间Dice系数UPGMA聚类法产生的聚类图 Fig. 2 Dendrogram resulting from UPGMA cluster analysis based on Dice coefficient |
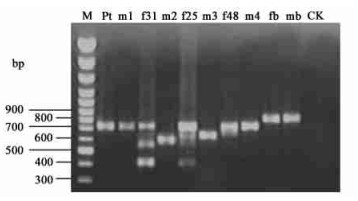
5对供试材料的ITS-PCR扩增结果(图 3)显示彩色豆马勃、白乳菇以及缘盖牛肝菌子实体及其分离菌株(Pt-m1, F48-m4, fb-mb)彼此的ITS片段长度基本一致,均介于700~850 bp。而正红菇子实体及其分离菌株(f31-m2)各有3个和1个长度不同ITS片段,香杯伞子实体及其分离菌株f25-m3也各有3个和1个ITS片段,且彼此片段的长度均不一致。
|
图 3 子实体及其分离菌株的ITS-PCR图谱 Fig. 3 ITS amplification patterns of the fruitbody and their isolates |
进一步对Pt-m1、f48-m4及fb-mb 3对供试材料的各3个ITS区段阳性单克隆进行测序,并对测序结果进行修正分析,得出二者的ITS序列。序列比较显示,3个子实体与其各自的组织分离菌株不仅ITS序列长度相同,而且碱基排列完全一致。其中Pt-m1为709 bp,f48-m4为73 2 bp,fb-mb为810 bp(图 4),这一结果再次表明彩色豆马勃、白乳菇以及缘盖牛肝菌的3个组织分离菌株皆为其各自的纯培养菌种。

|
图 4 子实体及其组织分离菌株的ITS序列图 Fig. 4 ITS sequences of the fruitbody and its isolate 两端阴影部分为ITS引物序列The shadowed part on the both ends of sequence is the ITS primer sequence. |
1) 真菌个体不同组织的DNA应该是均一性的,子实体组织与其组织分离的菌丝体是1个无性克隆系的的2种不同表达形态,应该具有完全相同的DNA组成。这是本研究利用DNA分子标记对子实体组织分离菌株进行“亲菌鉴定”的理论基础。
RAPD(William et al., 1990; Welsh et al., 1990)是一种能快速、简便而且准确地检测基因组DNA多态性的分子标记。通过选取足够多的随机引物对整个基因组进行地毯式的多态分析,理论上可揭示出2个基因组间的微小差异。引物越多,基因组DNA上被检测到的多态结合位点则越多,结果越真实可信(鲁亮等,1995)。通过优化RAPD-PCR反应体系,本研究共筛选了96个对供试材料的DNA能进行有效扩增且稳定性较好的随机引物。利用这些引物,对4个外生菌根菌、1个腐生菌子实体与其对应的组织分离菌株的基因组DNA进行RAPD指纹分析比较。分析结果显示,在所有筛选的随机引物中,彩色豆马勃、白乳菇和缘盖牛肝菌的子实体及其分离菌株彼此的RAPD指纹图谱极其相似,且皆在较高的Dice相似系数(0.949、0.953、0.969)水平上聚为一类,表明这3个子实体与其各自的组织分离菌株应该皆为同1个种;相反,在所有筛选的随机引物中,正红菇和香杯伞的子实体及其分离菌株彼此的RAPD指纹图谱几乎完全不同,且皆在极低的Dice相似系数(0.04、0.08)水平上才聚为一类,表明这2个子实体与其各自的组织分离菌株并非同一个种。
根据RAPD指纹图谱比较分析,本研究得出彩色豆马勃、白乳菇和缘盖牛肝菌的子实体及其分离菌株的Dice相似系数分别为0.949、0.953、0.969,贺冬梅等(1997)利用RAPD研究香菇不同部位子实体组织分离物与菌丝体的遗传相关性,得出二者的Dice相似系数为0.882、0. 971,相似系数均没有达到理论上1.0的水平。这表明尽管组织分离是一种无性繁殖技术,遗传变异小,但在无性繁殖过程中可能产生某些自然变异和突变,变异是绝对的,稳定是相对的。另外不容忽视的因素是,尽管RAPD标记具有经济、快捷、操作简单的优点,但RAPD同时也具有重复性和稳定性差的缺陷,也可能造成在扩增条带的判读上出现一定的偏差,进而导致相似系数偏低。
2) 真菌核糖体DNA(rDNA)的ITS区为高度可变区,进化速率较快且长度适中,在绝大多数真菌的属间和种间表现出了极为广泛的序列多态性。因而ITS序列的PCR扩增及RFLP分析一直被用于真菌的分类鉴定及系统发育关系研究(Royse et al., 1995;Bunyard et al., 1996)。为了进一步验证RAPD分子标记对5对供试材料的鉴定结果,笔者又对5对供试材料进行了rDNA ITS分析。分析结果显示,彩色豆马勃子实体及其分离菌株、白乳菇子实体及其分离菌株以及缘盖牛肝菌子实体及其分离菌株不仅ITS序列长度相同,而且碱基排列完全一致,这一结果再次表明3个组织分离菌株应该皆为其纯培养菌种。相反,正红菇和香杯伞子实体与其各自的组织分离菌株具有完全不同的ITS片段扩增图谱,体现在二者的ITS片段数量和大小皆不同,提示正红菇和香杯伞子实体中可能包含了至少三种“伴生”的微生物,而分离菌株则是另外一种微生物。
5对供试材料的rDNA ITS序列分析结果不仅与RAPD分析得出的结论相互支持,而且补充解释了正红菇和香杯伞2对材料彼此的RAPD指纹图谱几乎完全不同的机理在于其子实体中可能存在多种“伴生”的微生物,子实体的DNA模板实际上一个多种微生物的混合DNA模板,而分离菌株则是另外一种微生物的菌株。可见,对外生菌根菌子实体组织分离菌株的准确鉴定,IT S和RAPD二种分子标记的结合运用是一种高效可行的方法。
3) 研究表明,外生菌根菌鸡油菌(Cantharellus cibarius)(辛智海,1998)、腐生菌羊肚菌(Morchella sp.)(廖志勇等,2003)的子实体中皆有多种伴生细菌或真菌共存。银耳(Tremella fuciformis)的生长发育需要香灰菌的伴生(汪国莲等,2000),猪苓(Grifola umbellate)菌核的形成和持续生长发育离不开伴生菌和蜜环菌(邢晓科等,2003),研究还发现干巴菌(Thelephora ganbaju)的子实体中至少包含了毛霉、赤霉、交链孢霉等20余种真菌(杨大智等,1997)。这些伴生细菌或真菌的重要性显而易见,因此势必要借助DN A分子标记首先搞清伴生菌的数目、种类、归属、伴生菌与宿主以及多种伴生菌间的亲缘关系,进而才有可能对它们在外生菌根菌的菌根化甚至子实体形成中的具体作用进行深入研究。邢晓科等(2004)从ITS序列探讨猪苓与其伴生菌的亲缘关系,发现猪苓与其伴生菌的ITS序列同源性达99.36%。本研究结果也提示了ITS分子标记可以用来对外生菌根菌子实体中存在的伴生菌数目作出初步的评价,但要明确伴生菌的种类、归属甚至亲缘关系,尚需要做大量的研究工作,这将成为今后研究工作中一个新的方向。
贺冬梅, 陈明杰, 汪昭月, 等. 1997. 香菇子实体组织分离物与菌丝体的遗传相关性. 上海农业学报, 13(4): 85-88. |
花晓梅. 2001. 林木菌根生物工程. 世界林业研究, 14(1): 22-29. DOI:10.3969/j.issn.1001-4241.2001.01.004 |
黄亦存, 沈崇尧, 裘维蕃. 1992. 外生菌根的形态学、解剖学及分类学研究进展. 真菌学报, 11(3): 169-181. |
李海波, 吴学谦, 魏海龙, 等. 2005. 浙江丽水地区外生菌根菌资源调查初报. 中国食用菌, 24(5): 10-14. DOI:10.3969/j.issn.1003-8310.2005.05.003 |
李继东, 林跃鑫. 2002. RAPD技术在我国食用菌研究中的应用. 中国食用菌, 21(6): 6-8. DOI:10.3969/j.issn.1003-8310.2002.06.002 |
廖志勇, 苟才明, 曾先富, 等. 2003. 羊肚菌的人工驯化栽培研究初报. 阿坝科技, 1(71): 39-40. |
鲁亮, 归鸿. 1995. RAPD技术的特点及其在昆虫分类中的应用. 昆虫学报, 38(1): 117-122. DOI:10.3321/j.issn:0454-6296.1995.01.009 |
卯晓岚. 1988. 中国野生食用真菌种类及生态习性. 真菌学报, 7(1): 36-43. |
沙涛, 丁骅孙, 张汉波, 等. 2004. 利用松茸ITS特异性引物对松茸分离物进行鉴定. 云南植物研究, 26(5): 524-528. DOI:10.3969/j.issn.2095-0845.2004.05.008 |
孙文波, 李海鹰, 王桂文, 等. 2000. 应用RAPD鉴定红菇组织分离菌株的探索试验. 广西科学, 7(3): 222-224. DOI:10.3969/j.issn.1005-9164.2000.03.017 |
佟丽华, 连宾. 2005. 一株食用外生菌根菌乳牛肝菌的分子鉴定. 食品科学, 26(8): 318-320. DOI:10.3321/j.issn:1002-6630.2005.08.080 |
汪国莲, 陈立国, 陈明. 2000. 银耳菌丝体生长营养条件的初步研究. 中国食用菌, 20(4): 12-14. DOI:10.3969/j.issn.1003-8310.2000.04.006 |
王桂文, 孙文波. 2004. 广西红菇子实体及分离株的rDNA ITS序列分析. 广西科学, 11(3): 261-265. DOI:10.3969/j.issn.1005-9164.2004.03.025 |
辛智海. 1998. 鸡油菌及其伴生细菌的组织分离. 山地农业生物学报, 17(2): 92-95. |
邢晓科, 郭顺星. 2003. 猪苓、伴生菌及蜜环菌共培养的形态学研究. 菌物系统, 22(4): 653-660. DOI:10.3969/j.issn.1672-6472.2003.04.025 |
邢晓科, 郭顺星. 2004. 从ITS序列探讨猪苓与其伴生菌的亲缘关系. 微生物学通报, 31(2): 34-36. DOI:10.3969/j.issn.0253-2654.2004.02.008 |
熊涛, 肖满, 曾哲灵, 等. 2006. 松乳菇组织分离菌株的rDNA ITS序列分子鉴定. 微生物学通报, 33(4): 1-4. DOI:10.3969/j.issn.0253-2654.2006.04.001 |
杨大智, 朱启顺, 杨正斌, 等. 1997. 干巴菌子实体内伴生真菌的研究. 中国食用菌, 16(2): 8-9. |
于富强, 刘培贵. 2002. 外生菌根研究及应用的回顾与展望. 生态学报, 22(12): 2217-2226. DOI:10.3321/j.issn:1000-0933.2002.12.028 |
曾东方, 罗信昌. 2001. 应用RAPD分析快速鉴定外生菌根蘑菇分离物的真伪. 林业科学, 37(6): 59-65. DOI:10.3321/j.issn:1001-7488.2001.06.011 |
赵志鹏, 郭秀珍. 1989. 外生菌根真菌纯培养的生态学研究. 林业科学研究, 2(2): 136-141. |
曾大兴. 2003. 适于RAPD分析的真菌DNA提取方法. 生物技术, 13(2): 20-21. DOI:10.3969/j.issn.1004-311X.2003.02.014 |
Bunyard B A, Chaichuchote S, NichoIson M S, et al. 1996. Ribosomal DNA analysis for resolution of genotypic classes of Pleurotus.. Mycol Res, 100(2): 143-150. DOI:10.1016/S0953-7562(96)80112-2 |
Royse D J, Nicholson M S, Bunyard B A, et al. 1995. Ribosomal DNA for molecular systematics and genetic analysis of populations of edible fungi//Luo X C, Zang M.The Biology and Technology of Lentinula Mushroom. Beijing: China Agricultural Scientech Press, 121-127
|
Welsh J, McClelland M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucl Acids Res, 18: 7213-7218. DOI:10.1093/nar/18.24.7213 |
White T J, Bruns T D, Lee S, Taylor J W, 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics//Innis M A, Gelfand H, Sninsky J S, et al. PCR Protocols: A Guide to Methods and Applications. New York: Academic Press, 315-322
|
William J G K, Hanafey M K, Rubelik A R, et al. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucl Acids Res, 18: 6231-6235. DOI:10.1093/nar/18.21.6231 |
Zhang P, Chen Z H, Hu J S, et al. 2005. Production and characterization of Amanitin toxins from a pure culture of Amanita exitialis. FEMS Microbiol Lett, 252(2): 223-228. DOI:10.1016/j.femsle.2005.08.049 |
 2007, Vol. 43
2007, Vol. 43