文章信息
- 吴大强, 蔡诚, 魏国, 项艳.
- Wu Daqiang, Cai Cheng, Wei Guo, Xiang Yan
- 毛果杨全基因组NBS类型抗病基因分析
- Genome-Wide Analysis of NBS-Encoding Disease Resistance Genes in Populus trichocarpa
- 林业科学, 2009, 45(2): 152-157.
- Scientia Silvae Sinicae, 2009, 45(2): 152-157.
-
文章历史
- 收稿日期:2007-10-30
-
作者相关文章
病原菌威胁植物的生存繁殖,植物与病原菌之间存在着共进化关系(Holub,2001)。植物对外界病原菌的防御体系包括其自身固有和病原菌诱导,其中病原菌诱导的防卫系统的一个重要方面就是表达产物能特异性识别病原菌中相应无毒基因(avirulence gene,avr基因)编码产物的植物抗病基因(resistance gene,R基因)。阐明病原菌与寄主植物之间的相互作用关系,揭示植物抗病相关基因的结构、功能和作用机制,一直是植物病理学及植物抗病育种研究中的热点问题(Baker et al., 1997)。多数被识别抗病基因称为NBS抗病基因,属于物种特异抗性基因,其编码蛋白包含一个核苷酸结合位点(NBS)和亮氨酸的富集区(LRRs)。NBS区域广泛的被用于植物抗病基因的识别和分类,这个区域里面包含有一些非常保守的基序。以NBS-LRR结构编码的这一类基因(以下简称NBS-LRR基因)在植物中主要被分成2大类(Meyers et al., 1999)。一类是在编码蛋白N端包含一个TIR(Tolland interleukin-1 receptor,是与果蝇Toll蛋白及哺乳动物白细胞介素-1受体的胞外域相似的区域)结构,称作TIR-NBS-LRR类型基因或简称TNL类型基因。其他类型的NBS-LRR基因并不编码TIR结构区域(所以也称之为非TIR-NBS-LRR类型基因或者叫non-TNL类型基因),而这一类基因往往在它的N-端带有一个卷曲螺旋结构(CC,Coiled-coil),所以这些基因也称做CC-NBS-LRR类型基因或者简称CNL类型基因。
杨树是二倍体生物,基因组较小和生长速度较快(Stefan et al., 2007),因而作为一种林木模式生物研究倍受关注,其高密度的分子标记遗传图谱和物理图谱均已构建完成(Sterky et al., 2004)。而杨树的重要代表树种毛果杨(Populus trichocarpa)的全基因组测序已经完成(Tuskan et al., 2006)。杨树抗病基因作为杨树基因中的一个大家族,虽然目前已有文献报道分离出某些抗性位点(Lescot et al., 2004;Yin et al., 2004),Tuskan等(2006)也已对毛果杨抗病基因在连锁群上进行了定位,但是全基因组NBS类抗病基因的进一步分类和系统进化关系等方面的研究落后于模式生物拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)(Blake et al., 2003;Zhou et al., 2004)。本文通过生物信息学方法研究毛果杨全基因组中NBS类型的抗病基因总量,并对其进行分类和系统发生学分析,以期为加快克隆杨树的抗病基因、研究抗病机制和利用抗病基因加快林木抗病育种奠定基础。
1 材料与方法 1.1 试验材料本研究以毛果杨为研究对象,毛果杨全基因组数据和cDNA数据下载于毛果杨最新数据版本1.1。基因组网站http://genome.jgi-psf.org/Poptr1_1/Poptr1_1.home.html。
1.2 试验方法1)NBS类型基因的确认与分类 本研究在GeneBank中用抗病性关键词“resistance"、“disease"和“NBS”搜索抗病基因。将搜索到的基因通过Pfam数据库工具获取抗病基因NBS区域。再使用DNATOOLS软件进行tBLASTn(Altsxhul et al., 1990)(P value=0.001),在已建立的毛果杨基因组cDNA数据库中以抗病基因的NBS区域序列为递交序列进行BLAST寻找同源序列。BLAST结果中同源核苷酸序列通过Pfam(蛋白家族数据库,http://pfam.wustl.edu/hmmsearch.shtml)将无NBS结构的基因序列去除,结果通过CLUSTAL W(Thompson et al., 1994)方法进行多序列比对。通过Pfam和COILS(Lupas et al., 1991)推测出各个NBS类型基因的N端结构和LRR结构,根据这些结构的不同,将NBS类抗病基因进行分类。
2)NBS类型基因染色体物理定位 NBS类型基因通过NCBI数据库提供的染色体定位工具进行毛果杨染色体物理定位。首先利用NCBI中的毛果杨基因组BLAST工具进行BLAST比对,再使用“Genome View"工具,确认NBS类型基因的染色体位置。所有的NBS类型基因使用这种方法确认物理位置后,使用GenomePixelizer (http://www.niblrrs.ucdavis.edu/GenomePixelizer/GenomePix-elizer_Welcome.html)软件将所有含有完整NBS结构的标准抗病基因在毛果杨染色体上的物理位置绘制成图。
3)NBS类型基因复制分析 所有成簇NBS类型基因建立本地数据库,再将每一个簇上的基因利用DNATOOLS软件进行BLASTn比对,得出每一基因与其他基因的相似度,以确定是否存在基因复制现象。
4)NBS类型基因系统发生学分析 由于抗病基因的NBS结构的氨基酸序列具有很高保守性,根据排序结果,利用MEGA 3.1软件(http://www.megasoftware.net/)对所有毛果杨NBS抗病基因NBS结构区域氨基酸序列使用距离法(Neighbor-Joining method)(bootstrap=1 000)构建系统进化树。
2 结果与分析 2.1 NBS类型基因的鉴定在NCBI数据库中的搜索,可知双子叶植物为主的22种植物的共176个含NBS区域的核苷酸序列用于BLAST比对。经过本地BLAST,将每个递交序列的比对结果的重合序列去除,最终得到397个含有NBS结构的候选基因。然后通过对397个cDNA序列进行Pfam分析和多序列排序,去除无NBS结构区域的候选基因,最终得381个含NBS结构的基因。
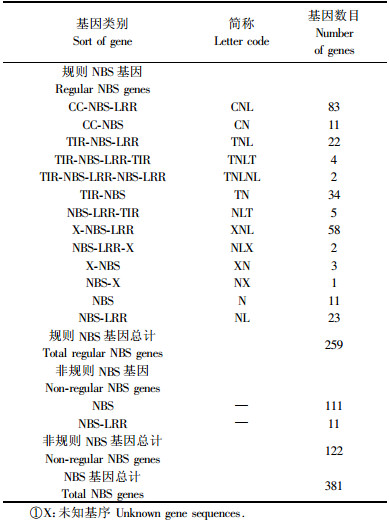
2.2 NBS类型基因的分类通过Pfam数据库共确定得到259个具有标准结构的NBS类型基因,其他122个基因虽然Pfam结果存在NBS结构但仍与标准NBS存在明显差异,主要是长度较短,所以归于非规则NBS基因。随后,标准结构NBS类型基因经过COILS和Pfam分析,共94个基因序列含有CC基序,28个基因序列含有TIR基序。
在拟南芥中,NBS类型基因可以根据它们的NBS区域清楚的划分成2大类,这样的区域划分在N端存在TIR结构或者不存在。而在毛果杨中存在大量的NBS结构不标准的NBS类型抗病基因。参考拟南芥和水稻的抗病基因分类方法(Blake et al., 2003;Zhou et al., 2004),按照N端和LRR区域基序的区别对这些NBS抗病基因进行了分类,共分为15类基因(表 1)。
|
|
图 1为标准NBS类型基因在毛果杨19条染色体上的分布,可看出这些基因在染色体上分布是不均衡的,如17号染色体无标准NBS类型基因而19号染色体有64条,未发现各类型的基因在染色体上的分布有明显规律性。根据Holub(2001)对基因簇的定义可以得出找到的基因中共169个基因(占65.3%)分布在22个基因簇中,平均每个基因簇含7.5个基因。由图可知第19号染色体基因簇最多达7个,抗病基因密度很高。除了基因簇外,还有11组两两之间分布很近的基因小簇和7组3个基因之间分布很近的基因小簇,共212个基因存在于基因簇或小组中,因此81.9%的NBS基因分布在一定的簇内。只有47个NBS类型基因单独分布于毛果杨染色体上,占标准NBS类型基因的18.1%,而拟南芥中这类基因占26.8%(Blake et al., 2003)、水稻占24.1%(Zhou et al.,2004)。因此,毛果杨基因组中单基因占NBS类型基因总数的比例比水稻和拟南芥中都小,说明毛果杨的NBS类型基因重复或重组事件的发生频率更高。

|
图 1 毛果杨NBS基因在染色体上物理位置 Figure 1 Physical locations of the NBS genes in Populus trichocarpa 染色体(灰色长条带)上方或下方的方块位置代表每一个NBS基因的物理位置。 Boxes above and below the chromosomes (chrm; gray bars) designate the approximate locations of each NBS gene. |
为证实杨树基因组中大量NBS类型基因是由于基因复制,我们确认基因复制通过以下2个标准(Gu et al. 2002):一是匹配的基因长度要大于80%,二是匹配上的区域要大于75%的序列相似性。总共18个存在复制基因的簇被确认,占22个基因簇的81.8%,如第18基因簇中的PX596330与第20基因簇的PX674676存在基因复制。这说明毛果杨抗病基因成簇分布并且大部分是由于基因复制现象造成,证实抗病基因复制是造成抗病基因在毛果杨基因组中扩充的原因。
2.5 NBS类型基因的系统发生分析分析NBS区域,毛果杨共有381个NBS类型基因,只有259个基因进行了NBS基序的系统发生分析,这是因为部分基因的NBS基序过短(小于正常的2/3长),所以没有把这一部分基因列入分析对象。
拟南芥的NBS结构域系统进化树有2个明显的分枝(Richly et al., 2002),而水稻的NBS区域没有明显簇的分类,共有192个小的分组,呈现星形的发散状系统分布(Zhou et al., 2004),毛果杨的NBS结构域的进化树(图 2)与水稻和拟南芥明显不同,按照30%的蛋白质序列差异度,杨树中共有68个分组,平均每组3.81个基因。但每组的基因数差异很大,其中32个组含1个基因,8个组含2个基因,5个组含3个基因,2个组含4个基因,21个组含5个以上的基因,最大的组含有26个基因。

|
图 2 毛果杨NBS基因NBS基序氨基酸序列系统发生学分析 Figure 2 Phylogenetic analyses of the NBS genes in Populus trichocarpa based on the amino acid sequences in the NBS domain 全局系统发生关系由MEGA 3.1软件的距离法分析NBS结构域的氨基酸序列得出。所有的259个序列包括83个CNL,11个CN,22个TNL,4个TNLT,2个TNLNL,34个TN,5个NLT,58个XNL,2个NLX,3个XN,1个NX,11个N,23个NL。 Universal phylogenetic relationship was analyzed using the neighbor-joining method based on amino acid sequences in the NBS domain by MEGA 3.1. Total 259 sequences contain 83 CNL, 11 CN, 22 TNL, 4 TNLT, 2 TNLNL, 34 TN, 5 NLT, 58 XNL, 2 NLX, 3 XN, 1 NX, 11 N, 23 NL. |
Flor(1971)提出抗病基因进化的“基因对基因”(gene for gene)假说,指出只有当植物抗病基因产物识别到病原菌无毒基因(avr)的产物之后,植物才能产生有效的抗病反应。这一进化模型的必然结果就是,病原菌在进化的压力之下形成多样性Avr蛋白,阻断其与植物中相应抗病蛋白的相互作用而逃避植物的识别,但这种多样性Avr蛋白必须能维持其核心功能而更进一步成功完成其寄生生活(Dangl et al., 2006)。相反,自然选择则倾向进化和维持植物抗病等位基因多样性从而扩大其对病原菌的特异性识别(Dangl et al., 2006)。
近年来对抗病基因的结构、功能和起源认识有了根本性的变化,基因重复是造成基因数目扩张的最主要原因(Gu et al., 2002),从图 1可知NBS基因在毛果杨不同染色体上的分布存在极大差异,17号染色体无分布,而19号染色体分布数达到64,因此,毛果杨NBS基因存在不均衡的特点。进一步分析表明NBS基因在染色体分布上存在热点分布染色体,如第19号和第1号染色体,而这些染色体上的NBS基因同源性较高,通过基因复制事件分析可知,在毛果杨的大部分抗病基因簇中,大部分是由于基因复制造成的,这与水稻、拟南芥相一致,这也进一步说明基因复制是造成抗病基因在基因组中扩张的主要原因。同时81.9%各种类型NBS基因分布在基因簇中,毛果杨NBS基因在染色体分布呈现多拷贝集中分布的特点,这与水稻(Zhou et al., 2004)和拟南芥(Richly et al., 2002)NBS基因分布的特征也非常类似,且远远大于水稻51%抗病基因位于簇里,比水稻和拟南芥有更密集的现象这种现象产生的原因可能是这些抗病基因通过基因重组、重基因复制等方式在原染色体附近位置进行扩张,以抵抗多变的病原菌,并最终形成一些染色体物理分布热点区域。
通过全基因组NBS基因系统进化树分析可以看到,毛果杨的进化树与水稻(Zhou et al., 2004)、拟南芥(Richly et al., 2002)差异较大,拟南芥有2个明显的分枝而水稻分组多达192个,毛果杨分组有68个,是介于两者之间的模式,说明杨树的抗病基因进化具有独特的特点,随着杨树抗病基因的克隆和功能验证才能得到进一步了解。
Altsxhul S F, Gish W, Miller W, et al. 1990. Basiclocal alignment search tool. J Mol Biol, 215: 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
Baker B, Zambryski P, Staskawicz B, et al. 1997. Signaling in plant-microbe interactions. Science, 276: 726-733. DOI:10.1126/science.276.5313.726 |
Blake C, Meyers B C, Alexander Kozik, et al. 2003. Genome-wide analysis of NBS-LRR-Encoding genes in Arabidopsis. Plant Cell, 15: 809-834. DOI:10.1105/tpc.009308 |
Dangl L J, McDowell J M. 2006. Two modes of pathogen recognition by plants. Proc Natl Acad Sci USA, 103: 8575-8576. DOI:10.1073/pnas.0603183103 |
Flor H. 1971. Current status of the gene-for-gene concept. Annu Rev Phytopathol, 9: 275-296. DOI:10.1146/annurev.py.09.090171.001423 |
Gu Z, Cavalcanti A, Chen F C, et al. 2002. Extent of gene duplication in the genomes of Drosophila, nematode, and yeast. Mol Biol Evol, 19: 256-262. DOI:10.1093/oxfordjournals.molbev.a004079 |
Holub E. 2001. Arms race is an ancient history in Arabidopsis, the wildflower. Nat Rev Genet, 2: 516-527. DOI:10.1038/35080508 |
Lupas A, Van Dyke M, Stock J. 1991. Predicting coled Coils from protein sequences. Science, 252: 1162-1164. DOI:10.1126/science.252.5009.1162 |
Lescot M, Rombauts S, Zhang J, et al. 2004. Annotation of a 95-kb Populus deltoides genomic sequence reveals a disease resistance gene cluster and novel class Ⅰ and class Ⅱ transposable elements. Theor Appl Genet, 109: 10-22. DOI:10.1007/s00122-004-1621-0 |
Meyers B C, Dickerman A W, Michelmore R W, et al. 1999. Plant disease resistance genes encode members of an ancient and diverse protein family within the nucleotide-binding superfamily. Plant J, 20: 317-332. DOI:10.1046/j.1365-313X.1999.t01-1-00606.x |
Richly E, Kurth J, Leister D. 2002. Mode of amplification and reorganization of resistance genes during recent Arabidopsis thaliana evolution. Mol Biol Evol, 19: 76-84. DOI:10.1093/oxfordjournals.molbev.a003984 |
Stefan J, Carl J Douglas. 2007. Populus: A Model System for Plant Biology. Annu Rev Plant Biol, 58: 435-458. DOI:10.1146/annurev.arplant.58.032806.103956 |
Sterky F, Bhalerao R R, Unneberg P, et al. 2004. A Populus EST resource for plant functional genomics. Proc Natl Acad Sci USA, 101: 13951-13956. DOI:10.1073/pnas.0401641101 |
Thompson J D, Higgins D G, Gibson T J. 1994. CLUSTAL W:Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res, 22: 4673-4680. DOI:10.1093/nar/22.22.4673 |
Tuskan G A, DiFazio S, Jansson S, et al. 2006. The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science, 313: 1596-1604. DOI:10.1126/science.1128691 |
Yin T M, DiFazio S P, Gunter L E, et al. 2004. Genetic and physical mapping of Melampsora rust resistance genes in Populus and characterization of linkage disequilibrium and flanking genomic sequence. New Phytologist, 164: 95-105. DOI:10.1111/j.1469-8137.2004.01161.x |
Zhou T, Wang Y, Chen J Q, et al. 2004. Genome-wide identification of NBS genes in Japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes. Mol Genet Genomics, 10: 1243-1254. |
 2009, Vol. 45
2009, Vol. 45