2. 中国疾病预防控制中心化学污染与健康安全重点实验室;
3. 应急管理部北戴河康复院
长链非编码RNA(Long non-coding RNA,LncRNA)是一类长度超过200 nt的RNA。LncRNA一般不具有编码蛋白的能力,通常以RNA的形式在多种层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达水平[1]。LncRNA CTBP1-AS1(CTBP1 antisense RNA1)位于人类4号染色体CTBP1基因AS区,有研究表明其作为转录辅调节因子发挥作用[2]。
甲基丙烯酸环氧丙酯(glycidyl methacrylate,GMA),又称2, 3-环氧丙烷甲基丙烯酸酯(2, 3-epoxypropyl methacrylate)。在环氧树脂、丙烯酸树脂的生产以及涂料、粘合剂中的加工与合成有广泛的应用。国际癌症研究中心近期完成了对GMA的致癌性评估,依据实验动物的“充分”证据和“强有力”的致癌机理证据被归类为“可能对人类致癌”(Group 2 A)[3-4]。研究表明,在一定的浓度范围,GMA可以诱发中国仓鼠肺细胞(V79)的微核率升高,导致V79细胞遗传损伤,具有遗传毒性[5]。王全凯等[6]的细胞毒性实验结果显示:采用(1~32) μg/mL剂量GMA染毒16HBE细胞,当GMA剂量为8 μg/mL,细胞相对存活率为93.7%;当GMA浓度高于16 μg/mL时,细胞相对存活率明显低于30%,呈现明显毒性剂量—反应关系。转化细胞生物学特性鉴定结果显示:8 μg/mL GMA染毒组第10代细胞开始对ConA的凝集敏感性增强,镜下可见细胞凝集成团但第10代未在软琼脂中形成集落,提示此时细胞膜表面的正常特性已发生改变;第30代转化细胞可在软琼脂中形成集落,而同代龄DMSO组细胞仍未形成集落,提示该细胞获得锚着非依赖性这一恶性转化的细胞生物学特征。杨敏等[7]研究结果表明:经GMA染毒处理后,16HBE细胞周期由G0/G1期向S期和G2/M期移动而呈现增殖性改变。因此选择8 μg/mL染毒细胞72 h,间隔24 h后再次染毒,连续3次,能够模拟GMA长期低剂量的暴露情况。本研究以8 μg/mL GMA作为细胞转化的染毒剂量,采用LncRNA Arraystar Human Microarray V 3.0芯片分析技术,检测GMA诱导的16HBE恶性转化过程中LncRNA表达的差异变化情况。结合LncRNA邻近编码基因信息及生物信息学手段预测LncRNA的可能靶向基因;实时荧光定量PCR(quantitative real-time PCR,qPCR)检测其表达量,探讨LncRNA在GMA诱导的16HBE恶性转化细胞中的表达及意义。
1 材料与方法 1.1 主要试剂与仪器GMA购于广州双键公司(美国陶氏化学公司,纯度>98.5%);16HBE细胞(美国加利福尼亚大学);胰蛋白酶(Tripsin 0.25%,美国Amresco);MEM培养基粉(美国Hyclone);胎牛血清(FBS,天津川页生化制品公司);双抗(美国Hyclone);EDTA-Na2及二甲基亚砜(DMSO)均购于Sigma公司;TRIzol试剂(美国Invitrogen),ViiA 7 qPCR反应体系(美国应用生物系统公司);安捷伦基因表达杂交试剂盒(5188-5242,美国Agilent)。
CO2培养箱(美国Thermo);台式高速低温离心机(德国Heraeus sepatech);小型台式常温离心机(上海安亭科学仪器厂);紫外可见分光光度计(UV-2800H,美国UNICO);7900HT Fast Real-Time PCR仪(美国Biosystems);水平电泳槽、电泳仪(中国北京六一仪器厂);凝胶成像系统(Tanon 1600,中国上海天能公司);Agilent Microarray Scanner(G2565BA,美国安捷伦);ND-1 000分光光度计(美国NanoDrop);Human LncRNA Microarray V3.0芯片(美国Arraystar)。
1.2 细胞培养在16HBE细胞的对数生长期,按照1 :3的比例接种。培养箱中培养24 h后,向培养液中加入终浓度为8 μg/mL的GMA进行染毒处理,另设1 μg/mL的DMSO作为溶剂对照。每次染毒72 h,间隔24 h后重复染毒3次后,即为第1代细胞。传代培养至第30代。收获第30代经GMA诱导的16HBE恶性转化细胞(第30代GMA染毒组细胞)与同代龄DMSO对照组细胞(第30代DMSO对照组细胞)。观察并记录细胞的生长及形态学变化。生物学特性鉴定选择ConA和软琼脂试验方法[8-9]。
1.3 LncRNA芯片分析 1.3.1 总RNA提取及质量检测使用TRIzol提取总RNA,RNAclean Kit纯化试剂盒对总RNA进行纯化处理,除去可能存在的基因污染。ND-1 000分光光度计评估总RNA的纯度,RNA的完整性由甲醛变性琼脂糖凝胶电泳进行检测。
1.3.2 cDNA的合成和标记使用安捷伦Quick Amp Labeling Kit标记逆转录生成的cDNA,与LncRNA芯片进行杂交,并使用NanoDrop检测荧光标记效率,具体实验操作步骤见参考文献[10]。
1.3.3 芯片数据采集和分析Agilent DNA Microarray Scanner扫描经过洗涤的LncRNA芯片。使用Agilent Feature Extraction(v11.0.1.1)、Agilent GeneSpring GXv12.1软件采集芯片信号值并进行标准化分析,得到LncRNA和mRNA的表达变化倍数。以差异表达倍数≥2.0为标准,筛选得到第30代GMA染毒组和DMSO对照组细胞之间差异表达的LncRNA和mRNA。
1.4 qPCR法检测LncRNA CTBP1-AS1的表达量收集第30代GMA染毒组和同代龄DMSO对照组细胞,Trizol试剂提取总RNA并进行质量检测,采用qPCR检测两组LncRNA CTBP1-AS1的表达量。使用管家基因GAPDH作为内参,以样品基因的表达量除以样品内参基因的表达量,得到的比值为LncRNA CTBP1-AS1相对含量。
10 μL ViiA 7 qPCR反应体系建立:5 μL的2 × Master Mix、2 μL的DNA模板、0.5 μL的PCR上游引物、0.5 μL的PCR下游引物以及2.0 μL的双蒸水,置于PCR反应管离心15 s。95 ℃预变性10 min,95 ℃变性10 s,60 ℃退火60 s,95 ℃延伸15 s。管家基因及LncRNA基因的引物序列见表 1。
| 基因名称 | 引物序列(5′→3′) | 产物大小(bp) |
| β-actin | 上游GGGAAACTGTGGCGTGAT | 73 |
| 下游GAGTGGGTGTCGCTGTTGA | ||
| CTBP1-AS1 | 上游ATGTAACTGAAATGTATCCCTTGAC | 126 |
| 下游ACAGAAGCAGAGGGTAGAATGG |
1.5 统计学分析
采用SAS 9.3进行统计分析,对第30代GMA染毒组和同代龄DMSO对照组细胞LncRNA CTBP1-AS1的相对表达量进行单因素方差分析。检验水准α=0.05。
2 结果 2.1 转化细胞生物学特性鉴定在GMA诱导的16HBE恶性转化过程中,分别对第10、20和30代细胞进行生物学特性鉴定。观察发现第30代GMA染毒组恶性转化细胞能够在软琼脂中形成明显集落,同时恶性转化细胞对ConA的凝集敏感性强,出现细胞凝集现象。软琼脂试验和ConA试验的结果提示,第30代GMA染毒组16HBE细胞具备恶性转化细胞的生物学特性(图 1)。
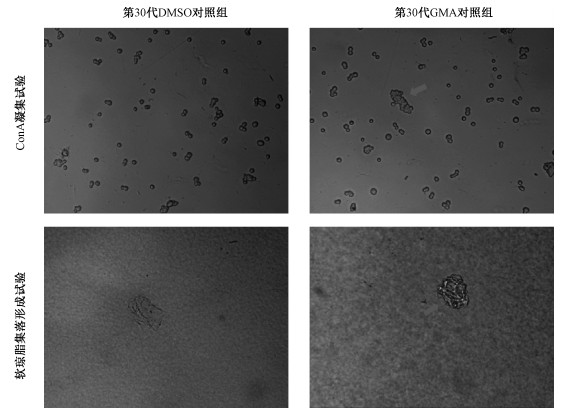
|
| 图 1 16 HBE恶性转化细胞软琼脂集落形成及ConA凝集试验(×100倍) |
2.2 LncRNA芯片分析结果
通过对第30代GMA诱导的16HBE恶性转化细胞相关差异表达的LncRNA进行筛选,与邻近编码基因进行联合分析确定出目标LncRNA CTBP1-AS1,两者的染色体位置关系见图 2。与DMSO对照组相比,GMA诱导的16HBE恶性细胞中LncRNA CTBP1-AS1上调2.20倍,其最可能的蛋白质编码基因CTBP1上调1.26倍(表 2)。
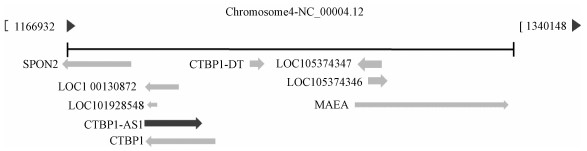
|
| 图 2 LncRNA CTBP1-AS1与CTBP1在4号染色体上的位置关系 |
| 名称 | 探针名称 | 变化情况(GMA组vs.DMSO组) | Fold change值 |
| CTBP1-AS1 | ASHGA5P051870 | 上调 | 2.20 |
| CTBP1 | ASHGA5P039298 | 上调 | 1.26 |
2.3 LncRNA CTBP1-AS1表达量的qPCR法检测结果
qPCR熔解曲线及扩增曲线提示扩增的特异性、可重复性良好(图 3)。qPCR结果显示,第30代GMA染毒组细胞的LncRNA CTBP1-AS1表达量为(91.70±0.70) ×10-5,而其同代龄DMSO对照组为(26.74±0.23) ×10-5,与芯片结果一致,GMA染毒组细胞LncRNA CTBP1-AS1的表达量明显上调,差异具有统计学意义(P < 0.05)。
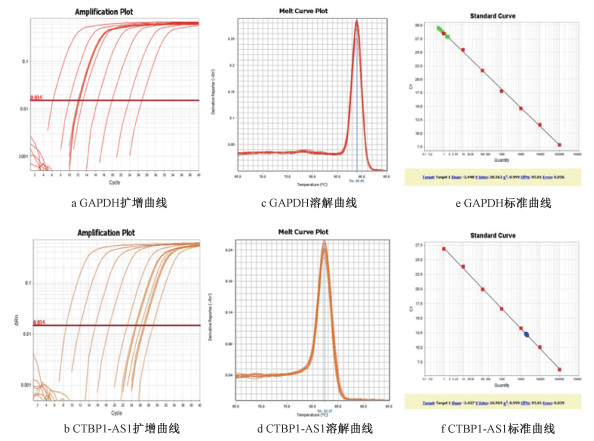
|
| 图 3 CTBP1-AS1及内参GAPDH的熔解曲线、扩增曲线和标准曲线 |
3 讨论
lncRNA是近年来研究的热点,它是一类长度大于200个核苷酸的非编码RNA,多数不具备蛋白质编码功能,曾一度被认为基因转录的“暗物质”[10],目前研究显示LncRNA可通过调节DNA甲基化、组蛋白修饰、染色质重构、mRNA降解、蛋白修饰及作为miRNA前体等多种方式发挥作用,主要参与调控细胞增殖、细胞周期、细胞分化、细胞凋亡生物学过程[11-12]。除此之外LncRNA的异常表达与包括癌症在内的多种疾病密切相关。研究发现,多种肿瘤如前列腺癌、胃癌、乳腺癌、肺癌中发现LncRNA异常表达,且这些LncRNA影响肿瘤的发生、发展、浸润、转移及复发等过程,发挥类似癌基因或抑癌基因的作用[13-14]。
Takayama等[2, 15]研究发现,LncRNA CTBP1-AS与前列腺癌的发病特征密切相关。其中LncRNA CTBP1-AS作为一种新的参与雄激素调节的LncRNA,在前列腺癌发生的过程中高表达,通过募集RNA结合转录阻遏物(PSF)和组蛋白脱乙酰基酶来直接抑制CTBP1的表达,从而促进前列腺癌的生长。此外,LncRNA CTBP1-AS被发现在乳腺癌中也发挥重要作用[16]。本研究通过LncRNA芯片和qPCR技术检测了LncRNA CTBP1-AS在GMA诱导的16HBE恶性转化细胞的表达改变。qPCR验证结果表明,第30代DMSO对照组和GMA染毒组相对表达量的结果与芯片分析结果一致,均为表达上调,提示LncRNA CTBP1-AS可能是GMA诱导16HBE细胞恶性转化中关键LncRNAs分子之一。
很多LncRNA可以通过调控邻近编码基因发挥生物学功能,与邻近基因进行联合分析可以为寻找LncRNA的功能研究提供线索。我们将差异LncRNAs与其临近(< 100 kb)的差异表达mRNAs进行整合,发现为LncRNA CTBP1-AS可能参与C-端结合蛋白1(C-terminminal binding protein 1,CTBP1)蛋白的编码调控。CTBP1是CTBP家族的重要成员之一,其在生长发育过程中具有多种功能,既可调节多种细胞过程,如调控EMT、细胞调亡、细胞周期进程、迁移、侵袭、血管形成及有丝分裂等[17],又可作为大量转录抑制因子的辅阻遏物而发挥转录辅抑制因子的功能,抑制多种肿瘤抑制基因的转录而影响肿瘤的发生发展[18-19]。本研究在通过生物学信息预测及相关文献报道初步确定CTBP1与LncRNA CTBP1-AS1可能存在调控关系。LncRNA芯片结果显示CTBP1表达上调1.26倍,同时全基因组表达谱芯片也显示CTBP1表达上调,与LncRNA芯片所获结果一致,因此推测CTBP1-AS1可能通过上调靶基因CTBP1而起作用。
在GMA诱导的16HBE恶性转化细胞中,LncRNA CTBP1-AS1可能通过上调靶基因CTBP1而起作用。因此,我们推测LncRNA CTBP1-AS1可能是GMA诱导的16HBE恶性转化细胞中相关特异分子标志之一。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Xu JY, Bai J, Zhang XX, et al. A comprehensive overview of lncRNA annotation resources[J]. Brief Bioinform, 2017, 18(2): 236-249. |
| [2] |
Takayama KI, Horie-Inoue K, Katayama S, et al. Androgen-responsive long noncoding RNA CTBP1-AS promotes prostate cancer[J]. Embo J, 2013, 32(12): 1665-1680. DOI:10.1038/emboj.2013.99 |
| [3] |
IARC Monographs Vol 125 Group. Carcinogenicity of some industrial chemical intermediates and solvents[J]. Lancet Oncol, 2020, 21(1): 25-26. DOI:10.1016/S1470-2045(19)30779-X |
| [4] |
Sharma RK, Kumar R. Functionalized cellulose with hydroxyethyl methacrylate and glycidyl methacrylate for metal ions and dye adsorption applications[J]. Int J Biol Macromol, 2019, 134: 704-721. DOI:10.1016/j.ijbiomac.2019.05.059 |
| [5] |
王全凯, 谢广云, 马顺鹏, 等. 甲基丙烯酸环氧丙酯的遗传毒性评价[J]. 癌变·畸变·突变, 2019, 31(6): 479-482. (In English: Wang QK, Xie GY, Ma SP, et al. Genotoxicity evaluation of glycidyl methacrylate[J]. Carcinog Teratog Mutagen, 2019, 31(6): 479-482.) |
| [6] |
王全凯, 谢广云, 刘红梅, 等. 甲基丙烯酸环氧丙酯致16HBE细胞恶性转化不同时期METTL9基因甲基化状态分析[J]. 癌变·畸变·突变, 2015, 27(6): 432-436. (In English: Wang QK, Xie GY, Liu HM, et al. METTL9 methylation status in malignant transformation of 16HBE cells induced by glycidyl methacrylate[J]. Carcinog Teratog Mutagen, 2015, 27(6): 432-436.) |
| [7] |
杨敏, 王全凯, 王雅文, 等. 甲基丙烯酸环氧丙酯对人支气管上皮细胞周期及凋亡影响的研究[J]. 卫生研究, 2007, 36(4): 404-406,413. (In English: Yang M, Wang QK, Wang YW, et al. Study on effect of glycidyl methacrylate on cell cycle and apoptosis in human bronchial epithelial cells[J]. J Hyg Res, 2007, 36(4): 404-406,413.) |
| [8] |
刘红梅. GMA诱导的16HBE恶性转化细胞相关差异LncRNA筛选及其研究[D].北京: 中国疾病预防控制中心, 2016. (In English: Liu HM. Screening and study on differentially expressed LncRNA in malignant transformation of 16HBE cells induced by glycidyl methacrylate[D]. Beijing: Chinese Center for Disease Control and Prevention, 2016.)
|
| [9] |
刘红梅, 王全凯, 谢广云, 等. 甲基丙烯酸环氧丙酯致16HBE细胞恶性转化中OPCML基因甲基化研究[J]. 中华劳动卫生职业病杂志, 2015, 33(11): 812-815. (In English: Liu HM, Wang QK, Xie GY, et al. Anti-oncogene of opioid binding protein/cell adhesion molecule-like methylation status in malignant transformation of human bronchial epithelial cells induced by glycidyl methacrylate[J]. Chin J Ind Hyg Occup Dis, 2015, 33(11): 812-815.) |
| [10] |
Schmitt AM, Chang HY. Long noncoding RNAs in cancer pathways[J]. Cancer Cell, 2016, 29(4): 452-463. DOI:10.1016/j.ccell.2016.03.010 |
| [11] |
Morlando M, Fatica A. Alteration of epigenetic regulation by long noncoding RNAs in cancer[J]. Int J Mol Sci, 2018, 19(2): 570. DOI:10.3390/ijms19020570 |
| [12] |
Li J, Li H, Lv XT, et al. Polymorphism in lncRNA AC016683.6 and its interaction with smoking exposure on the susceptibility of lung cancer[J]. Cancer Cell Int, 2018, 18: 91. DOI:10.1186/s12935-018-0591-2 |
| [13] |
Chang L, Hu Z, Zhou Z, et al. Linc00518 contributes to multidrug resistance through regulating the MiR-199a/MRP1 axis in breast cancer[J]. Cell Phys Biochem, 2018, 48(1): 16-28. |
| [14] |
Chen BB, Zhao QF, Guan LL, et al. Long non-coding RNA NNT-AS 1 sponges miR-424/E2F1 to promote the tumorigenesis and cell cycle progression of gastric cancer[J]. J Cell Mol Med, 2018, 22(10): 4751-4759. DOI:10.1111/jcmm.13726 |
| [15] |
Sung YY, Cheung E. Antisense now makes sense:dual modulation of androgen-dependent transcription by CTBP1-AS[J]. Embo J, 2013, 32(12): 1653-1654. DOI:10.1038/emboj.2013.112 |
| [16] |
Cui HB, Geng CZ. Molecular mechanisms of long chain non-coding RNA CTBP1-AS in regulation of invasion and migration of breast cancer cells[J]. J Biol Regul Homeost Agents, 2019, 33(3): 773-785. |
| [17] |
Blevins MA, Huang MX, Zhao R. The role of CtBP1 in oncogenic processes and its potential as a therapeutic target[J]. Mol Cancer Ther, 2017, 16(6): 981-990. DOI:10.1158/1535-7163.MCT-16-0592 |
| [18] |
Weichenhan D, Plass C. The evolving epigenome[J]. Hum Mol Genet, 2013, 22(R1): R1-R6. DOI:10.1093/hmg/ddt348 |
| [19] |
Yousef AF, Fonseca GJ, Cohen MJ, et al. The C-terminal region of E1A:a molecular tool for cellular cartography[J]. Biochem Cell Biol, 2012, 90(2): 153-163. |



