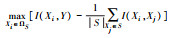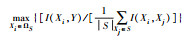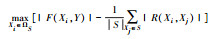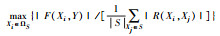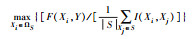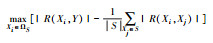2. 湖南农业大学植物病虫害生物学与防控湖南省重点实验室, 长沙 410128;
3. 湖南农业大学信息科学技术学院, 长沙 410128
2. Hunan Provincial Key Laboratory for Biology and Control of Plant Diseases and Insect Pests, Changsha 410128;
3. College of Information Science and Technology, Hunan Agricultural University, Changsha 410128
随着化学品的广泛应用, 生产杂质与废弃物排放量日趋增大, 其中不乏一些极难处理且有毒性的物质, 如工业溶剂中常用的醇类化合物(Hatipoğlu and Cinar, 2003)与工业废水中常用的酚类化合物(Fredlund et al., 1994).欧盟制定、我国2010年采用的《化学品的注册、评估、授权和限制》法规致力于在化合物进入市场前进行标准化处理与毒性评估(Mascarelli, 2012).尽管细胞培养特别是干细胞培养替代动物个体能缩短评估过程、提高实验灵敏度(Stadnicka Michalak et al., 2015; Uppal and Roquemore, 2013), 化合物毒性的实验评估仍耗时费力, 迄今有急性毒性记录的化合物尚不超过2%(Strempel and Scheringer, 2012; Verhaar et al., 2000).定量构效关系(quantitative structure activity relationship, QSAR)是化学与生物学的桥梁, 合理的QSAR模型是化合物毒性实验评估的有益补充(Puzyn et al., 2011).一个合理的QSAR模型需要满足:明确的活性指标, 不模糊的算法, 可定义模型的应用域, 具备良好的拟合性、鲁棒性和预测能力, 具备可解释性(Cumming et al., 2013).
QSAR包含分子描述符获取、特征选择、学习机器选择与模型解释4个关键环节(Taylor et al., 2014; 苏满秀等, 2012). ①描述符获取:化合物经量子化学计算可得数以千计的分子描述符, 包含大量无关特征与冗余特征. ②特征选择:可降低模型训练耗时, 提高预测精度, 增强模型解释性.一般的单变量过滤法仅能去除无关特征, 不能去除冗余特征.最小冗余最大相关(minimal redundancy maximal relevance, mRMR)同时考虑了特征的相关性与冗余性(Peng et al., 2005), 在多个特征选择领域表现优异(Ma and Sun, 2014; Unler et al., 2011).然而, 当前的mRMR仅适用于因变量为离散型变量的情形, 且自变量为连续型变量时其相关性测度与冗余性测度指标不可比. ③学习机器选择:与基于经验风险最小的多元线性回归、偏最小二乘(Sharma et al., 2013)和人工神经网络(Guha and durs, 2005)等相比, 基于结构风险最小的支持向量机(support vector machine, SVM)是机器学习领域的集大成者, 具非线性、适于小样本、能有效避免过拟合与维数灾难、泛化推广能力优异等诸多优点(Chang and Lin, 2011). SVM分为支持向量分类(support vector classification, SVC)和支持向量回归(support vector regression, SVR), 本文选用适于QSAR的SVR. ④SVM缺乏一个显性的表达式, 可解释性差.本室前期针对SVR已发展一套较完整的非线性回归解释性体系, 包括模型显著性测验、单因子显著性测验与单因子效应分析等(谭显胜等, 2009).
距离相关系数(distance correlation, dCor)是近年来提出的可表征2个连续型变量间非线性关联程度的新测度(Székely et al., 2007).本文针对QSAR研究中因变量、自变量均为连续型变量的情形, 发展了一种新的特征选择方法mRMR-dCor, 并以3个醇酚类化合物毒性的SVR独立预测结果验证了新方法的有效性, 结果报道如下.
2 材料与方法(Materials and methods) 2.1 数据来源与分子描述符本文选取3个数据集共227个醇酚类化合物作为研究对象. Dataset-1(表 1)为110种醇类有机小分子化合物对欧洲林蛙(Ranatemporaria)蝌蚪的毒性研究(苏强, 2013), Dataset-2(表 2)为50种酚类化合物对梨形四膜虫(Tetrahymenapyriformis)的麻醉毒性研究(郭明等, 1998), Dataset-3(表 3)为67种脂肪醇类化合物对梨形四膜虫的急性毒性研究(王新颖等, 2014).毒性实验值以50%生长抑制浓度的负对数-log (IGC50)表示, 单位均为mmol·L-1.
| 表 1 醇类化合物对欧洲林蛙蝌蚪的毒性(苏强, 2013) Table 1 Toxicities of alcohols to tadpoles of Ranatemporaria |
| 表 2 酚类化合物对梨形四膜虫的毒性(郭明和许禄, 1998) Table 2 Toxicities of phenols to Tetrahymenapyriformis |
| 表 3 醇类化合物对梨形四膜虫的毒性(王新颖等, 2014) Table 3 Toxicities of alcohols to Tetrahymenapyriformis |
将每种化合物的英文名称输入在线服务器http://www.ichemistry.cn/structure.asp进行结构转化, 整理好的SMILES文件输入PCLIENT在线软件http://146.107.217.178/lab/pclient/start.html (Tetko et al., 2005), 获得24组约3000个分子描述符(表 4).删除完全相同的描述符与缺失不全的描述符, 3个数据集的有效初始描述符分别为1778、1264、1466个.对Dataset-1, 按毒性值由低到高顺序排列化合物, 每4个样本取出1个作为独立测试样本, 即训练集:测试集=3:1;对Dataset-2, 按毒性值由低到高顺序排列化合物, 每5个样本取出1个作为独立测试样本, 即训练集:测试集=4:1;对Dataset-3, 为方便比较, 训练集与独立测试集同文献(王新颖等, 2014).
| 表 4 分子描述符的组名与解释 Table 4 Group name and explanation of molecular descriptors |
2个离散变量间的关联可用互信息I(X, Y)测度, I∈[0, +∞]; 一个离散变量与一个连续变量间的关联可用单因素不等重复方差分析的F-score (二分类时为t-score)测度, F∈[0, +∞]; 2个连续变量间的线性关联可用Pearson相关系数R测度, R∈[-1, 1](Ding and Peng, 2005).设自变量集合Ω={X1, X2, …, Xi, …, Xm}, 集合长度(元素个数)|Ω|=m.当前已引入特征集合为S, 未引入特征集合ΩS=Ω-S.当Y为离散型因变量时, Peng等(2005)总结了mRMR引入下一个特征的标准(表 5).
| 表 5 因变量Y离散时mRMR引入下一个特征的标准(Peng et al., 2005) Table 5 Different schemes to search for the next feature in mRMR while dependent variable Y is discrete |
当前的mRMR对Y为连续型变量并未给出解决方案, 且公式(3)、(4)中相关性测度F(Xi, Y)与冗余性测度R(Xi, Xj)不可比.本文先推广mRMR至Y为连续型变量情形, 其引入下一个特征的标准总结为表 6.
| 表 6 因变量Y连续时mRMR引入下一个特征的标准 Table 6 Different schemes to search for the next feature in mRMR while dependent variable Y is continuous |
表 6公式(5)、(6)中相关性测度F(Xi, Y)与冗余性测度I(Xi, Xj)仍不可比, 公式(7)、(8)中R(Xi, Y)不能反映非线性相关, R(Xi, Xj)不能反映非线性冗余.
dCor通过计算样本本身的欧几里得距离来衡量2个连续型变量间的关联程度.对2个连续型随机变量X、Y, 其总体距离系数dCor定义为:

|
(9) |
dCor∈[0, 1].当dCor (X, Y)=0时, X与Y相互独立, 相关性为0;dCor (X, Y)越大, 表示X与Y越相关. dCor能探测连续型变量间的非线性或非单调关系(樊嵘等, 2014), 不受总体是否呈正态分布约束, 并有较高的统计势(Székely et al., 2007).
2.3 mRMR-dCorQSAR数据中通常因变量Y与自变量X均为连续型变量.本文以dCor取代表 6公式(7)、(8)中的R(Xi, Y)和R(Xi, Xj), 使相关性测度与冗余性测度不但可比, 且能反映非线性相关与非线性冗余, 提出了新的特征选择方法mRMR-dCor, 其引入下一个特征的标准为:
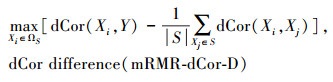
|
(10) |

|
(11) |
假定引入的第一个特征dCor (X1, Y)=0.6;现dCor (X2, Y)=0.2, dCor (X1, X2)=0.01; dCor (X3, Y)=0.5, dCor (X1, X3)=0.1.直觉上我们应优先引入X3, 但按公式(11), 由于dCor∈[0, 1], 作为分母的冗余性测度的作用被过分夸大, 最终X2优先引入.因此, 我们认为mRMR-dCor-Q是不合理的, 应选用mRMR-dCor-D; 参比模型应选用表 6公式(7)的mRMR-RCD.
2.4 前向选择最优特征子集与独立测试基于训练集以mRMR-dCor-D、mRMR-RCD分别得到全部特征的mRMR排序后, 以前向选择逐个引入特征.每引入一个特征, 对训练集以SVR进行5-fold交叉测试:若均方误差(mean square error, MSE)降低则新引入特征保留, 反之剔除(Zhang et al., 2014), 直到设定的引入特征上限B(本文中B=100).保留特征构成最优特征子集, 用于后续独立测试.SVR采用Libsvm3.1软件包(Chang and Lin, 2011), 核函数经试算采用径向基核(Radial Basis Function, RBF)与线性核(Linear), 参数c、g、p基于训练集由gridregression.py经5-fold交叉测试搜索自动获取.
2.5 模型评估独立测试评价标准采用Q2、R2与均方根误差(Root mean square error, RMSE).其中, ytest_i和ŷtest_i为测试集样本真值和预测值, ytrain和ytest分别为训练集和测试集样本真值的均值, n为测试集样本个数.
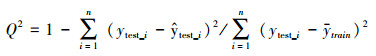
|
(12) |
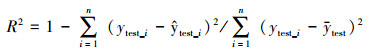
|
(13) |

|
(14) |
不同特征选择方法的SVR模型独立预测表现见表 7.以Q2为主要判断依据: ①不同数据集适合的核函数不同, Dataset-1与Dataset-2宜采用线性核, 而Dataset-3宜采用径向基核. ②对2种核函数3个数据集共6种情形, 与采用全部特征(不进行特征选择)相比, mRMR-RCD仅在3种情形下提高了独立预测精度, 而mRMR-dCor-D在6种情形下均提高了独立预测精度; 且6种情形下mRMR-dCor-D均优于mRMR-RCD, 体现了dCor能反映非线性关联的优点.
| 表 7 不同特征选择方法的SVR模型独立预测表现 Table 7 Independent test performances of SVR models with different feature selection methods |
由于分子描述符来源与评价指标不一, 本文仅给出基于mRMR-dCor-D的SVR模型独立预测与文献报道的简单比较:对Dataset-1, 本文Q2为0.954, 文献(苏强, 2013)的Q2为0.860;对Dataset-2, 本文RMSE为0.159, 文献(郭明和许禄, 1998)的RMSE为0.200;对Dataset-3, 本文Q2为0.981, 文献(王新颖等, 2014)的Q2为0.978.基于mRMR-dCor-D的SVR模型独立预测优于文献报道.图 1展示了3个数据集的毒性预测值与观察值分布, 可见多数样本位于对角线附近, 进一步直观说明基于mRMR-dCor-D的SVR模型预测结果优异.
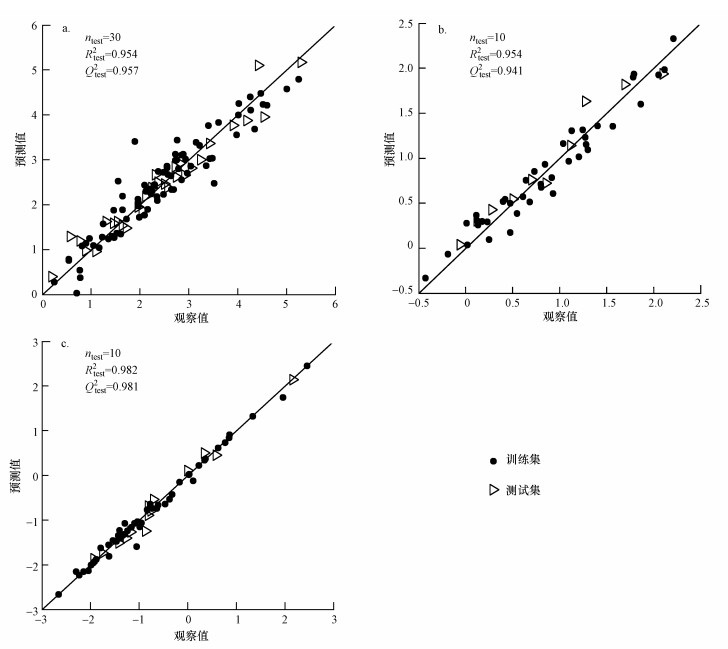 |
| 图 1 测试集样本毒性的观察值与预测值(a. Dataset-1(线性核),b. Dataset-2(线性核),c. Dataset-3(径向基核)) Fig. 1 Observed values and predicted values of testing samples in Dataset-1 (a), Dataset-2 (b), Dataset-3 (c) |
本室前期针对SVR已发展一套较完整的非线性回归解释性体系(谭显胜等, 2009; 苏满秀等, 2012).以Dataset-1为例, 全部110个样本、mRMR-dCor-D选择的17个保留描述符建立的SVR模型, 其F=70.48>F0.01(17, 92)=2.17, 表明所得模型非线性回归极显著.单因子非线性回归极显著的14个保留描述符(F>F0.01(1, 61)=7.07)见表 8.其中, MLOGP、MLOGP2、ALOGP、ALOGP2、BLTF96为用不同方法测得的正辛醇/水分配系数(Moriguchi et al., 1992), 是度量有机化合物在水中亲脂性的重要参数, 很多实验已证明其与化合物的多种毒理学性质强相关(赵元慧和何艺兵, 1995); EEig02d为原子的偶极矩参数, 实验证明偶极矩越小、亲水性越高、毒性越低(Gharagheizi, 2009; 焦健等, 1987); 范德华体积参数Mor01v、Mor04v、Mor08v (Goodarzi et al., 2011; Hemmateenejad et al., 2008), 原子极化性参数Mor01p (Gasteiger et al., 1996), 原子质量参数Mor04m (Habibi Yangjeh and Danandeh Jenagharad, 2009), 原子Sanderson极化性参数QYYe (Robinson et al., 1997)等对化合物毒性影响也有报道.值得注意的是, 尽管SPH (球面性参数)与JG13(平均拓扑电荷参数)未见有与化合物毒性相关的报道, 但本文结果显示其作用不可忽视.
| 表 8 Dataset-1中的14个极显著保留描述符 Table 8 14 remarkable descriptors retained in Dataset-1 |
单因子效应分析显示(图 2), 与醇类对蝌蚪毒性正相关的描述符包括: MLOGP2, ALOGP, MLOGP, EEig02D, SPH, JGI3, Mor04m, Mor08v, QYYe.与醇类对蝌蚪毒性负相关的描述符包括: BLTF96, ALOGP2, Mor01v, Mor04v, Mor01p.
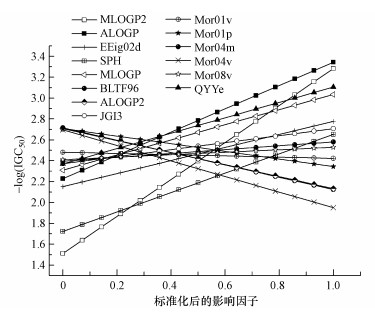 |
| 图 2 Dataset-1中14个极显著保留描述符的单因子效应(mRMR-dCor-D, 线性核) Fig. 2 Single-factor effects of the 14 remarkable descriptors retained in Dataset-1 |
量子化学计算获得的化合物分子描述符中存在诸多无关特征与冗余特征, 化合物毒性与描述符间往往存在非线性关系. mRMR同时考虑了特征的相关性与冗余性, 是当前Y为离散型变量时应用较广泛的特征选择方法.针对QSAR研究中Y与X均为连续型变量的情形, 本文首先将mRMR推广到适于Y为连续型变量, 再以非线性的dCor取代线性的相关系数R, 实现了相关性测度与冗余性测度的非线性可比, 提出了新的特征选择方法mRMR-dCor. 3个醇酚类化合物毒性QSAR数据集的独立预测表明, 基于mRMR-dCor选择特征的SVR模型明显优于参比模型与文献报道, 在化合物QSAR、定量构质关系等研究中有广泛应用前景.
| [${referVo.labelOrder}] | Chang C C, Lin C J. 2011. LIBSVM: a library for support vector machines[J]. ACM Transactions on Intelligent Systems and Technology, 2(3) : 27. |
| [${referVo.labelOrder}] | Cumming J G, Davis A M, Muresan S, et al. 2013. Chemical predictive modelling to improve compound quality[J]. Nature Reviews Drug Discovery, 12(12) : 948–962. DOI:10.1038/nrd4128 |
| [${referVo.labelOrder}] | Ding C, Peng H. 2005. Minimum redundancy feature selection from microarray gene expression data[J]. Journal of Bioinformatics and Computational Biology, 3(02) : 185–205. DOI:10.1142/S0219720005001004 |
| [${referVo.labelOrder}] | Fredlund D G, Xing A, Huang S. 1994. Predicting the permeability function for unsaturated soils using the soil-water characteristic curve[J]. Canadian Geotechnical Journal, 31(4) : 533–546. DOI:10.1139/t94-062 |
| [${referVo.labelOrder}] | 樊嵘, 孟大志, 徐大舜. 2014. 统计相关性分析方法研究进展[J]. 数学建模及其应用, 2014, 3(1) : 1–12. |
| [${referVo.labelOrder}] | Gasteiger J, Sadowski J, Schuur J, et al. 1996. Chemical information in 3D space[J]. Journal of Chemical Information and Computer Sciences, 36(5) : 1030–1037. DOI:10.1021/ci960343+ |
| [${referVo.labelOrder}] | Gharagheizi F. 2009. A QSPR model for estimation of lower flammability limit temperature of pure compounds based on molecular structure[J]. Journal of Hazardous Materials, 169(1) : 217–220. |
| [${referVo.labelOrder}] | Goodarzi M, Freitas M P. 2011. Linear and nonlinear quantitative structure-activity relationship modeling of the HIV-1 reverse transcriptase inhibiting activities of thiocarbamates[J]. Analytica Chimica Acta, 705(1) : 166–173. |
| [${referVo.labelOrder}] | Guha R, Jurs P C. 2005. Interpreting computational neural network QSAR models: a measure of descriptor importance[J]. Journal of Chemical Information and Modeling, 45(3) : 800–806. DOI:10.1021/ci050022a |
| [${referVo.labelOrder}] | 郭明, 许禄. 1998. 酚类化合物的QSAR研究[J]. 环境科学学报, 1998, 18(2) : 122–126. |
| [${referVo.labelOrder}] | Habibi Yangjeh A, Danandeh Jenagharad M. 2009. Application of a genetic algorithm and an artificial neural network for global prediction of the toxicity of phenols to Tetrahymenapyriformis[J]. Monatshefte Fur Chemie Chemical Monthly, 140(11) : 1279–1288. DOI:10.1007/s00706-009-0185-8 |
| [${referVo.labelOrder}] | Hatipoğlu A, Çinar Z. 2003. A QSAR study on the kinetics of the reactions of aliphatic alcohols with the photogenerated hydroxyl radicals[J]. Journal of Molecular Structure: Theochem, 631(1) : 189–207. |
| [${referVo.labelOrder}] | Hemmateenejad B, Shamsipur M, Safavi A, et al. 2008. Reversed-phase high performance liquid chromatography (RP-HPLC) characteristics of some 9, 10-anthraquinone derivatives using binary acetonitrile-water mixtures as mobile phase[J]. Talanta, 77(1) : 351–359. DOI:10.1016/j.talanta.2008.06.044 |
| [${referVo.labelOrder}] | 焦健, 毛志翔, 陈启琪, 等. 1987. O-乙基O-取代芳基N-异丙基硫代磷酰胺酯类化合物急性毒性与结构的相关性[J]. 高等学校化学学报, 1987, 8(2) : 128–132. |
| [${referVo.labelOrder}] | Ma X, Sun X J. 2014. Sequence-based predictor of ATP-binding residues using random forest and mRMR-IFS feature selection[J]. Journal of Theoretical Biology, 360 : 59–66. DOI:10.1016/j.jtbi.2014.06.037 |
| [${referVo.labelOrder}] | Mascarelli A. 2012. Environment: toxic effects[J]. Nature, 483(7389) : 363–365. DOI:10.1038/nj7389-363a |
| [${referVo.labelOrder}] | Moriguchi I, Hirono S, Liu Q, et al. 1992. Simple Method of Calculating Octanol/Water Partition Coefficient[J]. Chemical and Pharmaceutical Bulletin, 40(1) : 127–130. DOI:10.1248/cpb.40.127 |
| [${referVo.labelOrder}] | Peng H, Long F, Ding C. 2005. Feature selection based on mutual information criteria of max-dependency, max-relevance, and min-redundancy[J]. Pattern Analysis and Machine Intelligence, IEEE Transactions on Pattern Analysis and Machine Intelligence, 27(8) : 1226–1238. DOI:10.1109/TPAMI.2005.159 |
| [${referVo.labelOrder}] | Puzyn T, Rasulev B, Gajewicz A, et al. 2011. Using nano-QSAR to predict the cytotoxicity of metal oxide nanoparticles[J]. Nature Nanotechnology, 6(3) : 175–178. DOI:10.1038/nnano.2011.10 |
| [${referVo.labelOrder}] | Robinson D D, Barlow T W, Richards W G. 1997. Reduced dimensional representations of molecular structure[J]. Journal of Chemical Information and Computer Sciences, 37(5) : 939–942. DOI:10.1021/ci970424l |
| [${referVo.labelOrder}] | Sharma M C, Sharma S, Sahu N K, et al. 2013. QSAR studies of some substituted imidazolinones angiotensin Ⅱ receptor antagonists using partial least squares regression (PLSR) method based feature selection[J]. Journal of Saudi Chemical Society, 17(2) : 219–225. DOI:10.1016/j.jscs.2011.03.012 |
| [${referVo.labelOrder}] | Stadnicka Michalak J, Schirmer K, Ashauer R. 2015. Toxicology across scales: Cell population growth in vitro predicts reduced fish growth[J]. Science Advances, 1(7) : e1500302. DOI:10.1126/sciadv.1500302 |
| [${referVo.labelOrder}] | Strempel S, Scheringer M. 2012. Screening forPBTchemicals among the "existing" and "new" chemicals of the EU[J]. Environmental Science & Technology, 46(11) : 5680–5687. |
| [${referVo.labelOrder}] | 苏满秀, 王立峰, 代志军. 2012. 多肽一级结构表征与抗菌肽QSAM建模[J]. 高等学校化学学报, 2012, 33(11) : 2526–2531. |
| [${referVo.labelOrder}] | 苏强. 2013.基于数据挖掘算法的环境毒物QSAR研究[D].上海:上海大学. 83-86 |
| [${referVo.labelOrder}] | Székely G J, Rizzo M L, Bakirov N K. 2007. Measuring and testing dependence by correlation of distances[J]. The Annals of Statistics, 35(6) : 2769–2794. DOI:10.1214/009053607000000505 |
| [${referVo.labelOrder}] | 谭显胜, 王志明, 谭泗桥, 等. 2009. 支持向量回归可解释性体系的建立[J]. 系统仿真学报, 2009, 21(24) : 7795–7797. |
| [${referVo.labelOrder}] | Taylor K, Stengel W, Casalegno C, et al. 2014. Experiences of the REACH testing proposals system to reduce animal testing[J]. Altex(2) : 107–128. |
| [${referVo.labelOrder}] | Tetko I V, Gasteiger J, Todeschini R, et al. 2005. Virtual computational chemistry laboratory-design and description[J]. Journal of Computer-aided Molecular Design, 19(6) : 453–463. DOI:10.1007/s10822-005-8694-y |
| [${referVo.labelOrder}] | Unler A, Murat A, Chinnam R B. 2011. mr2PSO: a maximum relevance minimum redundancy feature selection method based on swarm intelligence for support vector machine classification[J]. Information Sciences, 181(20) : 4625–4641. DOI:10.1016/j.ins.2010.05.037 |
| [${referVo.labelOrder}] | Uppal H, Roquemore L. 2013. Harnessing stem cells for predictive toxicology: Meeting the challenges of drug discovery today[J]. Science, 341(6142) : 199–199. |
| [${referVo.labelOrder}] | Verhaar H J, Solbé J, Speksnijder J, et al. 2000. Classifying environmental pollutants: Part 3External validation of the classification system[J]. Chemosphere, 40(8) : 875–883. DOI:10.1016/S0045-6535(99)00317-3 |
| [${referVo.labelOrder}] | 王新颖, 张锦晖, 王丹丹, 等. 2014. 脂肪醇化合物对梨形四膜虫急性毒性的QSAR研究[J]. 计算机与应用化学, 2014, 31(6) : 732–736. |
| [${referVo.labelOrder}] | Zhang H, Li L, Luo C, et al. 2014. Informative gene selection and direct classification of tumor based on chi-square test of pairwise gene interactions[J]. BioMed Research International, Article ID589290, 1-9 . |
| [${referVo.labelOrder}] | 赵元慧, 何艺兵. 1995. 分子连接性指数与硝基芳烃理化参数的相关性[J]. 自然科学进展, 1995, 5(4) : 496–498. |
 2016, Vol. 36
2016, Vol. 36