2. 大连海洋大学, 大连 116023;
3. 大连理工大学环境学院, 大连 116023;
4. 厦门大学海洋与地球学院, 厦门 361005
2. Dalian Ocean University, Dalian 116023;
3. School of Environmental Science & Technology, Dalian University of Technology, Dalian 116023;
4. College of Ocean and Earth Science, Xiamen University, Xiamen 361005
固定CO2的细菌一般有两类:光能自养型细菌和化能自养型细菌(周集体等,1999).卡尔文循环是光能自养生物和化能自养生物同化CO2的主要途径,其中,核酮糖-1,5-二磷酸羧化酶/加氧酶(RubisCO)是卡尔文循环中的关键酶(Tabita et al.,1999).RubisCO酶含有大小两个亚基,最保守的位点位于大亚基,而RubisCO酶大亚基Ⅰ型和Ⅱ型分别由cbbL和cbbM基因编码,因此,可以用这两种基因进行保守区序列的扩增、比对和系统发育分析(Watson et al.,1997).cbbL基因也被许多学者用于环境样品的固碳细菌多样性研究,研究热点从陆地生态系统延伸到了水生生态环境(Yuan et al.,2012;Giri et al.,2004).与16S rRNA基因标记相比,功能基因可直接靶向环境中具有特定生理或生物地球化学功能的类群,从而在系统发育分析方面更具有优势(Paul et al.,1998),因此,通过研究cbbL和cbbM基因的多样性可以反映主要固碳细菌的群落结构.
近年来,用RubisCO基因(cbbL和cbbM)来反映固碳细菌多样性的研究主要集中在土壤(Yuan et al.,2012)、深海热液口(Hügler et al.,2010)、地下水(Alfreider et al.,2009)、湖泊(Giri et al.,2004),而在河口区(近海生态系统)其相关研究较少.与深海大洋相比,近海生态系统仅占全球海洋面积的7%~8%,但其有机碳埋藏量占整个海洋碳埋藏量的90%,并且至少有50%的颗粒无机碳沉积于此.此外,近海海洋环境易受人为活动影响,大量有机质和营养盐通过河流排入并汇集于此.因此,陆架边缘海域在全球碳循环中的作用不容忽视(Song et al.,2011).因南海北部是国内最大的边缘海之一,在北部的陆架区珠江口有大量淡水注入,使微生物组成、多样性受其影响,其独特的地理环境在很大程度上影响着该海域海洋水文和环流状况(袁梁英等,2005).因此,本文选取珠江口附近海域到南海北部水域的6个站位,采用高通量测序技术对cbbL、cbbM基因多样性进行分析,研究其固碳基因多样性组成和分布特征,并进一步探讨其与环境因子的关系.
2 方法与材料(Materials and methods) 2.1 样品采集和化学指标测定本研究搭载中国科学院南海海洋研究所“实验1”科考船,于2014年8月20日-9月4日进行样品采集,根据离岸距离布设6个站位:A9、J2、J4、I1、K3、Seats.具体采样站位如图 1所示.
 |
| 图 1 采样站位分布 Fig. 1 Distribution of sampling stations |
使用卡盖式便携采水器对表层海水进行采集,每个站位采集3个平行样,每个平行样采集2 L海水,充分混匀后,取2 L水经0.22 μm滤膜过滤,滤膜液氮保存,用于cbbL和cbbM基因的群落结构分析.
水温、盐度使用便携式水质分析仪YSI-556(USA)进行走航现场测定,硝酸盐、硅酸盐、亚硝酸盐、磷酸盐按照国标方法测定(国家海洋环境监测中心,2007);细菌生产力(BP)通过3H-亮氨酸示踪法测定(Kirchman et al.,1993).
2.2 细菌DNA提取、PCR扩增和高通量测序运用CTAB传统方法,对过滤过海水的微孔滤膜进行总DNA提取.通过分光光度仪测定DNA样品的浓度和纯度.
以总基因组DNA为模板,进行cbbL和cbbM基因的PCR扩增,采用两步法,cbbL第一轮扩增引物: F(5′-GACTTCACCAAAGACGACGA-3′)和R(5′-TCGAACTTGATTTCTTTCCA-3′),带有融合引物的cbbL二轮引物:F(5′-CATCATGTTCGACCAGGACT-3′)和R(5′-TCGAACTTGATTTCTTTCCA-3′)(Campbell et al.,2004);cbbM扩增引物:F(5′-TTCTGGCTGGG(G/T/C)GG(A/T/C)GA(C/T)TT(C/T)AT(C/T)AA-RAA(A/G)GACGA-3′)和R(5′-CCGTGRCC(A/G)GC(G/A/C)CG(A/G)TGGTA(A/G)TG-3′)(Elsaied et al.,2001).PCR反应体系为:2×Taq PCR Master mix 12.5 μL,引物(10 pmol·μL-1)各0.2 μL,DNA模板为2 μL,加ddH2O至25 μL.cbbL基因的PCR扩增反应条件:95 ℃预变性5 min;95 ℃变性1 min,52 ℃退火1 min,72 ℃ 延伸1 min,循环30次;72 ℃延伸45 min;第二轮扩增循环数为20次.cbbM基因的PCR扩增反应条件:94 ℃ 预变性2 min;94 ℃变性1 min,50 ℃退火1 min,72 ℃ 延伸10 min,循环30次;72 ℃ 延伸10 min.PCR产物用胶纯化试剂盒(AMPure XP PCR purification kit)纯化,等量混合后送至华大基因运用Illumina Miseq平台进行高通量测序.
2.3 生物信息分析下机数据经过数据过滤,通过检测3′端接头和引物,去除模糊碱基、单碱基高重复区及长度过短的低质量的Reads,剩余高质量的Clean Data用于后期分析;利用Reads之间的Overlap关系,运用FLASH软件将Reads拼接成Tags;然后通过USEARCH软件(v 7.0.1090)将拼接好的Tags在97%的相似度下聚类为OTU,利用RDP Classifer软件(v 2.2)将OTU代表序列与数据库比对进行物种注释;基于OTU和物种注释结果进行样品物种复杂度分析及组间物种差异分析.最后通过Mothur软件(v 1.31.2)计算样品的Alpha多样性值(Chao、Ace、Shannon和Simpson指数),并用R(v 3.0.3)软件做出相应的稀释曲线图.地理站位、相关性和冗余分析分别采用软件Surfer 8.0、SPSS 17.0和CANOCO 5.0完成.
3 结果(Results)对cbbL和cbbM基因进行扩增.所有样品的cbbL基因PCR扩增结果均为阳性,且浓度达到上机要求,可进行高通量测序(测序所得序列已提交到NCBI的SRA数据库,登录号为SRP070472);而cbbM基因的PCR扩增为阴性,未进行后续测序分析.
3.1 cbbL固碳基因的多样性通过计算稀释曲线来表明样本的取样大小是否合理,分析显示(图 2),各样点的稀释曲线都有着趋于平缓的趋势,表明本次实验测序深度已经能反映出该海域中的绝大多数含cbbL固碳基因的固碳菌群.
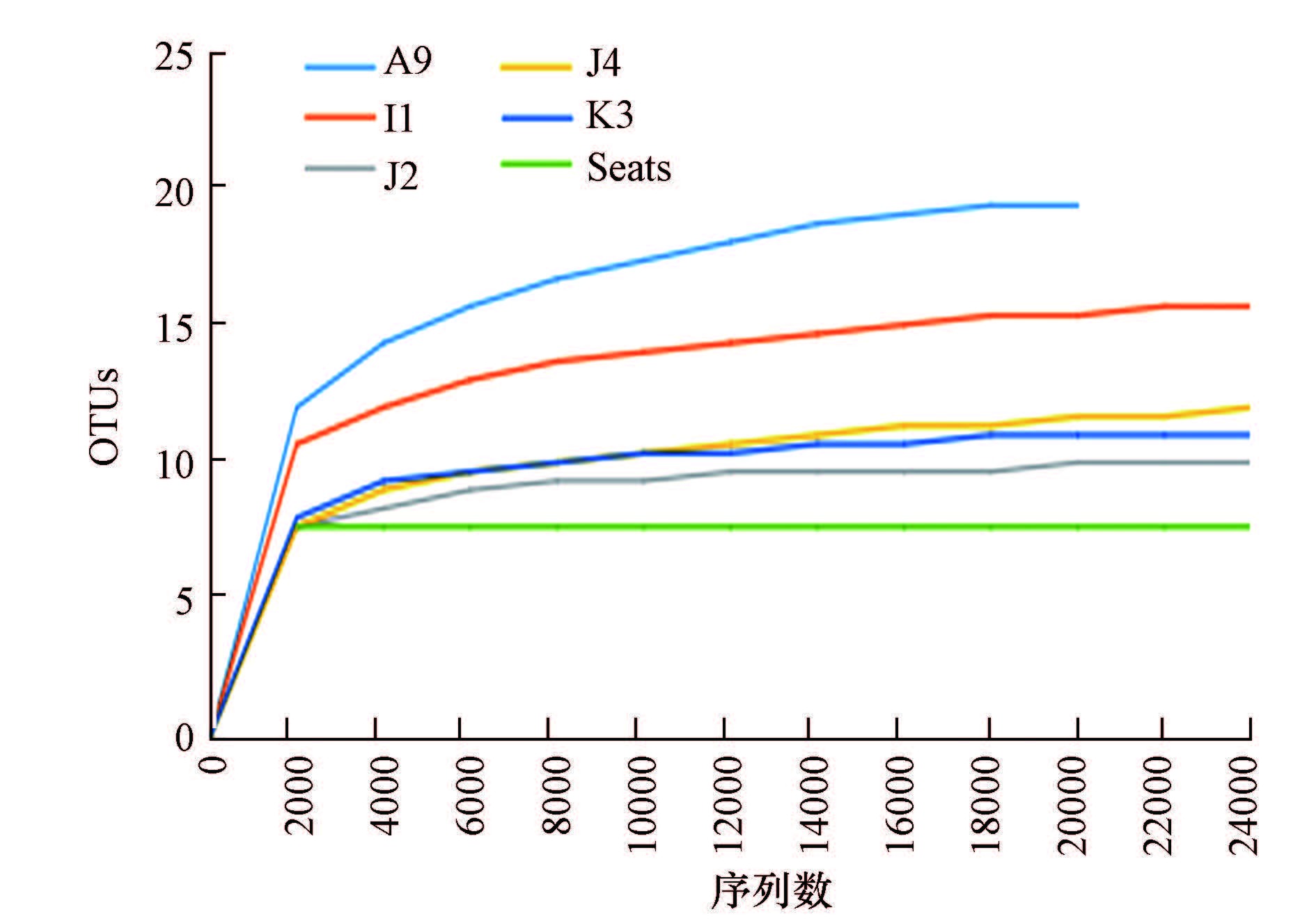 |
| 图 2 海水样品的稀释曲线 Fig. 2 Dilution curve of seawater samples |
在该海域中,A9站位的OTU数最多,为16个.通过RDP Classifier程序对不同样品的基因序列进行分类,用Ace、Chao1指数反映样品中群落的丰富度,用Shannon和Simpson指数来定量描述该水域的生物多样性(这两个指标反映的是Alpha多样性,Shannon指数与生物多样性成正比,而Simpson指数与之相反,值越大,生物多样性越低),结果见表 1.由表 1可知,A9站位OTU数最多,而其它几个站位变化不大;在含cbbL基因的固碳菌群中,A9站位物种最丰富;A9站位相较于其他站位具有较高的生物多样性.
| 表 1 cbbL基因序列分析 Table 1 cbbL gene sequence analysis |
本研究共获得3个门、5个纲、9个目、11个科、17个属.在该海域内优势亚群分别是γ-变形菌亚门(γ-Proteobacteria,45.3%)、蓝细菌(Cyanobacteria,30.9%)、β-变形菌亚门(β-Proteobacteria,23.8%).厚壁菌门(Firmicutes)只在A9站位发现,并且变形菌门在A9站位(98.9%)所占的比例与其他5个站位所占比例有很大差异(J2:43.4%,J4:76.7%,I1:62.9%,K3:73%,Seats:67.3%).
在属水平上细菌的主要组成见图 3,测序得到3个菌群中共包括17个属,其中,丰度较高的是外硫红螺菌属(Ectothiorhodospira,58.8%)、亚硝化单胞菌属(Nitrosomonas,17.7%)、硫杆菌属(Thiobacillus,13.6%),占所有菌群的90.1%.由图 3可知,在本实验所选取的6个站位中,离岸最近的A9站位菌群最丰富,并且与其他站位相比存在其独特的菌属:异着色菌属(Allochromatium)、硫杆菌属(Thiobacillus)、硫单胞菌属(Thiohalomonas).
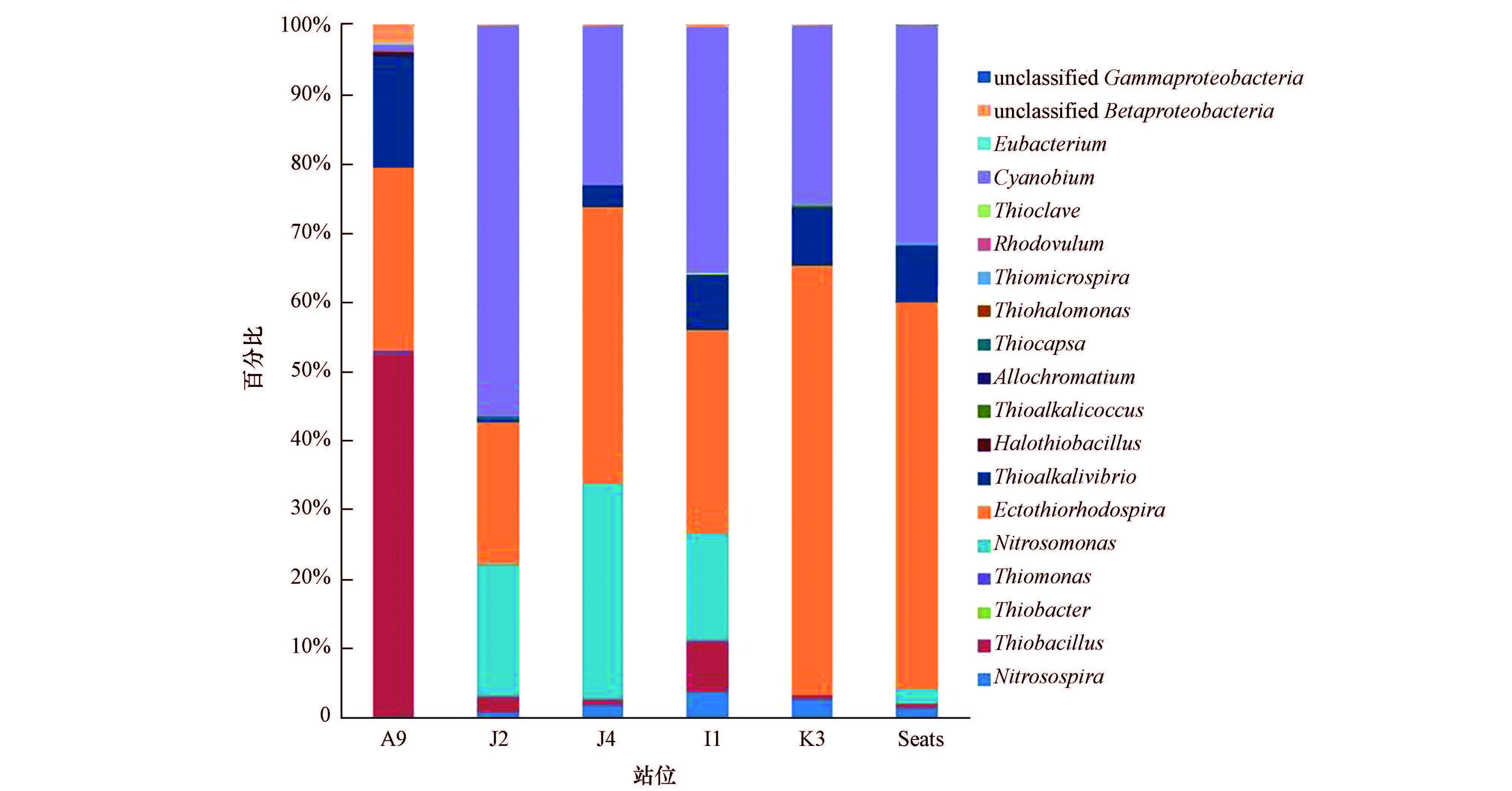 |
| 图 3 属水平上的群落组成 Fig. 3 Community composition on the genus level |
本研究采用Miseq高通量测序的方法,对南海北部近珠江口水域表层海水中含cbbL基因的细菌群落结构进行分析,共获得3个门,5个纲,9个目,11个科,17个属.在该海域内优势亚群分别是γ-变形菌亚门(γ-Proteobacteria,45.3%)、蓝细菌(Cyanobacteria,30.9%)、β-变形菌亚门β-Proteobacteria(23.8%).厚壁菌门(Firmicutes)只在A9站位发现,并且变形菌门在A9(98.9%)站位所占的比例与其他5个站位所占比例有很大差异(J2:43.4%、J4:76.7%、I1:62.9%、K3:73%、Seats:67.3%).
在属水平上细菌的主要组成见图 3,本研究所得到的3个菌群共包括17个属,其中丰度较高的是外硫红螺菌属(Ectothiorhodospira,58.8%)、亚硝化单胞菌属(Nitrosomonas,17.7%)、硫杆菌属Thiobacillus(13.6%),占所有菌群的90.1%.由图 3中可知,在本研究所选取的6个站位中,离岸最近的A9站位固碳基因多样性最丰富,并且与其他站位相比存在其独特的菌属:异着色菌属(Allochromatium)、硫杆菌属(Thiobacillus)、硫单胞菌属(Thiohalomonas).
此外,各站位中都含有一些无法确定其分类位置的序列,表明该海域存在一些未被认知的固碳菌群.
PCA分析是指运用方差分解的方法,将多组数据的差异反映在二维坐标图上,如果两个样品距离越近,则表示这两个样品的组成越相似.为进一步分析各站位之间细菌组成的差异和距离,对不同样品OTU(97%相似性)组成进行了PCA分析.由图 4可以看出,A9、J2站位与其他4个站位物种组成有较大的差异,J4、I1、K3、Seats距离较近,物种组成也较相似.
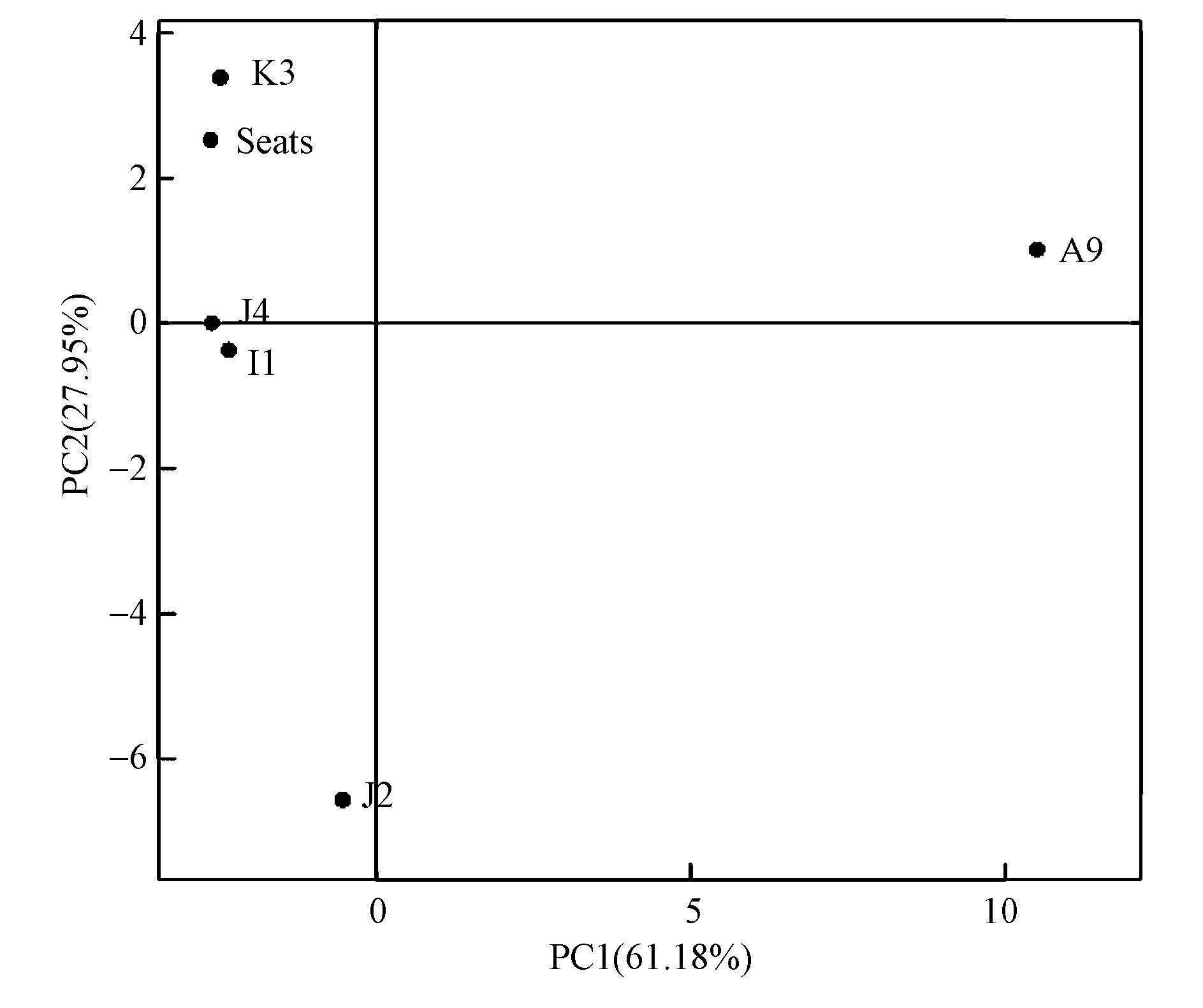 |
| 图 4 基于OTU丰度的PCA分析 Fig. 4 PCA analysis based on the abundance of OTU |
由上述分析可知,不同站位之间细菌群落结构差异较大.为了解这种差异的形成原因,对其主要影响因子进行了分析.夏季南海北部表层海水测得的环境因子如表 2所示.由表 2可知,近珠江口的A9站位的水温、盐度均低于其他5个站位,A9站位的硝酸盐、硅酸盐、亚硝酸盐和细菌生产力均高于离岸较远的其他5个站位.
| 表 2 不同站位主要环境因子 Table 2 Main environmental factors of different stations |
为研究该海域环境因子对含cbbL基因的固碳菌群群落组成的影响,对环境因子和主要固碳菌群进行了Pearson相关性分析和冗余分析(Redundancy Analysis,RDA),结果如图 5所示.由图 5可知,硫碱弧菌属(Thioalkalivibrio thiocyanodenitrificans)、硫杆菌属(Thiobacillus)、盐硫杆状菌属(Halothiobacillus)、异着色菌属(Allochromatium)、硫单胞菌属(Thiohalomonas)、硝化菌属(Thiobacter)与水温和盐度呈显著负相关(p<0.01),与硝酸盐、亚硝酸盐、硅酸盐、细菌生产力呈显著正相关(p<0.01).
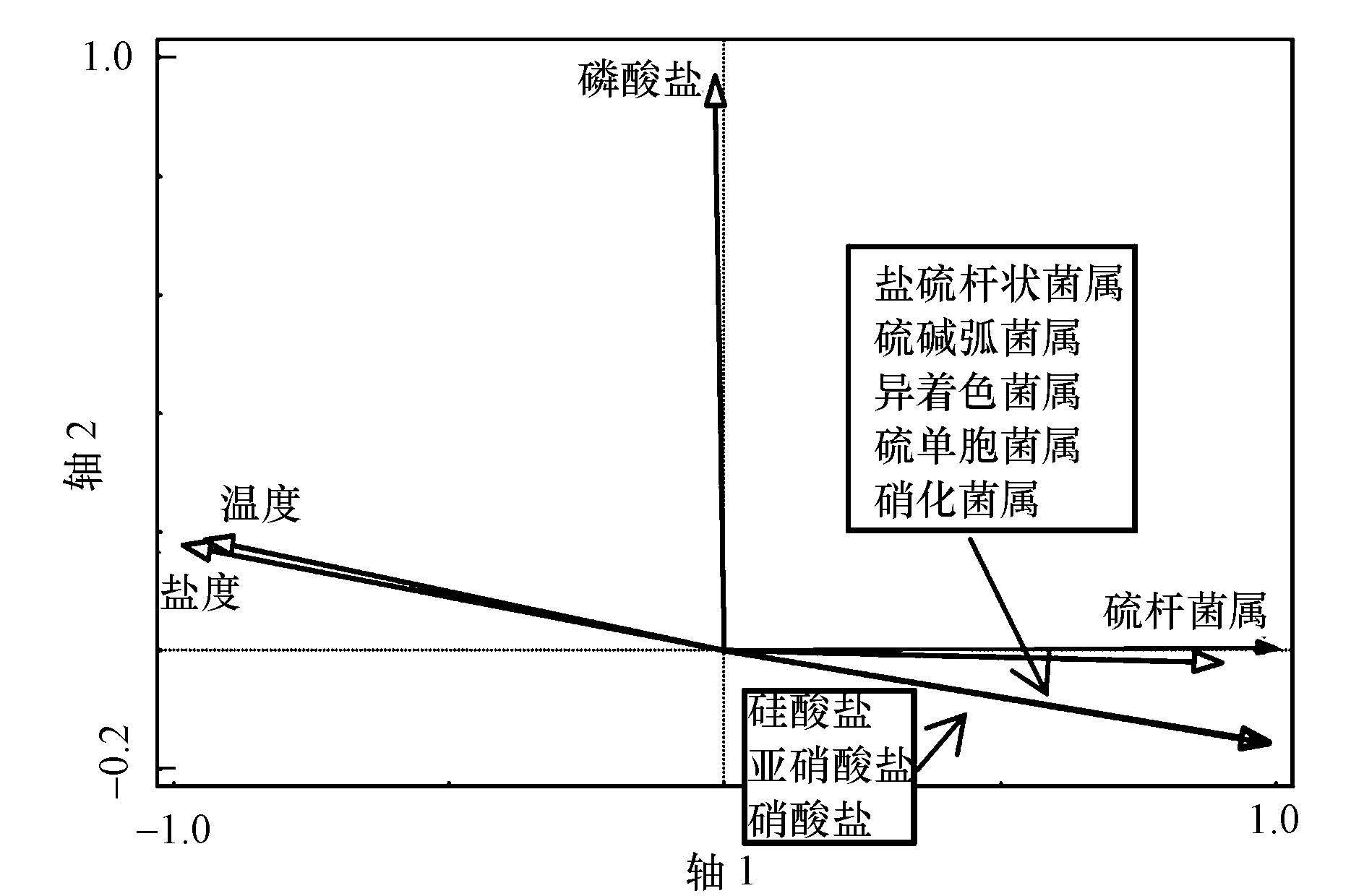 |
| 图 5 固碳细菌群落与环境因子的RDA分析 Fig. 5 Carbon sequestration bacterial communities and environmental factors RDA analysis |
本文以探索近珠江口到南海北部水域表层海水中含cbbL基因多样性为研究目的,获得了丰富的固碳细菌群落,这些菌群归属于3大门类:变形菌门、蓝藻门和厚壁菌门.其中,变形菌门在6个站位中都占有重要的比例(43.4%~98.9%),这一结果与其他学者的研究结果一致(Alfreider et al.,2012).在本研究中,变形菌门的γ-和β-亚群是该海域表层海水中的优势菌群,其中,γ-变形菌在细菌中所占的比例是目前所知最多的一类亚群,它包括一些重要的类群(Kovaleva et al.,2011);如在本研究各站位中都具有较大比例的外硫红螺菌属,该菌属可以利用光能,固定二氧化碳,这与其所发挥的生态功能十分吻合,与此同时,该菌属在本研究中也是γ-变形菌亚门最多的一个属.本研究中还发现了大量属于β-变形菌亚门的硫杆菌属,有学者利用不含有机碳源的硫代硫酸钠培养基进行硫杆菌的富集和分离,并进行功能基因的扩增得到了cbbL基因,推测可能是由于菌株利用无机碳进行固碳,从中取得能量并进行微弱生长(李少能等,2012).由此可见,这些变形菌群在海洋碳循环中具有重要作用.该研究水域中蓝藻门是第二大优势菌群,在6个站位中均有检出,它是一类产氧光合细菌,是海洋生态系统和细菌生产力的重要组成部分(白洁等,2014).蓝藻门在东海海水(Zeng et al.,2004)、地下水(国家海洋环境监测中心,2007)、潮间带和湖泊沉积物(Nigro et al.,2007)中均有发现.厚壁菌门是一类化能营养型菌群,在本研究中只出现在营养盐较高的A9站位,且存在数目较少(0.1%);已有学者证实少数厚壁菌门也参与卡尔文循环,其中,属于厚壁菌门的梭菌目还可以通过还原乙酰辅酶A途径固定CO2和产生甲烷生长所需要的醋酸(Caldwell et al.,2007).
4.2 未检测到cbbM固碳基因的原因分析本研究中对表层海水中cbbM基因的检测结果均为阴性,Giri等(2004)在美国莫诺德湖也未检测到该基因.分析原因可能有以下两方面:①含cbbM基因的固碳菌群广泛分布于厌氧的α-变形菌门中(English et al.,1992),而本研究采集的是南海北部海域表层海水,厌氧菌较少,同时分析本研究中cbbL基因多样性表明,γ-和β-变形菌亚门是该海域表层海水中的优势亚群,而非α-变形菌亚门,所以未能扩增出cbbM的目的条带;②Alfrider等(2012)和Videmšek等(2009)研究表明,低CO2浓度的环境有利于含cbbL固碳基因的固碳细菌的生长,但不利于含cbbM固碳基因的细菌生长.cbbL多在好氧细菌中被发现,而cbbM在低氧和高二氧化碳浓度下有较高活性(Badger et al.,2008);另有研究表明,南海海水中表层含氧量较高(刘小涯等,2006).因此可以推测,本研究中表层海水中未扩增出cbbM基因的原因可能与表层海水中溶解O2和CO2含量有关(Alfreider et al.,2012).
4.3 含cbbL基因的固碳细菌群落与环境因子的关系近岸营养盐丰富,硝酸盐、亚硝酸、磷酸盐和硅酸盐是细菌生长繁殖所必须的营养物质,加之细菌较大的表面积,对营养物质具有高亲和力,为固碳细菌的生存和发展提供了适宜的条件;并且前期已有研究表明,无机营养物可能是细菌生长的影响因子(Chrzanowski et al.,1995),因此,近岸A9具有较复杂的群落结构和高细菌生产力.通过Pearson相关性和冗余分析进一步分析表明,含cbbL基因的固碳细菌丰度与硝酸盐、亚硝酸盐、硅酸盐呈显著正相关(p<0.01),这一研究结果与前期研究一致(Gontero et al.,2014).此外,含cbbL基因的固碳细菌丰度与水温、盐度呈显著负相关(p<0.01),表明该海域固碳菌群还受水温、盐度的影响.在近珠江口区,由于陆源淡水注入,使离岸最近的A9站位构成了低盐、高营养盐特点,为异着色菌属(Allochromatium)、硫杆菌属(Thiobacillus)、硫单胞菌属(Thiohalomonas)提供了特有的生存条件,使得这些菌群只在该调查海域的A9站位附近的水域生存.
5 结论(Conclusions)1) 在珠江口到南海北部海域表层海水中,存在的固碳基因主要以cbbL为主,这说明含cbbL基因的固碳细菌是该海域表层海水中参与卡尔文循环途径的主要菌群.
2) 珠江口到南海北部水域中以cbbL为固碳基因的固碳菌群主要归属于3个类群,分别为变形菌门(69.1%)、蓝藻门(30.8%)和厚壁菌门(0.1%),其中,优势亚群为γ-变形细菌亚门(45.3%)、蓝细菌(30.9%)、β-变形菌亚门(23.8%).
3) 该海域水温、盐度、硝酸盐、亚硝酸盐、硅酸盐等因素是影响cbbL固碳细菌群落结构的主要因素.
致谢: 厦门大学海洋与地球学院为本研究提供了航次营养盐及温度盐度资料,中科院南海海洋研究所“实验1号”船长及全体船员在样品采集过程中给予了很大的帮助,在此一并表示感谢.| [1] | Alfreider A, Schirmer M, Vogt C. 2012. Diversity and expression of different forms of RubisCO genes in polluted groundwater under different redox conditions[J]. FEMS Microbiology Ecology , 79 (3) : 649–660. DOI:10.1111/fem.2012.79.issue-3 |
| [2] | Alfreider A, Vogt C, Geiger-Kaiser M, et al. 2009. Distribution and diversity of autotrophic bacteria in groundwater systems based on the analysis of RubisCO genotypes[J]. Systematic and Applied Microbiology , 32 (2) : 140–150. DOI:10.1016/j.syapm.2008.11.005 |
| [3] | 白洁, 刘小沙, 侯瑞, 等.2014. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学 , 2014, 34 (11) : 2950–2957. |
| [4] | Badger M R, Bek E J. 2008. Multiple Rubisco forms in proteobacteria:their functional significance in relation to CO2 acquisition by the CBB cycle[J]. Journal of Experimental Botany , 59 : 1525–1541. |
| [5] | Caldwell P E, MacLean M R, Norris P R. 2007. Ribulose bisphosphate carboxylase activity and a Calvin cycle gene cluster in Sulfobacillus species[J]. Microbiology , 153 (7) : 2231–2240. DOI:10.1099/mic.0.2007/006262-0 |
| [6] | Campbell B J, Cary S C. 2004. Abundance of reverse tricarboxylic acid cycle genes in free-living microorganisms at deep-sea hydrothermal vents[J]. Applied and Environmental Microbiology , 70 (10) : 6282–6289. DOI:10.1128/AEM.70.10.6282-6289.2004 |
| [7] | Chrzanowski T H, Sterner R W, Elser J J. 1995. Nutrient enrichment and nutrient regeneration stimulate bacterioplankton growth[J]. Microbial Ecology , 29 (3) : 221–230. DOI:10.1007/BF00164886 |
| [8] | Elsaied H, Naganuma T. 2001. Phylogenetic diversity of ribulose-1,5-bisphosphate carboxylase/oxygenase large-subunit genes from deep-sea microorganisms[J]. Applied and Environmental Microbiology , 67 (4) : 1751–1765. DOI:10.1128/AEM.67.4.1751-1765.2001 |
| [9] | English R S, Williams C A, Lorbach S C, et al. 1992. Two forms of ribulose-1,5-bisphosphate carboxylase/oxygenase from Thiobacillus denitrificans[J]. FEMS Microbiology Letters , 94 (1/2) : 111–119. |
| [10] | Giri B J, Bano N, Hollibaugh J T. 2004. Distribution of RuBisCO genotypes along a redox gradient in Mono Lake,California[J]. Applied and Environmental Microbiology , 70 (6) : 3443–3448. DOI:10.1128/AEM.70.6.3443-3448.2004 |
| [11] | Gontero B, Salvucci M E. 2014. Regulation of photosynthetic carbon metabolism in aquatic and terrestrial organisms by Rubisco activase,redox-modulation and CP12[J]. Aquatic Botany , 118 : 14–23. DOI:10.1016/j.aquabot.2014.05.011 |
| [12] | 国家海洋环境监测中心. 2007. GB17378.4-2007.海洋监测规范第4部分:海水分析[S].北京:中国标准出版社 |
| [13] | Hügler M, Gärtner A, Imhoff J F. 2010. Functional genes as markers for sulfur cycling and CO2 fixation in microbial communities of hydrothermal vents of the Logatchev field[J]. FEMS Microbiology Ecology , 73 (3) : 526–537. |
| [14] | Kirchman D L. 1993. Leucine incorporation as a measure of biomass production by heterotrophic bacteria[J]. Handbook of Methods in Aquatic Microbial Ecology , 509 . |
| [15] | Kovaleva O L, Tourova T P, Muyzer G, et al. 2011. Diversity of RuBisCO and ATP citrate lyase genes in soda lake sediments[J]. FEMS Microbiology Ecology , 75 (1) : 37–47. DOI:10.1111/fem.2010.75.issue-1 |
| [16] | 李少能.2012.南大西洋深海硫氧化细菌的分离鉴定与多样性初步分析[D].福州:福建农林大学 http://cdmd.cnki.com.cn/article/cdmd-10389-1012425412.htm |
| [17] | 刘小涯, 潘建明, 张海生, 等.2006. 南海海水中DO的平面,垂直分布以及海-气交换通量[J]. 海洋学研究 , 2006, 23 (4) : 41–48. |
| [18] | Nigro L M, King G M. 2007. Disparate distributions of chemolithotrophs containing form IA or IC large subunit genes for ribulose-1,5-bisphosphate carboxylase/oxygenase in intertidal marine and littoral lake sediments[J]. FEMS Microbiology Ecology , 60 (1) : 113–125. DOI:10.1111/fem.2007.60.issue-1 |
| [19] | Paul J H, Pichard S L. 1998. Phytoplankton Activity through the Measurement of Ribulose Bisphosphate Carboxylase Gene Expression (Rubisco)//Molecular Approaches to the Study of the Ocean[M]. Netherlands: Springe: 207 -225. |
| [20] | Ragsdale S W, Wood H G. 1991. Enzymology of the acetyl-CoA pathway of CO2 fixation[J]. Critical Reviews in Biochemistry and Molecular Biology , 26 (3/4) : 261–300. |
| [21] | Song J. 2011. Biogeochemical Processes of Biogenic Elements in China Marginal Seas[M]. Berlin: Springer Science & Business Media . |
| [22] | Tabita F R. 1999. Microbial ribulose 1,5-bisphosphate carboxylase/oxygenase:a different perspective[J]. Photosynthesis Research , 60 (1) : 1–28. DOI:10.1023/A:1006211417981 |
| [23] | Videmšek U, Hagn A, Suhadolc M, et al. 2009. Abundance and diversity of CO2-fixing bacteria in grassland soils close to natural carbon dioxide springs[J]. Microbial Ecology , 58 (1) : 1–9. DOI:10.1007/s00248-008-9442-3 |
| [24] | Watson G M F, Tabita F R. 1997. Microbial ribulose 1,5-bisphosphate carboxylase/oxygenase: a molecule for phylogenetic and enzymological investigation[J]. FEMS Microbiology Letters , 146 (1) : 13–22. DOI:10.1111/j.1574-6968.1997.tb10165.x |
| [25] | Yuan H, Ge T, Chen C, et al. 2012. Significant role for microbial autotrophy in the sequestration of soil carbon[J]. Applied and Environmental Microbiology , 78 (7) : 2328–2336. DOI:10.1128/AEM.06881-11 |
| [26] | 袁梁英.2005.南海北部营养盐结构特征[D].厦门:厦门大学 http://cdmd.cnki.com.cn/Article/CDMD-10384-2007055968.htm |
| [27] | Zakharchuk L M, Egorova M A, Tsaplina I A, et al. 2003. Activity of the enzymes of carbon metabolism in Sulfobacillus sibiricus under various conditions of cultivation[J]. Microbiology , 72 (5) : 553–557. DOI:10.1023/A:1026039132408 |
| [28] | Zeng Y H, Jiao N Z, Cai H Y, et al. 2004. Phylogenetic diversity of ribulose-1,5-bisphosphate carboxylase/oxygenase large subunit genes of bacterioplankton in the East China Sea[J]. Acta Oceanol Sin , 23 : 673–685. |
| [29] | 周集体, 王竞.1999. 细菌固定CO2的研究进展[J]. 环境科学进展 , 1999, 7 (1) : 1–9. |
 2016, Vol. 36
2016, Vol. 36


