我国土壤重金属污染已较为严重和普遍,重金属污染土壤的修复一直是国际上研究的难点和热点(骆永明等,2015; 宋伟等,2013).近年来,能源植物因其自身的优势在重金属污染土壤修复研究中引起越来越多研究者的关注.研究表明能源植物对重金属具有一定的耐受性,是重金属污染土壤修复的良好材料(Pandey et al.,2016; Van Ginneken et al.,20076; 余海波等,2011).由于矿山开采、重金属污水灌溉等原因,目前我国大面积土壤受重金属污染,选用能源植物作为修复植物既可以修复这些重金属污染农田、矿区废弃地,同时可以发展能源农业,缓解我国发展能源产业所面临的耕地压力,很好契合了"不与人争粮、不与粮争地"的能源农业发展原则,修复达标后的土地可以重新种植粮食作物,为治理和合理利用重金属污染土壤开辟了一条新的途径,同时可以改善生态环境,增加农民收入(Pandey et al.,2016; Weyens et al.,2009).
土壤中微生物种类多、数量大、生物活性高,在重金属污染土壤中修复植物的生长以及重金属的地球化学循环过程中起到重要作用(Teng et al.,2015; Weyens et al.,2009)).目前,在能源植物修复土壤重金属污染过程中根际土壤细菌群落组成和功能研究开展较少,相关工作亟待开展(Visioli et al.,2015).高通量测序为代表的新一代测序技术凭借低成本、高通量、流程自动化的优势为研究微生物群落结构提供了新的技术平台,大大推动了环境基因组研究的快速发展.但该方法无法揭示出微生物之间的相互作用,从而制约了微生物群落结构和功能之间关系的研究.生态网络分析技术(Network analysis)是一种基于数学模型分析生态系统中内在作用关系的系统分析方法,在研究生物群落物种之间的相互作用中显示出良好的效果(Faust&Raes,2012).进年来,研究者也将该方法应用于探索微生物生态网络结构,特别是高通量测序和基因芯片等分析技术的应用,为土壤微生物生态网络研究提供了坚实的数据基础(Zhou et al.,2010; Zhou et al.,2011).目前,分子生态网络分析已应用于海洋浮游细菌、牙齿表面生物膜、人体微生物、土壤等复杂生境,在微生物群落组成、微生物之间相互作用、关键种群的识别以及微生物群落结构对环境干扰的响应机制等研究中体现出不可替代的优势(Lu et al.,2013).
本研究选取生物量大、生长快且对重金属有一定耐受能力的油脂类能源植物大豆和碳水化合物类能源植物玉米(Jamal et al.,2002; Sheng et al.,2012),采用高通量测序方法研究大豆、玉米修复土壤镉污染过程中根际土壤细菌群落组成,基于高通量测序结果采用分子生态网络分析细菌相互作用,以期为重金属污染土壤-能源植物复合生境中细菌群落组成和功能、能源植物耐受重金属机制的阐明提供理论依据和技术支持.
2 材料与方法(Materials and methods) 2.1 盆栽种植土壤采自河南省南阳市东郊0~15 cm表层土(坐标为北纬32°99',东经 112°47'),土壤的基本理化性质为:黄棕壤,有机质10.29 g·kg-1、阳离子交换量CEC16.8 cmol·kg-1,全N 1.31 g·kg-1,全P 1.90 g·kg-1,速效P 18.27 mg·kg-1,pH 7.62,土壤中Cd未检测到.土壤风干后过20目筛,去除植物组织和石块,加入分析纯CdSO4·8H2O使土壤中Cd的浓度分别为50 mg·kg-1,充分混合并放置1个月后装盆,备用.因分子生态网络分析需要,每个样品设置7个重复,大豆品种中黄57种植于污染土壤(样品编号为S1~S7),玉米品种为郑单958(样品编号为Z1~Z7),以不种植物为对照组(CK1~CK7),大豆、玉米种子用30% NaClO(1∶1)表面消毒5 min后播入盆中,每盆5颗,出苗后每盆留苗3株.盆栽试验于南阳师范学院农业工程学院温室进行,种植时间为2015年4月至6月,共60 d,期间保持土壤含水量供植物生长所需的水分.采用抖落法收集根际土壤,将植株连同根系从土壤中挖出,然后轻轻抖动,抖落下来的土壤视为非根际土壤,黏附在根系上的土壤作为根际土壤,将收集的根际土壤混匀,用于后续分析.
2.2 重金属含量测定植物组织置于烘箱中105 ℃烘干,磨碎后准确称取样品于消煮管中用HClO4∶HNO3(优级纯,V∶V=13∶87)的混合酸液进行消解,电感耦合等离子发射光谱仪ICP-AES(Optima 2100 DV,Perkin Elmer)测定溶液中的Cd含量.
2.3 土壤DNA提取根际土壤细菌总DNA提取采用 FastDNA® Spin Kit for Soil试剂盒(MP Biomedicals,USA),按照说明书的提取步骤进行,将提取得到的土壤总DNA通过微量紫外分光光度计(NanoDrop® ND-1000,Wilmington,DE,USA)测定DNA浓度和纯度.
2.4 高通量测序采用通用引物(338F/806R)对土壤细菌16S rRNA基因的V3~V4区扩增,修饰后的通用引物含有不同的 Tag标签用以区分不同样品(Li et al.,2015).PCR 扩增体系为20 μL,其中含5×FastPfu Buffer 4 μL、2.5 mmol·L-1 dNTPs 2 μL、Forward Primer(5 μmol·L-1)0.8 μL、Reverse Primer(5 μmol·L-1)0.8μL、BSA(10 mg·mL-1)0.2 μL、FastPfu Polymerase 0.4 μL、DNA模板 10 ng,补ddH2O至20 μL.PCR 扩增的反应条件为: 94 ℃,5 min;30×(94 ℃,30 s;54 ℃,30 s;72 ℃,45 s);72 ℃,10 min.将不同样品的PCR扩增产物均一化至10 nmol·L-1后等体积混合,利用上海美吉生物医药科技有限公司的MiSeq PE300测序仪(Illumina Inc.,San Diego,CA,USA)完成序列测定.
2.5 高通量测序及其分子生态网络分析高通量数据的生物信息学分析采用 Qiime(quantitative insights into microbial ecology)进行,根据序列的相似度,将序列归为多个OTU(operational taxonomic unit),OTU 产出后,统计各个样品含有OTU情况及每个OTU中含有序列的数目,得到每个OTU的分类学信息(Ren et al.,2015).选取相似度在97%条件下的OTU生成预期的稀释曲线,并利用软件mothur计算丰富度指数Chao1和ACE,覆盖度指数(good's coverage)以及多样性指数Simpson和Shannon指数进行Alpha 多样性分析.利用PCoA、Correlation聚类法分别进行数据处理、细菌群落分布、主成分分析和聚类分析.
选择获得的高通量测序数据基于随机矩阵理论(random matrix theory,RMT)方法构建分子生态网络,网络构建、获取网络性质参数均在The Molecular Ecological Network Analyses Pipeline(MENAP)(http://ieg2.ou.edu/MENA/main.cgi)上完成(Deng et al.,2012,Zhou et al.,2010; Zhou et al.,2011).主要步骤如下:首先对需要分析样品OTU进行标准化,按MENAP要求排列并上传数据,MENAP计算任意2 个OTU的皮尔森相关性(Pearson correlation),并用成对相关系数的绝对值来检验相关矩阵,将相关矩阵转化为相似性矩阵,根据随机矩阵理论运用一个特定的阈值(similarity threshold),将相似性矩阵转换为邻接矩阵(OTU节点间的关联程度).上传数据后,点击MENAP中构建网络(Construct the network),其中阈值等参数设定以MENAP默认参数为准. 依次点击MENAP 网络分析(Analyze the networks)中"global network properties"、"individual nodes' centrality"、"module separation and modularity"选项进行分析,选择 "output for Cytoscape visualization",利用软件Cytoscape 3.1.1对网络进行可视化处理,得到可视网络结构图及相关信息,包括类别、模块化属性、相关性、模块枢纽等.在可视化网络结构图中,节点代表群落中的种群,节点之间的连线代表种群之间的相互作用关系(正、负相关性等)(Jiang et al.,2015; Lu et al.,2013; 汪峰等,2014)
2.6 数据分析采用主坐标分析(principal coordinate analysis,PCoA)和非参数检验方法Adonis(Permutational multivariate analysis of variance)来检验大豆、玉米种植和不种植条件下细菌整体群落组成的差异.PCoA和Adonis分析均通过 R 语言 vegan 程序包完成(Ren et al.,2015).数据以平均值±标准偏差表示,通过Excel独立样本t检验比较成对样本间差异显著性.运用SPSS 15.0统计软件进行数据的单因素方差分析(ANOVA),当差异达显著性水平时,以Duncan's多重比较进行不同处理间的显著性分析,检验标准为p<0.05或p<0.01.
3 结果(Results) 3.1 生物量及镉含量不同组织Cd浓度测定结果表明根部Cd浓度最高,大豆根部Cd浓度达到338.01 mg·kg-1,玉米根部Cd浓度也超过150 mg·kg-1,其次为玉米的茎部(85.25 mg·kg-1)和叶部(98.60 mg·kg-1),大豆地上部Cd浓度较低(图 1).结合不同组织生物量,可以得出玉米、大豆不同组织Cd积累总量,如图 1C所示,根部Cd积累量最高,玉米和大豆根部Cd积累量为植物Cd总积累的56.58%和73.82%,转移系数TF(植物地上部重金属含量与根中该元素含量的比值)分别为玉米0.78和大豆0.35.玉米和大豆Cd总积累分别为393 μg·pot-1和263μg·pot-1,土壤中Cd总量为75 mg·pot-1,因此,玉米和大豆的修复效率分别为0.524%和0.351%.
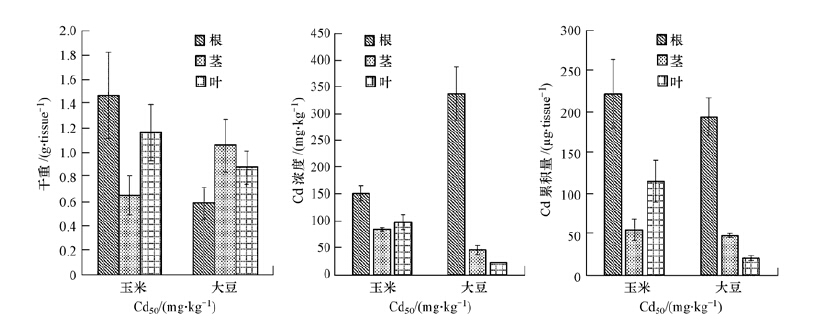 |
| 图 1 不同处理玉米、大豆组织中干重(A)、Cd浓度(B)和Cd积累量(C) Fig. 1 Dry weights(A),Cd concentrations(B)and total Cd uptake(C)of two plants with 50 mg·kg-1 Cd treatments |
高通量测序结果表明,21个实验样品文库覆盖率均在99%以上,能够反映样品中细菌群落的种类和结构.21个实验样品平均序列条数为32386,OTUs平均数1594,经过分析,主要为细菌的 31 个门,包括 Proteobacteria(变形菌门)、Acidobacteria(酸杆菌门)、Gemmatimonadetes(芽单胞菌门)、Bacteroidetes(拟杆菌门)、Candidate division TM7、Actinobacteria(放线菌门)、Chloroflexi(绿弯菌门)、Fibrobacteres(纤维杆菌门)、Firmicutes(厚壁菌门)等(图 2).其中属于 Proteobacteria、Acidobacteria、Gemmatimonadetes、Bacteroidetes门的序列总和占全部序列的 68.61%,这些微生物为优势种群.
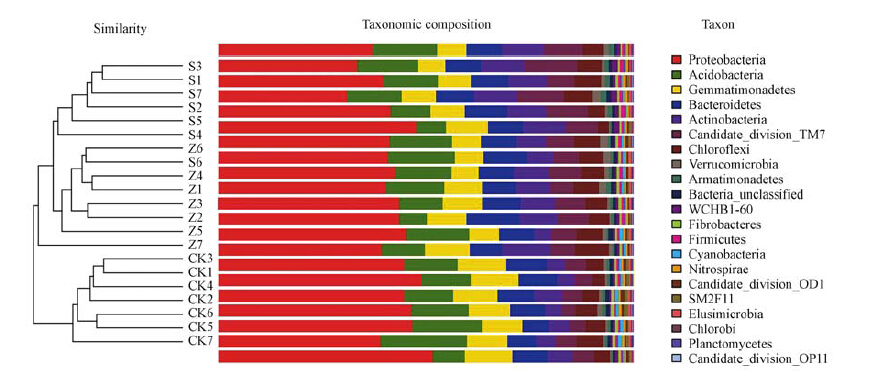 |
| 图 2 不同样品相似性聚类树(左图)和门水平上物种相对丰度分布图(右图) Fig. 2 Similarity tree(Left)and relative read abundance(Right)of different bacterial community structures at phylum levels in different treatments |
为了研究能源植物大豆和玉米修复Cd污染土壤对土壤细菌群落的影响,对相关实验样品细菌群落进行了PCoA分析.如图 3所示,横坐标PC1贡献度为40.46%,纵坐标PC2贡献度为11.18%,不种植物处理(CK1~CK7)聚集在一起,位于横坐标PC1左侧,与种植大豆处理(S1~S7)和种植玉米处理(Z1~Z7)相互分开,表明能源植物种植对土壤细菌群落结构产生影响.同时种植大豆处理(S1~S7)聚集在纵坐标 PC2上部,种植玉米(Z1~Z7)处理聚集在纵坐标PC2下部,表明不同能源植物之间土壤细菌群落组成也有差异.后续使用基于Unifra的UPGMA方法对相关实验样品细菌群落构成的相似性进行了聚类分析.通过样品加权的聚类分析,同样可以有效的将部不种植物组、种植大豆和种植玉米组实现组间聚类(图 2左图).Adonis分析结果表明,大豆、玉米种植与不种植物细菌群落比较,p值分别为0.002和0.003,达极显著水平(p <0.01),这表明大豆、玉米种植能显著改变土壤细菌群落组成.
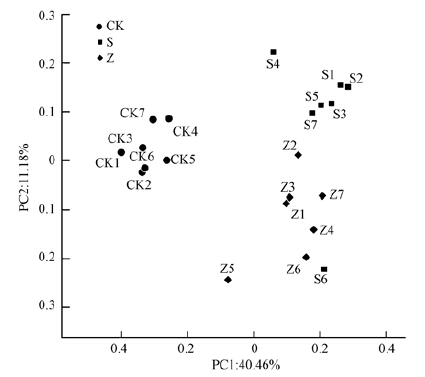 |
| 图 3 细菌群落多样性的主成分分析 Fig. 3 PCA analysis of bacterial community diversity |
经过分析,50 mg·kg-1 Cd污染土壤细菌分布于31 个门,421个属,大豆和玉米种植能影响这些细菌的相对丰度.421个属的细菌中,与不种植对照相比,大豆和玉米种植提高260和219个属细菌丰度,156和197个属细菌丰度有所降低.其中Candidate division TM7 norank、Acidimicrobiales norank、WCHB1-60 norank、Subgroup 6 norank、Rhizobium、Roseiflexus、Massilia、Streptomyces、Chitinophagaceae uncultured、Rhizomicrobium等为大豆种植处理相对丰度提升最多的细菌种群,比例从0.37%到4.60%,Sphingomonas、Gemmatimonas、Subgroup 7 norank、Nitrosomonadaceae uncultured、Acidobacteriaceae Subgroup 1 uncultured、Flavisolibacter、MLE1-12 norank、Anaerolineaceae uncultured、Xanthobacteraceae unculture、DB1-14 norank等为相对丰度下降最多的细菌种群,比例从0.47%到8.49%.Sphingomonas、Anaerolineaceae uncultured、Sphingobium、Subgroup 7 norank、Escherichia-Shigella、Pseudomonas、Ralstonia、Gemmatimonas、Xanthomonadales norank、Acidobacteriaceae Subgroup 1 uncultured等为玉米种植处理相对丰度提升最多的细菌种群,比例从0.22%到1.73%,Candidate division TM7 norank、Acidimicrobiales norank、Blastocatella、DB1-14_norank、SC-I-84_norank、WCHB1-60 norank、Bryobacter、Aquicella、Patulibacter、Niastella等为相对丰度下降最多的细菌种群,比例从0.22%到2.40%.
3.4 分子生态网络分析分别利用不种植物、种植玉米和种植大豆细菌相对丰度基于RMT方法构建了3个分子生态网络,可视化网络结构图显示了不同处理下土壤不同细菌在整个网络中的作用及其与其他细菌的相互联系(图 4).3个网络的相关性阈值均为0.92,高于大部分基于该方法构建网络的阈值,可以用于随后微生物相互关系的研究.描述网络的指标包括:连通度、路径长度、群聚系数和模块性,与不种植物相比,种植大豆的网络节点数、连接数和种植玉米的节点数均增加(表 1).此外,与对照相比,种植玉米和大豆网络的平均连通度较低、平均路径长度较大,表明种植大豆和玉米可以更好地缓冲环境变化对网络的干扰.
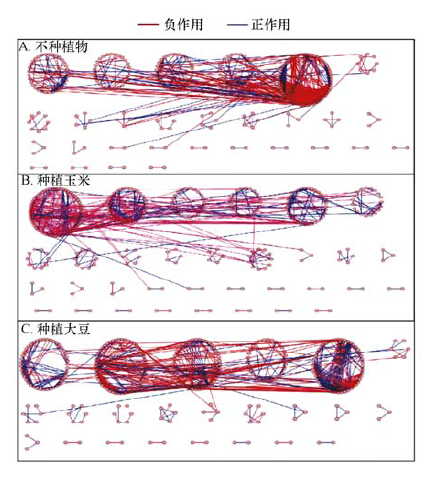 |
| 图 4 土壤细菌群落网络模型 Fig. 4 Overview of bacteria networks in soil samples |
| 表 1 分子生态网络的拓扑性质 Table 1 Topological characteristics of molecular ecological network |
同时,节点的拓扑角色可由模块内连通度(Zi)和模块间连通度(Pi)表征.模块内连通度描述了一个节点在模块中与其他节点的连接程度,而模块间连通度反映了一个节点连接到不同模块的程度.网络节点一般分为外围节点、连接器、模块枢纽和网络枢纽4 类.外围节点Zi和 Pi值较低,表明连接数很少且总是与模块内的节点相连.模块枢纽,Zi 高、Pi 较低,与本模块的许多节点高度连接,但与其他模块连接很少.连接器,Zi 低、Pi很高,表明在模块内部连接程度很低,但与某几个模块高度相连.网络枢纽,Zi 和Pi 较高,同时具有模块枢纽和连接器的作用.通过对不同处理生态网络节点的拓扑角色分析(图 5),不种植物处理网络节点绝大多数处于外围节点,只有1个节点处于模块枢纽内.与不种植物类似,种植大豆和玉米处理网络节点主要分布于外围节点,但在模块枢纽内有3和4个节点分布,连接器内有1和6个节点分布.种植大豆处理有2个节点分布于网络枢纽中.
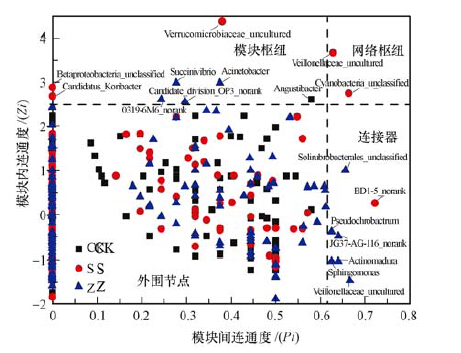 |
| 图 5 不同处理下土壤细菌网络的拓扑角色 Fig. 5 Zi-Pi plot showing the distribution of soil bacteria based on their topological roles |
综合以上结果表明种植大豆和玉米增加了细菌之间的相互作用,导致其网络结构更为复杂.
在分子生态网络的拓扑结构中,不同节点的拓扑学角色可以作为识别关键微生物的依据.参考Deng等(2012)研究,将Zi =2.5、Pi =0.62作为临界值.我们把Zi≥2.5或是Pi≥0.62的所有节点定为关键细菌(Key bacteria),这些细菌在各自的大模块中起着联系模块内部细菌的重要作用.不种植物处理的关键细菌为Angustibacter,种植大豆的关键细菌为 BD1-5 norank、Cyanobacteria unclassified、Veillonellaceae uncultured、Verrucomicrobiaceae uncultured、Betaproteobacteria unclassified、Candidatus Koribacter,种植玉米的关键细菌为Veillonellaceae uncultured、Solirubrobacterales unclassified、Pseudochrobactrum、Sphingomonas、JG37-AG-116_norank、Actinomadura、Succinivibrio、Acinetobacter、0319-6M6 norank、Candidate division OP3 norank(图 5).
4 讨论(Discussion)) 4.1 能源植物种植对土壤细菌群落的影响土壤中微生物种类多、数量大、生物活性高,在重金属污染土壤中修复植物的生长以及重金属的地球化学循环过程中起到重要作用,针对能源植物根际土壤细菌群落组成和功能研究为探明能源植物耐受、富集重金属及其调控提供帮助(Teng et al.,2015,Weyens et al.,2009).Chen等(2013)采用变性梯度凝胶电泳法(PCR-DGGE)的方法研究了能源植物油菜修复土壤镉污染过程种细菌群落变化,油菜根际土壤细菌主要由Actinobacteria、Alphaproteobacteria等5个门和12个属以及Unclassified细菌组成.目前,已开展的工作或采用传统平板分离法或采用非培养的PCR-DGGE等不全面和有选择性的方法(Visioli et al.,2015).本论文采用高通量测序的方法研究能源植物大豆和玉米修复Cd污染土壤细菌群落,结果表明大豆、玉米根际土壤细菌由Proteobacteria、Acidobacteria等31个门421个属细菌组成.分析表明,大豆和玉米种植能影响这些细菌的相对丰度.421个属的细菌中,与不种植对照相比,大豆和玉米种植提高260和219个属细菌丰度,156和197个属细菌丰度有所降低.
4.2 分子生态网络分析土壤细菌相互作用高通量测序技术的应用能很好揭示不同生境中微生物群落组成,但这些研究大多分析微生物物种的数量(丰度)以及多样性指数(如 Shannon和Simpson 指数)特征,无法揭示出微生物之间的相互作用,从而制约了微生物群落结构和功能之间关系的研究.生态网络分析技术的发展为该问题的解决提供了新的思路.汪峰等(2014)研究种植玉米对不同土壤氮素循环的影响,基于高通量基因芯片建立土壤核心氮转化基因的分子生态网络,表明种植玉米增加了土壤中大部分核心氮转化基因的丰度,显著提高了核心氮转化基因网络的复杂程度,土壤核心氮转化基因由裸地处理的固氮基因(nifH)转变为种植玉米处理的固氮(nifH)和反硝化基因(narG和nosZ).本实验基于高通量测序构建不种植物、种植玉米和种植大豆3个分子生态网络,结果表明种植大豆和玉米增加了细菌之间的相互作用,导致其网络结构更为复杂.同时,关键细菌从不种植物处理的1个增加到种植大豆的6个和种植玉米的10个.其中,种植大豆的关键细菌Sphingomonas既是大豆根际相对丰度排名靠前的细菌,也是丰度变化(与不种植物处理相比)排名靠前的.研究表明,Sphingomonas在土壤中广泛存在,很多具有植物促生特性,在有机污染降解、重金属污染植物修复中有着较多应用(Chen et al.,2014; Myresiotis et al.,2012),分析其可能在大豆生长、耐受、富集镉过程种起到重要作用.其他关键细菌Angustibacter、Pseudochrobactrum、Actinomadura等可能在玉米、大豆生长、耐受、富集镉过程种起到作用,需要继续深入研究.
4.3 高通量测序和分子生态网络分析指导功能菌株筛选研究表明重金属污染等逆境条件下植物根际土壤存在大量功能细菌,这些具有植物促生特性、污染物降解能力等功能的可培养细菌在植物生长、污染物降解以及植物耐受逆境胁迫等方面起到重要的作用(Chen et al.,2013; Li et al.,2016; Picard&Bosco.,2003; Timmusk et al.,2011).随着高通量测序及其分子生态网络分析技术在微生物生态研究中的应用,为复杂生境中微生物群落组成、关键种群识别、重要功能菌株的筛选提供了新的途径.Feng等(2015)利用高通量测序和克隆文库对我国华北平原潮土土壤微生物群落结构的研究发现,一种类Bacillus asahii细菌对长期施用有机肥的响应最为显著,而且它是潮土中的土著微生物;设计筛选方案,获得了该菌株,对其进行生理生化分析发现该物种有着不同于其它芽孢杆菌的生理特征,以及具有丰富的代谢多样性;回接实验显示该菌株能够加速和促进其它微生物对潮土有机质累积和磷素循环过程,在作物生长和土壤地力中起到领军性的作用.Duran-Pinedo等(2011)通过人类口腔微生物的分子生态网络分析,发现之前未能培养的重要菌株Tannerella sp. OT286与Prevotella oris OT311有很强的相互联系,在Prevotella oris OT311的辅助生长下获得Tannerella sp. OT286纯培养菌株.
本实验中高通量测序表明Sphingomonas既是大豆根际相对丰度排名靠前的细菌,也是丰度变化(与不种植物处理相比)排名靠前的.同时,生态网络分析表明Sphingomonas是种植大豆处理的关键细菌,分析其在大豆生长、耐受、富集镉过程中可能起到重要作用.针对高通量测序和分子生态网路分析确定的Sphingomonas等关键细菌,采用KOMODO数据库等方法重新设计和优化生长培养基,将有助于植物-微生物联合修复功能菌株筛选(Oberhardt et al.,2015; 袁志辉等,2014).高通量测序和分子生态网络分析在能源植物修复土壤重金属污染研究中的应用,为性能稳定、适应性强、能良好匹配土壤类型、重金属浓度和修复植物的土著促生细菌筛选创造良好条件,能较大程度改善植物促生细菌的筛选方案,提高能源植物-促生细菌联合修复实际修复效率.
5 结论(Conclusions)1) 50 mg·kg-1 Cd污染土壤中玉米、大豆根部Cd浓度和积累量最高,根部Cd积累量分别为玉米、大豆Cd总积累的56.58%和73.82%,转移系数TF分别为0.78和0.35.玉米和大豆的修复效率分别为0.524%和0.351%.
2) 高通量测序结果表明大豆、玉米根际土壤细菌由Proteobacteria、Acidobacteria等31个门细菌组成,大豆、玉米种植均能显著影响土壤细菌群落组成,能影响Candidate division TM7 norank、Acidimicrobiales norank、Sphingomonas、Gemmatimonas、Anaerolineaceae uncultured、Blastocatella等丰度.
3) 基于高通量测序的分子生态网络分析表明种植大豆和玉米增加了细菌之间的相互作用,导致其网络结构更为复杂,关键细菌从不种植物处理的1个增加到种植大豆的6个和种植玉米的10个.
致谢: 特别感谢南京农业大学博士生熊武、赵军和环境保护部南京环境科学研究所田伟博士在网络构建和分析上提供的帮助| [1] | Chen B, Shen J G, Zhang X C, et al. 2014.The endophytic bacterium, Sphingomonas SaMR12, improves the potential for Zinc phytoremediation by its host, Sedum alfredii[J]. PLoS ONE, 9 (9): e106826. |
| [2] | Chen Z J, Sheng X F, He L Y, et al. 2013.Effects of root inoculation with bacteria on the growth, Cd uptake and bacterial communities associated with rape grown in Cd-contaminated soil[J]. Journal of Hazardous Materials, 244 : 709–717. |
| [3] | Deng Y, Jiang Y H, Yang Y F, et al. 2012.Molecular ecological network analyses[J]. BMC Bioinformatics, 13 (1): 113. |
| [4] | Duran-Pinedo A E, Paster B, Teles R, et al. 2011.Correlation network analysis applied to complex biofilm communities[J]. PLoS ONE, 6 (12): e28438. |
| [5] | Faust K, Raes J. 2012.Microbial interactions: from networks to models[J]. Nature Reviews Microbiology, 10 (10): 538–550. |
| [6] | Feng Y Z, Chen R R, Hu J L, et al. 2015.Bacillus asahii comes to the fore in organic manure fertilized alkaline soils[J]. Soil Biology and Biochemistry, 81 : 186–194. |
| [7] | Jamal A, Ayub N, Usman M, et al. 2002.Arbuscular mycorrhizal fungi enhance Zinc and Nickel uptake from contaminated soil by soybean and lentil[J]. International Journal of Phytoremediation, 4 : 205–221. |
| [8] | Jiang Y J, Sun B, Li H X, et al. 2015.Aggregate-related changes in network patterns of nematodes and ammonia oxidizers in an acidic soil[J]. Soil Biology and Biochemistry, 88 : 101–109. |
| [9] | Li A J, Hou B L, Li M X. 2015.Cell adhesion, ammonia removal and granulation of autotrophic nitrifying sludge facilitated by N-acyl-homoserine lactones[J]. Bioresource Technology, 196 : 550–558. |
| [10] | Li Y, Wang Q, Wang L, et al. 2016.Increased growth and root Cu accumulation of Sorghum sudanense by endophytic Enterobacter sp[J]. K3-2: Implications for Sorghum sudanense biomass production and phytostabilization [J]. Ecotoxicology and Environmental Safety, 124 : 163–168. |
| [11] | Lu L H, Yin S X, Liu X, et al. 2013.Fungal networks in yield-invigorating and -debilitating soils induced by prolonged potato monoculture[J]. Soil Biology and Biochemistry, 65 : 186–194. |
| [12] | 骆永明, 章海波, 涂晨, 等.2015.中国土壤环境与污染修复发展现状与展望[J].中国科学院院刊, 30 (Z1):115–124. |
| [13] | Myresiotis C, Vryzas Z, Papadopoulou-Mourkidou E. 2012.Biodegradation of soil-applied pesticides by selected strains of plant growth-promoting rhizobacteria (PGPR) and their effects on bacterial growth[J]. Biodegradation, 23 (2): 297–310. |
| [14] | Oberhardt M A, Zarecki R, Gronow S, et al. 2015.Harnessing the landscape of microbial culture media to predict new organism-media pairings[J]. Nature Communications, 6 : 8493. |
| [15] | Pandey V C, Bajpai O, Singh N. 2016.Energy crops in sustainable phytoremediation[J]. Renewable and Sustainable Energy Reviews, 54 : 58–73. |
| [16] | Picard C, Bosco M. 2003.Soil antimony pollution and plant growth stage affect the biodiversity of auxin-producing bacteria isolated from the rhizosphere of Achillea ageratum L[J]. FEMS Microbiology Ecology, 46 (1): 73–80. |
| [17] | Ren W J, Ren G D, Teng Y, et al. 2015.Time-dependent effect of graphene on the structure, abundance, and function of the soil bacterial community[J]. Journal of Hazardous Materials, 297 : 286–294. |
| [18] | Sheng X F, Sun L N, Huang Z, et al. 2012.Promotion of growth and Cu accumulation of bio-energy crop (Zea mays) by bacteria: Implications for energy plant biomass production and phytoremediation[J]. Journal of Environmental Management, 103 : 58–64. |
| [19] | 宋伟, 陈百明, 刘琳.2013.中国耕地土壤重金属污染概况[J].水土保持研究, 20 (2):293–298. |
| [20] | Teng Y, Wang X M, Li L, et al. 2015.Rhizobia and their bio-partners as novel drivers for functional remediation in contaminated soils[J]. Frontiers in Plant Science, 6 : 32. |
| [21] | Timmusk S, Paalme V, Pavlicek T, et al. 2011.Bacterial distribution in the rhizosphere of wild barley under contrasting microclimates[J]. PLoS ONE, 6 (3): e17968. |
| [22] | Van Ginneken L, Meers E, Guisson R, et al. 2007.Phytoremediation for heavy metal-contaminated soils combined with bioenergy production[J]. Journal of Environmental Engineering and Landscape Management, 15 (4): 227–236. |
| [23] | Visioli G, D'Egidio S, Sanangelantoni A M. 2015.The bacterial rhizobiome of hyperaccumulators: future perspectives based on omics analysis and advanced microscopy[J]. Frontiers in Plant Science, 5 : 752. |
| [24] | 汪峰, ZhouJ Z, 孙波.2014.我国东部土壤氮转化微生物的功能分子生态网络结构及其对作物的响应[J].科学通报, 59 (4/5):387–396. |
| [25] | Weyens N, van der Lelie D, Taghavi S, et al. 2009.Exploiting plant-microbe partnerships to improve biomass production and remediation[J]. Trends in Biotechnology, 27 (10): 591–598. |
| [26] | 余海波, 宋静, 骆永明, 等.2011.典型重金属污染农田能源植物示范种植研究[J].环境监测管理与技术, 22 (3):71–76. |
| [27] | 袁志辉, 王健, 杨文蛟, 等.2014.土壤微生物分离新技术的研究进展[J].土壤学报, 51 (6):1183–1191. |
| [28] | Zhou J Z, Deng Y, Luo F, et al. 2010.Functional molecular ecological networks[J]. mBio, 1 (4): e00169–10. |
| [29] | Zhou J Z, Deng Y, Luo F, et al. 2011.Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2[J]. mBio, 2 (4): e00122–00111. |
 2016, Vol. 36
2016, Vol. 36


